DNA纳米结构设计、合成及应用研究进展简述
段金伟
(长安大学 理学院,陕西 西安 710064)
在原子尺度上操纵材料从而实现定制材料性能的可能性是化学和材料科学发展的重要目标之一,然而时至今日这依旧是一个巨大的挑战.在过去的几十年里,科学家们始终在寻找一些具有可控制且模块化性质的材料,希望能够推动纳米结构领域的快速发展.不同于传统的有机和无机材料,DNA具有良好的可寻址性、可控性、以及存在于DNA链之间的弱相互作用,使得DNA被认为是合成纳米材料的理想原材料之一[1-4].传统观点认为DNA仅仅是携带遗传信息的载体,但随着纽约大学N.C.Seeman教授的发现,这一观念彻底被颠覆.N.C.Seeman无意间发现能够利用DNA黏性末端将设计好的分支结构连接起来形成三维目标结构,于是在1991年首次人工合成了一个刚性的DNA立方体[5],打开了DNA纳米领域的研究大门[6-13].DNA纳米技术是利用Watson-Crick碱基互补配对的特异性和DNA自身的特殊性质,以自组装策略为基础,以构筑二维和三维方向上重复阵列为目标的新颖的分子纳米技术[1,14-15].
自Seeman提出以DNA为原材料合成纳米结构的设想后,科学家们先后开发了一系列帮助进行结构设计的算法和软件[16-23],优化、改进并提出了多种新的合成方法[7,10,24-29],并且尝试将DNA纳米结构应用到药物运输、疾病诊断和治疗、荧光成像等领域[30-37].
1 DNA结构设计软件
为了实现精确操作原子,达到合成目标DNA纳米结构的目的,需要根据目标结构来进行结构设计,这个过程不仅需要耗费大量的人力和物力,而且耗时极长.早期的DNA纳米结构设计软件使用的是pdb、mol等标准化结构文件的现有分子建模工具[38].这些工具允许设计者在原子级别上对DNA结构进行修改或修饰,结构建模大多是在序列层面上手动完成的.然而,这些程序设计过程中缺乏结构细节,限制了设计和操作多层三维结构的能力.而三维 DNA纳米结构的开发,需要大量的手工计算和熟练使用通用建模工具[38].
DNA纳米结构的构建通常包括一条长链的路径(大约8 000个核苷酸),钉书针的放置和序列的确定,对于大型纳米结构来说是一项挑战性任务.在功能化需求日益增高和结构设计复杂性逐渐提升的驱动下,为了促进新的DNA纳米结构发展,并让用户更加直观地了解DNA折叠的复杂性,科学家们基于不同用途开发了一系列建模工具和可视化程序,主要包括:Tiamat[38],CaDNAno[17],vHelix[39],NUPACK[40],ATHENA[41],Adentia[23]和MrDNA[22]等.DNA结构设计软件的界面对使用者越来越友好,功能也越来越强大,极大地降低了结构设计过程的成本.在降低定制DNA分子的生产成本和提升结构操纵的可能性方面取得了重大进展.本文中,选择性地介绍其中比较常用的4款软件.
1.1 CaDNAno
CaDNAno是Douglas等人2009年开发的一款支持利用DNA折纸技术进行结构设计的开源软件包,配套安装Python或Autodesk Maya运行,其官网地址为http://cadnano.org/.CaDNAno简化和增强了设计三维DNA折纸纳米结构的过程.通过用户友好的2D和3D界面,可以使设计者根据自己的需求进行任意设计创建.CaDNAno的嵌入规则与CanDo执行的有限元分析相结合,提高了结构稳定性的相对确定性.
CaDNAno是一个被广泛使用的软件,可用来帮助设计基于晶格的DNA纳米结构.该软件将设计的截面分为两种格子类型,即正方形和蜂窝,确保了设计的高度可靠性.这种截面类型可以确保交叉的适当位置,因此在体外DNA纳米结构呈现高折叠性.如图1所示,CaDNAno的建模过程包括六个主要步骤:①通过填充晶格上的空位置来定义纳米结构的平面形状,Honeycomb(蜂窝状)或Square(方形)二选一(图1a).②根据需要定义了单个螺旋的长度(图1c).③构建一个DNAorigami骨架链穿过纳米结构的二维图(图1d).④骨架链设计完成后,电极“AutoStaple”自动生成与骨架链绑定的订书针链(图1e).⑤点击订书针链根据用户需要调整其长度、连接和断开方式(图1e).⑥点击“Seq”按钮选择合适的骨架链类型,通常用M13mp18,确定骨架链和订书针链的DNA序列(图1b).⑦利用CaDNAno以.json文件格式导出DNA纳米结构的模型,以.csv格式导出DNA序列,以.svg格式导出示意图(图1f).
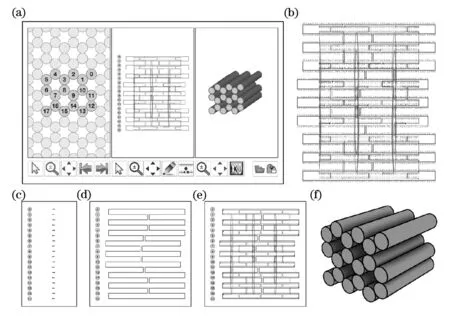
图1 CaDNAno设计DNA纳米结构示意图[17]
1.2 NUPACK
NUPACK是一个不断发展的DNA纳米结构序列设计在线分析软件,其官网为http://nupack.org/,主要用于核酸结构、设备和系统的分析和设计,使用nupack.org上的NUPACK web应用程序可以便捷地运行该软件的大部分功能.NUPACK web应用程序可以分析和设计一个或多个相互作用的核酸链的平衡碱基对的特性.主要功能包括分析、设计、用途.①分析:在稀溶液状态下,对核酸链的相互作用进行热力学分析.②设计:在平衡时,对目标二级结构的核酸链复合物的序列进行设计.③用途:对一组核酸链的平衡特性进行评估、显示和注释.

图2 NUPACK web应用程序操作界面[40]
1.3 Adentia
Adenita是一个交互式的3D设计软件,是作为SAMSON Connect(SAMSON Connect是一个3D建模图形框架)的一个开源插件开发的.Adenita提供半手动和高度模块化的设计方法,允许用户同时使用所有常见的DNA架构来减少早期DNA-origami软件解决方案的建模限制.Adenita通过调整模型的结构复杂性,实现模型的多尺度、适应性可视化,以适应当前设计目标[47]的需求.因此,可以在一个抽象图形或网格上设计大型的纳米结构.一个有经验的用户可以通过Adenita平台将所有现有设计结构整合到一个构型中,例如:腺嘌呤有助于有机和无机分子(包括蛋白质)的整合,可以被直接整合到DNA纳米结构中.Adenita平台可以简化纳米结构的功能化设计过程,赋予并提升用户研究和调节各种功能元素之间相互作用的能力.

图3 Adenita设计DNA纳米结构示意图[23]
1.4 MrDNA
虽然DNA结构纳米技术以惊人的速度发展,但从头设计复杂的3D纳米结构和功能器件仍然是一个费力且耗时的过程.其中一个原因是需要多次进行实验表征,以阐明设计选择对自组装物体实际形状和功能的影响.Aleksei Aksimentiev等人开发的MrDNA(图4),是一个基于Python开发的多分辨率的模拟框架,可以由社区进行扩展,并与DNA设计和分子图形工具集成的工具.MrDNA在30分钟或更短的时间内,可以构建一个自组装DNA纳米系统的原子分辨率结构.
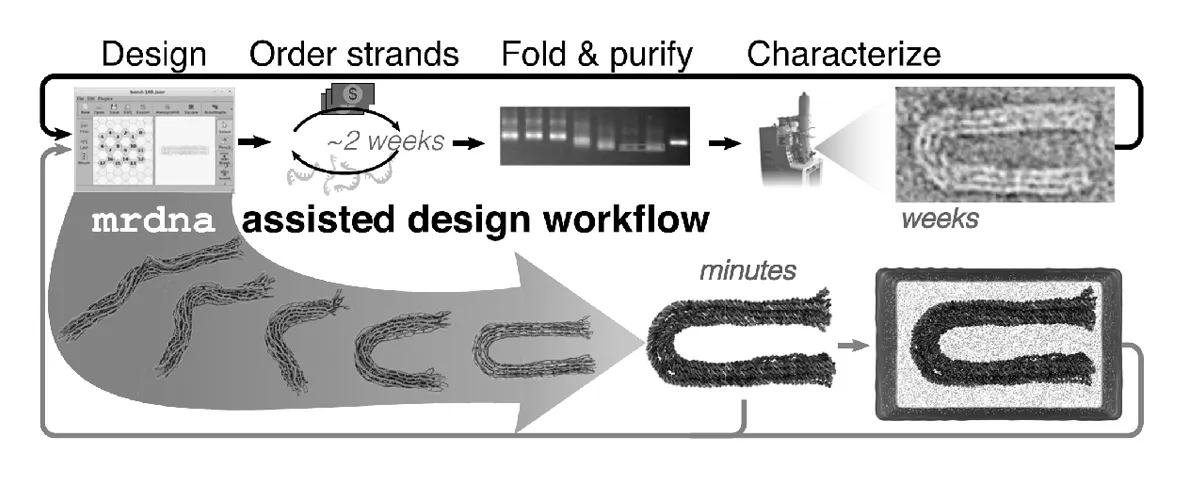
图4 MrDNA设计并合成DNA纳米结构示意图[22]
除此之外,其他的DNA纳米结构的在线设计及分析平台还包括,如Daedalus(http://daedalus-dna-origami.org/)[42],Perdix(http://perdix-dna-origami.org/)[20],TALOS(http://talos-dna-origami.org/)[19],METIS(https://metis-dna-origami.org/)[18],scadnano(https://scadnano.org/)[43]和TacoxDNA(http://tacoxdna.sissa.it/)[21]等,需要根据目标DNA纳米结构的不同,选择合适的设计软件.
2 DNA纳米结构合成方法
为了实现更精细的控制和合成更复杂的结构,科学家们先后提出了几种不同的组装方法,主要包括DNA tiles(瓦片)法[44]、DNAorigami(折纸)法[10]和DNAbricks(砖)法[45-46].
2.1 DNA瓦片法
基于碱基互补配对原则,DNA单链可以通过弱的作用力配对形成一定数目DNA双螺旋片段并列的瓦片结构,通常称为tiles或motifs.利用DNA瓦片裸露出来的黏性末端(图5a),可以将一定数量的DNA瓦片链接起来,形成从一维到三维的纳米结构.常见的瓦片结构包括:n-分支结构、DX-tile、星状结构模块等.
2.1.1n-分支结构(n-arm junction)
纽约大学Seeman教授受基因重组Holliday中间体的启发(如图5b所示),认为可以利用带有黏性末端的DNA双螺旋结构合成DNA分支结构.1983年,Seeman等人成功合成了DNA的四分支(four-armjunction)结构[47](如图5c所示).DNA四分支结构由四条DNA单链通过不完全互补配对构成,是每条DNA单链与相邻的两条单链部分结合形成的.DNA四分支结构与DNA双螺旋结构相比表现出更高的结构刚性,而且每个DNA四分支结构都有四个未配对的DNA黏性末端.这种黏性末端有可能利用DNA四分支结构进一步通过碱基互补配对构成更大规模的网格(图5c),可满足作为构建三维周期网络的基本构件的基本要求[48].随后,科学家们在实验室合成了一系列的n-分支结构,如5-分支结构[49]、6-分支结构[49]、8-分支结构[50]、12-分支结构等[50],但这些简单的结构灵活性较大,不利于相互连接得到更大的结构[51-52].

图5 n-分支结构示意图[22]
2.1.2 DX-tile
DNA构建模块需要有一定的刚性才能组装成更大的结构,但DNA分支结构显然不能满足这一要求.Seeman等人通过引入两个DNA双螺旋之间的交叉构筑了“DNA double-crossover molecule”(DX tile),主要包括5种DX-tile,3种平行取向的DPE、DPOW、DPON结构和2种反平行的DAE和DAO[44](如图6所示).其中DPE、DPOW、DPON结构不稳定,而DAE和DAO能以稳定的双交叉结构存在[53].DX tile因有两个稳定的crossover连接,因此结构刚性比线性的DNA双链更优[54],能够进一步通过设计黏性末端,就可以将不同类型的DX tile进行特异性结合,进而组装成周期性的结构[55].
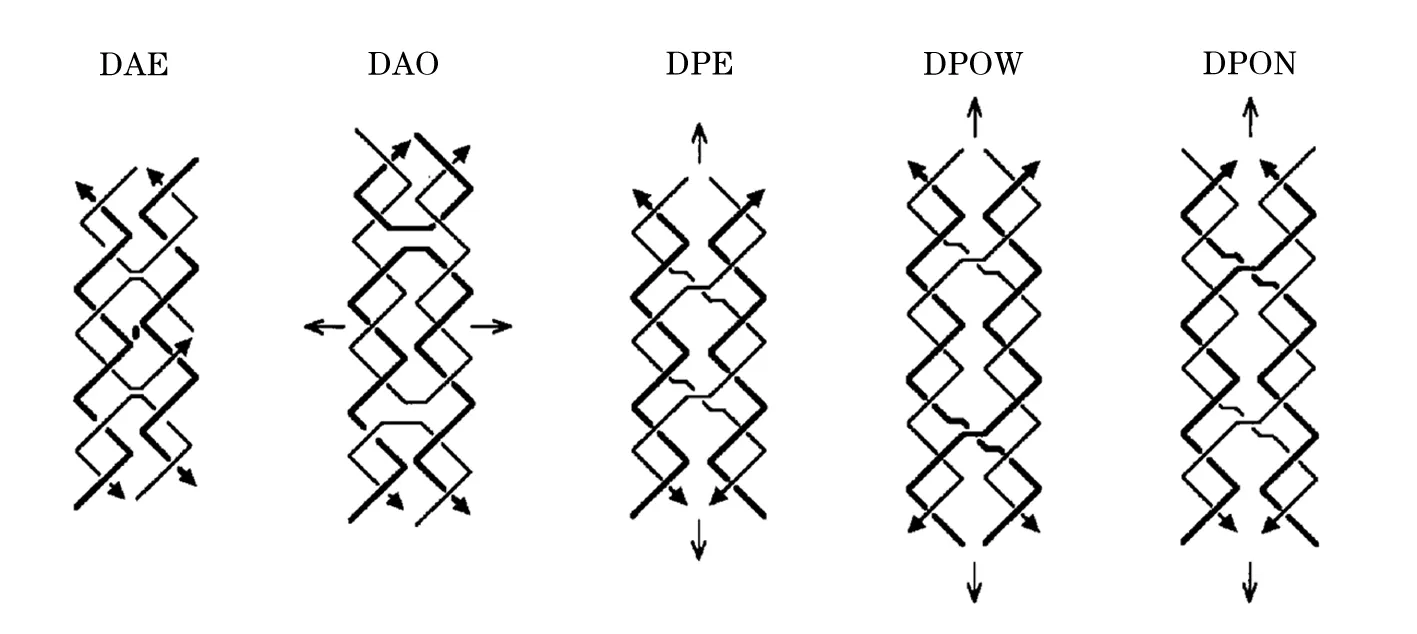
图6 5种不同排列的双交叉结构示意图 [44]
2.1.3 星状结构模块(n-point star)
为了合成复杂的DNA多面体,需要设计更加稳定的DNA结构模块,“n-point star”即“星状结构法”是科学家们在分支结构的基础上,通过超分子自组装策略实现的[56].根据目标多面体的顶点度数不同,设计不同的星状结构,如四面体的顶点度数是三度的,就必须设计3-point star.2008年毛承德等人设计了3-星状结构模块[56].该模块由7条DNA相互缠绕并从中心延伸出三个分支,每个分支由两条通过Holliday交叉连接的DNA双螺旋,剩余的1条DNA链在中心连接三个分支,通过调节中心连接的柔性,可再将设计好的星状结构组装成二聚物.这些二聚物会进一步组装成多面体结构,整个组装过程可以一步且高效完成.如图7所示,给出了利用“3-星状结构”合成DNA立方体的示意图[57].此外,还利用“3-星状结构”自组装得到了DNA四面体[56],十二面体和三十二面体[28],“4-星状结构”被用于合成DNA八面体[13],而“5-星状结构”被用于合成二十面体[58].

图7 “3-星状结构”及合成立方体示意图 [57]
2.2 DNA折纸术(DNA Origami)
DNA“折纸术”的合成策略是加州理工大学的P.W.K.Rothemund于2006年提出的[10].该方法构建了尺寸有限的二维和三维纳米结构,被视为一个里程碑.迄今为止,科学家们运用DNA折纸技术先后设计并合成了数量众多的大小可控、几何和拓扑结构各异、功能丰富的纳米结构.其设计原理(图8所示):设计一系列的合成寡核苷酸作为短的“订书钉链”,一条长的环状DNA单链作为“骨架链”混合,在特定位置通过碱基互补与杂交,让“订书钉链”与“脚手架链”结合,固定形状折叠出预先设计的具有固定边界和尺度的二维或三维纳米结构.通过折纸术方法可以扩展得到超大结构,能构建具有10 nm左右的微米尺寸器件.Rothemund利用M13mp18噬菌体的单链DNA(含有7 249个碱基,为目前产品化最高的DNA长单链)作为骨架链,含8个碱基的订书针链,自下而上组装得到了DNA Sierpinski三角结构[9].此外,还成功合成了方形、矩形、五角星、笑脸(图9)等对称图形[10].上海交通大学与中国科学院上海应用物理研究所合作用DNA折纸术构造了非对称的中国地图[59].

图8 DNA折纸术原理示意图 [10]

图9 DNA折纸术合成的纳米结构 [10]
DNA折纸目前有两个重要类别:一个是多层DNA折纸结构,另一个是线框DNA折纸结构.其中Rothemund提出的最原始的DNA折纸方法就属于多层折纸技术.尺寸较小的纳米结构可以将骨架链和订书针链通过一锅反应组装[60].然而,尺寸大且复杂的结构往往是由几个不同的支架链组装起来的,每个部分都必须单独组装并纯化后才能进行超级组装,实验过程复杂且需要高浓度的Mg2+才能保证结构的稳定性[61].而线框DNA折纸结构允许构筑任意的2D和3D几何形状[62-63].线框DNA折纸是基于DNA tile的DNA结构的一种变形,其优势在于只需要填充抽象几何物体边缘周围的区域,路径计算更简单且结构的最大尺寸可显著提高[64].与多层DNA折纸不同的是,线框DNA折纸结构在低浓度Mg2+和生理离子条件下也很稳定[65].
2.3 DNA砖块
DNA砖块法是一种模块化的多功能DNA纳米结构组装方式.每个DNA砖块类似于一块乐高积木,通过不同方式连接就可以构建任意形状的纳米结构,可组装高达1 GDa大小的结构[66],它们由成千上万个独特的部件组成,因此该策略特别适用于构建复杂的三维周期性纳米结构.
2012年,哈佛大学的尹鹏提出了DNA砖块法,这种方法依赖于数千个短寡核苷酸的组装[45].每个DNA砖块包含四个8 nt长的结构域,每块DNA砖长度是32 bp或者52 bp,能够结合其他四个DNA砖块(图10).纳米结构的稳定性和总尺寸依赖于单个砖结合域的长度[67].与DNA折纸术相比,DNA砖组装的结构具有更大的灵活性,设计策略不受骨架链长度的限制,而且模块化的方案也使该方法能通过简单增加或去除某些特定的链就可以构建出复杂的三维结构[68].

图10 DNA砖块法原理示意图 [45]
3 DNA纳米结构应用研究
DNA纳米技术使制造越来越复杂的纳米结构成为可能.这种制造方法是完全可编程和可复制的,因为它依赖于DNA碱基对相互作用的精确特异性.因此,DNA序列可以被合理地设计成具有明确尺寸、定制形状和多功能的结构.由于这种独特的设计适应性,DNA纳米技术已成为多种用途定制纳米材料的丰富来源.基于DNA tile或折纸术的DNA纳米结构已被用于细胞中进行药物传递[69]、生物成像[70]和病毒的检测、治疗[71]等.
4 总结与展望
随着DNA纳米技术的发展及功能强大的设计工具的研发,最终能实现无差错的DNA自组装.科学家们将进一步开发设计软件,改进合成技术实现对原子的精确操纵,从而制备纳米尺度的纳米构件和纳米器件的通用元件.DNA纳米技术已经发展成为很热门的研究领域.在纳米器件、基因诊疗、DNA计算机等领域将扮演越来越重要的角色.未来DNA纳米技术的推广和应用将带来巨大的经济效益,同时会推动相关领域的技术飞跃.当然,要实现这些目标还需要克服许多难题,还要经过漫长的产业化过程.

