采自青海湖的刚毛藻属一新种
——青海刚毛藻
赵志娟 朱 欢 熊 雄 何玉邦 敖鸿毅 吴辰熙 胡愈炘 刘国祥
(1. 南宁师范大学北部湾环境演变与资源利用教育部重点实验室, 南宁 530001; 2. 南宁师范大学广西地表过程与智能模拟重点实验室, 南宁 530001; 3. 中国科学院水生生物研究所淡水生态与生物技术国家重点实验室, 武汉 430072;4. 青海湖国家级自然保护区管理局, 西宁, 810007)
青海湖地处青藏高原东北部, 湖泊面积约4400 km2, 是我国最大的咸水湖泊(盐度约6‰—12‰)。青海湖因其独特的海拔、气候和地理位置成为我国西部著名的旅游风景区。但是自2013年以来, 每年7—9月青海湖均频频暴发丝状藻类水华, 大量黄绿色藻类漂浮于水体表面, 对水体生态系统造成了一定污染和危害。目前关于青海湖藻类的研究主要集中于浮游植物种类多样性调查、生物量监测及营养盐对藻类生长的影响等方面[1—5]。已报道过的丝状绿藻有转板藻、无隔藻、丝藻、水绵、双星藻和刚毛藻等,这些丝状藻类的调查主要依靠形态学观察, 极少结合分子序列进行分析[3]。
本研究对青海湖岸边暴发的水华丝状藻类进行样品采集, 并将其带回实验室开展显微形态观察和分子序列测定, 以分析丝状藻类所属种类及其典型特征, 以期为青海湖的水环境治理和生态建设提供理论参考和依据。
1 材料与方法
1.1 样品采集
本研究分别在2013—2015年8月对青海湖沿岸发生丝状藻类水华的位点进行样品采集, 采集后的样品同时进行3种方式保存: 10%的甲醛固定保存、干燥保存及低温新鲜样品保存。所有保存样品均带回实验室处理, 样品编号依次为QH1401、QH1402、QH1403、QH1404、QH1501、QH1502、QH1504和QH1505, 甲醛固定的样品保存于中国科学院水生生物研究所。
1.2 形态观察
显微形态: 在体式镜(KL1500 LCD; Zeiss, Göttingen, Germany)下用解剖针小心将目的藻丝分离,置于磷酸盐缓冲液(PBS, pH 7.0)中反复清洗3次, 去除表面附着的杂质。将干净藻丝置于光学显微镜下进行显微形态观察并测量拍照。关注的形态特征包含藻丝分枝类型、直径大小、细胞长宽比和有无固着器等, 同时收集干净藻丝以备DNA提取使用。
标本制作: 将新鲜藻样置于放有清水和标本纸的干净操作盘中, 在解剖镜下用解剖针轻轻分开各分枝, 吸走多余水分, 待标本纸处于半干状态时涂上适量胶水, 自然风干压制成标本, 做好的干标本可用于藻丝宏观形态观察, 标本保存于中国科学院水生生物研究所。
1.3 DNA提取
单细胞法提取藻丝DNA: 将收集的丝状绿藻用ddH2O反复洗涤3—4次, 置于显微镜下镜检。镜检后的藻丝转移至滴有缓冲液的载玻片上, 用解剖刀切割单个细胞, 使内含物释放; 用移液枪收集细胞内含物并转移至已灭菌的PCR管中。加入合适比例的蛋白酶K, 混匀, 置于55℃水浴0.5h; 最后高温(95℃, 5min)灭活蛋白酶K, 即获得样品总DNA。
1.4 PCR扩增及系统发育分析
PCR扩增核糖体小亚基编码基因(Small subunit ribosomal encoding gene, SSU)和大亚基编码基因(Large subunit ribosomal encoding gene, LSU)的方法参照Boedeker等[6]和Leliaert等[7]对刚毛藻目藻样的处理方法[6,7], 参照Hayakawa等[8]的方法扩增内转录间隔区ITS序列。PCR产物送生物公司(擎科生物, 武汉)测序。对于直接测序效果不佳的PCR产物, 用pMD18-T(宝生物, 大连, 中国)载体连接, 转化至大肠杆菌E.coliDH5α中, 培养筛选后重新PCR并测序。测序结果使用Seqman软件拼接(Swindell and Plasterer, 1997)[9]。利用Mafft 7.2(Katoh and Standley, 2013)进行序列比对[10], 使用Bioedit 7.0(Hall, 2004)手工校正[11], 人工校正后的序列提交GenBank数据库。对ITS序列(序列间差异较大)开展独立进化分析, 对于SSU和LSU序列(序列较为保守)开展联合进化分析。分析前首先进行SSULSU联合序列不相合长度差异检验,P>0.05, 表明数据集可联合分析。以上数据集分别采用似然法(ML)和贝叶斯算法(BI)对数据进行运算。前者通过RaxML(Stamatakis等[12])软件完成, 后者通过MrBayes 3.1.2完成(Huelsenbeck 和 Ronquist[13])。RaxML树和BI贝叶斯树分别在相应分支上获得自展支持值(BP)和后验概率值(PP)。
2 结果
2.1 新种确认
青海刚毛藻Cladophora qinghaiensisZ.J.ZHAO & G.X. LIU, sp. nov. 图版 Ⅰ-a—h
植物体生长于咸水环境, 盐度范围: 6‰—12‰。藻丝纤细柔软, 呈绿色或浅绿色, 分枝旺盛, 具明显的顶端生长和居间生长(图版Ⅰ-a—c)。叶绿体网状, 周生。大多数藻丝通过固着器着生于岸边沙石等基质上(图版Ⅰ-d)。细胞均匀, 多圆柱形, 顶细胞逐渐变细, 末端为钝顶末梢(图版Ⅰ-e)。细胞近顶端常产生分枝, 与主枝形成锐角(45°或更小), 典型的伪二歧分枝或者下方伪二歧分枝(图版Ⅰ-f)。随着生长, 有些细胞顶部可以产生3个、4个甚至5个分枝(图版Ⅰ-g)。有时植物体末端分枝或细胞会呈现轻微弯曲或卷曲现象(图版Ⅰ-h)。植物体细胞直径较小, 主轴细胞直径30.0—90.0 μm, 细胞长宽比2.4—8.0。分枝细胞直径25.0—70.0 μm, 长宽比4.0—12.3。顶端细胞直径25.0—50.0 μm, 长宽比3.6—12.0。温度变化、水流冲击或者植物体老化等因素可能导致植物体断裂或变短, 进而形成丝状团块漂浮于水面上, 颜色趋于黄色或浅黄色。漂浮的藻丝有时也继续保持顶端或居间生长。
生境及分布: 咸水(盐度6‰—12‰), 着生, 生长后期或漂浮。
新种词源学: 新种主要基于样品采集地命名。
模式标本: QH1501。
模式标本产地: 青海湖(N36°38′25″, E100°27′01″)。
模式标本保存地: 酒精及甲醛标本均保存于中国科学院水生生物研究所。
样品编号: QH1401, QH1402, QH1403, QH1404,QH1501, QH1502, QH1504和QH1505。
2.2 系统发育分析
基于SSU-LSU序列的联合进化分析(图 1): 序列比对产生了一个包含53条SSU-LSU rDNA序列的矩阵, 以细弱根枝藻Rhizoclonium subtile和分枝根枝藻R. ramosum作为外类群。序列手工校正后的矩阵长度为2230 bp。其中SSU全长1651 bp, 有保守位点(Conserved sites)1594个(96.5%), 45个简约信息位点(Parsimony-informative sites)。LSU全长579 bp, 有保守位点496个(85.7%), 57个简约信息位点。碱基组成分析结果显示平均的碱基组成为A=25.3%, T=23.2%, C=22.9%, G=28.7%, A+T的含量(48.4%)略低于G+C的含量(51.6%)。
基于ITS 序列的刚毛藻科系统发育分析(图 2):本研究共选取86条ITS序列用于刚毛藻科的系统进化分析, 选取细弱根枝藻R. subtile、厚壁根枝藻R.pachydermum和分枝根枝藻R. ramosum作为外类群。序列比对后产出一个长度为1484 bp的基因序列矩阵。其中有820个可变位点(Variable sites),713个简约信息位点(Parsimony-informative sites)。碱基组成分析结果显示平均的碱基组成为A=20.6%,T=21.9%, C=29.6%, G=27.9%, A+T的含量(42.5%)略低于G+C的含量(57.5%)。此外, 碱基的转换/颠换偏好为0.89。
基于SSU-LSU和ITS序列所构建的进化树中均包含了淡水、咸水和海洋刚毛藻样品, 这两个系统发育树的拓扑结构基本一致。其中淡水刚毛藻(以团集刚毛藻C. glomerata为代表)单独聚于一支, 位于进化树顶部, 与咸水刚毛藻(以散束刚毛藻C.vagabunda为代表)独立开来。而海洋刚毛藻(以苍白刚毛藻C. albida为代表)则位于进化树的基部。在SSU-LSU联合分析构建的进化树中, QH1401和QH1402与其他5株青海湖的刚毛藻样品存在一定遗传差异, 其中QH1401/QH1402的SSU序列与其他样品SSU序列存在1个碱基的差异、LSU序列存在4个碱基的差异。所有青海湖刚毛藻样品(QH1401、QH1402、QH1403、QH1404、QH1501、QH1502、QH1504和QH1505)的ITS序列100%一致, 无遗传差异。3个分子标记比对结果表明青海湖刚毛藻样品序列具有高度相似性, 碱基差异未达到种以上水平。SSU-LSU联合分析显示青海刚毛藻和达尔马提亚刚毛藻C.cf.dalmatica和帕里亚迪刚毛藻C.cf.parriaudii具有较近亲缘关系。青海刚毛藻与咸水刚毛藻聚为一个大支, 形成姐妹类群, 且支持率较高(1.00/88, 0.97/62)。在ITS进化树中, 青海刚毛藻C. qinghaiensis与史氏刚毛藻C. stimpsonii有较近的亲缘关系, 支持值1.00/95。综合比较构建的2个进化树, ITS构建的进化树显示了更大的基因序列差异, 各分支区分明显, 支持度高。
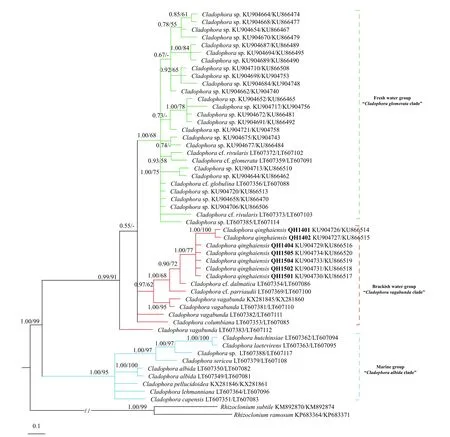
图 1 基于核糖体小亚基SSU和大亚基LSU联合构建的系统树Fig. 1 Molecular phylogenetic tree inferred based on sequences of SSU and LSU of the nuclear rDNA
3 讨论
3.1 青海刚毛藻和相似种的形态学比较
青海刚毛藻具有刚毛藻属的典型特征: 即植物体通过顶端分裂或居间分裂进行生长, 具分枝, 色素体紧密排列在细胞侧壁, 形成一个网状结构。细胞长宽比较大, 细胞核多数, 蛋白核多数[14,15]。同时青海刚毛藻也具有咸水/海水刚毛藻类群的一些独特特征, 如一个细胞顶部有时可同时形成4—5个分枝, 淡水类群一个细胞顶部通常则仅形成2—3个分枝[14]。此外, 青海刚毛藻藻丝分枝还具有内折或轻微弯曲现象, 呈镰刀形, 该特征也出现于散束刚毛藻、达尔马提亚刚毛藻和亮刚毛藻C. laetevirens等刚毛藻中[14]。但此特征并不在同一个种的所有样品或者培养样品中均出现, 说明分枝内折现象具有一定可塑性, 可能是受水流波动影响所导致。
通过和目前已知的咸水刚毛藻种进行比较, 青海刚毛藻具有一些较为细微的差异性特征。最明显的区别是细胞直径大小。史氏刚毛藻、散束刚毛藻、达尔马提亚刚毛藻、束生刚毛藻C. fascicularis和亮刚毛藻等的细胞直径均大于青海刚毛藻[14]。帕里亚迪刚毛藻植物体纤细, 主枝和分枝细胞(14—20 μm)直径小于青海刚毛藻[14]。此外, 分布、颜色及分枝特征也存在种间差异, 史氏刚毛藻主要分布于沿海和潮间带地区, 藻丝呈绢丝状, 分枝多偏生[16,17]。细弱刚毛藻C. gracilis主要生长与潮间带石沼中, 藻丝亮灰绿色, 密集丛生, 有较多侧生小枝[14,16,17]。
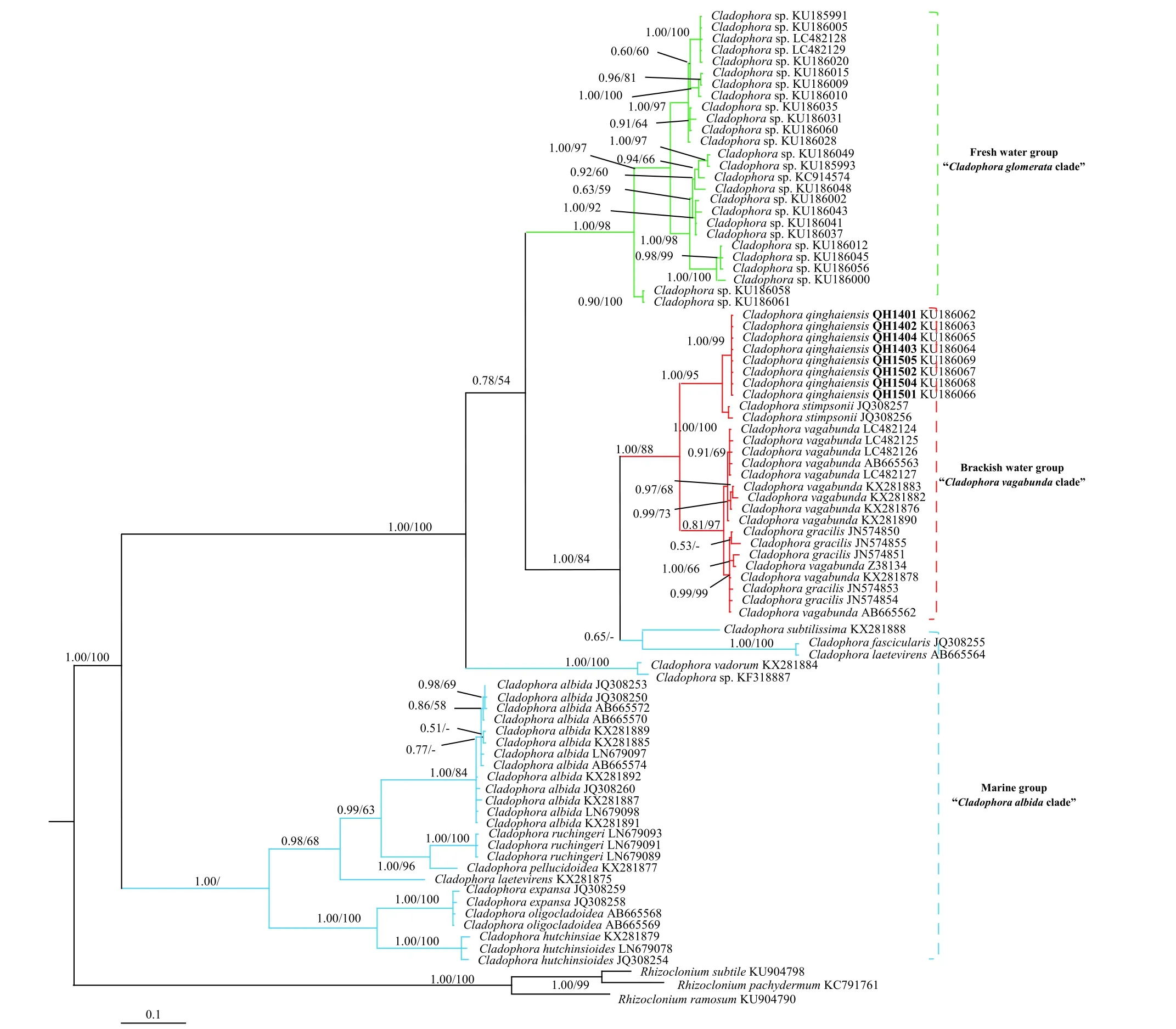
图 2 基于内转录间隔区ITS序列构建的系统树Fig. 2 Molecular phylogenetic tree inferred based on sequences of ITS
3.2 刚毛藻类群的系统进化分析
从系统发育树看, 刚毛藻属目前可划分为3大类群: 淡水类群(Freshwater group)、海洋类群(Marine water group)及咸水(过渡性)类群(Brackish water group)。海洋类群位于进化树的基部, 淡水类群位于进化树的顶部, 咸水支系则居于二者之间。这一进化结果进一步印证了刚毛藻是一个起源于海洋, 并逐渐向淡水过渡的类群[18—20]。Hayakawa等[8]对不同类型刚毛藻进行的盐度适应性实验显示团集刚毛藻在盐度低于10‰的水环境有较好的生长速率; 束生刚毛藻和亮刚毛藻在盐度较高的水环境(海洋)中适宜生长, 盐度低于16‰时, 生长速率降低;散束刚毛藻则对盐度的耐受范围较广。这表明刚毛藻在进化过程中, 面对不同生境产生了不同类型的种类, 这些种类具有各自的生理生态适应范围,这一范围反过来又会影响刚毛藻的分布。因此本研究推测, 青海刚毛藻这一种类的出现可能是刚毛藻适应青海湖这一特殊生境的结果。
与形态特征相比, 分子数据可以较好的用于区分不同的刚毛藻类群。朱欢等[21]曾借助分子手段将淡水刚毛藻分为8个分支。尽管借助形态特征来清晰区分青海刚毛藻、史氏刚毛藻和散束刚毛藻较为困难, 但在系统进化树上三者则明显分开, 形成不同种。此外, 刚毛藻属样品的SSU-LSU序列相对保守, 不同种间碱基差异较小, ITS序列却呈现了较大的序列差异, 说明在今后对刚毛藻进行系统发育分析时选择进化速度相对较快的ITS序列可能更为有效。
基于以上形态和分子结果, 本研究建议将采集于青海湖的刚毛藻作为一个新种, 同时定名为青海刚毛藻。尽管刚毛藻类群形态具有一定可塑性, 种的鉴定相对困难, 但相信在今后更多分子数据和形态数据提供的基础上, 刚毛藻的分类将日益清晰。

图版Ⅰ 青海刚毛藻PlateⅠ Cladophora qinghaiensis sp. nov.

