人源核酸烷基化损伤修复酶ALKBH3在肿瘤进展和治疗中的作用
胡静燕,张琳,张良
上海交通大学基础医学院药理学与化学生物学系,上海200025
维持天然的DNA结构对于细胞实现其正常生理功能至关重要。然而,来自环境和内源性的烷基化试剂不断通过产生各种烷基化碱基修饰来造成DNA损伤,导致基因突变,诱发细胞毒性,引起细胞死亡[1-2]。因此,预防和修复DNA碱基的烷基化损伤对于细胞的生存具有重要意义。人源核酸烷基化损伤修复酶ALKBH3(alphaketoglutarate-dependent dioxygenase homolog 3)隶属于亚铁(Fe2+)和α-酮戊二酸(α-ketoglutarate,α-KG)依赖型双加氧酶——烷基化DNA修复蛋白(alkylated DNA repair protein,AlkB)家族。该家族内大多数同工酶均属于核酸碱基去甲基化酶,对于保护细胞免受DNA损伤、调控基因表达和蛋白质翻译有重要作用。而ALKBH3作为AlkB家族中的一个成员,特异性识别单链DNA(ssDNA)或RNA上的N1-甲基腺嘌呤(1mA)和N3-甲基胞嘧啶(3mC)等烷基化修饰并进行去甲基化修复,在维持细胞基因组稳定和调控RNA转录中具有重要作用,其表达和功能的异常与多种癌症的发生发展密切相关。本文就人源ALKBH3的近年研究进展进行综述。
1 AlkB蛋白及其家族成员简介
1977年,Samson和Cairns发现大肠埃希菌基因组alkB基因编码的AlkB表达与其对烷基化试剂的适应性响应有关[3]。2001年,AlkB蛋白被证明是典型的Fe2+和α-KG依赖型双加氧酶,可通过氧化去烷基化来保护细菌基因组免受烷基化损伤[4]。AlkB对多种核酸烷基化损伤都具有广泛修复作用,主要催化ssDNA和双链DNA(dsDNA)上1mA和3mC的去烷基化修复反应[4]。另外,AlkB对dsDNA上N1-甲基鸟嘌呤(1mG)和N3-甲基胸腺嘧啶(3mT)烷基化修饰以及1,N6-亚乙烯基腺嘌呤和3,N4-亚乙烯基腺嘌呤环化修饰也有相对较弱的去甲基化活性[5-6]。这些烷基化修饰的共同点是会破坏dsDNA中碱基互补配对,进而破坏DNA结构,干扰基因复制、转录和翻译。因此可以推断,AlkB对于维持DNA结构和细菌正常生理功能至关重要,这也激发了人们对于研究AlkB在人体内同工酶及其生物功能的热情与兴趣。
截至目前,人体内共发现9类AlkB家族高度保守的同工酶,分别命名为ALKBH1~ALKBH8和FTO[7]。在氨基酸一级序列上,这些酶均包含His1-X-Asp/Glu-Xn-His2亚铁结合位点[8-9];而在蛋白质三级结构上,它们均具有羧基端果冻卷样反向平行β折叠层组成的催化核心区域、独特的核苷酸识别盖子结构域和氨基端的扩展结构域。尽管它们的一级序列和三级结构高度保守,研究[10]发现它们催化的生理底物及对应的生物学功能存在较大差异。ALKBH1催 化 位 于DNA转 录 泡(transcription bubble)ssDNA区域上N6-甲基腺嘌呤(6mA)及位于转运RNA(tRNAiMet)上m1A的甲 基 修饰 去 除[10-12];ALKBH5和FTO催 化 位 于 信 使RNA(mRNA)或 小 核RNA(snRNA)上的N6-甲基腺嘌呤(m6A)上甲基修饰的去除[13-15];ALKBH2和ALKBH3分别催化ssDNA/RNA或dsDNA上1mA和3mC等 烷 基 化 修 饰 的 去甲 基 化[16-17]。ALKBH4可能是催化肌动蛋白中单甲基化赖氨酸(K84me1)去甲基化酶[18]。ALKBH7可能与烷基化和氧化诱导的程序性坏死有关[19-20]。ALKBH8是tRNA去甲基化酶,与人类膀胱癌有密切的联系[21]。ALKBH6的生物学功能目前尚不清楚。
2 ALKBH3 的催化机制研究
在人体内的AlkB家族同工酶中,ALKBH3是由11号常染色体alkB3基因(Chr11∶43,880,807~43,920,275)编码的全长286个氨基酸的蛋白,同ALKBH2一起参与人体基因组上1mA和3mC烷基化修复并保护细胞免受烷基化损伤,是一种能补偿大肠埃希菌AlkB体内生物功能的酶。ALKBH3催化烷基化修复的分子机制普遍认为与AlkB的催化分子机制类似(图1A)。首先O2分子与Fe2+离子在活性中心结合,形成Fe3+-O2-超氧自由基阴离子复合物,并进一步作用于与复合物附近的α-KG,使其氧化脱羧生成琥珀酸和CO2。CO2从活性位点释放后,反应产生的Fe4+-超氧配体复合物迁移到核酸底物上目标烷基基团附近,将其羟基化形成不稳定的羟基化中间体,并迅速脱醛基回到未修饰的碱基状态,完成对DNA烷基化的修复[8,22]。
尽管ALKBH2和ALKBH3都催化1mA和3mC的烷基化修复,但是它们在底物的识别上具有截然不同的选择性。通过测定人源和鼠源ALKBH3和ALKBH2对人工合成的含有1mA或3mC修饰的ssDNA和dsDNA催化的稳态动力学数据显示,ALKBH3对含有修饰的ssDNA或RNA具有显著的识别和催化作用,且对3mC修饰的修复活性要略高于1mA。然而ALKBH3对含有修饰的dsDNA几乎无催化活性[23]。相比之下,人源和鼠源的ALKBH2对dsDNA上1mA和3mC修饰的催化活性最高,对ssDNA上修饰的催化活性较弱,对RNA则无显著催化作用[24]。这些实验结果证实,ALKBH3具有与其同工酶ALKBH2截然不同的底物选择性,它对ssDNA和RNA上的1mA和3mC修饰具有特异性识别和催化的生物功能,但是决定其底物选择性差异的分子机制至今还不明晰。
为了阐明ALKBH3的催化和底物选择分子机制,2006年Sundheim等[25]解析并报道了ALKBH3催化结构域截短体(N端截短69个氨基酸)与配体[Fe2+和α-KG类似物2-氧戊二酸盐(2-OG)]的复合物晶体结构(pdb号:2IUW)(图1B)。该结构显示,ALKBH3催化结构域由14个β-链和2个α-螺旋构成,其中8条β链(β7~β14)形成了AlkB家族经典的果冻卷样反向平行β折叠层结构,这是包括Fe2+和α-KG依赖型双加氧酶家族在内的许多金属结合和非金属结合蛋白的典型特征。其中,β1位于果冻卷折叠中稍小的β-片层的底端;β2、β3和β6分别位于另一个更大的β片层两端;β4和β5形成了一个独特的发卡结构;紧靠在β6和α3之间的短α2螺旋形成了活性位点上方的盖子形状,这是AlkB蛋白家族特有的核酸识别盖结构,且在家族内不同同工酶之间呈现出了一定的差异性,可能是该家族酶实现不同底物选择性的关键结构域;带正电荷的凹槽穿过β4-β5发夹与果冻卷中心之间的活性部位,构成了DNA/RNA结合沟槽。ALKBH3的活性依赖于与Fe2+和α-KG相互作用的关键氨基酸,例如与Fe2+形成配位螯合键的高度保守氨基酸His191、Asp193和His257;与α-KG类似物2-OG上5号位羧基形成盐桥和氢键相互作用的氨基酸Arg269、Tyr181和Asn271;与2-OG上1号位羧基形成氢键相互作用的氨基酸Arg275等等。除此以外,值得注意的是,与2-OG上1号位羧基形成疏水相互作用的氨基酸Leu177被发现存在自羟基化,推测其可能在没有初级底物时发挥一定的抑制功能,保护细胞免受有害的氧化副反应。
为了探究ALKBH3不同于ALKBH2的底物选择性分子机制,解析ALKBH3-ssDNA或RNA的复合物结构是最直接的技术手段。然而由于AlkB家族蛋白普遍存在与底物结合力弱的特点,ALKBH3-ssDNA复合物至今尚未被解析[8]。但是Yang等[26]通过发展能共价交联ALKBH2的非天然核苷酸技术,解析了ALKBH2共价交联含有1mA的dsDNA复合物结构。随后,Chen等[16]进一步对ALKBH3和ALKBH2的蛋白序列和结构进行了分析(图1C)。该研究发现,这2种蛋白质在结构上最显著的差异是核苷酸识别盖子和β-发夹结构。ALKBH2的氨基酸V101、F102和G103的侧链选择性地插入dsDNA的主沟槽中,起到了稳定底物的作用。将ALKBH3结构叠合到ALKBH2-dsDNA复合结构上后,发现ALKBH3相应位置的氨基酸R122、E123和D124与dsDNA发生了严重的空间位阻冲突,意味着ALKBH3上这3个具有强亲水性的氨基酸(缩写为RED)所在的发卡结构可能形成一个庞大的盐桥,选择性地阻止了dsDNA结合并进入底物结合位点和进一步底物碱基的翻转。研究进一步将ALKBH3中的RED氨基酸与ALKBH2中对应的VFG氨基酸进行交换,发现ALKBH3获得了与ALKBH2类似的对dsDNA的结合能力以及对dsDNA上修饰碱基的催化 酶 活。另 外,Monsen等[27]在 交 换ALKBH3和ALKBH2的β-发夹结构后得到了对于dsDNA具有更强催化活性的ALKBH3杂交蛋白,证实了该发夹结构对于去甲基的酶活性和底物选择特异性非常重要,ALKBH3 β-发夹结构上的正电荷和极性氨基酸会阻止dsDNA结合与识别。
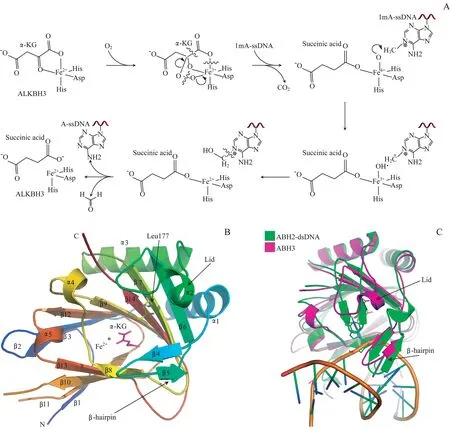
图1 ALKBH3的催化机制和结构Fig 1 Catalytic mechanism and structure of ALKBH3
3 ALKBH3 的生物功能研究
针对ALKBH3底物选择性的生物化学和结构研究结果均显示,ALKBH3主要参与人体内ssDNA和RNA上的1mA和3mC烷基化修饰的修复。然而,在人体基因组主要以dsDNA的形式存在,那么如何体现ALKBH3针对ssDNA烷基化的修复功能?Dango等[28]发现,ALKBH3与激活信号协整复合物(activating signal cointegrator complex,ASCC)可共同作用形成复合物。ASCC3作为ASCC最 大的 亚 基编 码了3′-5′DNA解 旋 酶,可 催化dsDNA解旋产生ssDNA,从而使ALKBH3发挥对ssDNA的特异性修复。对于ASCC的进一步研究揭示了一条泛素依赖的ALKBH3介导的DNA去甲基化修复的信号通路,其中ASCC2具有泛素结合结构域,通过上游的泛素化作用将ALKBH3-ASCC3招募到DNA的烷基化损伤位点,从而实现其修复作用[29]。这一发现首次提出了细胞内ALKBH3发挥DNA修复作用的上游信号通路,对进一步理解和阐明ALKBH3的生理功能和调控机制有重要的意义。
不同于ALKBH2仅仅出现在细胞核内,ALKBH3在细胞核和细胞质内均有分布[7],这可能与ALKBH3更为丰富的生理功能有关。除ssDNA外,ALKBH3对RNA也具有显著的去甲基活性。尽管目前对于RNA甲基化损伤可能会造成的影响尚不清楚,然而,研究人员发现核糖体RNA中异常的m1A修饰会影响密码子-反密码子的结合[30],导致逆转录酶错误编码;m1A和m3C对碱基互补配对的干扰还可能会影响翻译过程[24]。总之,RNA的异常甲基化可能会破坏细胞正常代谢,因此细胞自身的防御系统应当会对这些甲基化损伤进行主动的修复。Li等[31]发现,ALKBH3可以降低mRNA的m1A修饰水平,其特异性识别的m1A位点主要分布于mRNA的5′非翻译区,这不仅提示ALKBH3具有RNA烷基化修复功能,更暗示ALKBH3可能还具有不同于目前已知的烷基修复酶功能的转录调节功能。此外,Ueda等[32]确证了ALKBH3能够有效地对RNA的m1A和m3C进行去甲基化修复,并通过体外翻译实验证实由ALKBH3去甲基化的tRNA具有更高的翻译效率。Chen等[33]则进一步证实ALKBH3是tRNA的m1A和m3C去甲基化酶,并具有促癌细胞增殖、迁移和侵袭的生物功能。其具体机制猜测为ALKBH3针对tRNA的m1A去甲基修复造成了tRNA稳定性下降,从而在反密码子区域周围产生许多tRNA-来源的小RNA(tDRs),这种tDRs可以加强核糖体的组装并且防止细胞凋亡,从而促进肿瘤生长和侵袭。
基因组DNA在受到外界因素影响产生的1mA、3mC、1mG、3mT等烷基化修饰会破坏Watson-Crick碱基互补配对,从而破坏DNA的空间结构,影响基因表达,产生遗传突变或者细胞毒性作用[34]。而ALKBH3是人体内主要对这些烷基化损伤进行修复的酶,在维持DNA正常结构、实现细胞正常的生理功能上有积极正面的重要意义[5-6,17]。在癌症治疗中应用较为广泛的多种化学治疗(化疗)药物,如用于治疗白血病的白消安(busulfan)和用于治疗脑瘤的替莫唑胺(temozolomide)等,是通过使DNA发生烷基化损伤从而诱发癌症细胞的死亡。而ALKBH3在癌症细胞内的存在和表达会修复外源性药物造成的烷基化损伤,使得癌症细胞得以抵抗药物的作用继续生存,这又体现了ALKBH3相对的负面作用。有研究[35]发现ALKBH3可以与一些高活性基因启动子的转录起始位点相结合(如启动子近端暂停RNA聚合酶Ⅱ和增强子的位置),抑制高表达基因转录相关的DNA损伤,从而在ALKBH3过表达癌细胞中维持基因组完整性。
从上述的内容中可以发现,ALKBH3不仅仅可能通过DNA烷基化损伤修复的功能促进癌细胞的生存与增殖,也可以通过对RNA甲基化修饰的催化作用调控转录、翻译的过程,进而影响癌细胞的命运。因此,对ALKBH3生理作用以及调控机制的深入研究在人类对抗癌症的斗争中也许有着非常重要的意义。
4 ALKBH3 与癌症
临床研究发现,ALKBH3在多种人类实体瘤中均呈现出高表达。Konishi[36]在对影响人前列腺癌的基因进行研究的过程中,发现一个基因在前列腺癌中表现出较高的mRNA表达,将其命名为前列腺癌抗原-1(PCA-1)。进一步数据库分析和比较发现,PCA-1即ALKBH3。ALKBH3的表达水平与非激素依赖性前列腺癌呈显著正相关,而ALKBH3 shRNA敲低的前列腺癌细胞形成的肿瘤明显小于对照,这提示ALKBH3是潜在的抗前列腺癌,尤其是抗对激素治疗和化疗具有较强抵抗力的去势耐药前列腺癌(castration-resistant prostate cancer,CRPC)的全新药物靶点。靶向ALKBH3开发CRPC治疗剂将具有广大的临床应用前途[37]。
除前列腺癌外,ALKBH3还被发现在头颈部鳞状细胞癌(head and neck squamous cell carcinoma,HNSCC)、胰腺癌、肺癌、卵巢癌和乳腺癌等多种人类实体瘤中高表达。ALKBH3的表达水平与HNSCC肿瘤的大小有直接的关系[38]。Yamato等[39]发现ALKBH3通过支持凋亡抵抗和血管生成而促进胰腺癌的发生,ALKBH3的沉默可以下调血管内皮生长因子(vascular endothelial growth factor,VEGF)的表达,抑制体内血管生成。siRNA诱导的ALKBH3沉默可以有效地诱导肺腺癌细胞在体内和体外的细胞衰老和生长抑制[40]。Woo等[41]还发现在卵巢癌和乳腺癌中,ALKBH3对细胞因子CSF-1 mRNA上的m1A的去甲基化作用增加了它的稳定性、延长它的半衰期,而这种细胞因子CSF-1的作用通常与卵巢癌和乳腺癌的预后不良相关;过表达ALKBH3在增加CSF-1表达的同时也增加了癌细胞的侵袭程度,表明ALKBH3对RNA的去甲基作用可能会间接地影响肿瘤的发展。除此之外,临床病例的研究还发现ALKBH3对人肾细胞癌[42]、肝细胞癌[43]的发生、发展具有重要的作用。因此,ALKBH3不仅是抗肿瘤的潜在药物靶标,还是多种实体瘤的全新临床病理生物学标志物[40]。
由于ALKBH3在肿瘤发生发展中体现出的重要作用,靶向ALKBH3设计和发现抑制剂小分子成为抗肿瘤药物发现的新策略。目前共有3类ALKBH3非共价抑制剂小分子被报道,但由于缺乏ALKBH3-抑制剂复合物晶体结构数据,其抑制机理并未完全明晰。2014年,Nakao等[44]通过随机化合物库筛选和结构优化改造的方法得到了第一个ALKBH3小分子抑制剂,命名为HUHS015;体外实验显示该抑制剂在浓度为10μmol/L时,能达到ALKBH3酶活性61%的抑制效果;体内实验发现HUHS015能够抑制皮下移植人前列腺癌细胞DU145的小鼠模型肿瘤生长,且未发现显著不良反应。这是首次报道的靶向ALKBH3的小分子抑制剂。2018年,芳基化吲哚酮衍生物也被报道可抑制ALKBH3的烷基修复作用,通过设计、合成、筛选和评价,得到的候选物5c被认为可与ALKBH3的活性位点特异性结合,从而竞争性抑制DNA底物。细胞实验进一步证实该化合物能够抑制人肺癌细胞株A549的增殖[45]。近期,Li等[46]报道了靶向AlkB家族的广谱小分子抑制剂——大黄酸(rhein),证实了它可以在体外抑制AlkB、ALKBH2和ALKBH3的酶活性,还增加细胞对主要产生1mA和3mC病变的甲基甲烷磺酸盐的敏感性。通过结合复合物结构和体内外的实验结果,认为大黄酸是特异性的靶向ALKBH介导的DNA修复作用的抑制剂。这些ALKBH3的小分子抑制剂的发现和研究,清晰地显示出了发展ALKBH3特异性抑制剂作为全新低毒副作用抗肿瘤治疗药物的潜力,也为日后相关药物的进一步开发和研究奠定了的前期基础。
5 总结与展望
随着对ALKBH3分子机制和生物功能的研究,ALKBH3在DNA和RNA的烷基化修复,维持DNA结构稳定,调控基因复制、转录和翻译,促进肿瘤增殖和侵袭,协助肿瘤抵抗化疗试剂烷基化损伤等方面的生物功能逐步被揭示,显示了其独特而强大的生物功能,以及作为新的临床生物标志物和药物靶标在临床癌症治疗中的巨大潜力。本文对ALKBH3在这些方面的研究进展进行了综述,以期为靶向ALKBH3的抗肿瘤先导小分子开发提供思路。
目前,对ALKBH3的研究尚有几个亟待解决的问题:①对于ALKBH3底物选择特异性的研究仅仅建立在氨基酸突变和催化活性比较的基础上,其与ssDNA和RNA的特异性结合与识别的分子机制仍然有待更具有说服力的直接证据进行阐明。通过发展能共价交联ALKBH3的非天然核苷酸技术,获得ALKBH3-核酸共价交联复合物,解析其晶体结构可能是解决该问题的发展方向。②对于ALKBH3的生理功能以及调控机制的研究仍不完善,发现ALKBH3调节通路中的上下游因子可以为靶向ALKBH3的治疗提供更多的可能性。③众多的证据表明了ALKBH3可以作为癌症治疗中一个全新的药物发现靶点,ALKBH3特异性小分子抑制剂的研究才刚起步,进一步加大对靶向ALKBH3的小分子抑制剂研发有希望成为日后抗癌药物的重要研发方向之一。
参·考·文·献
[1]Sedgwick B.Repairing DNA-methylation damage[J].Nat Rev Mol Cell Biol,2004,5(2):148-157.
[2]Taverna P,Sedgwick B.Generation of an endogenous DNA-methylating agent by nitrosation in Escherichia coli[J].J Bacteriol,1996,178(17):5105-5111.
[3]Samson L,Cairns J.A new pathway for DNA repair in Escherichia coli[J].Nature,1977,267(5608):281-283.
[4]Trewick SC,Henshaw TF,Hausinger RP,et al.Oxidative demethylation by Escherichia coli AlkB directly reverts DNA base damage[J].Nature,2002,419(6903):174-178.
[5]Chen FY,Tang Q,Bian K,et al.Adaptive response enzyme AlkB preferentially repairs 1-methylguanine and 3-methylthymine adducts in double-stranded DNA[J].Chem Res Toxicol,2016,29(4):687-693.
[6]Delaney JC,Smeester L,Wong C,et al.AlkB reverses etheno DNA lesions caused by lipid oxidation in vitro and in vivo[J].Nat Struct Mol Biol,2005,12(10):855-860.
[7]Liu Y,Yuan Q,Xie L.The AlkB family of Fe(II)/alpha-ketoglutaratedependent dioxygenases modulates embryogenesis through epigenetic regulation[J].Curr Stem Cell Res Ther,2018,13(2):136-143.
[8]Yi C,Yang CG,He C.A non-heme iron-mediated chemical demethylation in DNA and RNA[J].Acc Chem Res,2009,42(4):519-529.
[9]Hausinger RP.Fe(II)/alpha-ketoglutarate-dependent hydroxylases and related enzymes[J].Crit Rev Biochem Mol Biol,2004,39(1):21-68.
[10]Liu FG,Clark W,Luo GZ,et al.ALKBH1-mediated tRNA demethylation regulates translation[J].Cell,2016,167(7):1897.
[11]Tian LF,Liu YP,Chen LQ,et al.Structural basis of nucleic acid recognition and 6mA demethylation by human ALKBH1[J].Cell Res,2020,30(3):272-275.
[12]Zhang M,Yang SM,Nelakanti R,et al.Mammalian ALKBH1serves as an N6-mA demethylase of unpairing DNA[J].Cell Res,2020,30(3):197-210.
[13]Xu C,Liu K,Tempel W,et al.Structures of human ALKBH5demethylase reveal a unique binding mode for specific single-stranded N6-methyladenosine RNA demethylation[J].J Biol Chem,2014,289(25):17299-17311.
[14]Gerken T,Girard CA,Tung YC,et al.The obesity-associated FTO gene encodes a 2-oxoglutarate-dependent nucleic acid demethylase[J].Science,2007,318(5855):1469-1472.
[15]Zhang X,Wei LH,Wang YX,et al.Structural insights into FTO′s catalytic mechanism for the demethylation of multiple RNA substrates[J].Proc Natl Acad Sci USA,2019,116(8):2919-2924.
[16]Chen BE,Liu HC,Sun XX,et al.Mechanistic insight into the recognition of single-stranded and double-stranded DNA substrates by ABH2and ABH3[J].Mol Biosyst,2010,6(11):2143-2149.
[17]Duncan T,Trewick SC,Koivisto P,et al.Reversal of DNA alkylation damage by two human dioxygenases[J].Proc Natl Acad Sci USA,2002,99(26):16660-16665.
[18]Li MM,Nilsen A,Shi Y,et al.ALKBH4-dependent demethylation of actin regulates actomyosin dynamics[J].Nat Commun,2013,4:1832.
[19]Fu D,Jordan JJ,Samson LD.Human ALKBH7is required for alkylation and oxidation-induced programmed necrosis[J].Genes Dev,2013,27(10):1089-1100.
[20]Jordan JJ,Chhim S,Margulies CM,et al.ALKBH7drives a tissue and sexspecific necrotic cell death response following alkylation-induced damage[J].Cell Death Dis,2017,8(7):e2947.
[21]Ohshio I,Kawakami R,Tsukada Y,et al.ALKBH8promotes bladder cancer growth and progression through regulating the expression of survivin[J].Biochem Biophys Res Commun,2016,477(3):413-418.
[22]Fedeles BI,Singh V,Delaney JC,et al.The AlkB family of Fe(II)/αketoglutarate-dependent dioxygenases:repairing nucleic acid alkylation damage and beyond[J].J Biol Chem,2015,290(34):20734-20742.
[23]Lee DH,Jin SG,Cai S,et al.Repair of methylation damage in DNA and RNA by mammalian AlkB homologues[J].J Biol Chem,2005,280(47):39448-39459.
[24]Aas PA,Otterlei M,Falnes PO,et al.Human and bacterial oxidative demethylases repair alkylation damage in both RNA and DNA[J].Nature,2003,421(6925):859-863.
[25]Sundheim O,VågbøCB,Bjørås M,et al.Human ABH3structure and key residues for oxidative demethylation to reverse DNA/RNA damage[J].EMBO J,2006,25(14):3389-3397.
[26]Yang CG,Yi CQ,Duguid EM,et al.Crystal structures of DNA/RNA repair enzymes AlkB and ABH2bound to dsDNA[J].Nature,2008,452(7190):961-965.
[27]Monsen VT,Sundheim O,Aas PA,et al.Divergentß-hairpins determine double-strand versus single-strand substrate recognition of human AlkBhomologues 2 and 3[J].Nucleic Acids Res,2010,38(19):6447-6455.
[28]Dango S,Mosammaparast N,Sowa ME,et al.DNA unwinding by ASCC3helicase is coupled to ALKBH3-dependent DNA alkylation repair and cancer cell proliferation[J].Mol Cell,2011,44(3):373-384.
[29]Brickner JR,Soll JM,Lombardi PM,et al.A ubiquitin-dependent signalling axis specific for ALKBH-mediated DNA dealkylation repair[J].Nature,2017,551(7680):389-393.
[30]Yoshizawa S,Fourmy D,Puglisi JD.Recognition of the Codon-anticodon helix by ribosomal RNA[J].Science,1999,285(5434):1722-1725.
[31]Li XY,Xiong XS,Wang K,et al.Transcriptome-wide mapping reveals reversible and dynamic N1-methyladenosine methylome[J].Nat Chem Biol,2016,12(5):311-316.
[32]Ueda Y,Ooshio I,Fusamae Y,et al.AlkB homolog 3-mediated tRNA demethylation promotes protein synthesis in cancer cells[J].Sci Rep,2017,7:42271.
[33]Chen ZJ,Qi MJ,Shen B,et al.Transfer RNA demethylase ALKBH3promotes cancer progression via induction of tRNA-derived small RNAs[J].Nucleic Acids Res,2019,47(5):2533-2545.
[34]Moore LD,Le T,Fan G.DNA methylation and its basic function[J].Neuropsychopharmacology,2013,38(1):23-38.
[35]Liefke R,Windhof-Jaidhauser IM,Gaedcke J,et al.The oxidative demethylase ALKBH3marks hyperactive gene promoters in human cancer cells[J].Genome Med,2015,7(1):66.
[36]Konishi N,Nakamura M,Ishida E,et al.High expression of a new marker PCA-1 in human prostate carcinoma[J].Clin Cancer Res,2005,11(14):5090-5097.
[37]Koike K,Ueda Y,Hase H,et al.Anti-tumor effect of AlkB homolog 3 knockdown in hormone-independent prostate cancer cells[J].Curr Cancer Drug Targets,2012,12(7):847-856.
[38]Pilžys T,Marcinkowski M,Kukwa W,et al.ALKBH overexpression in head and neck cancer:potential target for novel anticancer therapy[J].Sci Rep,2019,9(1):13249.
[39]Yamato I,Sho M,Shimada K,et al.PCA-1/ALKBH3contributes to pancreatic cancer by supporting apoptotic resistance and angiogenesis[J].Cancer Res,2012,72(18):4829-4839.
[40]Tasaki M,Shimada K,Kimura H,et al.ALKBH3,a human AlkB homologue,contributes to cell survival in human non-small-cell lung cancer[J].Br J Cancer,2011,104(4):700-706.
[41]Woo HH,Chambers SK.Human ALKBH3-induced m1A demethylation increases the CSF-1 mRNA stability in breast and ovarian cancer cells[J].Biochim Biophys Acta Gene Regul Mech,2019,1862(1):35-46.
[42]Hotta K,Sho M,Fujimoto K,et al.Clinical significance and therapeutic potential of prostate cancer antigen-1/ALKBH3in human renal cell carcinoma[J].Oncol Rep,2015,34(2):648-654.
[43]Wang Q,Wang G,Wang Y,et al.Association of AlkB homolog 3 expression with tumor recurrence and unfavorable prognosis in hepatocellular carcinoma[J].J Gastroenterol Hepatol,2018,33(9):1617-1625.
[44]Nakao S,Mabuchi M,Shimizu T,et al.Design and synthesis of prostate cancer antigen-1(PCA-1/ALKBH3)inhibitors as anti-prostate cancer drugs[J].Bioorg Med Chem Lett,2014,24(4):1071-1074.
[45]Nigam R,Babu KR,Ghosh T,et al.Indenone derivatives as inhibitor of human DNA dealkylation repair enzyme AlkBH3[J].Bioorg Med Chem,2018,26(14):4100-4112.
[46]Li Q,Huang Y,Liu XC,et al.Rhein inhibits AlkB repair enzymes and sensitizes cells to methylated DNA damage[J].J Biol Chem,2016,291(21):11083-11093.

