西南地区小麦种质资源白粉病抗性的全基因组关联分析
高 煜,程 斌,丁延庆,曹 宁 ,高 旭,张立异,
(1.贵州大学农学院,贵州贵阳 550025; 2.贵州省农业科学院旱粮研究所,贵州贵阳 550006)
小麦(TriticumaestivumL.)是世界上最重要的农作物之一,全球近35%的人口以小麦为主食[1]。小麦白粉病是由白粉菌(Blumeriagraminisf. sp.Tritici)引起的危害小麦植株地上部分的真菌病害,在世界各麦区都有发生[2-4]。西南地区独特的气候条件,使其成为我国白粉病发生普遍且危害较为严重的地区。农药是减轻白粉病发生的一项措施,但过度使用会造成环境污染,影响小麦的品质,所以培育抗病品种是防治白粉病最为经济有效且环境友好的措施[5]。
发掘新的抗病基因是防治白粉病的常用育种策略。1930年,澳大利亚学者Waterhouse首次发现并报道了第一个抗白粉病基因[6]。此后,Sears等[7]在加拿大小麦品种Axminster的7AL染色体上也定位一个抗白粉病基因Pm1a。迄今为止,在小麦及其近缘种属中已有90多个抗白粉病基因(Pm1~Pm65)被正式命名[8-10]。但是白粉病菌的快速变异,导致大部分抗病基因丧失抗性[11]。目前,抗白粉病基因Pm21在我国广泛使用,它来源于簇毛麦6VS染色体上,具有很好的广谱抗性和持久性[12]。但是单一抗病基因的过度利用,也会对小麦生产造成威胁。因此,不断挖掘新的抗病基因对防治白粉病具有重要意义。
全基因组关联分析(genome-wide association study, GWAS)技术可以直接鉴定出与表型变异显著关联的SNP或者基因位点,目前已经大量应用于小麦重要性状的功能性等位变异检测及基因研究中[13]。随着基因芯片和测序技术的快速发展,基于SNP标记的GWAS在抗病基因定位中得到广泛应用[14-15]。通过GWAS分析,Li等[16]成功定位了位于2B染色体上的三个白粉病抗性位点,并进行了验证;Liu等[17]用185个美国冬小麦品种确定了9个与白粉病抗性显著相关的SNP标记。
本研究以主要来源于我国西南地区的120个小麦品种(系)为材料,利用小麦90K SNP芯片对白粉病抗性进行GWAS分析,确定抗白粉病基因或QTL,并发掘与已知白粉病基因紧密连锁的分子标记,以期为小麦白粉病的遗传改良和种质资源的有效利用提供理论依据。
1 材料与方法
1.1 试验材料及白粉病抗性鉴定
120个普通小麦品种(系)分别来源于贵州(62个)、四川(36个)和国内其他地区(河南、湖北、甘肃、江苏等,共22个)(表1)。于2012―2013年度分别种植于贵州省贵阳市、贵州省赫章县和四川省绵阳市,于2013―2014种植于贵州省贵阳市,形成四个不同环境条件。试验采用随机区组设计,3次重复,行长1 m,行距30 cm。以高感白粉病的铭贤169为诱发材料,每隔五行种植一行,确保发病充分。常规大田管理,以环境中白粉病菌混合生理小种诱发材料自然发病。待铭贤169充分发病时调查记录白粉病反应型,采用0~4级分级标准,其中0表示免疫,0;表示近免疫,1表示高抗,2表示中抗,3表示中感,4表示高感[18]。
1.2 DNA提取、90K SNP芯片分型和标记扫描
采用改良的CTAB法[19]提取供试材料单株幼嫩叶片DNA。利用小麦90K iSelect SNP芯片(包括81 587个SNP标记)对120个小麦品种(系)进行SNP分型。芯片检测在中玉金标记公司完成。利用与抗白粉病基因Pm21紧密连锁的分子标记SCAR1265(R: 5′-CACTCTCCTCAAACCTTGCAAG-3′;L:5′-CACTCTCCTCCACTAACAGAGG-3′)对这120份小麦材料进行PCR扩增,并用琼脂糖凝胶电泳进行检测,统计在1 265 bp处的扩增片段,有DNA条带的记为1,无带记为0。
1.3 数据分析
群体结构分析:使用Genome Studio软件进行SNP分型,剔除数据缺失频率>20%、最小等位基因频率<5%的SNP标记,保留高质量的SNP标记进行数据分析。利用Structure 2.3.4 软件对该群体进行群体结构分析[20],设置亚群数目K为1~12,5次模拟运算,模拟参数迭代(length of bum-in period)和蒙特卡罗迭代(markov chain monte carlo,MCMC)设置为 10 000次循环,在混合模型和频率相关模型下运算。输出后验概率值的结果输入Structure Harvester(http://taylor0.biology.ucla.edu/struct-ure Harvester/),计算2个连续的后验概率值的变化速率(ΔK)和每个材料在1~12亚群的Q值,以Q=0.5为分界线,最终确定群体的数目和结构。
主成分分析(principal component analysis,PCA):运用R语言包GAPIT对自然群体进行PCA群体结构分析和亲缘关系评估,计算亲缘关系K矩阵。亲缘关系代表两个特定材料之间的遗传相似度与任意材料之间遗传相似度的相对值,当供试材料之间的亲缘关系值为负值时,将其定义为0[21]。

表1 120个小麦品种(系)的名称、来源及其在主成分分析(PCA)中的类群Table 1 Names, origins and PCA(principal component analysis) groups of 120 wheat varieties(lines)
全基因组关联分析(GWAS):利用质控后得到的37 790个高质量的SNP标记,运用R语言包GAPIT中的FarmCPU模型(http://zzlab.net/FarmCPU/index.html)[22]对4个环境下小麦品种的抗白粉病表型分别进行GWAS,阈值为 -log10(2.6×10-7)(以显著性水平为0.01,质控后得到的37 790个高质量SNP标记来计算阈值),判定SNP标记与目标性状关联的显著性。
相关性分析:利用SPSS v20软件,计算Pm21连锁标记SCAR1265与6A染色体上显著关联SNP标记的Pearson相关系数。
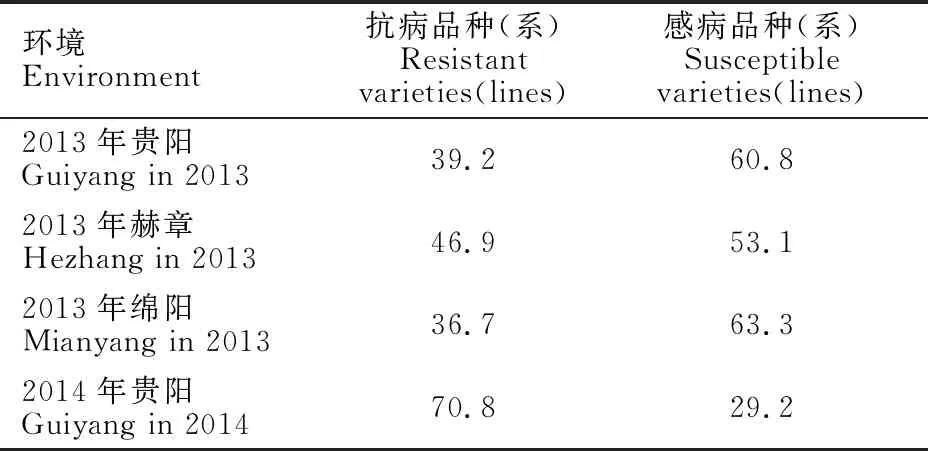
表2 4个环境下120个小麦品种(系)中抗病 和感病品种(系)的比例Table 2 Ratio of resistant and susceptible varieties(lines) to 120 wheat varieties(lines) in four environments %
2 结果与分析
2.1 小麦品种(系)成株期抗白粉病鉴定结果
从表2可以看出,不同环境下抗病材料所占比例不同,其中,2014年贵阳环境条件下,白粉病表现抗性的品种(系)占比最高(70.8%),2013年绵阳环境条件下占比最低(36.7%)。36个品种(系)(30%)在四种环境下均表现为抗病,其中,贵州省品种(系)30个,四川省品种(系)4个,其他地区品种(系)2个(表3)。另外有18个品种(系)在四个环境下都表现为感病,包括贵州省品种(系)4个,四川省品种(系)9个,其他地区品种(系)5个。在不同环境下,抗病品种(系)中来自贵州省的都最多,占比均超过50%,其次是四川省,其他地区抗病品种(系)比例较低(表4)。由此可见,本试验中贵州省小麦品种(系)的抗病性表现较好。
2.2 群体结构分析
从图1可以看出,当K=6时,ΔK取得最大值,因此,试验群体可分为6个类群。其中,第一类群[20个品种(系)]、第二类群[27个品种(系)]和第四类群[20个品种(系)]的小麦材料主要来自贵州省和四川省,第三类群[13个品种(系)]和第五类群[20个品种(系)]的小麦材料主要来自贵州省,第六类群[20个品种(系)]的小麦材料来自其他地区。
为了进一步了解这些小麦材料的亲缘关系,对供试材料进行主成分分析。结果(图2)表明,120个小麦品种(系)按地理来源聚集形成两个类群。PC1解释了全部遗传变异的12.8%,PC2解释了全部遗传变异的8.4%。位于图2左上侧的类群主要由来自四川省和其他地区的小麦品种(系)组成,在PC2水平上分布较为集中。来源于贵州省的小麦品种(系)以及铭贤169聚集在右下侧,在PC1水平上分布较分散,其中由贵州大学选育的小麦品种(系)(包括贵农系列、TP2、硬葡1等)分布更分散。
2.3 全基因组关联分析(GWAS)
利用37 790个SNP标记对120个小麦品种(系)在四个环境中的白粉病抗性分别进行GWAS,共检测到16个与白粉病抗性显著关联的SNP标记(图3、表5),分布于1A(2)、1D(2)、2A(2)、3A(2)、4B(1)、5B(1)、6A(3)、6D(2)和7B(1)染色体上。
在2013年贵阳环境条件下,检测到6个与白粉病抗性显著关联的SNP标记,分别位于1A、1D、3A、4B、5B和6A染色体上。在2013年赫章环境条件下,只检测到1个与白粉病抗性显著关联的SNP,位于6A染色体上。在2013年贵阳环境条件下,检测到6个与白粉病抗性显著关联的SNP标记,分别位于1D、3A、4B、6A和6D染色体上。在2014年贵阳环境条件下,共检测到6个与白粉病抗性显著关联的SNP标记,分别位于1A、2A、6A、6D和7B染色体上。
有多个染色体区段在2~4个环境中均被检测到。其中,位于6AS染色体大约44 cM处的区段,在4个环境中均被检测出与白粉病抗性显著关联,包含3个SNP位点,其中,Jagger_c4823_169在2013年贵阳和2013年绵阳两个环境中均被检测到,位于6A染色体43.85 cM处,P值分别为 7.24E-53和5.76E-27;Tdurum_contig55201_928在2013年赫章环境中被检测到,位于6A染色体44.06 cM处,P值为2.39E-08;Tdurum_contig12215_89在2014年贵阳环境中被检测到,位于6A染色体44.11 cM处,P值为4.17E-30,这3个SNP标记均显示与白粉病抗性显著关联。位于3A染色体43.44 cM处的SNP(Excalibur_c3510_1888)和位于4B染色体49.44 cM处的SNP(BS00011148_51)在2013年贵阳和2013年绵阳两个环境中均被检测到,且与白粉病抗性显著 关联。
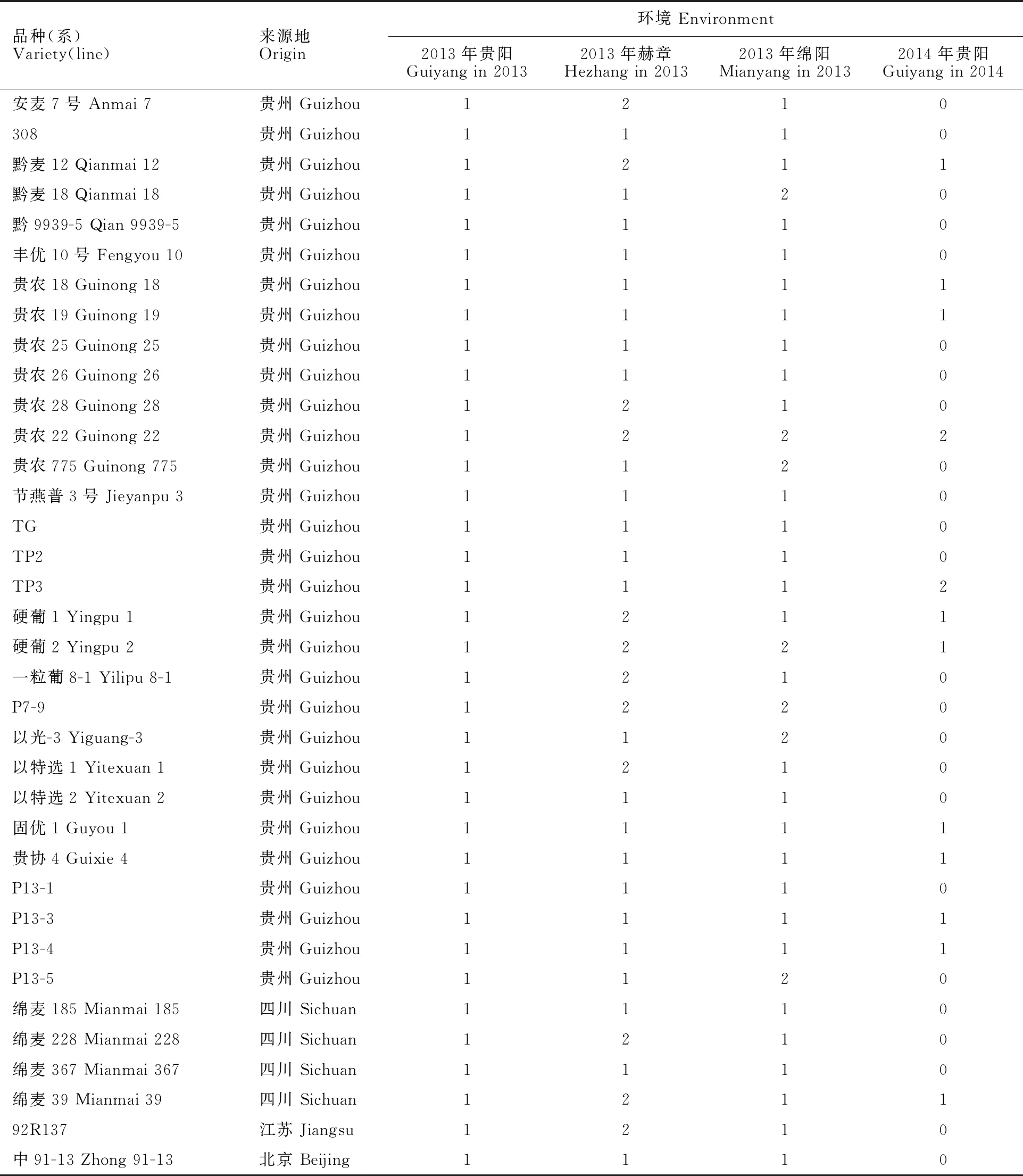
表3 四种环境下均表现抗病的品种(系)Table 3 Resistant varieties(lines) in four environments
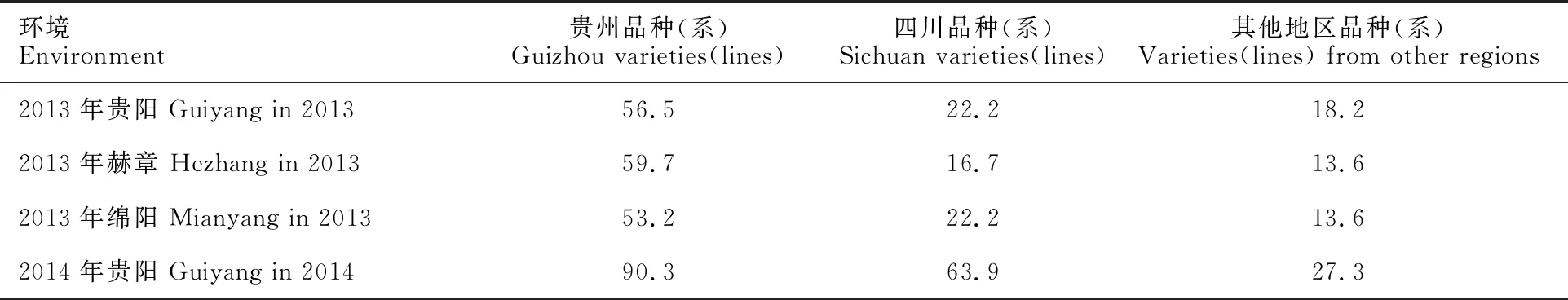
表4 4种环境中不同地区抗病品种(系)的频率Table 4 Percentage of resistant varieties(lines) from different regions in four environments %
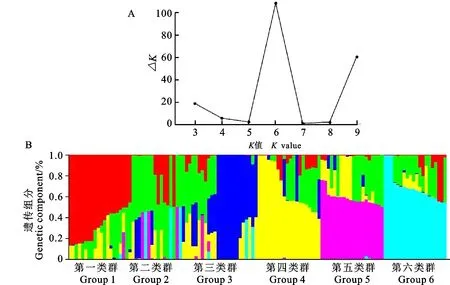
A:△K折线图;B:群体结构分析。
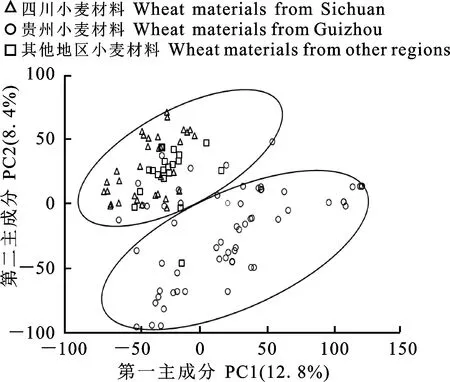
图2 120个小麦品种(系)的主成分分析Fig.2 Principal component analysis of 120 wheat varieties(lines)
2.4 相关性分析
为了进一步验证6A染色体上的重要位点与抗白粉病基因Pm21的关系,利用与Pm21紧密连锁的分子标记SCAR1265对这120个小麦品种(系)小麦材料进行扫描,部分小麦品种的PCR扩增结果如图4所示。分型结果显示,这3个SNP标记(Jagger_c4823_169,Tdurum_contig55201_928,Tdurum_contig12215_89)与SCAR1265的Pearson相关系数R值分别为0.85、0.77和0.87(P<0.01),说明这3个SNP标记与Pm21分子标记位点紧密连锁。
3 讨 论
3.1 西南地区小麦白粉病抗性
小麦白粉病常发生在潮湿的地区,而西南地区因其特有的气候地理特征,使得白粉病菌能够安全越夏,并加快生理小种的变异,这是西南地区小麦白粉病及其他病害高发的重要原因。
本研究抗性鉴定结果显示,2014年贵阳环境条件下抗白粉病的小麦品种(系)占比最高,原因可能是由于当年气候较为干燥,导致白粉病发病不充分。在120个小麦品种(系)中,有36个在四个环境下都表现为抗病,其中,来自贵州省的小麦品种(系)中抗病品种比例最高,原因可能是贵州麦区白粉病菌的毒力强,而在植物-病菌协同进化过程中,育种家通过人工选择,使该地区小麦品种具有较好的抗病性[23]。这对贵州地区小麦抗白粉病品种抗性基因资源的利用和推广有重要的 意义。
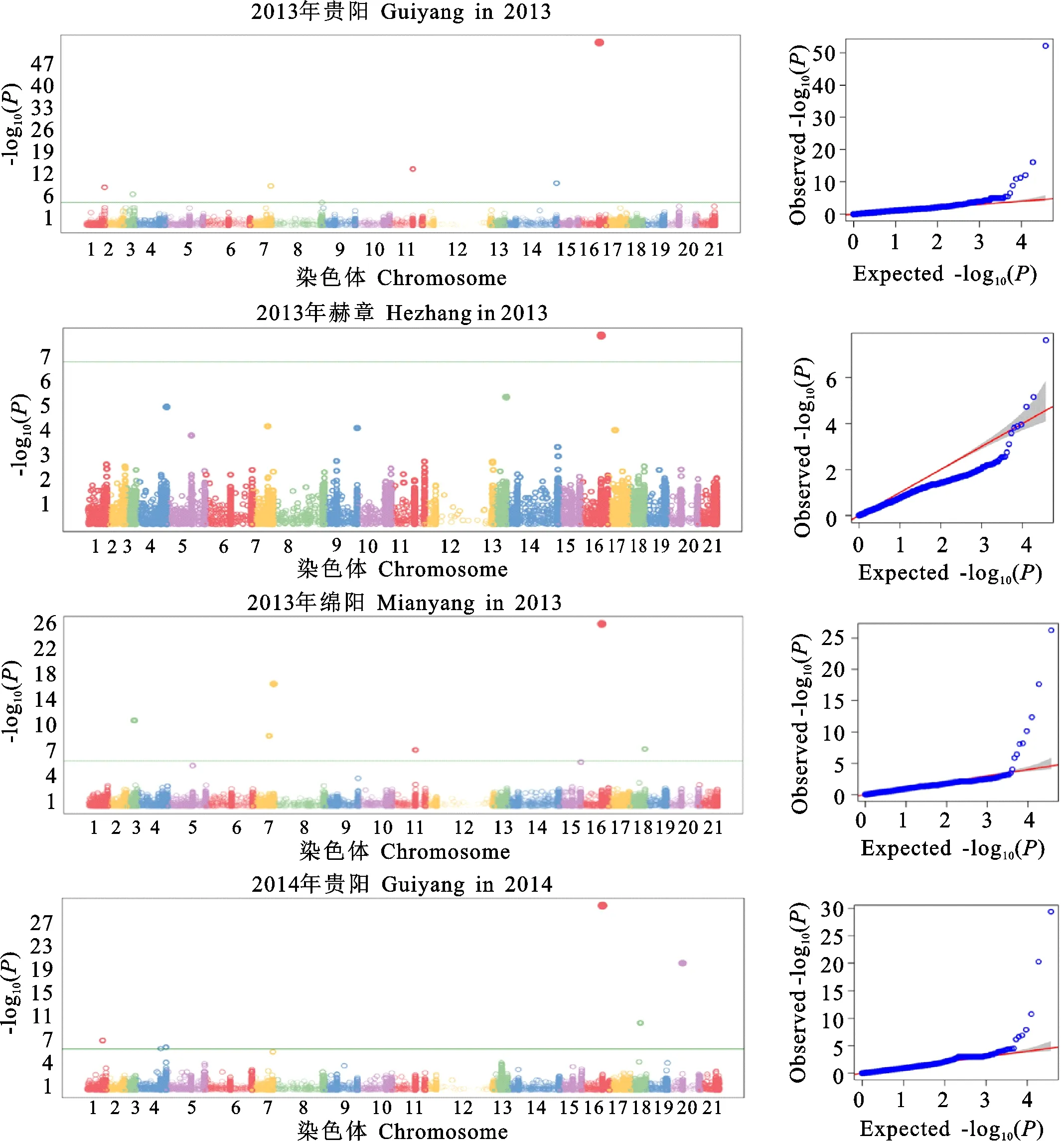
绿色线为SNP显著性的阈值,等于-log10(2.6×10-7)。
3.2 西南地区小麦白粉病的全基因组关联分析(GWAS)
本研究全基因组关联分析结果显示,16个SNP标记与白粉病抗性显著关联,分布在1A、1D、2A、3A、4B、5B、6A、6D和7B染色体上,其中,3A、4B和6A染色体上分别有1个重要的染色体区段在2或4个环境中均能被检测到。
本研究在2013年贵阳和2013年绵阳两个环境条件下,分别在3A(约43.44 cM)和4B(约 49.44 cM)染色体的两个区段检测到与白粉病抗性显著关联的SNP位点。目前,3A染色体上已鉴定出的抗白粉病基因为Pm44,而4B染色体上暂无报道[24-25]。Lillemo等[26]利用Saar和Avocet构建的113个重组自交系群体,在3AS和4BL染色体上分别检测到一个抗白粉病QTL,均与本研究检测到的位点不同。Keller等[27]利用Forno和Oberkulmer构建的226个重组自交系,分别在3A(40~62 cM)和4B(84~88 cM)染色体上检测到一个抗白粉病QTL,推测本研究在3A染色体上检测到的位点位置与前人研究结果相似,而在4B染色体上检测到的位点位置不同。Liang等[28]利用Fukuho-komugi和Oligoculm构建的107个DH群体,在4BL(41.38~44.59 cM)染色体上检测到一个抗白粉病QTL,推测与本研究在4B染色体上的SNP位点位置相似。本研究供试小麦品种(系)中能够同时检测到这两个位点的品种(系)有硬葡1、硬葡2和一粒葡8-1等材料,均来自贵州省。目前,这些材料对小麦白粉病仍然表现出较好的抗病性,在育种中具有一定的利用价值。
本研究在四个环境条件下,在6AS染色体43.85~44.11 cM区段检测到3个与白粉病抗性显著相关的SNP标记,分别为Jagger_c4823_169、Tdurum_contig55201_928和Tdurum_contig12215_89。6AS染色体上已鉴定出的抗白粉病基因为Pm21,与Pm21紧密连锁的分子标记为SCAR1265[29]。本研究检测到的3个SNP标记也与Pm21紧密连锁。在120个小麦品种(系)中,能够同时检测到这4个SNP标记的有33个,其中来自贵州省的有26个,包括安麦7号、308、黔麦12、黔麦18、黔麦19、夏繁28、黔9939-5、贵农18、贵农22、贵农25、贵农26、贵农28、贵农775、节燕普3号、TG、TP2、TP3、P5-1、以光-3、以特选1、以特选2、固优1、贵协4、P13-1、P13-3和P13-5;来自四川省的有5个,包括绵麦185、绵麦228、绵麦367、绵麦37和绵麦39;来自其他地区的有2个,包括92R137和南农24。Jagger_c4823_169、Tdurum_contig55201_928、Tdurum_contig12215_89这3个SNP位点和分子标记SCAR1265在西南地区小麦品种(系)中出现的频率较高,分别为37.8%、34.7%、35.7%和 41.8%,说明西南地区小麦品种(系)中含有Pm21基因的品种(系)较多。江 峥等[30]利用开发的Pm21特异性标记对662个小麦品种(系)进行检测,发现西南地区小麦材料中Pm21的使用频率最高 (34.4%);邹景伟[31]利用Pm21的连锁标记对参试品种进行检测,发现有15个品种可能含有Pm21基因,其中,有11个来自西南冬麦区,与本研究结果一致。说明Pm21基因在西南地区小麦品种(系)中分布频率较高,有加大病原菌定向选择压力的风险,对西南地区小麦生产造成威胁。因此,在今后的小麦抗病育种工作中,要加强白粉病新抗源的发掘,同时通过多个抗病基因的聚合,选育抗病性更为持久的小麦品种。
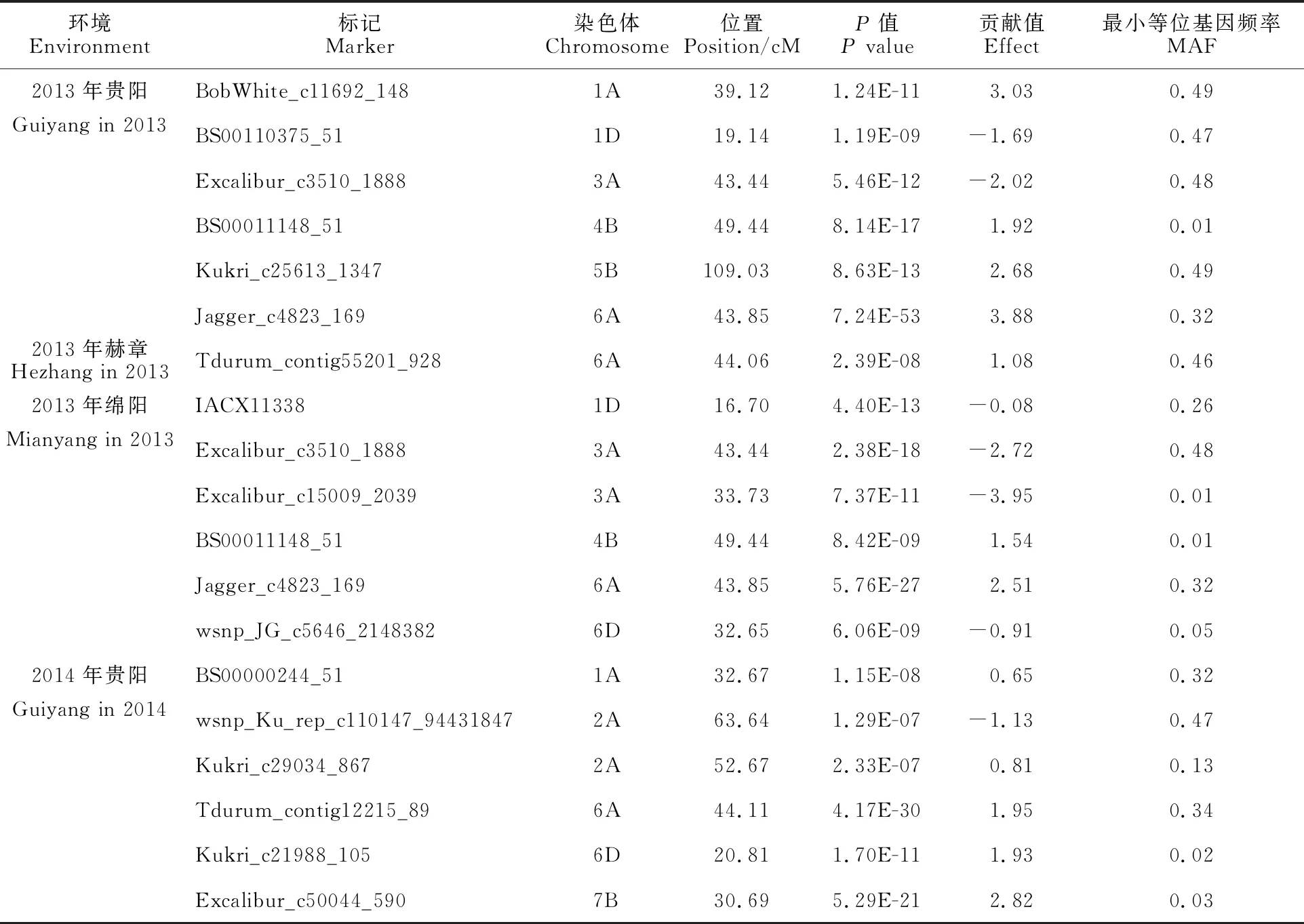
表5 小麦白粉病抗性显著相关的SNP标记Table 5 Significant SNP markers associated with wheat powdery mildew resistance
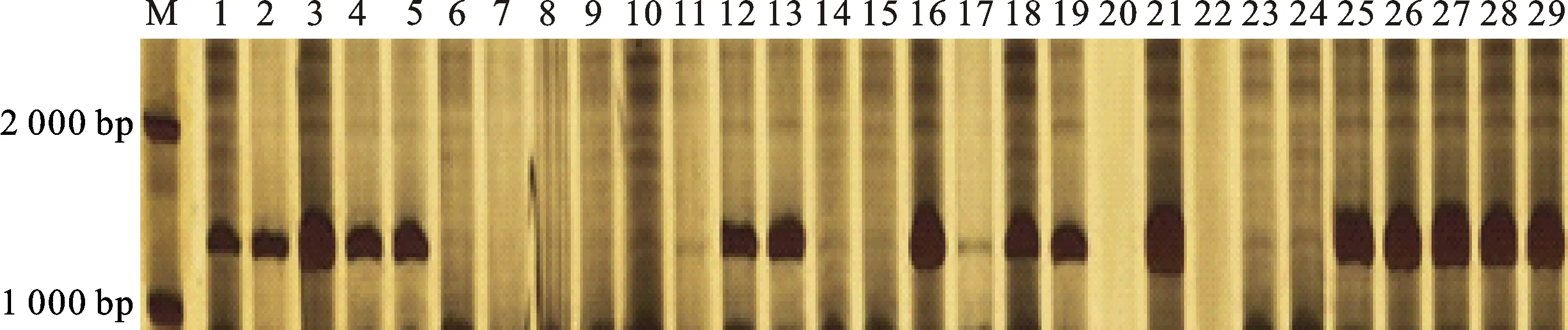
M: DL2000;1:黔麦17;2:黔麦18;3:黔麦19;4:夏繁28;5:黔9939-5;6:丰优1号;7:丰优2号;8:丰优3号;9:丰优6号;10:丰优7号;11:丰优8号;12:丰优9号;13:丰优10号;14:丰优92212;15:兴育7号;16:兴育823;17:CN04-1;18:淮麦18;19:92R137;20:南农04y10;21:南农24;22:南农06y603;23:小偃54;24:绵麦1403;25:绵麦185;26:绵麦228;27:绵麦367;28:绵麦37;29:绵麦39。
4 结 论
本试验对主要来源于西南地区的120个小麦品种(系)的白粉病抗性进行GWAS,获得16个与白粉病抗性显著相关的SNP位点。在6A染色体上有3个SNP标记,遗传距离小于1 cM,与Pm21基因紧密连锁。本研究结果说明,Pm21在西南地区小麦白粉病抗性育种中发挥了巨大作用。但在今后的小麦抗病育种中应该使用更多不同的抗病基因,以降低Pm21过度使用给西南地区的小麦生产带来风险。

