转移性结直肠癌患者ctDNA基因突变检测方法的比较及影响因素分析
陈馨宁,黄 斐,沈敏娜,杨轶慧,王蓓丽,2,郭 玮,2
1.复旦大学附属中山医院检验科,上海 200032 2.复旦大学附属中山医院厦门医院检验科,福建 厦门 361015
循环肿瘤DNA(circulating tumor DNA,ctDNA)是肿瘤细胞体细胞DNA经脱落或当细胞凋亡后释放进入循环系统,存在于血液、尿液、脑脊液等体液中。ctDNA分析能够快速甄别细胞信号通路上的基因突变情况并且能够定量ctDNA的突变丰度[1-3]。
常规基因测序技术如Sanger测序、焦磷酸测序只能检测出高负荷肿瘤或ctDNA含量较高的患者体内的肿瘤突变片段。随着DNA测序技术的快速发展,已实现对痕量DNA分子进行定量分析。常见的ctDNA检测技术有二代测序(next generation sequencing,NGS)、微滴式数字聚合酶链反应(droplet digital polymerase chain reaction,ddPCR)、MALDI-TOF等。
本文建立了NGS和MALDI-TOF两个分析平台,检测转移性结直肠癌(metastatic colorectal cancer,mCRC)患者血浆ctDNA中KRAS、NRAS、BRAF、PIK3CA基因的常见突变;分析两个平台在检测血浆ctDNA突变中的性能及血浆ctDNA在指导临床诊疗方面的潜在价值。
1 资料和方法
1.1 研究对象
选取2016年6月—2017年1月复旦大学附属中山医院收治的30例mCRC患者。
1.2 入选标准
患者均经活组织病理学检查确诊为结直肠腺癌,临床分期为Ⅳ期(即转移性癌)且均有3处及以上转移灶,所有患者的活检组织都接受过分子诊断,明确了KRAS、NRAS、BRAF、PIK3CA基因的突变状态。每例患者收集2管10 mL乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA)抗凝全血,明确采血时间和采血时的患者状态。
1.3 方法
1.3.1 标本预处理
抽取10 mL外周血全血(EDTA抗凝管)。采用两步离心法,1 600×g,4 ℃离心10 min后吸取离心后的血浆,16 000×g,4 ℃再次离心10 min,吸取上清,-80 ℃冻存备用。使用QIAmp Circulating Nucleic Acid Kit(德国Qiagen公司)抽提ctDNA。ctDNA定量使用Qubit dsDNA HS Assay Kit核酸定量分析试剂盒(美国Invitrogen公司),由ctDNA-Qubit 3.0得到具体浓度后用高灵敏度DNA分析试剂盒(5067-4678)在Agilent 2100 Bioanaylzer上进行样本质量控制。剩余标本-20 ℃保存。
1.3.2 病史获取
通过病史查询的方式获取由复旦大学附属中山医院病理科为患者组织标本进行的分子病理学检查结果和复旦大学附属中山医院检验科为患者血液进行的癌胚抗原(carcinoembryonic antigen,CEA)、糖类抗原19-9(carbohydrate antigen 19-9,CA19-9)、糖类抗原125(carbohydrate antigen 125,CA125)检测结果。
1.3.3 ctDNA-NGS检测
采用美国Illumina公司NGS平台MiSeq,配合上海安可济生物科技有限公司的Comet结直肠癌试剂盒(AccuraGen Accu-Kit)完成对上述标本的检测。最终测序的结果中将包含所检测的基因、外显子、碱基和对应氨基酸的改变方式,并附上每种基因突变的拷贝数及丰度作为定量结果,突变丰度的计算公式如下:突变丰度=(突变型拷贝数)/(突变型拷贝数+野生型拷贝数)×100%。
1.3.4 ctDNA-MALDI-TOF检测
使用美国Agena公司UltraSEEKTM Colon Panel试剂盒进行MALDI-TOF检测。出具的结果由软件进行自动分析和处理,最后进行对靶目标序列的定性和半定量分析。
1.3.5 ctDNA-ddPCR检测
使用QX100 ddPCR™系统完成对上述标本的检测。QX100系统包括QX100微滴生成仪和QX100微滴分析仪,另配备美国Bio-Rad公司的96深孔反应模块PCR仪。检测试剂盒选用美国Bio-Rad公司的商品化试剂盒(货号:Lot1863110~1863115及Lot1863102),包括突变位点KRASG12A、KRASG12C、KRASG12D、KRASG12R、KRASG12S、KRASG12V、BRAFV600R。系统将计算阳性和阴性微滴的数量,从而以数字格式对靶DNA进行绝对定量分析。
1.4 统计学处理
采用SPSS 11.0统计软件分析数据,采用配对资料t检验进行分析。以总符合率、阳性预测值和阴性预测值作为评判两个平台的主要参数指标,计算公式:总符合率=[(真阳性+真阴性)/总样本数]×100%;阳性预测值=[真阳性/(真阳性+假阳性)]×100%;阴性预测值=[真阴性/(真阴性+假阴性)]×100%。P<0.05为差异有统计学意义。
2 结果
2.1 入组患者的基本信息
本研究共入组30例CRC患者(表1),其中21例男性,9例女性,平均年龄为59.63岁。30例患者根据TNM分期均处于结直肠癌Ⅳ期,即mCRC且所有患者均进行过肿瘤组织(原发灶/转移灶)KRAS、NRAS、BRAF、PIK3CA基因突变检测并获得了检测结果。血清中位CEA水平为46.65 ng/mL(2.90~710.60 ng/mL),血清中位CA19-9水平为37.50 ng/mL(8.40~7 506.00 ng/mL),血清中位CA125水平为17.05 ng/mL(4.80~1 141.00 ng/mL)。
2.2 患者组织和血浆中KRAS、NRAS、BRAF、PIK3CA基因突变的情况
本研究比较了30 例患者组织和血浆中KRAS、NRAS、BRAF、PIK3CA基因突变的情况(图1)。ADx-ARMS中获得的组织结果同通过NGS获得的血浆ctDNA结果比较,22例患者组织和血浆结果完全匹配;但由于组织和血浆采样时间不同以及期间接受治疗,有8例患者出现了组织和血浆结果不匹配的情况。ADx-ARMS中获得的组织结果同通过MALDI-TOF获得的血浆ctDNA结果比较,18例患者组织和血浆结果完全匹配;同理,有12例患者出现了组织和血浆结果不匹配的情况。
根据以上结果可见,NGS与组织结果匹配度较高(73.3%),但由于患者组织标本和患者血浆标本获取的最长时间间隔达3年6个月,最短时间间隔为6 d,尽管组织ARMS结果存在一定参考价值,但该匹配度可能与临床实际情况不符。因此,本研究查阅病史,结合患者的治疗、手术情况,并且引入ddPCR对突变位点进行复核(表2)。

表1 患者基本信息Tab.1 Patient characteristics[n (%)]

图1 组织结果与血浆ctDNA结果匹配情况Fig.1 Correlation between ctDNA results and tumor ARMS results
2.3 MALDI-TOF和NGS的符合率、阳性预测值及阴性预测值
MALDI-TOF和NGS的符合率分别为76.67%和86.67%,阳性预测值为86.67%和83.33%,阴性预测值为66.67%和91.67%(表3)。
2.4 提取的ctDNA的量与原发灶切除与否的关系
从患者血浆中提取的ctDNA的量与原发灶切除与否无相关性(P=0.724 7);突变位点丰度与原发灶切除与否显著相关(P=0.001,图2)。
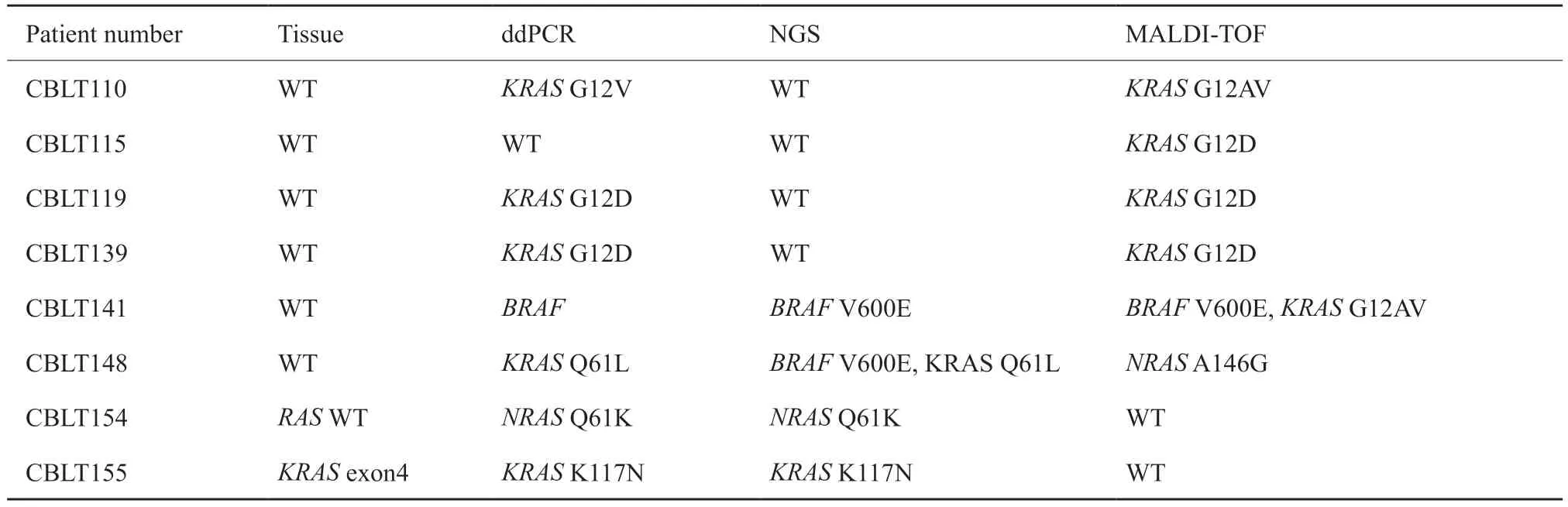
表2 采用ddPCR复核数据Tab.2 Data of re-detection using ddPCR

表3 MALDI-TOF和NGS的符合率、阳性预测值及阴性预测值Tab.3 The coincidence rate,positive predictive value and negative predictive value of MALDI-TOF and NGS
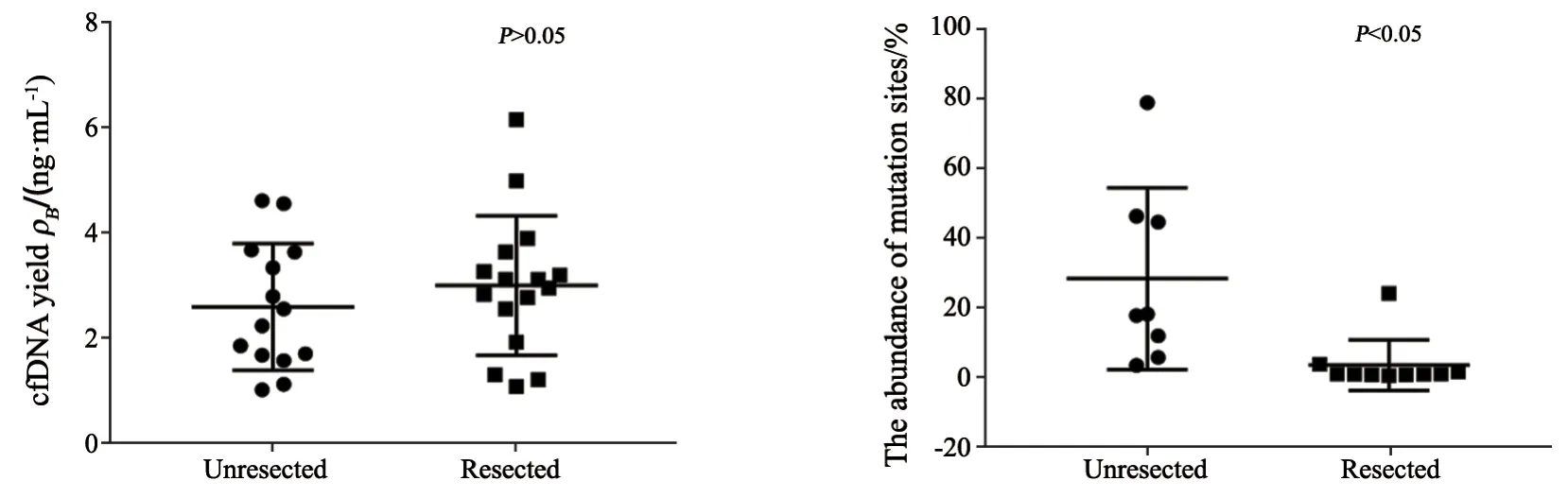
图2 原发灶切除与否与患者突变位点丰度及ctDNA的量的关系Fig.2 Correlation between primary tumor resection and the abundance of mutation sites/the amount of ctDNA
2.5 提取的ctDNA的量与患者接受治疗与否的关系
从患者血浆中提取的ctDNA的量与患者接受治疗(化疗、放疗、靶向治疗)与否无相关性(P=0.871 9);突变位点丰度与患者接受治疗(化疗、放疗、靶向治疗)与否显著相关(P=0.000 6,图3)。
组织活检后血浆采集前使用爱必妥治疗的患者6例,其中组织结果为野生型,血浆结果为突变型有4例,占66.7%。2例为BRAFV600E突变,1例为KRASQ61L突变,1例为NRASQ61K突变。
本实验还统计了不同突变位点及其突变位点丰度分布(图4),剔除突变发生较少的PIK3CA和BRAF位点。
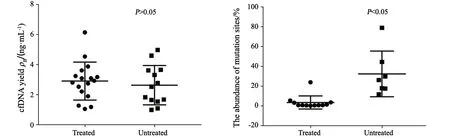
图3 是否接受治疗与患者突变位点丰度及ctDNA的量的关系Fig.3 Correlation between treatment and the abundance of mutation sites/the amount of ctDNA

图4 各突变位点丰度情况Fig.4 The abundance of each mutation site
3 讨 论
本研究纳入了30例mCRC患者,利用NGS和MALDI-TOF检测入组患者血浆ctDNA中常见突变的性能比对。NGS使用边合成边测序的方法对ctDNA进行检测[4-5]。MALDI-TOF利用质谱分析对PCR衍生的扩增子进行分析[6]。
本研究发现NGS的符合率和阴性预测值较好,而MALDI-TOF的阳性预测值较好。在一项利用NGS检测108例mCRC患者ctDNA中KRAS、BRAF、BRAS突变状态代替组织检测的可行性实验[7]中发现,血液、组织KRAS突变检测整体一致性为65.26%,BRAF突变检测整体一致性为93.68%,NRAS突变检测整体一致性为96.84%。本研究或需纳入更多的病例来判断NGS在结直肠癌ctDNA突变检测中的可行性及其他性能。血液样本检测结果在肺癌中可以指导临床用药已经获得证实。在一项利用MALDI-TOF检测结直肠癌肿瘤组织的石蜡包埋标本中KRAS基因突变情况的实验[8]中,MALDI-TOF较常规测序检测的检出率和阳性预测值更高,具有一定临床价值。仍未见以上两种方法的阳性预测值、阴性预测值等在结直肠癌ctDNA突变检测表现方面的大样本量数据。
组织ARMS虽然是ctDNA-NGS临床验证的有效参考方法,但仍有相当比例样品存在两种方法结果不一致的情况。本研究引入了ddPCR,作为有效的补充检测手段,协助分析组织和血检结果不一致的情况。同时,样品临床信息也有助于分析组织和血检结果不一致的原因。在应用西妥昔单抗之前,临床上需要进行常规的肿瘤组织RAS基因状态检测。本实验中,6例获得组织标本前未使用西妥昔单抗,而采血前使用西妥昔单抗的患者中,有4例患者出现了基因突变。2例为BRAFV600E突变,1例为KRASQ61L突变,1例为NRASQ61K突变。其中发生RAS基因突变是患者对西妥昔单抗的耐药标志[9]。而产生BRAF基因突变一般被认作是不良预后的因素之一[10-13]。鉴于本实验样本量较少且研究时间较短,无法确定由野生型转为突变型的患者的预后情况,以及突变位点是否会对患者后续治疗产生影响。
本实验中ctDNA的量与患者是否接受肿瘤原发灶的切除以及是否接受治疗(包括化疗、放疗、靶向治疗)无关。但Tie等[14]报道,化疗过程中mCRC患者的ctDNA发生了变化,在治疗第2个周期之前就观察到的48例ctDNA和组织伴有突变的样本中有41例ctDNA水平显著降低。推测本研究的样本量或实验设计可能产生一定影响,本研究样本量较少,并且在实验设计时增加了对同一患者治疗前后的血浆ctDNA含量的比较。
在12例KRAS突变和2例NRAS突变患者中,发现RASctDNA突变位点丰度低于1%的患者的肿瘤原发灶均发生在左半结肠;与此同时RASctDNA突变位点丰度高于25%的患者的肿瘤原发灶均发生在右半结肠,结直肠癌中,肿瘤原发灶位于左半结肠通常认为比位于右半结肠预后更好,但仍需对患者进行随访,了解其肿瘤进展情况和生存期来验证以上假设。另外,Morelli等[15]研究发现,基线高的RASctDNA突变位点丰度与生存率低相关。本研究发现,ctDNA突变位点丰度水平与患者是否接受肿瘤原发灶的切除以及是否接受治疗(包括化疗、放疗、靶向治疗)有关。接受手术干预和治疗的患者的突变位点丰度普遍较低。ctDNA水平和ctDNA突变位点丰度可以在治疗开始前为RAS基因突变的患者提供评估信息。
本研究存在部分缺陷,目前来看急需更大样本量及更长随访周期来观察来自不同平台ctDNA的数据对于临床应用的价值。此外,应用ARMS方法获得的组织分子分型结果的说服力较低,在有条件的情况下使用大基因检测组合的NGS进行组织分型,将有利于平台比对以及后续研究的开展。
综上所述,两个平台均可用于检测mCRC患者ctDNA中的常见突变,但NGS的阴性预测值要优于MALDI-TOF。患者突变位点丰度与患者的原发灶是否手术切除以及患者是否接受过治疗(包括化疗、放疗、靶向治疗)有关。

