覆盖作物对猕猴桃园土壤氨氧化微生物丰度和群落结构的影响
张玲玲,李青梅,贾梦圆,张艳军,赵建宁,杨殿林,王华玲,王 慧*
(1 农业农村部环境保护科研监测所,天津 300191;2 十堰市经济作物研究所,湖北十堰 442000)
覆盖作物一般指除目标作物外被人为种植的草本植物,用作杂草的控制或裸露地面的覆盖。果园行间种植覆盖作物是可持续农业系统中一种被广泛使用的有益管理模式[1-2],可以提高水分渗透[3],改变土壤温度[4],减少径流及土壤侵蚀[5],减少氮浸出[6],控制杂草和害虫。覆盖作物品种通常选择一年生或多年生豆科或禾本科草本植物,如白三叶草 (Trifolium repensL.)、毛叶苕子 (ViciavillosaRoth)、黑麦草(LoliumperenneL.) 和高羊茅 (FestucaelataKengexE.Alexeev) 等,其具有较强的生物固氮能力、耐阴、耐踩踏、能诱引益虫及较短的生育期等特性[7]。
近年来,覆盖作物对土壤理化性质和微生物群落的影响受到广泛关注。有研究表明,果园种植覆盖作物后能够有效改善园区地温和气温,提高园区空气相对湿度,增强土壤保水能力,通过土壤理化性质的改善,影响土壤微生物的生长环境条件[8]。微生物在氮循环过程中起着重要作用,作为氮循环的关键步骤,硝化作用能够将铵态氮转化成硝态氮,其第一步和限速步骤是由氨氧化微生物 (氨氧化古菌AOA和氨氧化细菌AOB) 完成的氨氧化过程[9]。土壤生态系统中,硝化因子和硝化速率与植被类型、位置和环境条件有着紧密联系[10]。植物种类和覆盖类型对土壤性质和微生物性质有着根本性的影响[11],植物 [如宽叶蒿 (ArtemisialatifoliaL.)、白刺 (Nitraria sibirica)和猪毛菜 (Salsolaferganic)]是改变微生物群落的驱动因素,植物可能通过其有机质输入和对硝化潜势的影响,改变氨氧化微生物的群落组成[12]。因此,种植不同种类或组合种植覆盖作物可能对土壤氨氧化微生物群落产生不同的影响。
目前,关于覆盖作物的研究主要集中在覆盖作物单种对果园节肢动物多样性、土壤水肥流失、土壤理化性状[12]、果园小气候[13]等方面的影响,并且在苹果园[14]、桃园[15]、梨园[16]应用较多。而针对猕猴桃园豆科与禾本科覆盖作物混播处理下硝化作用 (连接固氮作用和反硝化作用) 的研究较少。硝化作用不仅反映了植物对氮素的利用状况,还能反映因过量施用化肥引起的土壤酸化和硝酸盐淋失所引起的水体污染及氧化亚氮 (N2O) 释放等一系列环境问题[17]。因此,以猕猴桃果园为研究对象,通过设置不同覆盖作物处理模式,研究其对土壤氨氧化微生物基因丰度、群落多样性及组成的影响,其结果将为探索覆盖作物对氮循环过程的影响机制提供理论指导,并对果园覆盖作物生态研究、果园高效覆盖作物利用技术的推广及示范应用提供理论和实践指导。
1 材料与方法
1.1 试验设计
试验地位于湖北省十堰市农业科学院西沟乡猕猴桃基地 (32°37'10''N,110°38'2''E),该地区属亚热带大陆性季风气候,海拔550 m,年降雨量800 mm,无霜期244 天,年平均气温15℃。该基地的果树品种为树龄一致、生长整齐的5年生金魁猕猴桃,株行距为 2.5 m×4.5 m。试验地土壤质地为砂壤土,本底基本理化性质:有机质10.77 g/kg、全氮0.39 g/kg、pH 6.65。
供试覆盖作物包括白三叶草 (TrifoliumrepensL.)、毛苕子 (ViciavillosaRoth)、黑麦草 (Lolium perenneL.) 和高羊茅 (FestucaelataKengexE.Alexeev)。白三叶草和毛苕子为豆科绿肥,黑麦草生物量大,高羊茅控制杂草能力强[18]。试验采用随机区组试验设计,共设置5个处理:1) 单种白三叶草(W);2) 白三叶草+黑麦草混种 (WR);3) 白三叶草+黑麦草+毛苕子混种 (WRV);4) 白三叶草+毛苕子+黑麦草+高羊茅混种 (WRVF);5) 清耕对照(CK)。各处理在区组中随机排列,共3个区组,即每个处理3次重复。覆盖作物于2017年9月进行行间播种,每个小区总播种量为4 g/m2,豆科与禾本科混播比例为2∶3且同一科覆盖作物播种比例为1∶1,覆盖作物距猕猴桃树0.5 m。每年4月份和7月份的雨季分别撒施硫酸钾复合肥2.58 kg和尿素710 g,其中硫酸钾复合肥N、P2O5、K2O 的质量比为14∶16∶15,尿素含氮46%,即每个小区的施氮量为80 kg/hm2。试验小区呈矩形,长43 m、宽2 m,面积为86 m2。覆盖作物每年刈割2~3次,将其覆盖于猕猴桃树行间自然腐解,清耕区定期进行人工除草。各处理区的生态条件和田间管理措施保持一致。
1.2 植物和土壤样品采集
2019年11月采集植物和土壤样品。每个小区随机选取3个1 m×1 m的样方,采集样方内植物地上部。同时,用直径5 cm的土钻在每个小区以“S”形采集10钻0—20 cm耕层土壤,混合后剔除土壤中可见的石块和动植物残体,分成两部分放入塑封袋中带回实验室,一部分土样放入带有冰袋的盒子,用于分子生物学分析、硝化潜势及含水量的测定;另一部分土样风干过筛后用于土壤基本理化性质的测定。
1.3 测定方法
1.3.1 植株生物量和土壤理化性质分析 植物地上部在 65℃下烘干至恒重,称重记为地上部生物量。新鲜土样在105℃烘箱中烘干12 h测定土壤含水量。使用AA3流动分析仪 (SAN++,Skalar,Holland) 测定鲜土铵态氮 (NH4+-N) 和硝态氮 (NO3–-N)含量。利用 pH 计 (Delta 320,Mettler-Toledo Instruments Co.,Shanghai,China) 以土水比 1∶2.5(g/mL) 测定土壤pH。采用常规重铬酸钾容量法—外加热法,测定土壤有机碳含量。采用硫酸消煮—凯氏定氮法测定土壤全氮含量。
1.3.2 土壤硝化潜势的测定 通过测定短期内土壤硝化速率估算土壤硝化潜势是评价土壤硝化活性的方法之一,本研究参考Yao等[9]的方法并略有改进。采用悬浮液培养法,培养基中含有1 mmol/L (NH4)2SO4的磷酸盐缓冲液 (PBS)[19]。称取10.0 g新鲜土壤置于250 mL白色塑料瓶中,每种土壤3个重复。向白色塑料瓶中分别加入120 mL的液体培养基,置于摇床上 (180 r/min) 振荡培养 24 h。培养期间,分别于 2、4、16、22和24 h采集20 mL摇匀的土壤悬浮液,过定量滤纸,滤液在–20℃保存,使用AA3流动分析仪 (SAN++,Skalar,Holland) 测定 NO3–-N及NH4+-N浓度。
土壤硝化潜势计算以培养时间为横坐标、提取液中NO3–-N含量为纵坐标,求出斜率,即可得单位时间内NO3–-N含量的增长速率。计算公式如下:
Np=R×(0.12 +V1)×24/m[20]
式中:Np为土壤硝化潜势[mg/(kg·d)];R为NO3–-N含量的增长速率[mg/(L·h)];0.12为缓冲液体积(L);V1为土壤样品中水分体积 (L);m为烘干土质量 (kg)。
1.3.3 土壤DNA提取和实时定量PCR 称取0.3 g冻干土,按照 DNeasy® PowerSoil® Kit试剂盒(Promega,USA) 所提供的方法提取土壤DNA。DNA的浓度和纯度采用NanoDrop分光光度计(NanoDrop Technologies,USA) 测定。AOA-amoA和AOB-amoA的定量测定在iCycler iQ5仪器(Bio-Rad Laboratories,Inc.,USA) 上完成,所用的引物序列如表1所示。AOA-amoAqPCR反应条件[21]:95℃ 预变性 5 min;95℃ 变性 30 s,60℃ 退火 45 s,72℃延伸30 s,38个循环。AOB-amoAqPCR反应条件[22]:95℃ 预变性 30 s;95℃ 变性 30 s,58℃ 退火 45 s,72℃ 延伸 30 s,40 个循环。采用 SYBR Green染料法进行荧光定量PCR,反应体系为20 μL,包括 2 μL DNA 样品,1 μL 上下游引物 (10 μmol/L),10 μL 2×SYBR Premix Ex TaqⅡ,其余用 RNase-Free ddH2O补足。各基因都在延伸阶段收集荧光,得到CT值。各样品重复3次。
利用标准曲线法计算基因拷贝数。用含AOA-amoA和AOB-amoA基因的重组PGEM-T载体为标准质粒。计算出标准质粒的拷贝数,按10倍浓度梯度稀释,以10–2~10–7浓度梯度稀释的标准质粒进行荧光扩增,设置阴性对照。以标准质粒拷贝数的log值为横坐标,不同浓度梯度质粒的CT值为纵坐标,建立标准曲线。本试验AOA-amoA和AOB-amoA基因的扩增效率分别为93.6%、91.7%,R2>0.99。
1.3.4 末端限制性片段长度多态性 (T-RFLP) 土壤DNA的PCR扩增引物如表1所示,其中正向引物的 5’末端用 6-carboxyfluorescein (FAM) 标记。AOA-amoAT-RFLP反应条件[21]:94℃预变性5 min;94℃ 变性 30 s,55℃ 退火 45 s,72℃ 延伸1 min,35个循环。AOB-amoAT-RFLP 反应条件[22]:94℃ 预变性 3 min;94℃ 变性 30 s;53℃ 退火 45 s,72℃ 延伸 1 min,30 个循环。用 Wizard SV Gel and PCR Clean-Up System 试剂盒 (Promega,USA) 对PCR产物进行纯化。选取适当的限制性内切酶酶切纯化后的产物,即AOA-amoA基因和AOB-amoA基因采用的内切酶分别是HhaI (TaKaRa Biotechnology,Japan)和MspI (Takara Biotechnology,Japan)。AOA的酶切体系为20 μL,包括7 μL 灭菌超纯水,2 μL 10×M buffer,10 μL DNA,1 μLHhaⅠ;AOB 的酶切体系为 20 μL,包括 5 μL 灭菌超纯水,2 μL 10×T buffer,2 μL 0.1% BAS,10 μL DNA,1 μLMspⅠ。酶切时间为 6 h。
酶切样品送至生工生物工程 (上海) 股份有限公司,使用 ABI Prism 3700 Genetic Analyzer (Applied Biosystems,USA) 进行毛细管电泳,检测酶切片段。使用 GeneMarker软件 (SoftGenetics,USA) 分析得到的图谱。末端限制性片段(terminal restriction fragments, T-RFs)的相对丰度用每个片段的峰面积除以总的峰面积表示。
1.3.5 克隆测序与系统发育树分析 使用ArchamoAF/Arch-amoAR和amoA-1F/amoA-2R 引物 (表 1)分别对每个样本中AOA和AOB的amoA基因进行PCR 扩增。PCR 反应 (50 μL) 包含 4 μL DNA 样品,2 μL 上下游引物 (10 μmol/L),5 μL 10×buffer,4 μL dNTPs,0.5 μL TaqHs,其余用 RNase-Free ddH2O 补足。将AOA和AOB的PCR产物单独混合,分别构建混合的AOA和AOB克隆文库。混合PCR产物用Wizard SV Gel and PCR Clean-Up System 试剂盒(Promega,USA) 进行凝胶纯化。纯化后的PCR产物连接到 PGEM-T Easy Vector (Promega,USA) 中,然后转化到大肠杆菌 JM109 (Takara Biotechnology)。从AOA和AOB克隆文库中分别随机选取120和170个阳性克隆进行测序,并对所得序列利用ClustalX2和Mothur软件进行同源性分析,显示超过97%的一致性序列被划分为相同的操作分类单位(OUT),每个OTU的代表性序列与NCBI BLAST比对分析,获取相近典型菌株的基因序列。利用MEGA5.0 中的邻接法 (Neighbor-Joining) 进行系统发育分析。

表 1 氨氧化微生物功能基因的引物及序列Table 1 Primers and sequences of ammonia-oxidizing microorganism functional genes
1.4 数据处理
微生物群落多样性指数按如下公式进行计算:
丰富度指数 (S)=物种数
香农-维纳多样性指数 (Shannon-Weinner指数,H')=–∑(Pi×lnPi)
式中,Pi为第i个物种相对丰度。
均匀度指数 (Pielou 指数,E)=H'/(lnS)
使用SPSS 22.0软件,对植物地上部生物量、土壤理化性质、AOA-amoA和AOB-amoA基因拷贝数以及群落多样性指数进行单因素方差分析与Pearson相关性分析。使用Canoco 5软件,对土壤理化性质和AOA-amoA、AOB-amoA基因群落结构进行冗余分析 (redundancy analysis,RDA)。利用 Mega 5.0 构建系统发育树。
2 结果与分析
2.1 不同覆盖作物处理模式对土壤理化性质、硝化潜势及地上部植物生物量的影响
如表2所示,WR模式显著提高了土壤铵态氮和硝态氮含量以及土壤含水率,同时降低了土壤pH。不同覆盖作物种植模式下土壤硝化潜势提高,变化范围 4.54~7.73 mg/(kg·d),且在 W 模式下最高,与CK对照相比提高了121.49%。不同覆盖作物种植模式对植物地上部生物量、土壤有机质、全氮含量影响不显著。

表 2 不同覆盖作物下猕猴桃园土壤理化性质 (n=3)Table 2 Physiochemical properties in soils of kiwifruit orchard under different cover crops
2.2 不同覆盖作物处理模式对土壤AOA-amoA和AOB-amoA基因丰度的影响
图1显示,覆盖作物种类对土壤AOA-amoA和AOB-amoA基因丰度的影响不同。土壤AOA-amoA基因丰度范围为 1.86×105~2.75×105个拷贝数/g,W处理的AOA-amoA基因丰度最高,比CK显著提高了2.85%,WR、WRV及WRVF处理的AOA-amoA基因丰度也有一定的增加,但与CK间差异没有达到显著水平。土壤AOB-amoA基因丰度范围为 2.00×104~4.40×104个拷贝数/g,W 和WR处理的AOB-amoA基因丰度比CK分别增加了4.95%、6.42%,WRV和WRVF两个处理的AOB-amoA基因丰度没有显著变化。
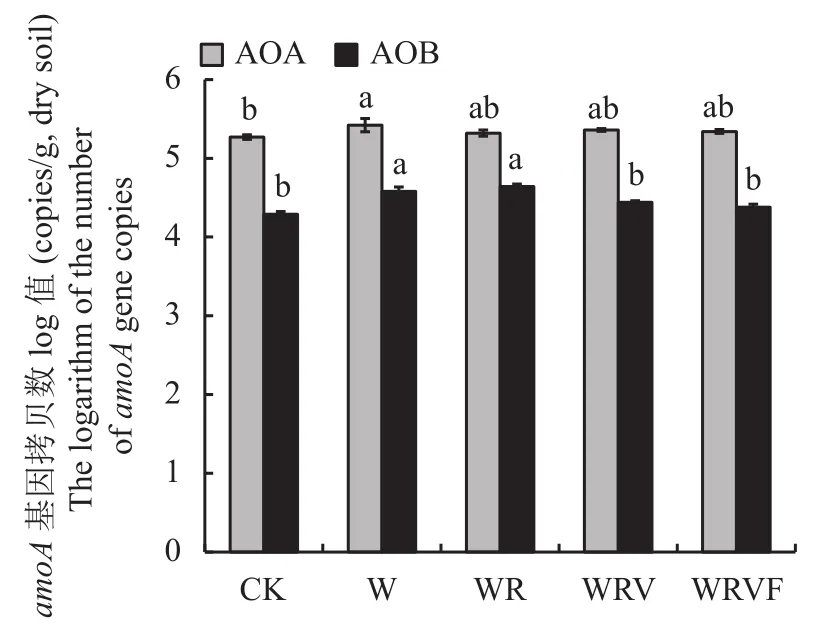
图 1 不同覆盖作物下猕猴桃园土壤AOA-amoA和AOB-amoA基因的丰度Fig.1 Abundance of AOA-amoA gene and AOB-amoA gene in kiwifruit orchard soils under different cover crops
2.3 不同覆盖作物处理模式对土壤氨氧化古菌(AOA) 与氨氧化细菌 (AOB) 群落结构的影响
2.3.1 末端限制性片段长度多态性 (T-RFLP) 分析如图2所示,相对丰度不到1%的未参与计算,相差为 ± 1 bp的片段为同一T-RF,大于10%的TRFs为优势片段[23]。AOA基因经过酶切主要得到5个末端限制性片段 (T-RFs),其中165、267、542、635 bp所代表的T-RFs片段为优势片段,在W、WR模式中片段542 bp的百分含量显著升高,在WRV和WRVF模式中,片段635 bp的百分比含量显著降低,说明不同T-RFs所代表的AOA在不同种植模式中所占比例发生改变,但AOA群落结构未发生明显改变。AOB经过酶切主要得到3个末端限制性片段 (T-RFs),分别是 T-RFs 55、155、234 bp,W处理的土壤中未检测到T-RFs 55 bp片段,且234 bp 片段的百分含量显著升高 (P< 0.05),而 WR、WRV、WRVF模式均检测到T-RFs 55、155、234 bp片段,这说明不同T-RFs所代表的AOB在不同种植模式下发生变化,其氨氧化细菌 (AOB) 群落结构可能发生改变。

图 2 不同覆盖作物下猕猴桃园土壤AOA-amoA和AOB-amoA基因T-RFs相对丰度Fig.2 Relative abundance of T-RFs of AOA-amoA gene and AOB-amoA gene in kiwifruit orchard with different cover crops
覆盖作物处理对土壤AOA多样性指数和均匀度指数以及AOB多样性指数有显著性影响(表3)。WR处理显著降低了AOA的多样性指数,WR和WRVF处理显著降低了AOA均匀度指数,而WRV和WRVF模式显著提高了AOB的多样性指数。

表 3 不同覆盖作物下猕猴桃园土壤氨氧化微生物群落多样性指数Table 3 Ammonia-oxidizing microbial community diversity index in kiwifruit orchard with different cover crops
2.3.2 克隆测序及系统发育树分析 从混合AOA和AOB克隆文库中分别随机选取120和170个阳性的AOA和AOB克隆进行测序。最终分别获得107和165条可用的AOA和 AOB序列,分别被划分为21和7个OTUs。利用Mega软件对AOA和AOB各自所有的OTUs及已知序列进行系统发育树的构建(图3),结果表明覆盖作物下猕猴桃园土壤AOA为Nitrososphaera(44.2%)和Nitrosotalea(45.8%),其中属于Nitrososphaera的片段包括T-RFs 165、255、267、542、635 bp,而 T-RFs 255、542、635 bp 剩余部分属于Nitrosotalea,不同覆盖作物种类处理AOA两个菌属的丰度与克隆文库没有显著差异。覆盖作物下猕猴桃园AOB为Nitrosospira(96.98%) 和Nitrosomonas(3.02%),其中属于Nitrosospira的片段有 T-RFs 155、55、234 bp,而 T-RFs 55、234 bp 剩余部分属于Nitrosomonas,每个处理中均含T-RFs 234 bp片段,不同覆盖作物处理AOB的群落组成没有显著差异。

图 3 不同覆盖作物下猕猴桃园土壤氨氧化古菌 (AOA) 和氨氧化细菌 (AOB) 系统发育树Fig.3 Phylogenetic trees of ammonia-oxidizing archaea (AOA) and ammonia-oxidizing bacteria (AOB)in kiwifruit orchard with different cover crops
2.4 土壤理化性质、硝化潜势、土壤氨氧化微生物群落多样性的相关分析
对土壤理化性质、硝化潜势与土壤微生物群落多样性进行Pearson分析 (表4),结果表明AOA-amoA基因丰度与土壤全氮含量呈显著正相关,AOA 多样性指数 (H′)分别与土壤 NO3−-N含量和土壤含水率呈显著正相关和负相关,AOA均匀度指数(E) 与土壤 NH4+-N含量和土壤有机质含量呈显著正相关。AOB-amoA基因丰度与土壤有机质、全氮含量及硝化潜势 (Np) 呈显著正相关,而其它3个多样性指数与测定的土壤性状无显著相关性。

表 4 氨氧化微生物多样性与土壤理化性质间的相关性分析Table 4 Correlation analysis of ammonia-oxidizing microorganisms diversity and soil physicochemical properties
微生物群落多样性和土壤理化性质的关系应用冗余分析 (RDA),结果 (图 4) 表明,轴一和轴二对AOA-amoA基因群落结构的解释量分别为38.54%和4.64%,WR模式分布在第二象限,基因群落结构主要受土壤含水率、铵态氮、硝态氮和有机质含量影响。轴一和轴二对AOB-amoA基因群落结构的解释量分别为52.11%和0.89%,轴一明显的区分了CK和WRVF模式,而WR与CK模式下基因群落结构更为相似,且土壤pH (P=0.016) 对AOB-amoA基因群落结构有显著性影响。

图 4 不同覆盖作物下猕猴桃园土壤AOA-amoA和AOB-amoA基因群落结构和环境因子的冗余分析Fig.4 Biplot of redundancy analysis (RDA) on relationship between environmental factors and community structure of AOA-amoA gene and AOB-amoA gene in kiwifruit orchard with different cover crops
3 讨论
植物通过根系吸收土壤养分,不同植物吸收利用养分的情况不同。有研究表明,果园种植不同覆盖作物、种植覆盖作物的时间长短以及生态环境的差异都会对土壤养分状况产生影响[24]。孙霞等[25]的研究结果表明,2年豆科覆盖作物种植能够提高酸性土壤有机质、速效氮含量及土壤含水率,但土壤全氮含量却有所下降且pH变化并不明显。焦润安等[26]的研究结果表明,3年自然生草提高了碱性土壤有机质含量,降低了土壤pH;而李华等[27]的研究结果表明,1年或者2年的覆盖作物种植均提高了碱性土壤pH。潘介春等[28]的研究结果表明,短时间覆盖作物种植降低了酸性土壤全氮含量及pH,并提高了土壤含水率。本研究中,WR模式显著提高了土壤含水率 (P< 0.05),而其他3种种植模式的土壤含水率与CK 没有显著性差异 (P> 0.05,表 2)。与前人研究结果不尽一致,可能原因在于单种白三叶草不能完全覆盖裸露地面,导致土壤蒸发量较高;随着覆盖作物种类和地表覆盖度提高到一定程度,土壤蒸散量降低,有利于稳定土壤墒情,土壤含水量相对较高。本研究中,WR 模式显著降低了 pH (P< 0.05),而其他3种种植模式与CK相比无显著性差异,与前人研究覆盖作物种植降低土壤pH以及pH变化不明显的研究结果一致,造成这种现象的原因可能在于不同的覆盖作物在腐解过程中释放了不同含量的碱性物质[29]。土壤中的速效养分受耕作措施的影响很大,如施肥量、地面植被类型等都会对土壤中铵态氮和硝态氮等的含量产生直接影响,豆科植物可以通过固氮作用增加土壤中的铵态氮,不同豆科植物的固氮能力不同,已有研究表明紫花苜蓿固氮能力要高于白三叶草和毛苕子[15, 30],而本研究中混种白三叶草+黑麦草区域土壤铵态氮含量显著高于混种白三叶草+黑麦草+毛苕子的区域 (P< 0.05),可能原因在于:1) 白三叶的固氮能力要高于毛苕子[31];2)WR模式下的土壤固氮能力高于WRV模式下,不同的覆盖作物会产生不同的土壤固氮菌群[32]。
植被类型影响土壤环境因子 (土壤pH、土壤铵态氮和土壤有机质含量),而氨氧化微生物对土壤环境因子具有特异的选择性,因此土壤环境因子的变化会对氨氧化过程乃至整个氮循环产生影响[33]。硝化潜势在一定程度上可以反映土壤环境中氨氧化微生物的活性[34]。Souto等[35]的研究结果表明,植物分泌的酚类化感物质能够刺激氨氧化微生物的活性,进而影响了氨氧化微生物群落结构。在本研究中,种植不同的覆盖作物,土壤硝化潜势会稍有不同,W模式显著提高了土壤硝化潜势和AOA-amoA、AOB-amoA基因丰度 (P< 0.05),而 W 模式和 CK 对照模式下的土壤理化因子没有显著性差异 (P>0.05),可能原因在于白三叶草根系分泌物对氨氧化古菌 (AOA) 和氨氧化细菌 (AOB) 的活性具有促进作用,进而影响不同覆盖作物处理下土壤硝化潜势,植物群落和土壤养分循环的互作关系通常是通过进入土壤中的根系分泌物和植株凋落物来实现的[36]。
前人研究结果表明,在酸性土壤中氨氧化古菌(AOA) 起主导作用,在中碱性土壤中氨氧化细菌(AOB) 起主导作用。Jia等[37]通过微宇宙、分子指纹图谱及DNA同位素探针技术等试验方法,发现中性土壤环境中氨氧化细菌 (AOB) 占主导地位;Xia等[38]通过DNA探针同位素标记微宇宙试验表明,碱性土壤环境中氨氧化细菌 (AOB) 占主导地位;Zhang等[39]的研究结果表明,酸性土壤环境中氨氧化古菌(AOA) 占主导地位且AOA-aomA基因丰度与硝酸盐浓度显著正相关。本研究中,土壤pH始终在6.7附近,且Pearson相关分析发现AOA-amoA基因丰度与土壤硝化潜势无显著关系,而AOB-amoA基因丰度与土壤硝化潜势显著正相关,说明氨氧化细菌(AOB) 在本试验中性土壤环境中占主导地位,同时曾有研究发现在农业土壤中虽然氨氧化古菌 (AOA)基因丰度显著高于氨氧化细菌 (AOB) 基因丰度,但是起主导作用的是氨氧化细菌 (AOB) 而不是氨氧化古菌 (AOA)[37],本研究与前人研究结果一致。WR模式显著提高了 AOB-amoA基因丰度 (P< 0.05),而AOA-amoA基因丰度无显著性变化 (P> 0.05),造成这一结果的原因可能在于:1) 在中性土壤中,AOA-amoA基因丰度尽管在土壤中保持很高的水平,但氨氧化细菌 (AOB) 对氨的响应更为敏感;2) 研究发现,影响氨氧化微生物丰度的并非植被的种植,而是土壤养分含量的变化[40],WR模式显著提高了土壤铵态氮和硝态氮含量,而低铵态氮环境有利于氨氧化古菌(AOA) 的生长,高铵态氮环境有利于氨氧化细菌(AOB) 的生长[41],故进而影响了AOB-amoA基因丰度;3) 土壤水分在调节氨氧化微生物群落结构方面起着重要作用,能够缓解水分压力[42],有利于氨氧化细菌 (AOB) 的生长[43]。Zhou 等[44]对紫色土的研究得出,氮和磷可能是影响氨氧化细菌 (AOB) 群落组成和土壤硝化潜势的重要因素,而土壤pH可能对紫色土壤氨氧化古菌 (AOA) 群落结构和硝化潜势起重要作用。Bao等[45]对北运河沉积物的研究表明,土壤全氮、有机质、铵态氮和pH是影响氨氧化微生物群落结构的重要因素。在本研究中,Pearson相关分析发现,氨氧化古菌 (AOA) 多样性指数分别与硝态氮(NO3–-N) 含量和土壤含水率呈显著正相关和负相关(P< 0.05),并且RDA分析发现土壤pH显著影响氨氧化细菌 (AOB) 群落组成,而对氨氧化古菌 (AOA)群落结构的影响不显著,由此说明由覆盖作物种植引起的土壤理化性质变化是导致氨氧化微生物群落结构显著变化的主要因素。
本研究结合各处理的T-RFLP结果分析,WR模式下优势片段T-RFs 542 bp所代表的优势菌群数量显著升高,同时显著降低了氨氧化古菌 (AOA) 多样性和均匀度指数,可能原因在于T-RFs 542 bp所代表的优势菌群的生长抑制其他氨氧化菌群的生长[46]。在不同种植模式中AOB-amoA基因中的优势片段TRFs 155 bp 是属分类水平中的Nitrosospira,即 AOB-amoA基因中的优势菌属为Nitrosospira,与土壤生态系统中氨氧化细菌 (AOB) 群落结构组成以Nitrosospira为主的研究结果[47]相一致。
总之,种植覆盖作物后较多的植物根量和凋落物进入到土壤,它们的输入和分解促进土壤有机质的形成和积累,增加土壤养分含量,改变土壤微生物的生长环境和食物来源,影响土壤微生物的繁殖能力[48]。覆盖作物根系分泌物为土壤微生物提供碳源,不同的覆盖作物分泌不同的物质,为土壤微生物种类及数量的增加提供了基础[49]。豆禾混播促进了土壤微生物的生长,且微生物的生长又促进了土壤有机物的矿化、矿质营养含量的提高,进而形成覆盖作物与微生物、矿质营养在土壤中的相互依存相互促进的关系[37]。
综上所述,本研究以连续种植不同覆盖作物的猕猴桃果园为研究对象,探讨了猕猴桃园覆盖作物种植对土壤氨氧化微生物丰度、群落结构以及土壤生态系统的影响,结果表明两种混种模式显著改变了猕猴桃园土壤理化性质,从而改变土壤氨氧化微生物丰度和群落结构,影响了氮循环过程中的氨氧化作用,进而影响了整个氮循环。本研究探讨了果园种植不同覆盖作物对土壤氨氧化微生物丰度和群落结构的影响机制,为理解覆盖作物种植对土壤氨氧化微生物群落结构和土壤生态系统的影响提供了理论依据。本研究采用的土壤氨氧化微生物分析方法为荧光定量 PCR (fluorescence quantitative PCR)、末端限制性片段长度多态性 (terminal restriction fragment length polymorphism,T-RFLP) 以及克隆测序技术,但是只能分析土壤氨氧化微生物部分种群,需要采用更为先进的土壤微生物分析方法,如高通量测序、同位素示踪技术等,对土壤氨氧化微生物全体进行研究;且本研究仅为2年,不同年限的覆盖作物种植会引起不同的环境变化,进而可能对研究结果产生影响,因此要深入研究覆盖作物种植对土壤氨氧化微生物的影响机制,还需要做长期的监测研究。
4 结论
1) 与清耕相比,覆盖作物种植对氨氧化微生物(AOA-amoA和AOB-amoA) 基因丰度有显著性影响,白三叶草单种可显著提高AOA-amoA基因丰度,白三叶草单种或者白三叶草和黑麦草混种可以显著提高AOB-amoA基因丰度。
2) 不同 T-RFs 所代表的 AOA 在不同种植模式中所占比例发生变化,但AOA群落结构未发生明显改变,不同T-RFs所代表的AOB在不同种植模式下发生变化,其氨氧化细菌 (AOB) 群落结构可能发生改变。覆盖作物处理对土壤AOA多样性指数、均匀度指数以及AOB多样性指数均有显著影响。
3) 覆盖作物种类对氨氧化微生物群落组成没有显著性影响,各覆盖作物处理的猕猴桃园土壤氨氧化古菌 (AOA) 均为Nitrososphaera和Nitrosotalea,氨氧化细菌 (AOB) 均为Nitrosospira和Nitrosomonas。
4) 影响氨氧化微生物群落结构变化的主要因素是土壤pH、含水率、铵态氮和硝态氮含量。白三叶草和黑麦草混合种植显著提高了土壤含水率、铵态氮和硝态氮含量,降低了土壤pH,白三叶草单种显著提高了土壤硝化潜势。

