苦荞FtCAD-1和FtCAD-2基因克隆及组织表达分析
段迎 杨晓琳 蔡苏云 贺润丽 尹桂芳 王艳青 卢文洁 孙道旺 王莉花

摘要:【目的】克隆苦荞肉桂醇脱氢酶(CAD)基因并分析其组织表达特性,为深入研究CAD基因在苦荞果壳形成中的分子调控机制提供理论依据。【方法】基于苦荞转录组测序结果,从苦荞厚果壳品种云荞1号和薄果壳品种小米荞克隆CAD基因,对其序列进行生物信息学分析,并利用实时荧光定量PCR(qRT-PCR)检测CAD基因在不同果壳厚度类型(厚果壳苦荞和薄果壳苦荞)不同组织的表达情况。【结果】从薄果壳苦荞和厚果壳苦荞中均克隆获得2条苦荞CAD基因,且这2条基因序列在薄果壳苦荞与厚果壳苦荞中均完全一致,命名为FtCAD-1和FtCAD-2。FtCAD-1基因的开放阅读框(ORF)长度为876 bp,编码291个氨基酸残基,为疏水性的稳定酸性蛋白,定位于细胞核和细胞质;FtCAD-2基因的ORF长度为1083 bp,編码360个氨基酸残基,为亲水性的稳定酸性蛋白,定位于细胞质。FtCAD-1和FtCAD-2蛋白均具有CAD蛋白3个典型的保守结构域,且不具有跨膜结构域和信号肽,属于非分泌蛋白。FtCAD-1与数据库目标蛋白2cf5.1.B的结构相似度为74.74%,而FtCAD-2与数据库目标蛋白5z0c.1.A的结构相似度为63.03%。FtCAD-1与拟南芥的第一类CAD蛋白(AtCAD4和AtCAD5)的亲缘关系较近;FtCAD-2与拟南芥的第二类CAD蛋白(AtCAD2、AtCAD3、AtCAD6等)的亲缘关系较近。FtCAD-1和FtCAD-2基因均在种仁中的相对表达量最高,且厚果壳苦荞与薄果壳苦荞间无显著差异(P>0.05)。FtCAD-1基因在厚果壳苦荞叶、花和果壳中的相对表达量显著(P<0.05,下同)或极显著(P<0.01)高于薄果壳苦荞,尤其是在厚果壳苦荞果壳中相对表达量是薄果壳苦荞的16倍。FtCAD-2基因除了在薄果壳苦荞果壳中相对表达量显著高于厚果壳苦荞外,在其他组织中的相对表达量均表现为厚果壳苦荞高于薄果壳苦荞。【结论】FtCAD-1属于第一类CAD基因,具有明显的组织表达特异性,推测其在苦荞木质素生物合成过程中发挥重要调控作用。
关键词: 苦荞;肉桂醇脱氢酶(CAD);RT-PCR克隆;生物信息学分析;实时荧光定量PCR
中图分类号: S517.035.3 文献标志码: A 文章编号:2095-1191(2021)12-3340-10
Cloning and tissue expression analysis of FtCAD-1 and FtCAD-2 genes from tartary buckwheat
DUAN Ying1, YANG Xiao-lin1, CAI Su-yun1, HE Run-li1*, YIN Gui-fang2,
WANG Yan-qing2, LU Wen-jie2, SUN Dao-wang2, WANG Li-hua2*
(1School of Traditional Chinese Medicine and Food Engineering,Shanxi University of Traditional Chinese Medicine,Taiyuan 030619, China; 2Yunnan Academy of Agricultural Sciences, Biotechnology and Genetic Germplasm Resources Research Institutes/Yunnan Provincial Key Laboratory of Agricultural Biotechnology/Key Laboratory of Southwestern Crop Gene Resources and Germplasm Innovation,Ministry of Agriculture and Rural Affairs, Kunming 650201, China)
Abstract:【Objective】To clone the cinnamyl alcohol dehydrogenase(CAD) gene of tartary buckwheat and analyze its tissue expression characteristics, in order to provide a theoretical reference for further study of the molecular mechanism underlying CAD gene regulation during the formation of the thin husk of tartary buckwheat. 【Method】The CAD gene of tartary buckwheat with thin husk (Yunqiao 1) and tartary buckwheat with thick husk (rice buckwheat) was cloned based on the related fragments of tartary buckwheat transcriptome and analyzed by bioinformatics. The expression of CAD in different tissues of the different husk types(thin husk and thick husk) was studied by real-time fluorescence quantitative PCR. 【Result】Two CAD genes of tartary buckwheat were cloned from tartary buckwheat with thin and thick husks,and found to be completely identical to FtCAD-1 and FtCAD-2. The open reading frame(ORF) of FtCAD-1 gene was 876 bp in length and encoded 291 amino acid residues. It was predicted to be a hydrophobic stable acidic protein and was located in the nucleus and cytoplasm. The ORF of FtCAD-2 gene was 1083 bp in length and encoded 360 amino acid residues predicted to be a hydrophilic and stable acidic protein located in the cytoplasm. Both proteins had three typical conserved domains of CAD proteins,but did not have transmembrane domains or signal peptides, and so belong to non-secreted proteins. The structural similarity between FtCAD-1 and the database target protein 2cf5.1.B was 74.74%, while the structural similarity between FtCAD-2 and the database target protein 5z0c.1.A was 63.03%. FtCAD-1 was closely related to the first type of protein (AtCAD4, AtCAD5) in Arabidopsis lignin synthesis, but FtCAD-2 was closely related to the second type of CAD protein (AtCAD2, AtCAD3, AtCAD6 et al.) in Arabidopsis. The relative expression levels of FtCAD-1 and FtCAD-2 genes were the highest in the seed kernels, where there was no significant difference observed between the thick-husk and the thin-husk tartary buckwheat types(P>0.05). The relative expression of FtCAD-1 gene in the leaves,flowers and husks of tartary buckwheat with thick husks was significantly (P<0.05, the same below) or very significantly (P<0.01) higher than those in tartary buckwheat with thin husks. This was most notable in the husks,where the relative expression level of FtCAD-1 thick husked tartary buckwheat was 16 times higher than that in tartary buckwheat with thin husk. In contrast,the relative expression level of the FtCAD-2 gene was significantly higher in thin husks than that in thick husks, while in other tissues FtCAD-1 showed a higher relative expression level in thick husked tartary buckwheat than that in thin husked tartary buckwheat. 【Conclusion】The FtCAD-1 gene belongs to the first type of CAD gene, it has tissue expression specificityand plays an important role in the regulation of lignin biosynthesis in tartary buckwheat.
Key words:tartary buckwheat; cinnamyl alcohol dehydrogenase(CAD); RT-PCR cloning; bioinformatics analysis; real-time fluorescence quantitative PCR
Foundation item: Construction Project National Technical System of Oat and Buckwheat Industry(CARS-07-C-2);Key Research and Development Project of Shanxi(201803D221012-6); Shanxi Province Natural Science Research General Project(20210302123231)
0 引言
【研究意义】苦荞[Fagopyrum tataricum(L.) Gaertn]具有利耳目、续精神、益气力等功效,是我国药食同源文化的典型体现(王世霞等,2016;周良等,2019)。苦荞果壳较厚,壳比率可达20%~30%,果壳十分坚韧,脱壳困难,从而难以实现生产整粒的苦荞米,无法高效获得具有高芦丁含量的麸皮层全营养苦荞米,很大程度上降低了苦荞米的营养功效(陈庆富等,2015)。研究发现,薄果壳苦荞的果壳木质素含量较厚果壳苦荞低(吴朝昕等,2020),更受人们的青睐。虽然目前关于苦荞薄果壳形成的原因尚不清楚,但研究证实肉桂醇脱氢酶(CAD)是木质素特异合成途径中最后一步的催化酶,在木质素生物合成过程中发挥重要作用。因此,克隆苦荞CAD基因并进行生物信息学分析,对探究CAD在苦荞薄果壳形成的作用机制及培育薄果壳苦荞均具有重要的意义。【前人研究进展】CAD属于NADPH依赖性酶家族,含有CAD蛋白家族的甘氨酸NADP(H)辅酶结构域GLGGV(L)G及2个Zn2+结合位点:GHE(X)2G(X)5G(X)2V和GD(X)9,10C(X)2C(X)2C(X)7C(Chao et al.,2014)。自Knight等(1992)首次从烟草茎部克隆获得CAD基因以来,陆续从番薯[Ipomoea batatas (L.) Lam.](Kim et al.,2010)、马尾松[Pinus massoniana Lamb.](张逢凯等,2014)、高梁(Sorghum vulgare Pers.)(王丽华等,2015)、香椿[Toona sinensis (A. Juss.) Roem.](隋娟娟等,2019)等多种植物中克隆获得。韩国粉(2014)通过转录组测序分析,筛选出软核山楂与硬核山楂间的差异表达基因,结果发现CAD基因在软核山楂果实中的相对表达量较硬核山楂显著下调。龚凌燕(2014)对不同石榴品种的籽粒中PgCAD基因的表达水平进行分析,结果发现PgCAD基因在红玉石籽、白玉石籽、粉皮、会理软籽和蒙自甜石榴4个石榴品种籽粒中的相对表达量与籽粒种皮总木质素含量呈正相关。张颖俊(2015)研究发现干旱胁迫下油菜CAD基因表达水平与茎秆和根系中的木质素含量均呈显著正相关。可见,CAD基因在木质素生物合成过程中发挥着重要调控功能,将芥子醛、松柏醛和香豆醛催化还原成木质素单体的前体物质芥子醇、松柏醇和香豆醇(Pandey et al.,2011)。近年来,从苦荞克隆获得的基因主要包括查尔酮合成酶基因(FtCHS1和FtCHS2)(孙朝霞等,2014)、类黄酮3′-羟化酶基因(FtF3′H)(李双江等,2014)、4-香豆酸辅酶A连接酶基因(Ft4CL)(凌瑶等,2015)、肉桂酸-4-羟基化酶基因(C4H)(刘荣华等,2017)等,其中大部分基因属于黄酮生物合成支路,而在木质素生物合成支路的基因研究鲜见报道。【本研究切入点】本课题组前期对厚果壳品种云荞1号和薄果壳品种小米荞进行转录组测序分析,结果发现大多数木质素生物合成相关基因在云荞1号的表达量高于小米荞,说明这些基因在苦荞果壳形成过程中发挥重要的调控作用。但对于这些基因的克隆及表达分析等相关研究鲜见报道。【拟解决的关键问题】基于前期苦荞转录组测序结果,克隆厚果壳苦荞品种云荞1号和薄果壳苦荞品种小米荞的CAD基因(FtCAD-1和FtCAD-2),对其序列进行生物信息学分析,并利用实时荧光定量PCR(qRT-PCR)檢测CAD基因在不同果壳类型苦荞不同组织的表达模式,为研究苦荞薄果壳形成的分子调控机制提供理论依据。
1 材料与方法
1. 1 试验材料
供试的薄果壳品种小米荞为云南地方品种。供试的苦荞厚果壳品种云荞1号为云南省农业科学院生物技术与种质资源研究所选育品种。分别取2个供试品种不同发育期的果实,剥离果壳,按品种混合在一起作为基因克隆材料。采用云荞1号和小米荞结实期的叶、花、茎、果壳和种仁等不同组织用于qRT-PCR检测检测。主要试剂:Trizol提取试剂盒(B511321)、柱式DNA胶回收试剂盒(B518131)、M-MuLV Reverse Transcriptase和RNase H(B500517)均购自生工生物工程(上海)股份有限公司;TaKaRa LA Taq(DRR02AG)、琼脂糖B(BBI,A600014)、4S Red Plus 核酸染色剂(10000×水溶液,BBI,A606695)、2×SG Fast qPCR Master Mix(BBI,B639271)等。主要仪器设备:PCR反应扩增仪(加拿大,BBI)、YXJ-2离心机(湘仪离心机仪器有限公司)、DYY-8型稳压稳流电泳仪(上海琪特分析仪器有限公司)、SW-CJ-1D洁净工作台(江苏苏洁净化设备厂)、FR980凝胶成像系统(上海复日科技有限公司)、U-3010紫外-可见分光光度计(Hitachi)、SMA4000微量分光光度计(Merinton Instrument,Inc)、HC-2518R高速冷冻离心机(安徽中科中佳仪器有限公司)和LightCycler480型荧光定量PCR仪(Rotkreuz,Switzerland)等。
1. 2 试验方法
1. 2. 1 总RNA提取及cDNA合成 利用Trizol提取试剂盒提取云荞1号和小米荞不同发育期的果壳混合材料的总RNA,并以1.5%琼脂糖凝胶电泳检测RNA的完整性。采用cDNA合成试剂盒反转录合成cDNA。
1. 2. 2 基因克隆 基于苦荞转录组分析结果,根据CAD基因序列设计2对特异引物(表1)。以cDNA为模板进行PCR扩增。反应体系为:2×GC Buffer Ⅰ 12.5 μL,上、下游引物(10 ?mol/L)各0.5 μL,40 ng/μL的DNA模板1.0 μL,dNTPs(10 mmol/L)0.2 μL,Taq DNA聚合酶(5 U/μL)0.2 μL,双蒸馏水补充至25.0 μL。扩增程序:95 ℃ 3 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 90 s,进行33个循环;72 ℃ 7 min。PCR产物用1%琼脂糖凝胶电泳进行检测,再和用柱式DNA胶回收试剂盒进行目的片段回收纯化,并委托生工生物工程(上海)股份有限公司进行测序。
1. 2. 3 生物信息学分析 利用NCBI数据库的ORFfinder和Conserved domains对开放阅读框(ORF)及保守功能结构域进行预测分析。通过ProtParam对蛋白的理化性质进行预测分析;利用ProtScale对蛋白的亲疏水性进行预测分析。通过TMHMM Server v.2.0、SignalP 5.0 Server、NetPhos 3.1 Server和Psort对蛋白的跨膜结构域、信号肽、磷酸化位点和亚细胞定位进行预测分析。利用SOPMA和SWISS-MODEL进行蛋白的二、三级结构预测。利用DNAMAN 9.0对GenBank中的同源蛋白进行氨基酸序列多重序列比对分析。同时,采用MEGA 6.0的邻接法构建不同物种CAD蛋白的系统发育进化树。
1. 2. 4 qRT-PCR检测 采用Trizol总RNA抽提试剂盒分别提取厚果壳和薄果壳苦荞的不同组织(叶、花、茎、果壳和种仁)总RNA,并利用Maxima Reverse Transcriptase试剂盒反转录合成cDNA。以cDNA为模板进行qRT-PCR检测。反应体系:2×SG Fast qPCR Master Mix 10.0 μL,10 μmol/L上、下引物(表1)各0.4 μL,40 ng/μL cDNA模板2.0 μL,双蒸馏水补充至20.0 μL。扩增程序:95 ℃预变性3 min;95 ℃ 5 s,60 ℃ 30 s,进行45个循环。以苦荞基因H3为内参,采用2-ΔΔCt法计算基因的相对表达量。
2 结果与分析
2. 1 总RNA提取及基因克隆结果
分别提取云荞1号和小米荞不同发育期的果壳混合材料总RNA,并利用1.5%琼脂糖凝胶电泳检测,结果(图1)显示,RNA条带清晰可见,28S RNA和18S RNA亮度比为1∶1~2∶1,表明RNA基本未降解,可用于基因克隆。利用RT-PCR均从薄果壳苦荞和厚果壳苦荞中克隆获得2条片段,利用1.0%琼脂糖凝胶电泳检测,结果显示,2条片段的大小约1000 bp(图2)。对这2条片段进行回收测序,结果发现2条CAD基因序列在薄果壳苦荞与厚果壳苦荞中均完全一致,命名为FtCAD-1(GenBank登录号:MW455112)和FtCAD-2(GenBank登录号:MW455113)。FtCAD-1基因的开放阅读框(ORF)长度为876 bp,编码291个氨基酸残基;FtCAD-2基因的ORF长度为1083 bp,编码360个氨基酸残基(图3)。
2. 2 生物信息学分析结果
2. 2. 1 理化性质预测分析结果 ProtParam预测结果显示,FtCAD-1蛋白的分子式C1401H2238N372O416S15,理论分子量31430.33 Da,理论等电点(pI)6.14,亲水性平均系数0.093,不稳定系数35.00,为疏水性的稳定酸性蛋白;FtCAD-2蛋白的分子式C1743H2759N465O515S21,理论分子量39142.15 Da,pI 6.75,亲水性平均系数-0.036,不稳定系数25.52,为亲水性的稳定酸性蛋白(表2)。ProtScale预测结果显示,FtCAD-1蛋白的最高分值(2.278)出现在多肽链中的第211位氨基酸,说明该氨基酸疏水性最强;最低分值(-2.678)出现在多肽链中的第49位氨基酸,说明该氨基酸亲水性最强。FtCAD-2蛋白的最高分值(2.100)出现在多肽链中的第98位氨基酸,说明该氨基酸疏水性最强;最低分值(-3.211)出现在多肽链中的第35位氨基酸,说明该氨基酸亲水性最强。NetPhos3.1 Server預测结果显示,FtCAD-1蛋白的磷酸化位点共有27个,其中丝氨酸、苏氨酸和酪氨酸分别为14、9和4个;FtCAD-2蛋白的磷酸化位点有30个,其中丝氨酸、苏氨酸和酪氨酸分别为16、12和2个。推测这些位点氨基酸发生磷酸化反应,从而对FtCAD-1和FtCAD-2蛋白的活性和生物学功能发生调控作用。TMHMM预测结果显示,FtCAD-1和FtCAD-2蛋白均不具有跨膜结构域。SignalP 5.0 Server预测结果显示,FtCAD-1和FtCAD-2蛋白均不具有信号肽,属于非分泌蛋白。Psort预测结果显示,FtCAD-1主要分布于细胞核和细胞质中;FtCAD-2主要分布于细胞质中。
2. 2. 2 结构域预测分析结果 对FtCAD-1和FtCAD-2蛋白的保守结构域(Conserved domains)进行预测分析,结果显示,FtCAD-1属于PLNO2514超基因家族,FtCAD-2属于PLNO2586超基因家族(图4)。
2. 2. 3 蛋白结构预测 通过SOPMA对蛋白的二级结构进行预测,结果发现FtCAD-1蛋白的二级结构由无规则卷曲(占37.80%)、α-螺旋(占26.46%)、延伸链(占25.77%)和β-折叠(占9.97%);FtCAD-2蛋白的二级结构由无规则卷曲(占44.44%)、α-螺旋(占23.89%)、延伸链(占24.72%)和β-折叠(占6.94%)(图5)组成。FtCAD-1和FtCAD-2蛋白的三级结构预测,结果如图6所示。FtCAD-1蛋白与数据库目标蛋白2cf5.1.B的结构相似度为74.74%,QMEAN值为-1.22,GMQE值为0.85,说明建模可信度较高。FtCAD-2蛋白与数据库目标蛋白5z0c.1.A的结构相似度为63.03%,QMEAN值为-0.87,GMQE值为0.82,说明建模可信度较高。
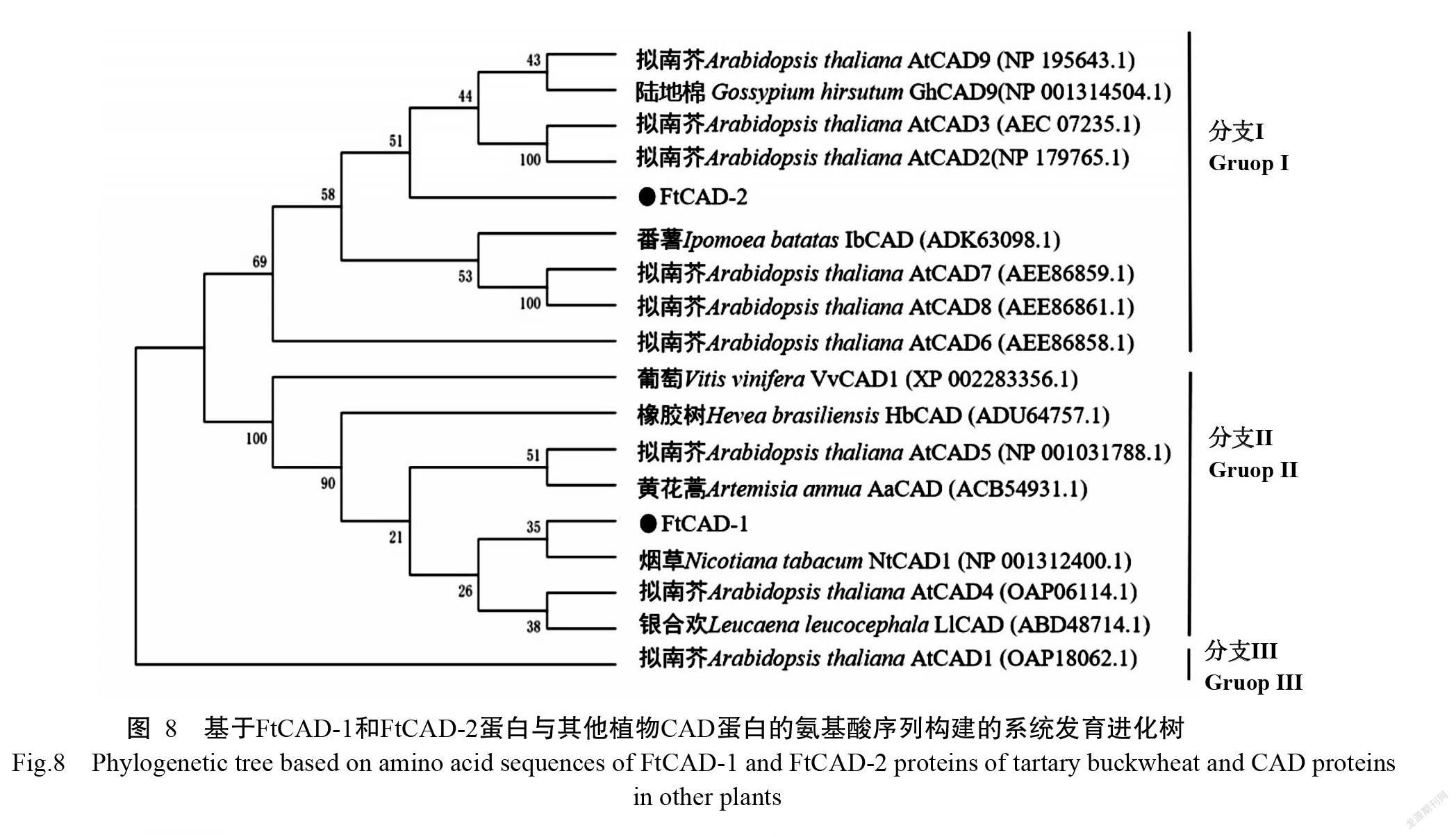
2. 2. 4 氨基酸序列分析及系统发育进化树构建
利用NCBI数据库中的BLASTp搜索FtCAD-1和FtCAD-2蛋白的同源序列,结果显示,FtCAD-1蛋白与GenBank中的同源序列包括芜青(Brassica rapa L,XP—009101881.2)、欧洲油菜(B. napus L,XP—013644950.1)、番茄(Solanum lycopersicum L,XP—004235066.2)、麻风树(Jatropha curcas L,XP—012075299.1)、澳洲棉(Gossypium australe,KAA345 7323.1)、橡胶树[Hevea brasiliensis(Willd. ex A. Juss.) Muell. Arg.,XP—021692374.1]、梅(Prunus mume,XP—008236378.1)、胡桃(Juglans regia L,XP—018 827699.1)、美洲棉(G. raimondii,KJB4736 1.1)和陆地棉(G. hirsutum L,ABZ01817.1)的CDA氨基酸序列相似性为75.26%~79.73%;FtCAD-2蛋白与GenBank中上述同源序列的氨基酸序列相似性为74.04%~79.89%。利用DNAMAN 9.0将FtCAD-1和FtCAD-2与其他物种的同源序列进行比对分析,结果显示FtCAD-1和FtCAD-2与其他植物的CAD蛋白均含有3个CAD蛋白家族的典型保守结构域(图7)。
运用MEGA 6.0构建拟南芥[Arabidopsis thaliana(L.) Heynh.]的9个AtCAD蛋白、其他7种植物的CAD蛋白及FtCAD-1和FtCAD-2的系统发育进化树,结果如图7所示。这些蛋白可分成3支,FtCAD-1和FtCAD-2蛋白分别聚在不同分支。其中,FtCAD-1与拟南芥AtCAD4和AtCAD5、葡萄(Vitis vinifera L.)VvCAD1、黄花蒿(Artemisia annua L.)AaCAD、橡胶树HbCAD、烟草(Nicotiana tabacum L.)NtCAD1、银合欢[Leucaena leucocephala(Lam.) de Wit]LlCAD聚为一支,属于第一类CAD蛋白。由于AtCAD4和AtCAD5蛋白在拟南芥木质素的合成中发挥重要作用(Jourdes et al.,2007),由此推測FtCAD-1蛋白在苦荞木质素的合成中发挥重要作用。由于FtCAD-2与拟南芥的AtCAD2、AtCAD3、AtCAD6、AtCAD7、AtCAD8、AtCAD9及陆地棉GhCAD9和番薯[Ipomoea batatas (L.) Lam.]IbCAD聚为一个分支,属于第二类CAD蛋白。AtCAD1蛋白单独聚为一个分支,属于第三类CAD蛋白。
2. 3 基因组织表达特性分析结果
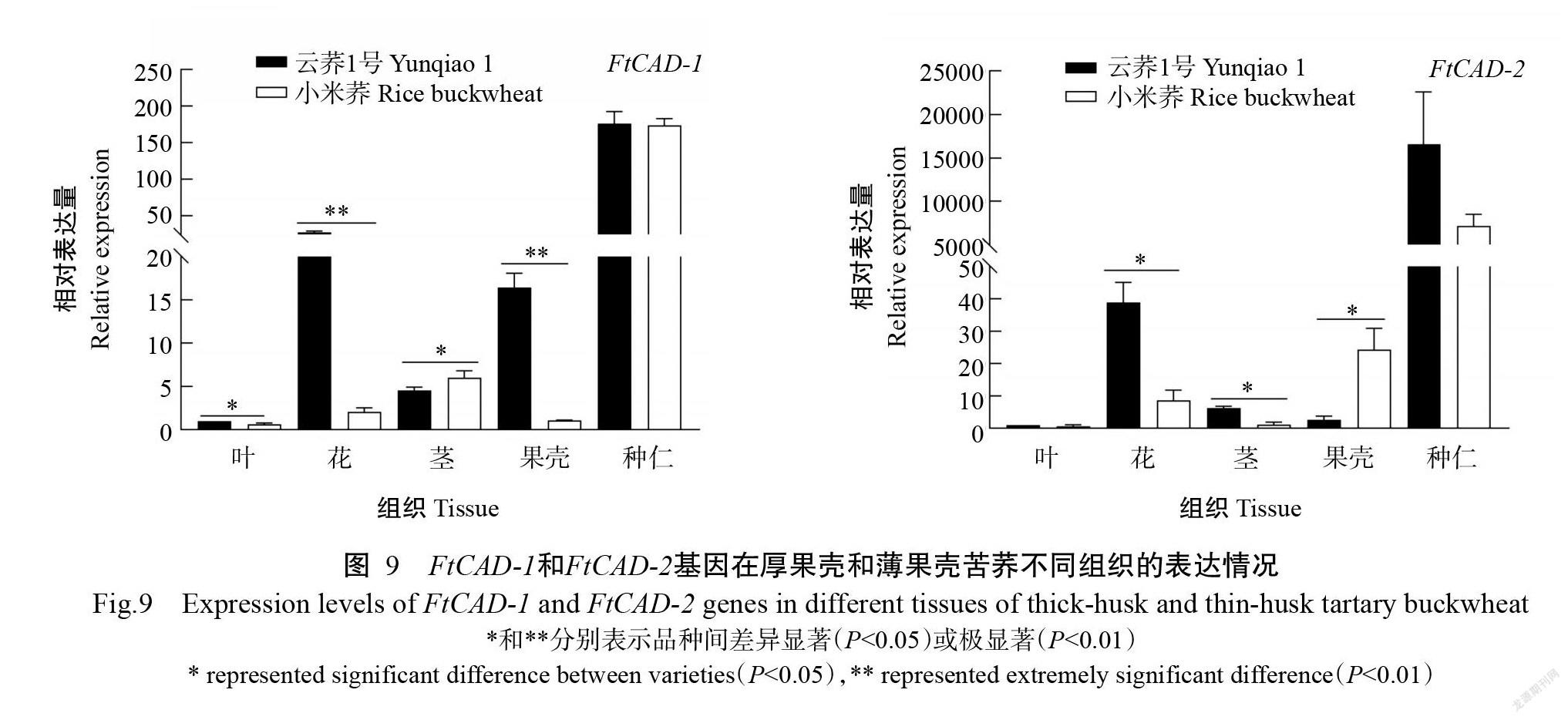
qRT-PCR检测苦荞FtCAD-1和FtCAD-2基因在厚果壳与薄果壳苦荞不同组织中的表达水平,结果如图9所示。FtCAD-1和FtCAD-2基因在不同组织的表达水平存在明显差异。FtCAD-1和FtCAD-2基因均在种仁中的相对表达量最高,且厚果壳与薄果壳苦荞间无显著差异(P>0.05)。FtCAD-1基因在厚果壳苦荞叶、花和果壳中的相对表达量显著(P<0.05,下同)或极显著(P<0.01,下同)高于薄果壳苦荞,尤其是在厚果壳苦荞果壳中相对表达量是薄果壳苦荞的16倍,但在薄果壳苦荞茎中的相对表达量显著高于厚果壳苦荞。FtCAD-2基因除了在薄果壳苦荞果壳中相对表达量显著高于厚果壳苦荞外,在其他组织中的相对表达量均表现为厚果壳苦荞高于薄果壳苦荞,尤其是在厚果壳苦荞花和茎的相对表达量显著高于薄果壳苦荞。由此推测FtCAD-1基因为控制苦荞厚薄果壳性状的关键基因。
3 讨论
苦荞有“五谷之王”“三降食品”的美称,营养价值和药用功效极高。但目前市面上的栽培苦荞大部分难以脱壳,严重影响了有效成分的利用。通过基因工程的方法探究厚果壳形成的调控机制已成为研究热点。CAD作为植物木质素合成过程中的重要限速酶,能将不同的肉桂醛转化为相应的肉桂醇,进而影响木质素的结构及形成,是非常理想的调控木质素合成的控制酶(Tobias and Chow,2004)。已有较多研究表明,CAD基因在木质素合成途径中发挥重要调控作用,且其表达量与木质素含量成正相关(刘莉等,2013;胡丹等,2015;郭永翠等,2020)。本研究从厚果壳苦荞品种云荞1号和薄果壳苦荞品种小米荞均克隆获得FtCAD-1和FtCAD-2基因,qRT-PCR检测发现二者在叶、花、茎、果壳和种仁中均有表达,但相对表达量存在明显差异。FtCAD-1基因在厚果壳苦荞果壳中的相对表达量是薄果壳苦荞的16倍,二者的差异达极显著水平,而FtCAD-2基因在薄果壳苦荞的相对表达量显著高于厚果壳苦荞。通过序列比对分析发现,厚果壳苦荞的FtCAD-1和FtCAD-2基因序列与薄果壳苦荞的FtCAD-1和FtCAD-2基因序列完全一致,而基因表达受到多种因素的影响,如启动子、转录因子的激活、光照、时间、温度等,推测FtCAD-1基因可调控苦荞果壳的木质素合成进而影响果壳厚度。
前人研究发现,CAD以多基因家族的形式存在,不同植物的CAD基因数目不同,功能也不同。例如模式植物拟南芥中主要存在9个CAD基因,其中AtCAD4和AtCAD5属于第一类CAD基因,均参与维管束的发育过程,在木质素的生物合成过程中将松柏醛和芥子醛还原为相应的醇;AtCAD2、AtCAD3、AtCAD6、AtCAD7、AtCAD8和AtCAD9属于第二类CAD基因,AtCAD1属于第三类CAD基因(Kim et al.,2004)。本研究的系统进化分析结果显示,FtCAD-1与拟南芥的AtCAD4和AtCAD5聚在一支,故推测FtCAD-1基因属于第一类CAD基因,参与调控木质素生物合成;FtCAD-2与拟南芥的AtCAD2、AtCAD3、AtCAD6、AtCAD7、AtCAD8、AtCAD9等聚在一支,故推测FtCAD-2属于第二类CAD基因。相对第一类CAD基因而言,第二和三类CAD基因在木质素生物合成中的作用并不突出,是第一类CAD基因的冗余基因(Kim et al.,2004)。因此,推测FtCAD-2基因在木质素合成方面作用不是很突出。多序列比对结果发现FtCAD-1和FtCAD-2虽然均含有3个CAD蛋白的典型保守结构域,但序列不完全一致。且FtCAD-1和FtCAD-2蛋白的三级结构也明显不同,其中FtCAD-1与2cf5.1.B的结构相似度较高,而FtCAD-2与5z0c.1.A的结构相似度更高。由于蛋白结构决定其功能,证明二者功能存在明显差异。因此本研究获得的苦荞FtCAD-1和FtCAD-2基因在薄果壳苦荞木质素生物合成途径的调控功能还需进一步验证。
4 结论
FtCAD-1基因属于第一类CAD基因,具有明显的组织表达特异性,推测其在苦荞木质素生物合成过程中发挥重要调控作用。
参考文献:
陈庆富,陈其饺,石桃雄,黄凯丰,梁龙兵,饶庆琳,唐链,张启迪. 2015. 苦荞厚果壳性状的遗传及其与产量因素的相关性研究[J]. 作物杂志,(2):27-31. [Chen Q F,Chen Q J,Shi T X,Huang K F,Liang L B,Rao Q L,Tang L,Zhang Q D. 2015. Inheritance of tartary buckwheat thick shell character and its relationships with Yield Factors[J]. Crops,(2):27-31.] doi:10.16035/j.issn.1001-7283.2015.02. 006.
龚凌燕. 2014. 石榴籽粒木质素合成相关基因的克隆及表达分析[D]. 合肥:安徽农业大学. [Gong L Y. 2014. Clo-ning and expression analysis of genes related with lignin synthesis in pomegranate seed[D]. Hefei:Anhui Agricultural University.]
郭永翠,秦江南,袁雨婷,张锐. 2020. 新疆核桃WJ-CAD和ZJ-CAD基因克隆及时空表达表达分析[J]. 基因组学与应用生物学,39(3):1215-1221. [Guo Y C,Qin J N,Yuan Y T,Zhang R. 2020. Cloning and expression analysis of WJ-CAD and ZJ-CAD genes of walnut in Xinjiang[J]. Genomics and Applied Biology,39(3):1215-1221.] doi:10.13417/j.gab.039.001215.
韩国粉. 2014. 软核山楂与硬核山楂基因表达差异分析及木质素合成相关转录因子基因的克隆与鉴定[D] 沈阳:沈阳农业大学. [Han G F. 2014. Screening differentially expressed genes between soft-endocarp and hard-endocarp hawthorns and cloning and identification of transcription factors regulating lignin biosynthesis[D]. Shenyang:Shen-yang Agricultural University.]
胡丹,刘星贝,汪灿,杨浩,李鹤鑫,阮仁武,袁晓辉,易泽林. 2015. 不同抗倒性甜荞茎秆木质素合成关键酶基因的表达分析[J]. 中國农业科学,48(9):1864-1872. [Hu D,Liu X B,Wang C,Yang H,Li H X,Ruan R W,Yuan X H,Yi Z L. 2015. Expression analysis of key enzyme genes in lignin synthesis of culm among different lod-ging resistances of common buckwheat(Fagopyrum esculentum Moench)[J]. Scientia Agricultura Sinica,48(9):1864-1872.]
李双江,朱冬寅,秦东,李成磊,陈惠,吴琦. 2014. 苦荞类黄酮3′-羟化酶基因的克隆及其冷胁迫下的组织表达[J]. 中草药,45(9):1300-1306. [Li S J,Zhu D Y,Qin D,Li C L,Chen H,Wu Q. 2014. Cloning of flavonoid 3′-hydroxylase gene from Fagopyrum tataricum and its tissue-specific expression under cold stress[J]. Chinese Traditional and Herbal Drugs,45(9):1300-1306.]
凌瑶,高飞,王安虎,李成磊,陈惠,吴琦. 2015. 苦荞4-香豆酸辅酶A连接酶基因(Ft4CL)的克隆及序列分析[J]. 广西植物,35(5):728-732. [Ling Y,Gao F,Wang A H,Li C L,Chen H,Wu Q. 2015. Cloning and sequence analysis of 4-coumarate:CoA ligase gene from Fagopyrum tatarium[J]. Guihaia,35(5):728-732.]
刘莉,孙虹丽,程召阳,贾兵,刘普,叶振风,朱立武,衡伟. 2013. 砀山酥梨褐皮芽变果皮中木质素生物合成相关基因克隆与表达分析[J]. 华北农学报,28(6):88-92. [Liu L,Sun H L,Cheng Z Y,Jia B,Liu P,Ye Z F,Zhu L W,Heng W. 2013. Cloning and expression of enzyme genes related to lignin biosynthesis in the pericarp of russet mutant of Dangshansuli[J]. Acta Agriculturae Boreali-Sinica,28(6):88-92.]
刘荣华,王丽,孙朝霞,侯思宇,李红英. 2017. 苦荞C4H基因的cDNA克隆及生物信息学分析[J]. 山西农业大学学报(自然科学版),37(11):767-773. [Liu R H,Wang L,Sun C X,Hou S Y,Li H Y. 2017. Cloning and bioinformatics analysis of C4H gene in tatary buckwheat[J]. Journal of Shanxi Agricultural University(Natural Science Edition),37(11):767-773.] doi:10.13842/j.cnki.issn1671-8151. 2017.11.002.
隋娟娟,邓红祥,杨京霞,孙晶晶,屈长青. 2019. 香椿TsCAD1基因的克隆与非生物胁迫下的表达特性[J]. 基因组学与应用生物学,38(10):4617-4625. [Sui J J,Deng H X,Yang J X,Sun J J,Qu C Q,2019. Cloning and expression characteristics of TsCAD1 gene under abiotic stress condition in Toona sinensis[J]. Genomics and Applied Biology,38(10):4617-4625.] doi:10.13417/j.gab.038.00 4617.
孫朝霞,侯思宇,郭彬,令狐斌,黄可盛,许冬梅,韩渊怀. 2014. 苦荞查尔酮合成酶基因序列特征及分子进化分析[J]. 分子植物育种,12(4):772-779. [Sun C X,Hou S Y,Guo B,Linghu B,Huang K S,Xu D M,Han Y H. 2014. Characterize of sequences and molecular evolution analysis of chalcone synthase in Fagopyrum tartaricum[J]. Molecular Plant Breeding,12(4):772-779.] doi:10. 13271/j.mpb.012.000772.
王丽华,李杰勤,詹秋文,樊飞飞. 2015. 高粱SbCAD4基因的克隆与序列分析及原核表达分析[J]. 湖南农业大学学报(自然科学版),41(6):595-601. [Wang L H,Li J Q,Zhan Q W,Fan F F. 2015. Cloning,sequence analysis and prokaryotic expression of gene SbCAD4 from Sorghum bicolor[J]. Journal of Hunan Agricultural University(Natural Sciences),41(6):595-601.] doi:10.13331/j.cnki.jhau.2015.06.004.
王世霞,李笑蕊,贠婷婷,幺杨,任贵兴,綦文涛. 2016. 不同品种苦荞麦营养及功能成分对比分析[J]. 食品与机械,32(7):5-9. [Wang S X,Li X R,Yuan T T,Yao Y,Ren G X,Qi W T. 2016. Comparable analysis of nutrition and functional active ingredients in different varieties of tartary buckwheat[J]. Food and Machinery,32(7):5-9.]doi:10.13652/j.issn.1003-5788.2016.07.002.
吴朝昕. 2020. 薄壳苦荞果壳结构及其发育中的转录组学分析[D]. 贵阳:贵州师范大学. [Wu C X. 2020. Shell structure of rice tartary buckwheat and its transcriptome analysis during development[D]. Guiyang:Guizhou Normal University.]
张逢凯,潘婷,盛璐,季孔庶. 2014. 马尾松CAD基因的克隆及其编码蛋白质的结构预测[J]. 分子植物育种,12(4):638-645. [Zhang F K,Pan T,Sheng L,Ji K S. 2014. Cloning and coding protein structure prediction on cDNA encoding cinnamyl alcohol dehydrogenase from Pinus massoniana[J]. Molecular Plant Breeding,12(4):638-645.] doi:10.13271/j.mpb.012.000638.
张颖俊. 2015. 干旱胁迫下油菜木质素含量及其关键基因表达的时空动态变化[D]. 重庆:西南大学. [Zhang Y J. 2015. Temporal and spatial dynamic changes of the lignin content and the expression of key genes of Brassica napus under drought stress[D]. Chongqing:Southwest University.]
周良,黄小燕,王炎,李振宙,吴兴慧,李振东,孔德章,陈庆富,黄凯丰. 2019. 钾肥对苦荞灌浆特性、根系形态及充实度的影响[J]. 河南农业大学学报,53(2):175-179. [Zhou L,Huang X Y,Wang Y,Li Z Z,Wu X H,Li Z D,Kong D Z,Chen Q F,Huang K F. 2019. Effects of potassic fertilizer on filling characteristics,root morphology,and plumpness of tartary buckwheat[J]. Journal of Henan Agricultural University,53(2):175-179.]
Chao N,Liu S X,Liu B M,Li N,Jiang X N,Gai Y. 2014. Molecular cloning and functional analysis of nine cinnamyl alcohol dehydrogenase family members in Populus tomentosa[J]. Planta,240(5):1097-1112. doi:10. 1007/s00425-014-2128-9.
Jourdes M,Cardenas C L,Laskar D D,Moinuddin S G A,Davin L B,Lewis N G. 2007. Plant cell walls are enfeebled when attempting to preserve native lignin configuration with poly-p-hydroxycinnamaldehydes:Evolutionary implications[J]. Phytochemistry,68(14):1932-1956. doi:10. 1016/j.phytochem.2007.03.044.
Kim S J,Kim M R,Bedgar D L,Moinuddin S G A,Cardenas C L,Davin L.B,Kang C H,Lewis N.G. 2004. Functional reclassification of the putative cinnamyl alcohol dehydrogenase multigene family in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America,101(6):1455-1460. doi:10.1073/pnas.0307 987100.
Kim Y H,Bae J M,Huh G H. 2010. Transcriptional regulation of the cinnamyl alcohol dehydrogenase gene from sweetpotato in response to plant developmental stage and environmental stress[J]. Plant Cell Reports,29(7):779-791. doi:10.1007/s00299-010-0864-2.
Knight M E,Halpin C,Schuch W. 1992. Identification and characterisation of cDNA clones encoding cinnamyl alcohol dehydrogenase from tobacco[J]. Plant Molecular Bio-logy,19(5):793-801. doi:10.1007/bf00027075.
Pandey B,Pandey V P,Dwivedi U N. 2011. Cloning,expression,functional validation and modeling of cinnamyl alcohol dehydrogenase isolated from xylem of Leucaena leucocephala[J]. Protein Expression and Purification,79(2):197-203. doi:10.1016/j.pep.2011.06.003.
Tobias C M,Chow E K. 2004. Structure of the cinnamyl-alcohol dehydrogenase gene family in rice and promoter activity of a member associated with lignification[J]. Planta,220(5):678-688. doi:10.1007/s00425-004-1385-4.
(責任编辑 陈 燕)

