西瓜OSCA基因家族全基因组鉴定及胁迫响应分析
张瑜 吴才君 苏文桢 周聪 滕丽姚 涂志伟 朱强龙

摘要:【目的】對西瓜OSCA基因家族进行成员鉴定、系统发育分析以及胁迫响应分析,为揭示西瓜OSCA基因生物学功能和抗逆分子机制提供参考依据。【方法】运用生物信息学的方法从西瓜全基因组数据库中鉴定出西瓜OSCA基因家族成员,根据染色体定位信息进行命名。对基因家族成员染色体位置、基因结构、共线性、启动子、蛋白结构和系统发育等进行全面分析。利用转录组数据和qRT-PCR分析西瓜OSCA基因在胁迫条件下的表达情况。【结果】西瓜OSCA基因家族有10个成员,不均等地分布在6条染色体上。西瓜OSCA蛋白分子量变化范围为24.59~91.97 kD,氨基酸数量为214~809 aa,多数定位于质膜上。共线性分析结果表明,西瓜OSCA基因在进化过程中发生了片段复制事件。西瓜OSCA基因家族分为三大类群,同一类群成员的外显子一内含子的组成模式、蛋白保守基序排列、蛋白二级结构数量比例和蛋白三级结构模型均相似。西瓜、水稻、番茄和拟南芥的OSCA基因被分为6个亚族,每个亚族均有西瓜OSCA基因分布。西瓜OSCA基因启动子区域含有厌氧诱导、冷胁迫应答、光反应和干旱胁迫应答等多种非生物胁迫响应元件。在干旱、低温和盐胁迫下,西瓜OSCA基因家族各成员表达量均有不同程度变化,其中ClaOSCA3和ClaOSCA5在3种不同胁迫条件下表达量均有显著差异(P<0.05)。【结论】西瓜OSCA基因在进化过程中具有一定保守性,与拟南芥、水稻和番茄OSCA基因存在较近亲缘关系。西瓜OSCA基因家族内部存在功能分化,ClaOSCA3和ClaOSCA5可能是胁迫响应机制中的重要抗逆基因。
关键词: 西瓜;OSCA基因;胁迫响应;生物信息学
中图分类号: S651 文献标志码: A 文章编号:2095-1191(2021)12-3330-10
Genome-wide identification and stress response analysis
of OSCA gene family in watermelon
ZHANG Yu, WU Cai-jun, SU Wen-zhen, ZHOU Cong, TENG Li-yao,
TU Zhi-wei, ZHU Qiang-long*
(College of Agronomy, Jiangxi Agricultural University, Nanchang 330045, China)
Abstract:【Objective】The member identification, phylogenetic analysis and stress response analysis of watermelon OSCA gene family were carried out to provide reference for revealing the biological function and molecular mechanism of stress resistance of watermelon OSCA gene. 【Method】The members of watermelon OSCA gene family were identified by bioinformatics method from watermelon genome database, and named according to chromosome location information. Chromosome position, gene structure, collinearity, promoter, protein structure and phylogeny of gene family members were analyzed comprehensively. Transcriptome data and qRT-PCR were used to analyze OSCA gene expression in watermelon under stress. 【Result】Watermelon OSCA gene family had 10 members, which were unevenly distributed on 6 chromosomes. The molecular weight of watermelon OSCA protein varied from 24.59 to 91.97 kD, and the number of amino acids ranged from 214 to 809 aa, most of which were located in the plasma membrane. The results of collinearity analysis showed that the fragment replication event occurred during the evolution of watermelon OSCA gene. The watermelon OSCA gene family was divided into three groups, and the exon-intron composition pattern, conserved motif arrangement of protein, the number proportion of secondary structure and the tertiary structure model of the members of the same group were similar. The OSCA genes of watermelon, rice, tomato and Arabidopsis thaliana were divided into 6 subgroups, watermelon OSCA gene was distributed in each subgroup. The promoter region of OSCA gene in watermelon contained several abiotic stress response elements such as anaerobic induction, cold stress response, light response and drought stress response. Under drought, low temperature and salt stress, the expression levels of OSCA gene family members in watermelon all changed to different degrees, and the expression levels of ClaOSCA3 and ClaOSCA5 all changed significantly under three different stress conditions(P<0.05). 【Conclusion】Watermelon OSCA gene is conserved in the evolutionary process, and is closely related to OSCA gene of A. thaliana, rice and tomato. Functional differentiation exists within the OSCA gene family in watermelon, and ClaOSCA3 and ClaOSCA5 may be important stress resistance genes in the stress response mechanism.
Key words: watermelon; OSCA gene; stress response; bioinformatics
Foundation item: National Natural Science Foundation of China(31960607)
0 引言
【研究意义】植物OSCA是一种能感知细胞渗透势变化的钙离子非选择性阳离子通道蛋白,在感受机制中调控信号转导以应对自然界中各种不利环境条件(Zhu,2002;Bartels and Sunkar,2005)。西瓜[Citrullus lanatus(Thunb.) Matsum. et Nakai]是富含多种维生素的夏季水果,市场需求量不断增加,抗逆育种研究是我国西瓜产业基础研究的重要内容(王超楠等,2018)。研究西瓜OSCA基因的功能及胁迫响应分子机制对培育优良抗逆西瓜品种有重要意义。【前人研究进展】植物在生长发育过程中,会受到自然界中许多不良环境因素的影响。其中干旱、低温、高温和盐等非生物胁迫会使得植物难以维持正常的细胞结构和离子平衡。在严重的逆境胁迫情况下,植物的生理机制会受到严重破坏。在植物应对外界不良环境条件的保卫机制中,作为第二信使的钙离子会在钙离子通道被激活后浓度快速升高,形成应激的钙离子信号。钙离子信号在下游转移过程中调控植物细胞产生一系列的生理变化(李建伟等,2017),诱导出植物的抗逆性来应对外界的非生物胁迫(朱强龙等,2017),使得植物的生命系统继续正常运转。钙信号的传导与植物体中钙离子通路蛋白密切相关,2014年钙离子通道蛋白OSCA首次在模式植物拟南芥中被发掘报道(Yuan et al.,2014)。钙离子通道蛋白在拟南芥中参与高渗胁迫响应(Batistic and Kudla,2012;Hou et al.,2014)。在前人对拟南芥OSCA基因家族进行的系统分析与鉴定中,基因家族分为4个亚族,15个家族成员均含DUF221功能结构域,在干旱或高盐等高渗胁迫下,DUF221作为钙离子依赖性通道的结构域发挥着重要作用。此外,OSCA基因被分别在水稻(Li et al.,2015)、梨(顾小雨,2017)等植物中鉴定和分析,验证了OSCA基因在不同胁迫响应中的生物学功能。【本研究切入点】目前为止,尚未见关于运用生物信息学手段对西瓜等葫芦科植物OSCA基因进行分析的研究报道。【拟解决的关键问题】基于西瓜全基因组数据库运用生物信息学的方法鉴定出OSCA基因家族成员,对西瓜OSCA基因的特征进行全面分析,利用转录组数据对西瓜OSCA基因在不同胁迫条件下的表达模式进行分析,筛选出重要抗逆基因并通过qRT-PCR对表达量进行检测,为西瓜OSCA基因生物學功能研究提供参考。
1 材料与方法
1. 1 试验设计
以江西农业大学农学院保存的LSW-177西瓜品种为试验材料,对四叶一心的LSW-177西瓜幼苗分别进行干旱(20% PEG6000)、低温(4 ℃)和盐(3 mmol/L KCl)胁迫处理。在处理后的0和6 h分别对西瓜幼苗的第2片真叶进行采样,设置3次生物学重复,将样品用液氮速冻后放入-80 ℃超低温冰箱中保存。
1. 2 西瓜OSCA基因家族成员鉴定
在拟南芥基因组数据库TAIR获取拟南芥OSCA基因家族的氨基酸序列,并将其作为参考序列,与葫芦科基因组数据库(http://cucurbitgenomics.org/)中的西瓜全基因组氨基酸序列进行比对(E-value小于1e-10,Identity大于60%)(朱强龙等,2017),筛选得到符合标准的西瓜OSCA家族候选基因。运用生信软件TBtools中的Simple HMM Search功能输出得到西瓜OSCA基因家族候选基因(Chen et al.,2020),与Blast分析结果进行比对后筛查矫正,利用PFAM(http://pfam.xfam.org/)对候选基因进行预测分析,含完整DUF221结构域的候选基因为西瓜OSCA基因家族成员。利用ExPASy(https://web.expasy.org/protparam/)分析西瓜OSCA基因家族成员的蛋白序列,获取蛋白理化信息,通过Softberry的ProtComp程序对西瓜OSCA蛋白进行亚细胞定位预测。
1. 3 染色体定位、共线性及选择压力分析
在西瓜基因组数据库中获取OSCA基因位置信息后使用MapGene2绘制染色体定位图,利用TBtools对西瓜OSCA基因家族进行共线性分析,并计算重复基因对之间非同义突变率与同义突变率的比值(Ka/Ks)(Wang et al.,2012),以此衡量环境选择压力,根据同义突变率估算重复基因对的离散时间(Yuan et al.,2015),每年每个同义位点的替换量T=Ks/2X(X=6.56×10-9)。
1. 4 基因结构及蛋白保守基序分析
将已鉴定出的西瓜OSCA基因的核苷酸序列与编码序列同时输入到GSDS 2.0分析网站(http://gsds.gao-lab.org/)(赵阳等,2015;Hu et al.,2015),利用MEGA 7.0内置的ClustalW(Larkin et al.,2007)程序将编码区的蛋白质序列进行多重比对后(Kumar et al.,2016),采用邻接法(Neighbor-joining,NJ)构建西瓜OSCA基因家族同源进化树,重复运算1000次。整理图形可获取基因家族成员的内含子与外显子的分布情况,以及OSCA基因结构进化关系。通过MEME(https://meme-suite.org/meme/)对西瓜OSCA基因家族成员蛋白基序类型和排列顺序进行分析,Motif长度设置为6~50,查找Motif数量为20。
1. 5 蛋白跨膜结构、二级和三级结构分析
通过TMPred和TMHMM分析西瓜OSCA蛋白的跨膜区(雷召雄等,2019),使用SOPMA分析西瓜OSCA蛋白二级结构(赵钟毓等,2020;赵训超等,2021);通过SWISS-MODEL(https://www.swissmo-del.expasy.org/interactive)对西瓜OSCA蛋白三级结构的模型进行预测分析。
1. 6 OSCA基因系统进化和顺式元件利用
在Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html#)数据库下载已报道的拟南芥、水稻和番茄的OSCA蛋白序列,并与西瓜OSCA蛋白序列进行多序列比对,构建系统发育进化树(Bootstrap值设定为1000),使用ITOL在线网站(https://itol.embl.de/)编辑美化进化树。提取西瓜OSCA基因转录起始位点上游2000 bp长度序列,利用PlantCare分析启动子区域顺式作用元件(Lescot et al.,2002)。
1. 7 基于转录组测序数据的胁迫响应分析
从葫芦科基因组数据库下载转录组数据(PRJNA326331、PRJNA328189和PRJNA209092),以转录组分析通用的RPKM值作为基因表达量,分析西瓜OSCA基因家族各成员在干旱、低温和盐胁迫条件下的表达情况,筛选重要抗逆OSCA基因。
1. 8 重要抗逆基因的qRT-PCR检测
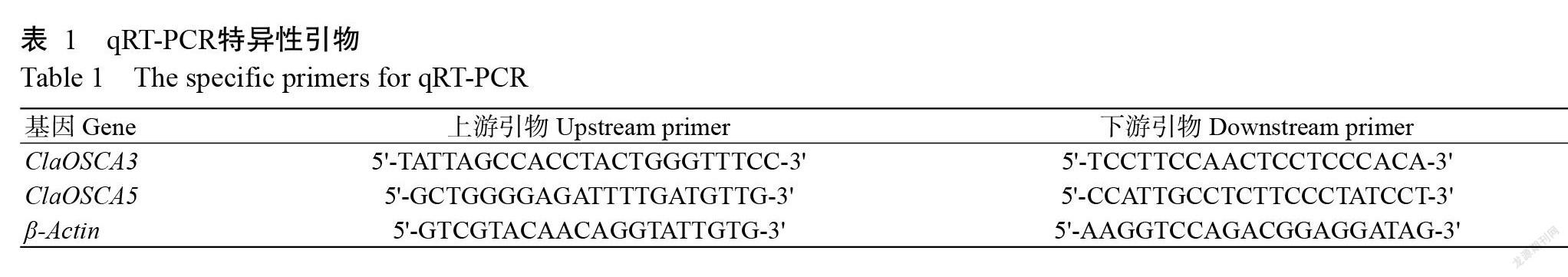
使用Promega试剂盒提取样品RNA,cDNA的合成采用TaKaRa试剂盒。利用Primer3 Plus设计特异性引物,选择β-Actin基因为内参(表1)。qRT-PCR反应体系20.0 μL:SYBR Green Master Mix 10 μL,cDNA 0.5 μL,上、下游引物各0.5 μL,ddH2O 8.5 μL。反应程序:95 ℃ 3 min,95 ℃ 3 s,60 ℃ 30 s,进行40个循环;72 ℃ 5 min;熔解曲线65 ℃ to 95 ℃,温度增量0.5 ℃ 0.05 s,终点读板。
1. 9 统计分析
采用2-ΔΔCt法计算基因的相对表达量,运用Excel 2013和SPSS 17.0对数据进行整理分析。
2 结果与分析
2. 1 西瓜OSCA基因家族鉴定
运用生物信息学的方法鉴定出10个OSCA基因成员,根据染色体定位信息分别命名为ClaOSCA1~ ClaOSCA10。西瓜OSCA基因家族10个成员不均等地分布在6条染色体上,1号染色体(Chrom1)上4个,5號染色体(Chrom5)上2个。ClaOSCA9编码氨基酸数量为214 aa,其余成员氨基酸数量范围在714~809 aa。西瓜OSCA蛋白理化性质的分析结果(表2)表明,蛋白分子量变化范围为24.59~91.97 kD,ClaOSCA10的等电点(PI)最小,10个OSCA蛋白均为疏水的碱性蛋白,即亲水系数(GRAVY)均大于0、PI均大于7.00。亚细胞定位结果表明,西瓜OSCA蛋白多数定位于质膜。
2. 2 西瓜OSCA基因染色体分布及加倍事件分析
10个OSCA基因分布在1、2、5、8、9和10号染色体上(图1),其余染色体上无OSCA基因。ClaOSCA5和ClaOSCA9在1号染色体上遗传距离相对较近,ClaOSCA6和ClaOSCA8在5号染色体上遗传距离相对较近。此外,西瓜OSCA基因在不同染色体组中分布不均等,表明OSCA基因在进化过程中可能发生了基因的复制或丢失。通过共线性分析发现,位于5号染色体的ClaOSCA6(Cla97C05G102470.1)与位于8号染色体(Chrom8)的ClaOSCA7(Cla97C08 G160460.1)存在片段重复事件(图2),片段重复基因对的Ks值为2.71719,分离时间在207Mya(Million years ago),Ka/Ks值为0.24741,小于1.00000,则说明西瓜OSCA基因在进化过程中受纯化选择作用。
2. 3 西瓜OSCA基因家族基因结构与蛋白保守基序分析
西瓜OSCA基因编码氨基酸的多重序列比对结果(图3)表明,在编码序列中含有P、C、F等多个绝对保守位点,推测其在渗透胁迫下受到钙离子渗透适应力作用。此外,对西瓜OSCA基因家族基因结构的分析结果(图4)表明,10个ClaOSCA基因主要可分为三大类群,ClaOSCA1、ClaOSCA5、ClaOSCA2、ClaOSCA3、ClaOSCA4和ClaOSCA9这6个成员为同一类群,ClaOSCA7、ClaOSCA8和ClaOSCA6归属同一类群,ClaOSCA10单独为一类群。此外,ClaOSCA7、ClaOSCA8和ClaOSCA6属于同一类群,内含子数目接近,外显子与内含子的组成模式相似,表明基因结构的保守性与基因进化关系是基本一致的。ClaOSCA10所在类群基因的长度较短且无内含子,属于内含子缺失,结合进化树来看ClaOSCA10基因在进化过程中可能存在一些非必要功能的丢失。
利用MEME在西瓜OSCA基因中预测到20个基序(图5),基因同源性越高,基序排列相似性越强。10个OSCA基因均含有Motif13;除ClaOSCA10外其余9个基因均含Motif2;除ClaOSCA9和ClaOSCA10外其余8个ClaOSCA基因均含Motif1;ClaOSCA1、ClaOSCA2、ClaOSCA3、ClaOSCA4、ClaOSCA5和ClaOSCA9均含Motif3;ClaOSCA1、ClaOSCA5、Cla-OSCA2和ClaOSCA4具有相似基序且排列顺序相同;ClaOSCA8、ClaOSCA6和ClaOSCA7属于同源性较高的同一类群,具有相同基序且排列顺序相同。
2. 4 西瓜OSCA基因家族蛋白跨膜结构、二级和三级结构分析
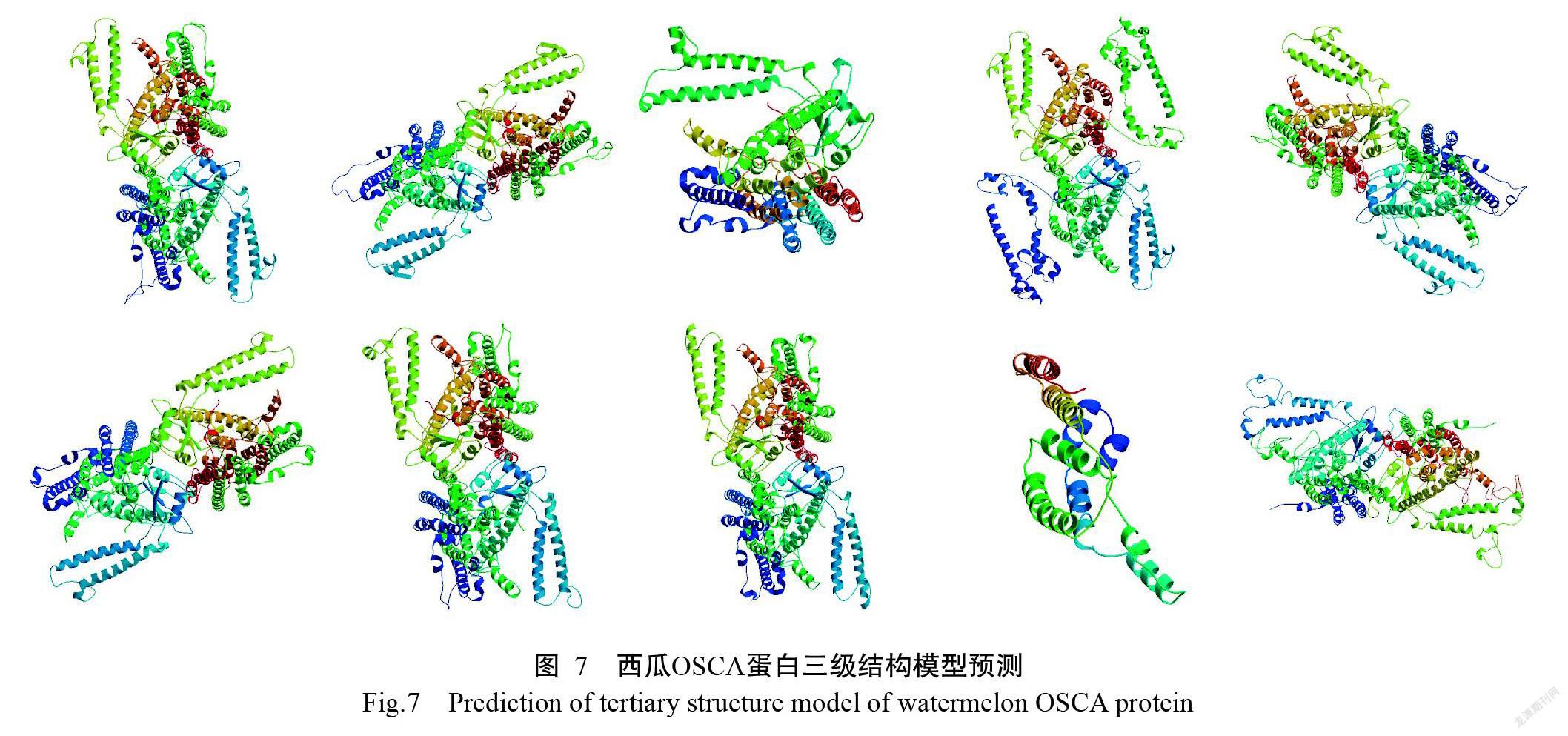
通过TMHMM与TMPred的分析结果发现,除ClaOSCA9蛋白相对分子质量较小外,其余ClaOSCA蛋白均具有7~10个潜在的跨膜结构域(图6),且西瓜OSCA蛋白均具有10种潜在的跨膜拓扑结构,表明西瓜OSCA蛋白均为膜蛋白,进化高度保守。西瓜OSCA基因家族蛋白二级结构分析结果(表3)表明,西瓜OSCA家族基因编码10个蛋白的二级结构中主要是无规则卷曲结构和α螺旋结构,此外在西瓜OSCA蛋白二级结构中还有一小部分是β-转角结构和延伸链结构;α螺旋结构数量比例为36.45%~54.62%,延伸链结构数量比例为12.00%~21.96%,β-轉角结构数量比例为1.51.%~7.01%,无规则卷曲结构数量比例为29.69%~36.58%。ClaOSCA6、ClaOSCA7和ClaOSCA8亲缘关系较近,蛋白三级结构模型高度相似,ClaOSCA5和ClaOSCA1的蛋白三级结构模型高度相似(图7)。
2. 5 西瓜OSCA基因家族系统进化分析和启动子顺式作用元件分析
西瓜、拟南芥、水稻和番茄的OSCA基因家族的系统进化分析结果(图8)表明,OSCA基因进化树主要分为6个亚族,分别命名I~Ⅵ。上述4种植物OSCA基因在6个亚族中均有分布,亲缘性较近。西瓜OSCA基因家族中存在5个明显的垂直同源基因对,分别是I亚族的ClaOSCA3与SlOSCA9,II亚族的ClaOSCA10与SIOSCA4,Ⅲ亚族的ClaOSCA6与SIOSCA1,Ⅳ亚族的ClaOSCA8与SlOSCA5,Ⅴ亚族的ClaOSCA2与AtOSCA8,以及Ⅵ亚族的ClaOSCA4与AtOSCA7、ClaOSCA1与SlOSCA8。西瓜OSCA基因家族中平行同源基因对为Ⅵ亚族的ClaOSCA5和ClaOSCA9。Ⅳ亚族和Ⅵ亚族中OSCA基因数量较多,表明亚家族Ⅳ和Ⅵ中OSCA基因可能发生了功能分化。此外系统进化树中亚家族I、II、Ⅲ和Ⅴ中OSCA基因数量较少,但这部分OSCA基因在进化中得以保留,说明在某些生物学过程中发挥关键作用。
顺式作用元件对于旁侧基因的功能调控具有重要作用,本研究提取西瓜OSCA基因转录起始位点上游2000 bp的序列进行启动子区域分析,发现存在多个与胁迫应答相关的顺式作用元件(表4)。西瓜OSCA基因启动子区域顺式作用元件主要包括响应植物激素与非生物逆境胁迫的顺式元件,其中,响应非生物胁迫的顺式作用元件主要包括厌氧诱导顺式元件(ARE)、冷胁迫应答顺式元件(LTR)、光反应顺式元件(G-box)、干旱胁迫应答顺式元件(MBS)及防卫和逆境应答顺式元件(TC-rich repeats)。西瓜OSCA基因家族中有9个OSCA基因含厌氧诱导顺式元件ClaOSCA1、ClaOSCA4、ClaOSCA5、ClaOSCA6和ClaOSCA8含有涉及低温诱导性的冷胁迫应答顺式元件。ClaOSCA4、ClaOSCA5和ClaOSCA7含有顺式元件,说明这3个成员与应激反应有关。
2. 6 西瓜OSCA基因家族基于转录组测序数据的胁迫响应分析
利用下载的转录组测序数据对西瓜OSCA基因家族的表达模式进行分析,结果(图9)显示,在干旱、低温和盐胁迫下,西瓜OSCA基因家族各成员表达量均有不同程度变化,表现出不同的表达模式。在20% PEG6000胁迫下处理6 h后,ClaOSCA6和ClaOSCA8表达量上调,此外ClaOSCA1、ClaOSCA3和ClaOSCA5表达量显著下调(P<0.05,下同),而ClaOSCA4和ClaOSCA6表达量变化较小(图9-A),说明ClaOSCA4和ClaOSCA6表达一般不受干旱胁迫的影响。经过4 ℃低温胁迫条件诱导6 h后ClaOSCA7表达量上调(图9-B),其他9个基因均下调表达,其中,ClaOSCA2、ClaOSCA3和ClaOSCA5表达量出现显著下调。而在3 mmol/L KCl胁迫下处理6 h后ClaOSCA1、ClaOSCA3和ClaOSCA5表达量显著上调,ClaOSCA4表达量基本不变,ClaOSCA7、ClaOSCA8和ClaOSCA10表达量下调(图9-C)。综合上述胁迫响应情况表明,西瓜OSCA基因家族内部在进化过程中可能发生了功能分化。此外,对西瓜OSCA基因表达情况进行分析与筛选,发现亲缘关系较近的ClaOSCA3和ClaOSCA5在3种不同胁迫条件下表达量变化明显,胁迫响应结果与2.5中2个基因家族成员的启动子顺式作用元件分析相对应,由此推测ClaOSCA3和ClaOSCA5可能为重要抗逆OSCA基因。
2. 7 重要抗逆基因的表达分析
运用qRT-PCR技术对重要抗逆基因ClaOSCA3和ClaOSCA5进行相对表达量检测,结果(表5)表明:经过20% PEG6000胁迫条件诱导6 h后,ClaOSCA3和ClaOSCA5表达量显著下调;在4 ℃下处理6 h后ClaOSCA3和ClaOSCA5表达量显著下调;在经过3 mmol/L KCl胁迫条件诱导6 h后ClaOSCA3和ClaOSCA5均响应强烈。上述结果与转录组测序数据的分析结果基本相符合,进一步验证ClaOSCA3和ClaOSCA5在胁迫响应过程中发挥重要作用。
3 讨论
钙离子在诱导植物抗逆性应对非生物逆境胁迫条件过程中发挥着重要作用,而钙离子信号传导与能感知细胞渗透势变化的钙离子通道蛋白OSCA密切相关,因此对西瓜OSCA基因家族的研究为西瓜遗传改良提供了依据。本研究基于西瓜基因数据库对OSCA基因家族进行了全面分析,运用生物信息学的方法鉴定出10个OSCA基因。基因复制事件对基因家族拓展尤其重要,对西瓜OSCA基因家族进行共线性分析的结果表明在基因家族中存在片段重复,推测片段复制事件发生在单双子叶分化时期(200~205 Mya)之后。基于系统发育分析,西瓜OSCA基因家族被分为3个类群。从基因结构分析来看同一个类群的外显子一内含子组成模式相似,与大豆OSCA基因家族的研究报道(李建伟等,2017)一致,也表明保守的基因复制现象出现在同一类群的OSCA基因家族拓展中。根据蛋白结构分析发现,同一类群成员的蛋白二级结构数量比例和蛋白三级结构模型比较相似。此外,西瓜OSCA基因家族成员均含有完整的DUF221特征结构域,与前人对拟南芥(Yuan et al.,2014)和番茄(王傲雪等,2019)等植物OSCA基因家族保守结构域的研究报道相一致。OSCA蛋白此前在被认为是跨膜通道蛋白和早期脱水应激蛋白(Yuan et al.,2014),本研究中西瓜OSCA蛋白的亚细胞定位分析结果与蛋白跨膜结构域分析结果相符合,表明西瓜OSCA蛋白均为膜蛋白。综上所述,西瓜OSCA基因在进化过程中具有一定保守性。
西瓜、水稻、番茄和拟南芥OSCA基因家族的系统进化树显示西瓜与番茄之间存在4个垂直同源基因对,表明西瓜OSCA基因与番茄OSCA基因的亲缘关系相对更近。此外,在西瓜OSCA基因启动子区域中发现了特异且与光反应有关的光反应顺式作用调节元件,但目前没有在其他植物中发现,需要进一步探究。前人研究表明,OSCA是一种高渗胁迫响应通道蛋白,且初步验证了OSCA基因与植物的逆境调节相关(Yuan et al.,2014)。本研究发现ClaOSCA3的启动子区域不含冷胁迫应答顺式作用元件,然而ClaOSCA3表达量在低温胁迫下变化明显,由此可推测单一胁迫条件诱导下,其他顺式元件也能起到关键调控作用,或者启动子区域存在未被鉴定出的相关顺式元件。ClaOSCA6和ClaOSCA7存在片段复制现象,这2个基因家族成员的启动子区域包含不同的顺式元件,两者的表达模式也有差异,说明基因组复制的过程中也存在功能分化现象。ClaOSCA3和ClaOSCA5在干旱、低温和盐胁迫条件下响应强烈,由此推测ClaOSCA3和ClaOSCA5应答3种胁迫的反应通路中存在交互性。已有研究表明,逆境下植物体内钙信号会迅速增加,高渗胁迫感受蛋白ClaOSCA的应激反应特性需进一步研究,而目前钙信号调控下的西瓜OSCA胁迫响应分子机制尚未被解析完整,仍需在今后的研究中进行验证。
4 结论
西瓜OSCA基因在进化过程中具有一定保守性,与拟南芥、番茄和水稻OSCA基因存在较近亲缘关系。西瓜OSCA基因家族内部存在功能分化,ClaOSCA3和ClaOSCA5可能是胁迫响应机制中的重要抗逆基因。
参考文献:
顧小雨. 2017. 梨OSCA基因家族分析及PbrOSCA2.6和PbrOSCA3.2的功能验证[D]. 南京:南京农业大学. [Gu X Y. 2017. OSCA family analysis and functional validation of PBOSCA2.6 and PBOSCA3.2 in Pyrus[D]. Nanjing:Nanjing Agricultural University.]
雷召雄,柏雪,林亚秋,李健,字向东,熊显荣,熊燕. 2019. 牦牛Lkb1基因编码区克隆及其在骨骼肌的表达分析[J]. 农业生物技术学报,27(1):71-79. [Lei Z X,Bai X,Lin Y Q,Li J,Zi X D,Xiong X R,Xiong Y. 2019. Cloning region cloning of Yak(Bos grunniens) Lkb1 gene and its expression analysis in skeletal muscle[J]. Journal of Agricultural Biotechnology,27(1):71-79.] doi:10.3969/j.issn. 1674-7968.2019.01.008.
李建伟,杨珺凯,贾博为,孙明哲,刘瑀,殷奎德,孙晓丽. 2017. 大豆基因组中OSCA基因家族的进化和表达分析[J]. 中国油料作物学报,39(5):589-599. doi:10.7505/j.issn.1007-9084.2017.05.002. [Li J W,Yang J K,Jia B W,Sun M Z,Liu Y,Yin K D,Sun X L. 2017. Evolution and expression analysis of OSCA gene family in soybean[J]. Chinese Journal of Oil Crops Sciences, 39(5):589-599.] doi:10.7505/j.issn.1007-9084.2017.05.002.
王傲雪,张可为,张瑶,陈秀玲,刘佳音. 2019. 番茄OSCA基因家族鉴定及不同胁迫条件下表达分析[J]. 东北农业大学学报,(1):19-28. [Wang A X,Zhang K W,Zhang Y,Chen X L,Liu J Y. 2019. Identification of tomato OSCA gene family and expression analysis under different stress conditions[J]. Journal of Northeast Agricultural University,(1):19-28.] doi:10.19720/j.cnki.issn.1005-9369.2019. 01.003.
王超楠,朱强龙,崔浩楠,崔博铭,王欣,栾非时. 2018. 西瓜CDPK基因家族鉴定与特征分析[J]. 北方园艺,(17):1-6. [Wang C N,Zhu Q L,Cui H N,Cui B M,Wang X,Luan F S. 2018. Identification and characteristic analysis of CDPK gene family[J]. Northern Horticulture,(17):1-6.] doi:10.11937/bfyy.20180692.
赵训超,魏玉磊,丁冬,刘梦,盖胜男,张今杰,邵文静,李嘉欣,徐晶宇. 2021. 甜荞麦脂肪酸脱氢酶基因(FeFAD)家族的鉴定与分析[J]. 东北农业科学,46(1):36-41. [Zhao X C,Wei Y L,Ding D,Liu M,Gai S N,Zhang J J,Shao W J,Li J X,Xu J Y. 2021. Genome-wide identification and bioinformatics analysis of fatty acid desaturase gene (FeFAD) family in common buckwheat[J]. Journal of Northeast Agricultural Sciences, 46(1):36-41.] doi:10. 16423/j.cnki.1003-8701.2021.01.010.
趙阳,王玉,蔡慧林,程备久,马庆. 2015. 玉米脱水素基因家族的鉴定与分析[J]. 安徽农业大学学报,42(5):657-665. [Zhao Y,Wang Y,Cai H L,Cheng B J,Ma Q. 2015. Identification and characterization of the dehydrin gene family in maize[J]. Journal of Anhui Agricultural University,42(5):657-665.] doi:10.13610/j.cnki.1672-352x.20150825.028.
赵钟毓,侯丹,胡秋涛,魏涵天,郑颖,林新春. 2020. 毛竹PeNAC047基因的克隆与表达分析[J]. 农业生物技术学报,28(1):58-71. [Zhao Z Y,Hou D,Hu Q T,Wei H T,Zheng Y,Lin X C. 2020. Cloning and expression analysis of PeNAC047 gene in Phyllostachys puercens[J]. Journal of Agricultural Biotechnology,28(1):58-71.] doi:10. 3969/j.issn.1674-7968.2020.01.006.
朱强龙,赵玉龙,吕慧玲,栾非时,高鹏. 2017. 西瓜CBL家族基因的鉴定与特征分析[J]. 北方园艺,(15):18-24. [Zhu Q L,Zhao Y L,Lü H L,Luan F S,Gao P. 2017. Identification and characterization of CBL family genes in watermelon[J]. Northern Horticulture,(15):18-24.] doi:10. 11937/bfyy.20170580.
Bartels D,Sunkar R. 2005. Drought and salt tolerance in plants[J]. Critical Reviws in Plant Sciences,24(1):23-58. doi:10.1080/07352680590910410.
Batistic O,Kudla J. 2012. Analysis of calcium signaling pathways in plants[J]. Biochimica et Biophysica Acta(BBA)- General Subjects,1820(8):1283-1293. doi:10.1016/j.bbagen.2011.10.012.
Chen C J,Chen H,Zhang Y,Thomas H R,Frank M H,He Y H,Xia R. 2020. TBtools:An integrative toolkit deve-loped for interactive analyses of big biological data[J]. Mollecular Plant,13(8):1194-1202. doi:10.1016/j.molp. 2020.06.009.
Hou C C,Tian W,Kleist T,He K,Garcia V,Bai F L, Hao Y L,Luan S,Li L G. 2014. DUF221 proteins are a family of omosensitive calcium-permeable cation channels conserved across eukaryotes[J]. Cell Research,24(5):632-635. doi:10.1038/cr.2014.14.
Hu B,Jin J P,Guo A Y,Zhang H,Luo J C,Gao G. 2015. GSDS 2.0:An upgraded gene feature visualization server[J]. Bioinformatics,31(8):1296. doi:10.1093/bioinformatics/btu817.
Kumar S,Stecher G,Tamura K. 2016. MEGA7:Molecularevolu-tionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution,33(7):1870-1874. doi:10.1093/molbev/msw054.
Larkin M A,Blackshields G,Brown N P,Chenna R,McGettigan P A, McWilliam H,Valentin F,Wallence I M, Wilm A,Lopez R,Thompson J D,Gibson T J,Higgins D G. 2007. Clustal W and Clustal X version 2.0[J]. Bioinformatics,23(21):2947-2948. doi:10.1093/bioinformatics/btm404.
Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouzé P, Rombauts S. 2002. PlantCare, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,30(1):325-327. doi:10. 1093/nar/30.1.325.
Li Y S,Yuan F,Wen Z H,Li Y H,Wang F,Zhu T,Zhuo W Q,Jin X,Wang Y D,Zhao H P,Pei Z M,Han S C. 2015. Genome-wide survey and expression analysis of the OSCA gene family in rice[J]. BMC Plant Biology, 15:261-273. doi:10.1186/s12870-015-0653-8.
Wang Y P,Tang H B,DeBarry J D,Tan X,Li J P,Wang X Y,Lee T H,Jin H Z,Marler B,Guo H,Kissinger J C,Paterson A H. 2012. MCScanX:A toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Rescarch,40(7):e49. doi:10.1093/nar/gkr1293.
Yuan F,Yang H M,Xue Y,Kong D D,Ye R,Li C J,Zhang J Y ,Theprungsirikul L ,Shrift T,Krichilsky B. 2014. OSCA1 mediates osmotic-stress-evoked Ca2+ increases vital for osmosensing in Arabidopsis[J]. Nature,514(7522):367-367. doi:10.1038/nature13593.
Yuan S X,Xu B,Zhang J,Xie Z N,Cheng Q,Yang Z M,Cai Q S,Huang B R. 2015. Comprehensive analysis of CCCH-type zinc finger family genes facilitates functional gene discovery and reflects recent allopolyploidization event in tetraploid switchgrass[J]. BMC Genomics,16:129-145. doi:10.1186/s12864-015-1328-4.
Zhu J K. 2002. Salt and drought stress siginal transduction in plants[J]. Annual Review of Plant Biology, 53:247-273. doi:10.1146/annurev.arplant.53.091401.143329.
(責任编辑 邓慧灵)

