毛干检材STR基因分型成功率的影响因素研究
郑阳阳,郑文彦,吴伟斌,杜蔚安
(广东华美众源生物科技有限公司,广东 佛山 528000)
自然脱落毛发是法医常见物证检材之一,在案件侦破中具有重要线索与证据价值[1]。带毛囊毛发的短串联重复序列(STR)分型技术成熟、可靠,已广泛用于个体识别、亲缘关系鉴定等[2-3]。而在案发现场采集到的毛发样本70%是处于休眠期的脱落毛发,毛囊退化或只剩下毛干,DNA分型难度较大,目前,常用办法是测序分析线粒体DNA高度变异D-环区[4]。虽然线粒体DNA含量比核DNA丰富,检出率更高[5-6],但存在母系遗传、异质性、分析数据无法进入数据库检索比对等缺点[7-8],导致其应用范围和案件线索作用有限。本研究从毛干DNA含量、DNA降解程度、黑色素抑制3个方面入手,设计实验分析和验证了无毛囊毛干检材在STR分型过程存在的难点,并初步研究了解决策略,以提高无毛囊毛干的分型成功率。
1 材料与方法
1.1材料
1.1.1样本采集 2019年10月收集15名健康志愿者毛干样本,均为健康志愿者自然脱落毛发(未经染烫等化学处理);用木梳轻轻梳理头皮,收集存留在梳子上的毛发,肉眼观察毛根部位体积较小、呈干瘪坚硬状,显微镜下观察毛球萎缩、无根鞘附着。单根毛干实验时尽量选择长度为20 cm的毛发,若单根长度小于20 cm可选择多根混合使用,计算叠加长度。采集健康志愿者口腔唾液样本作为对照样本(毛干样本与唾液对照样本分型结果一致的基因座计为成功分型基因座)。标准对照采用人类基因组DNA 9947A。
1.1.2主要试剂与仪器 Qubit-iT dsDNA HS Assay试剂盒(ABI,美国)、AGCU Expressmarker 16CS STR分型试剂盒(无锡中德美联,中国)、黑色素标准品(Sigma-aldrich,美国)、Qubit荧光定量系统(ABI,美国)、Mastercycler®nexus聚合酶链反应(PCR)仪(Eppendorf,德国)、3130XL型遗传分析仪(ABI,美国)、GeneMapper ID-X数据分析软件(ABI,美国)等。引物合成由上海生工生物工程公司完成。
1.2方法
1.2.1样本前处理与DNA提取 从靠近毛根端剪去2 cm后将毛干样本剪至长度0.5 cm若干段,用10%次氯酸钠溶液清洗[9-10],再加入1%十二烷基硫酸钠溶液800 μL浸泡5 min,期间不断涡旋混匀,用1 mL无水乙醇与无菌水各清洗1次,室温晾干。加入600 μL裂解液(0.01 M三羟甲基氨基甲烷-盐酸(pH 8.0)、2%十二烷基硫酸钠、0.005 M乙二胺四乙酸、100 mM氯化钠、300 mM二硫苏糖醇、1 mg/mL蛋白酶K等56 ℃消化2 h;采用chelex-100螯合法或磁珠法进行提取纯化。
1.2.2DNA定量 使用Qubit荧光定量系统对毛干提取液进行DNA定量分析,基于高灵敏荧光染料与核酸分子结合后被激发产生的荧光信号强度与结合的核酸分子数呈正比的原理,利用已知质量浓度的DNA标准品绘制标准曲线,具体操作参照Qubit-iT dsDNA HS Assay kit说明书进行。根据DNA定量结果与初始检材量计算每毫克毛发DNA含量及单根毛干DNA含量。
1.2.3DNA降解分析 根据定量结果筛选质量浓度较高的DNA提取产物,利用3组引物进行同时扩增,对比不同大小片段的扩增效果以验证评估DNA降解情况。扩增引物根据人类基因组3个不同遗传多态性位点自主设计、产物片段大小分别为80~88、142~145、212~266 bp 3个区间。引物序列见表1。PCR扩增产物通过3130XL遗传分析仪进行分离分析,将12μL去离子甲酰胺与0.5 μL分子质量内标Marker SIZ-500混合。加入1 μL PCR反应产物,涡旋混匀,3 000 r/min离心2 min,95 ℃变性3 min,冰浴3 min,进行毛细管电泳。利用GeneMapper®ID-X软件分析扩增产物电泳峰图,比较不同大小区域的片段出峰情况以评估DNA降解程度。以无降解的人类基因组标准DNA9947A作为对照。
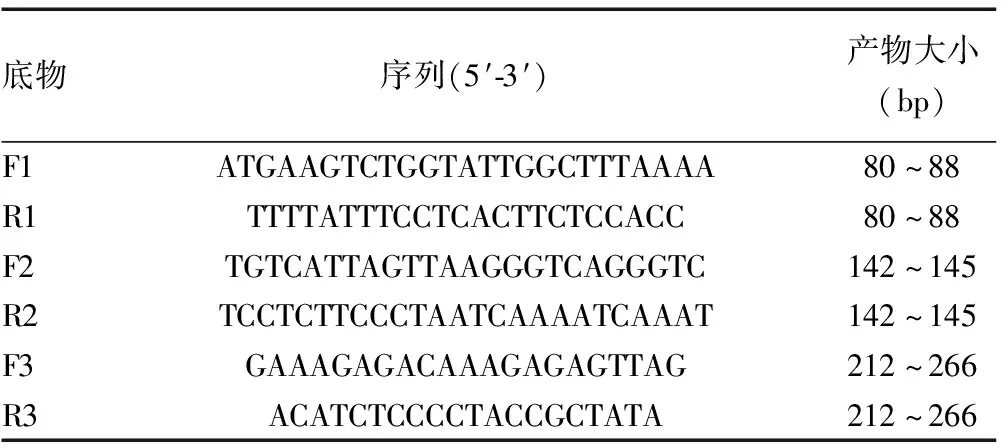
表1 不同长度扩增产物引物序列
1.2.4毛发黑色素对STR的影响测定 设计单因素实验测定毛发黑色素对STR分型的影响及产生影响的临界质量浓度。将人类基因组DNA 9947A溶液加入一系列不等量的毛发黑色素标准品,配制黑色素终质量浓度为28、32、36、40、44、48、52、56、60、64 ng/μL的PCR反应体系,DNA模板量维持为0.5 ng/μL,按AGCU Expressmarker 16CS STR分型试剂盒说明书进行PCR扩增,统计各基因座出峰情况。产物毛细管电泳与数据分析方法与1.2.3项相同。
1.2.5STR分型条件优化 采用Chelex-100螯合法提取毛干DNA,利用Microcon100超滤柱进行浓缩,所得产物利用人类常染色体mini-STR分型试剂盒进行基因分型。此外,通过尝试增加PCR体系中镁离子(Mg2+)、DNA聚合酶浓度或将DNA聚合酶与牛血清清蛋白(BSA)预孵育处理等途径缓解黑色素抑制作用,进而优化单根毛干样本STR分型条件。
2 结 果
2.1单根毛干DNA含量 初始毛干检材用量为30~50 mg,1 mg头发相当于单根毛干(长度20 cm)重量[11],据此将DNA提取产物的定量数值换算为单根毛干平均DNA含量值。随机抽取的8名健康志愿者中除1名DNA含量高达0.78 ng外,其余DNA含量为0.01~0.35 ng。8名健康志愿者单根毛干DNA含量具有较大差异。见图1。
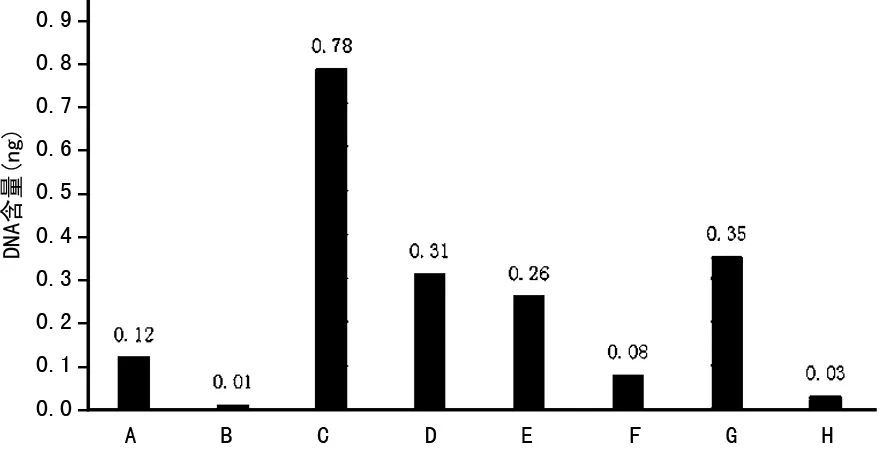
图1 8名健康志愿者单根毛干DNA含量比较
2.2毛干DNA降解评估 除个别样本(图2a)可得到80、142、212 bp 3个产物峰外,其余5名健康志愿者毛干样本均只能在小片段区域得到1个或2个产物峰,而大片段区域的扩增全部失败(图2b),而标准对照9947A则能成功扩增到3个目标区域的相应产物峰(图2c)。说明毛干DNA存在降解。
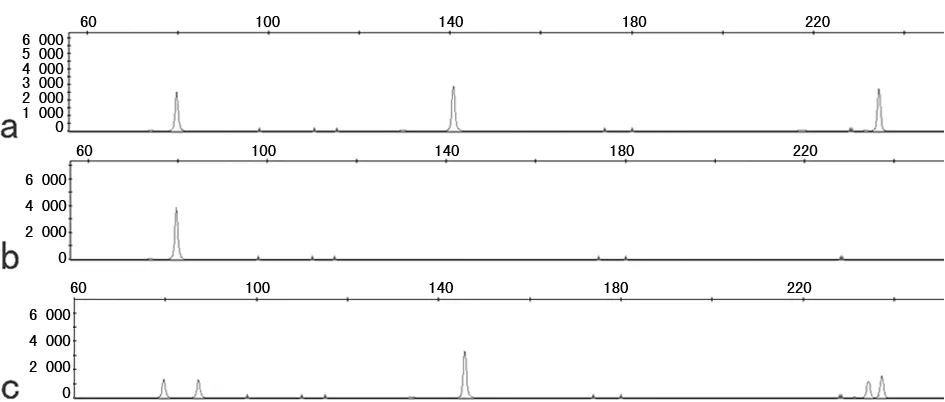
a.健康志愿者C毛干样本;b.健康志愿者A、D、E、F、G毛干样本;c.标准对照9947A。
2.3毛发黑色素对STR分型的影响 当PCR反应体系中黑色素质量浓度大于或等于36 ng/μL时个别基因座出现等位基因丢峰现象;当PCR体系中黑色素质量浓度大于或等于60 ng/μL时16个基因座所有等位基因全部扩增失败,同时试剂盒自带的内参基因座IC也无法正常出峰,说明此时黑色素对PCR扩增反应产生了强烈的抑制作用。见图3。

注:红色表示扩增后丢峰的基因座;绿色表示分型正确的基因座。
2.4单根毛干STR分型优化结果 实验优化前毛干样本STR分析的基因座检出率为41.2%。增加Mg2+质量浓度对分型效果无改善作用,但适当增加DNA聚合酶质量浓度能提高基因座检出率,且当DNA聚合酶质量浓度为0.4 U/μL时基因座检出率提高至58.8%,改善效果最为明显。见表2。在反应前先采用10 μM BSA对DNA聚合酶于56 ℃条件下预处理30 min基因座检出率可进一步提高至70.6%。见图4。随机选取4名健康志愿者单根毛干样本(长20 cm)进行分型测试,优化前基因座检出率分别为35.3%、41.2%、41.2%、47.1%;其中3名健康志愿者单根毛干样本成功分型基因座大于或等于10个,基因座检出率分别为58.8%、70.6%、64.7%;1名健康志愿者单根毛干样本优化后基因座检出率为52.9%。毛干样本STR分型成功率具有个体差异。见图5。
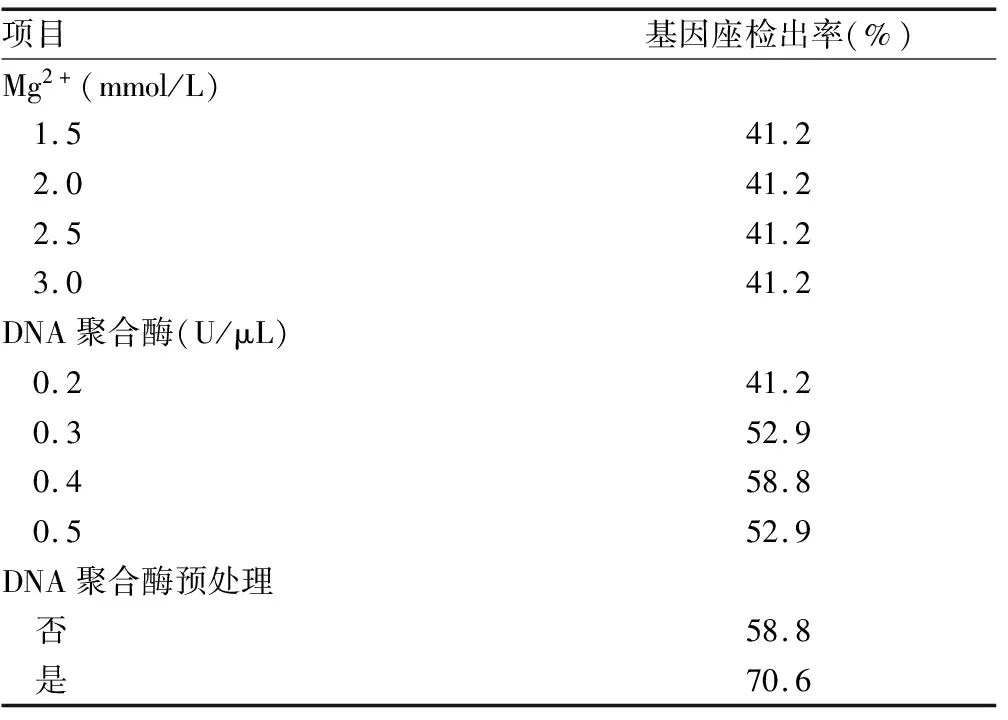
表2 不同质量浓度Mg2+、DNA聚合酶基因座检出率比较

图4 单根毛干优化后STR分型图谱
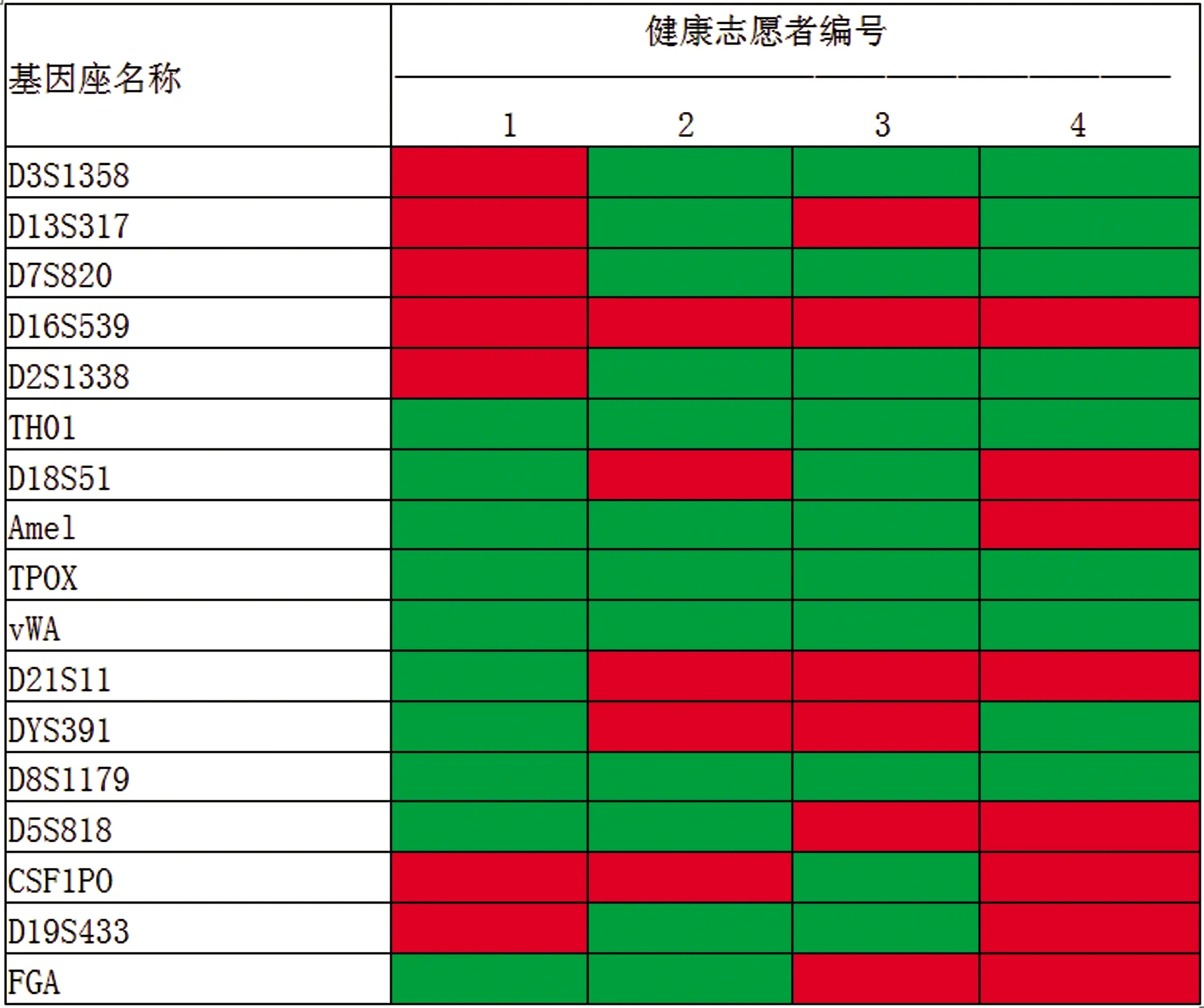
注:红色表示扩增后丢峰的基因座;绿色表示分型正确的基因座。
3 讨 论
由于毛干DNA含量极低,提取产物的定量分析难度较大,因此,本研究将初始检材量提高至30~50 mg,并选用Qubit荧光定量系统、利用荧光染料与双链DNA的特异性结合提高定量的灵敏度与准确性;单根毛干DNA含量计算结果与HEYWOOD等[12]得出的0.40 ng DNA结论较为接近。此外,不同个体毛干DNA含量差异较大,推测这也是导致毛干检材DNA分型失败率高的一个重要因素。
法医实验室常用的商业化试剂盒的检测灵敏度为0.05~0.10 ng;在实际测试过程中0.05 ng标准品模板常能够得到完整分型,而同等甚至更高模板量的毛干DNA却无法得到大片段产物峰,说明毛干STR分型失败不仅是由毛干提取产物模板量太低所致,也与DNA降解或抑制物密切相关。为克服DNA降解问题,本研究利用mini-STR技术原理,通过在更靠近核心重复序列的两端侧翼区设计引物,使PCR产物的片段大小缩短至300 bp以内,以提高小片段基因组DNA的检测成功率。
由于毛干DNA含量极少,如极力追求特殊的、针对去除黑色素的纯化效率高的纯化方案,势必会严重影响DNA得率[13]。因此,为减少DNA损失考虑采用chelex-100提取法,但此法对黑色素去除没有太大效果。相关研究表明,1 mg人毛发中黑色素含量为0.055 mg,据此计算,单根毛发STR分型体系中黑色素质量浓度为48.8 ng/μL[14];基于研究结果,此质量浓度的STR分型成功率会受到严重抑制,基因座检出率小于47.1%。因此,若要提高毛干STR分型成功率必须改善黑色素的抑制作用[15-16]。本研究在经过多轮实验摸索后发现,将反应体系中聚合酶质量浓度提高至0.4 U/μL并利用BSA对DNA聚合酶预处理能有效改善反应效果,使基因座检出率提高至70.6%;然而需要注意的是,同一实验条件下不同个体来源样本的基因座检出率差异仍较大,分析原因可能是由于不同个体毛干DNA含量的差异及角质细胞中DNA降解程度的差异所致。因此,尚需进一步研究,以使毛干检材发挥更大的检测价值。

