桃小食心虫化学感受蛋白CSP16配体结合特性
刘孝贺,仇贵生,佟兆国,张怀江,闫文涛,岳强,孙丽娜
桃小食心虫化学感受蛋白CSP16配体结合特性
刘孝贺1,仇贵生1,佟兆国2,张怀江1,闫文涛1,岳强1,孙丽娜1
1中国农业科学院果树研究所,辽宁兴城 125100;2西昌学院农业科学学院,四川西昌 615013
【】桃小食心虫()是我国北方果树生产中的重要害虫之一,本文通过体外表达桃小食心虫化学感受蛋白CsasCSP16,明确其与寄主挥发物分子和性信息素分子的结合特性,并通过生物信息学预测CsasCSP16与挥发物分子结合的关键氨基酸位点,为揭示桃小食心虫嗅觉的分子机理提供理论依据。通过原核表达系统获得CsasCSP16蛋白,并使用Ni-NTA柱纯化重组蛋白。采用荧光竞争结合的方法,以N-苯基-1-萘胺(N-pheny-1-naphthylamine,1-NPN)为荧光探针,从33种寄主挥发物和2种性信息素分子中筛选出与CsasCSP16结合亲和性高的配体分子。通过对CsasCSP16进行同源建模获得其三维结构模型,以CSPMbraA6(PDB ID:1N8U)为模板成功构建CsasCSP16的三维结构模型。使用Autodock Vina软件将CsasCSP16与亲和性高的配体分子进行对接,构建蛋白-配体复合物。然后使用GROMACS(2019.3)软件对复合物进行动力学模拟,从动力学模拟的平衡状态中选取100个构象,使用g_mmpbsa软件计算CsasCSP16与气味分子的结合能,并通过能量分解的方法预测关键氨基酸残基。成功构建了CsasCSP16的克隆表达载体,通过大肠杆菌原核表达系统获得高纯度的重组蛋白。与35种配体分子的荧光竞争结合表明,CsasCSP16与水杨酸甲酯、6-甲基-5-庚烯-2-酮、十五烷、庚酸丁酯和-蒎烯有较强的结合活性,其Ki值分别为6.59、6.25、3.50、6.73和4.47 µmol·L-1。分子对接的结果表明,CsasCSP16与水杨酸甲酯、6-甲基-5-庚烯-2-酮、十五烷、庚酸丁酯和-蒎烯的Vina Score值分别为-6.1、-5.3、-5.8、-5.2和-6.6。动力学模拟结果表明,CsasCSP16与气味分子复合物在50 ns内达到平衡状态,通过g_mmpbsa计算复合物的结合能,CsasCSP16-水杨酸甲酯、CsasCSP16-6-甲基-5-庚烯-2-酮、CsasCSP16-十五烷和CsasCSP16-庚酸丁酯的结合自由能分别为-50.264、-65.551、-136.035和-93.805 kJ·mol-1;最后,通过分解每个氨基酸贡献的结合自由能表明异亮氨酸49(Ile49)、缬氨酸71(Val71)、异亮氨酸72(Ile72)和酪氨酸90(Tyr90)贡献的结合能>3 kJ·mol-1,因此推测这4种氨基酸在CsasCSP16结合气味分子的过程中发挥关键作用。桃小食心虫CsasCSP16能与寄主植物的多种气味分子结合,推测其可能在桃小食心虫对寄主植物的定位过程中发挥重要作用。异亮氨酸49(Ile49)、缬氨酸71(Val71)、异亮氨酸72(Ile72)和酪氨酸90(Tyr90)可能是CsasCSP16与配体分子结合的关键氨基酸位点。
桃小食心虫;化学感受蛋白;原核表达;荧光竞争结合;分子对接;动力学模拟
0 引言
【研究意义】桃小食心虫()属鳞翅目(Lepidoptera)蛀果蛾科(Carposidae),雌虫将卵产在苹果、桃、梨、山楂等果实的萼洼处或者果面,幼虫孵化后蛀入果实内部纵横串食,造成“豆沙馅”似的虫果,给果农造成严重的经济损失[1]。目前对桃小食心虫的防控主要依靠化学农药,但是大量化学农药的使用导致天敌被误杀并引起环境问题。随着对昆虫嗅觉机制研究的逐渐深入,引诱剂和趋避剂的开发为害虫防控提供了新的思路和策略。研究桃小食心虫化学感受蛋白(chemosensory protein,CSP)与寄主挥发物的识别和结合分子机制,对明确该虫产卵定位和取食选择以及建立基于昆虫行为学的生态防控具有重要理论意义。【前人研究进展】嗅觉在昆虫生理活动中发挥关键作用,例如定位产卵场所、寻找寄主植物和避免天敌危害等[2-3]。气味受体(odorant receptor,OR)或离子型受体(ionotropic receptor,IR)具有分辨和识别不同气味信号的功能。然而,疏水性的气味分子到达神经元树突激活气味受体或离子型受体之前,通常需要与气味结合蛋白(odorant binding protein,OBP)或化学感受蛋白相结合以复合物的形式穿过亲水性的淋巴液,到达嗅觉神经元附近刺激嗅觉受体,化学信号转化为电信号[4]。尽管OBP或CSP在嗅觉形成过程中的具体功能尚未完全探明,但是,在基因组和转录组中发现大量的OBP和CSP基因可能发挥关键作用[5-8]。CSP通常由100—120个氨基酸残基组成,结构紧凑,主要由6个-螺旋形成一个疏水性的空腔。不同种类昆虫的CSP都含有4个保守的半胱氨酸残基,会形成两个二硫键用以保持CSP三维结构的稳定[9-10]。与OBP相比,CSP除了具有嗅觉功能外,还具有其他生理功能。CSP被鉴定出分布在不同的组织中。CSP首先在黑腹果蝇()中被发现,命名为OS-D和A10,但当时关于CSP的功能还不清楚[11]。目前,有关CSP功能的信息在许多昆虫中得到验证。(1)CSP参与昆虫的再生和发育。在螳螂中,CSP10参与螳螂若虫断足的再生;蜜蜂的CSP5与受精卵和表皮的发育有关[12-13]。(2)使用亚致死浓度的菊酯类农药处理小菜蛾()的研究中发现,有3个CSP(CSP3、CSP4和CSP8)基因表达量上调,推测CSP参与昆虫抗药性的形成[14-15]。(3)蝗虫由散居型变成聚居型时,体内的CSP基因表达量提高,CSP可能与蝗虫的聚集状态改变有关[16]。(4)一些CSP在昆虫喙和眼中作为营养吸收和视觉色素的载体[17]。(5)有研究表明,CSP在性腺或生殖器官中结合化学信息素信号,并协助它们释放到环境中[18-20]。最后,CSP结合气味分子和信息素分子,参与昆虫嗅觉的形成过程,CSP在蚊类的淋巴液中富集并且对化学信息素具有很高的亲和性;在蜜蜂触角中高表达的CSP3可以与信息素结合[21]。CSP如此广泛的表达谱和结合谱说明其在昆虫的生命活动中发挥重要的作用。因此,有关CSP的具体生理功能需要深入研究。【本研究切入点】笔者所在课题组前期从桃小食心虫的触角转录组中鉴定29个OBP基因,并发现2个GOBP和3个PBP基因,研究结果表明酯类、醛类和萜类等气味分子是潜在的吸引物质[22-23]。前期研究发现,CsasCSP16在雌、雄成虫的触角和翅中均有表达[24]。本研究采用原核表达和荧光竞争结合试验,研究CsasCSP16与35种小分子化合物的结合特性。使用分子对接和动力学模拟技术,预测CsasCSP16与小分子结合的关键位点。【拟解决的关键问题】研究桃小食心虫CsasCSP16与相关化合物的结合特性,并通过三维建模和分子动力学模拟方法研究CsasCSP16与配体结合的分子机制,为桃小食心虫的绿色防控技术提供新的思路和理论依据。
1 材料与方法
试验于2018年7月至2020年3月在中国农业科学院果树研究所完成。
1.1 供试昆虫
供试种群于2018年7月采自中国农业科学院果树研究所(40.61°N,120.73°E)试验园。采集被桃小食心虫幼虫蛀入带有“泪滴”危害状的金冠苹果,带回实验室,于(25±1)℃、相对湿度70%±5%、光周期L﹕D=15 h﹕9 h条件下饲养[25]。
1.2 总RNA提取及cDNA第一链合成
分别取桃小食心虫雌、雄成虫全虫以及触角(100对)、头部(去触角,20头)、胸部(10个)、腹部(10个)、足(20头×6对)、翅(20对)等组织,按照TRIzol(TaKaRa)试剂盒说明书提取总RNA,用RNase-free水溶解,在检测其浓度和纯度后,再根据Prime ScriptTMRT Reagent Kit with gDNA Eraser(perfect real-time)试剂盒反转录合成cDNA第一链,-20℃保存备用或直接进行下一步试验。
1.3 重组蛋白的原核表达和纯化
根据CsasCSP16的开放阅读框序列[24],于http:// www.cbs.dtu.dk/services/SignalP/网站预测信号肽,去除信号肽后,设计带有酶切位点HI正向引物和RI的反向引物,分别为CGATGCGCCC CGAAGAGCACT和TGTTATGGCCTTGAC GGTGCG。纯化的PCR产物克隆到pMD-19T 载体,测序成功的pMD-19T/质粒和表达载体pET-28a进行双酶切,按照T4 DNA连接酶试剂盒说明书将目的片段连接到pET-28a表达载体上,转化到BL21(DE3)大肠杆菌细胞中,并将阳性菌落接种到LB液体培养基中,37℃过夜振荡培养。当LB培养基的OD600值达到0.6—0.8时,加入IPTG至终浓度为1 mmol·L-1,37℃培养4 h诱导重组蛋白表达。通过离心(8 000×,4℃,20 min)收集菌体并进行超声处理(100 W,超声16 min),收集上清和沉淀,经SDS-PAGE电泳检测重组蛋白主要在包涵体中,用10 ml的无咪唑的8 mol·L-1尿素溶液重悬分散沉淀,在冰水浴下100 W超声10 min,留20 µL进行电泳检测,4℃,5 000 r/min离心15 min收集上清[26]。
使用Ni-NTA亲和层析柱对表达在沉淀中的重组蛋白进行纯化后,放入透析袋中,经过(6、4、3、2、1、0 mol·L-1)梯度尿素缓冲液进行透析复性,每种梯度透析2 h[27]。透析缓冲液配方:5%甘油,1% L型精氨酸,0.1 mol·L-1EDTA,尿素,使用1×PBS溶解。
1.4 荧光竞争结合试验
N-苯基-1-萘胺(1-NPN)用作CSP结合亲和性研究的探针,选择35种小分子化合物,包括33种苹果挥发物和2种桃小食心虫信息素组分。使用色谱级甲醇将1-NPN和气味物质配制成1 mmol·L-1的工作液,蛋白用50 mmol·L-1的Tris-HCl缓冲液(pH 7.4)稀释成2 µmol·L-1,设定激发波长为337 nm,扫描发射光波长范围350—600 nm。在浓度为2 µmol·L-1的CsasCSP16蛋白溶液中,逐一加入终浓度为0—20 µmol·L-1的1-NPN,直至荧光强度达到饱和,根据Scatchard方程计算CsasCSP16与1-NPN的结合常数。然后测定气味物质与CsasCSP16的结合能力。将气味物质加入CsasCSP16与1-NPN的混合液体系中,气味标样浓度依次从2 µmol·L-1梯度递增。每种气味物质重复测试3次。使用公式Ki= [IC50]/(1+[1-NPN]/K1-NPN)计算结合常数,其中IC50为荧光强度降至一半时加入的配基浓度,[1-NPN]是游离的1-NPN浓度,K1-NPN为重组蛋白CsasCSP16与1-NPN的结合常数[27]。
1.5 同源建模和分子对接
使用Modeller(version-9.19)构建CsasCSP16的三维结构模型,选择CsasCSP16的氨基酸序列作为探针在PDB蛋白数据库中搜索,根据晶体结构的R-因子、序列连续性和结构相似性选择最佳模板。使用Align2D软件比对CsasCSP16与模板序列。Modeller中的自动建模程序生成100个构象,首先用共轭梯度法(conjugate gradient,CG)优化每个构象。其次使用DOPE(discrete optimized protein energy)值来衡量CsasCSP16构象的稳定性,最后使用Profile-3D方法和在线工具Swiss-model(https://swissmodel.expasy. org/assess)生成拉氏图评价三维模型的合理性。
运用openbabel(https://sourceforge.net/projects/ openbabel/)工具将气味分子转换为pdb格式,并使用Autodock tools对气味分子去水加氢。使用Autodock vina 软件构建CsasCSP16与配体分子的蛋白复合物[28]。运用Autodock tool 1.5.7软件包对CsasCSP16蛋白去水加氢,并生成坐标文件保存为PDBQT格式。配体分子结构的键设定为任意旋转,设定网格参数为40点×40点×40点,格点间距为0.0375 nm,采用CB-DOCK软件预测CsasCSP16表明可能的结合口袋,选择前5个结合口袋进行对接[29]。通过局部能量搜索和拉马克遗传算法相结合的方法寻找对接构象。选择默认的Vina-score作为打分值。最终,采用vina-score评价对接结果。使用PyMOL-2.3.0(http:// www.pymol.org/)显示对接结果。
1.6 分子动力学模拟
使用GROMACS(2019.3)进行能量最小化和分子动力学模拟[30]。蛋白的拓扑结构使用CHARMM36力场自动生成[31]。配体的拓扑结构使用CGenFF服务器(https://cgenff.umaryland.edu/)生成。进行动力学模拟每个系统在截断八面体周期边界条件下使用TIP3P水模型进行溶剂化,截止距离为10,其中包含简单电荷模型(sample point charge,SPC)的水分子,并在体系中加入抗衡离子(Na+和Cl-)使体系保持电中性。首先采用最陡下降法对复合物进行50 000步能量优化,消除能量重叠。然后分别进行100 ps的正则系综(NVT)和100 ps的等温等压系综(NPT)模拟,使体系温度保持在300 K,压强保持在1×10-5pa的平衡状态,采用周期性边界条件和PME(Particle Mesh Ewald)方法处理长程静电作用。通过V-rescale和Parrinello Rahman控制系统的温度和压力。使用LINCS算法固定所有原子之间的相对距离。模拟过程中采用的步长为2 fs,每1 ps保存一次轨迹用于后续的数据分析。CsasCSP16复合物动力学模拟的质量由主链原子的均方根位移(root mean square deviation,RMSD)评价。
1.7 CsasCSP16蛋白复合物结合自由能计算
从分子动力学模拟过程中得到的配体与受体蛋白的相对结合自由能通常比分子对接得分具有更高的试验结果相关性[32]。运用MM-PBSA方法计算得到CsasCSP16与配体分子之间的相对结合自由能ΔGbind[33]。
ΔGbind=ΔH-TΔS≈ΔEMM+ΔGsol-TΔS (1)
ΔEMM=ΔEint-ΔEele+ΔEvdW(2)
ΔGsol=ΔGPB+ΔGSA(3)
相对结合自由能计算中熵的变化可以忽略,ΔEint为配体与蛋白间的相互作用,ΔEele为静电作用力,ΔEvdW为范德华力,ΔGsol为溶剂化作用,ΔGPB为极性溶剂化作用,ΔGSA为非极性溶剂化作用。非极性化与极性化作用计算采用APBS软件,溶剂的介电常数设为80,复合物的介电常数设为1。从分子动力学模拟40—50 ns轨迹中每隔100 ps取一个构象,获得相对结合自由能值,配体分子与CsasCSP16蛋白的相对结合自由能表明配体与蛋白结合能力的强弱,也反映配体分子存在于活性口袋中保持稳定性的能力,这种能力往往是决定CsasCSP16与配体分子是否结合的关键因素。
1.8 CsasCSP16蛋白质复合物关键氨基酸能量分解
基于残基分解的方法,对配体分子与氨基酸残基之间的相互作用进行分析,定性描述配体分子与CsasCSP16的结合机理[34]。复合物中每个残基分解能可以进一步分解为主链、侧链和总的分解能。每个配体分子-残基对(ΔGCsasCSP16-residue)的相互作用表示为:
ΔGCsasCSP16-residue=ΔEint-TΔS+ΔEvdW+ΔEele+ΔGPB+ ΔGCAVITY(4)
使用g_mmpbsa计算出配体分子与每个CsasCSP16残基之间的范德华力(ΔEvdW)和静电作用力(ΔEele)。ΔGPB表示由PB模型计算得出的对溶剂化自由能的静电贡献。通过基于相应SASA的经验模型获得了对溶剂化自由能(ΔGCAVITY)的非极性贡献。内部能量(ΔEint)为零,因为它基于单个轨迹并且忽略了熵项(TΔS)。PB非极性溶剂化能量目前不可分解。
2 结果
2.1 CsasCSP16重组蛋白的表达与纯化
通过终浓度为1 mmol·L-1IPTG 诱导结果显示,在14 kD左右出现一条蛋白条带。SDS-PAGE电泳显示CsasCSP16重组蛋白主要在包涵体中表达,因此对包涵体进行复性处理后过柱纯化获得了CsasCSP16重组蛋白。在SDS-PAGE条带上的大小与预测的分子量大小一致(图1)。
2.2 CsasCSP16与配基的结合
为了进一步确定CsasCSP16在嗅觉方面的潜在作用,选择33种气味的化合物和2种信息素作为配体,运用荧光竞争结合试验验证CsasCSP16的结合特性(表1)。荧光探针1-NPN与CsasCSP16有良好的结合特性,其解离常数(Kd)为9.408 µmol·L-1,表明1-NPN可用于后续的竞争结合试验。根据荧光竞争结合曲线(图2)计算出CsasCSP16与配体分子结合的IC50和Ki值。结果表明,CsasCSP16与水杨酸甲酯、6-甲基-5-庚烯-2-酮、十五烷、庚酸丁酯和-蒎烯有较强的结合活性,其Ki值分别为6.59、6.25、3.50、6.73和4.47 µmol·L-1。

A:CsasCSP16 cDNA核苷酸序列及其推导的氨基酸序列,信号肽序列用下划线标注,保守的半胱氨酸残基用圆圈标注,终止密码子用星号表示 Nucleotide and deduced amino acid sequence of CsasCSP14 cDNA. Signal peptide is underlined, conserved cysteine residues are marked in circles, the stop codons are denoted by asterisks [25]。B:CsasCSP16在大肠杆菌中的表达Induciton of the CsasCSP16 in E. coli;1:蛋白分子量标签 Protein molecular weight marker;4:未诱导对照组 Control group without induction;2、3和5:IPTG诱导的表达产物 Expressed products induced by IPTG。C:超声破菌结果The result of sonication;1:蛋白分子量标签 Protein molecular weight marker;2:重组菌的上清蛋白The soluble fraction of recombinant E. coli;3:包涵体 Inclusion body。D:CsasCSP16重组蛋白的纯化Purification of the CsasCSP16 recombinant protein;1:蛋白分子量标签 Protein molecular weight marker;2:上柱前蛋白 The proteins before purified by NTA-Ni affinity chromatography;3:流出液Outflow;4:30 mmol·L -1咪唑Imidazole at 30 mmol·L -1;5:300 mmol·L-1咪唑Imidazole at 300 mmol·L-1
2.3 CsasCSP16蛋白质三维模型构建
选择一致性(69%)最高的CSPMbraA6(登录号:1N8U)晶体结构为模板构建CsasCSP16蛋白质的三维模型(图3)。拉氏图评价结果表明有94.4%的氨基酸残基在最适区域内,5.6%的氨基酸残基在允许区域内(图 4)。Profile-3D的方法得到Verify Score值为37.81,远远高于要求的最低Verify Score值。CsasCSP16与CSPMbraA6叠合非常好,RMSD值为0.159(图3-B)。综上,CsasCSP16的分子三维模型是合理的,可用于后续的研究工作。
将CsasCSP16与水杨酸甲酯、6-甲基-5-庚烯-2-酮、十五烷、庚酸丁酯和-蒎烯5种气味分子进行分子对接,得到的Vina Score值分别为-6.1、-5.3、-5.8、-5.2和-6.6。分子对接的结果与试验数据相吻合。由CB-DOCK预测的结合口袋得出,5种气味分子与CsasCSP16的结合部位均在疏水性结合腔内部的同一位置(图5)。
2.4 CsasCSP16与气味分子的动力学模拟
为了使CsasCSP16与配体分子复合物的结构与晶体结构更接近,将CsasCSP16与配体复合物放置于TIP3P显性溶剂中进行了50 ns的动力学模拟。以CsasCSP16与配体分子在加热阶段的构象作为参考结构,使用RMSD值评估复合物的稳定性。结果如图6所示,CsasCSP16与水杨酸甲酯形成的复合物在10 ns时已经收敛达到平衡,RMSD值在0.2 nm左右波动;CsasCSP16与6-甲基-5-庚烯-2-酮形成的复合物在30 ns时达到平衡状态,RMSD值稳定在0.3 nm左右波动;CsasCSP16与十五烷形成的复合物在30 ns时达到收敛平衡,RMSD值在0.4 nm左右波动,CsasCSP16与十五烷的RMSD值波动较大,这可能与十五烷在结合腔内结构变化较大有关;CsasCSP16与庚酸丁酯在20 ns处达到收敛平衡,RMSD值稳定在0.25 nm左右。由于-蒎烯在形成拓扑文件时,结构罚分值超过50,故未能进行动力学模拟。以上结果说明复合物已经达到平衡状态,动力学模拟的结果达到期望值。

A、B:CsasCSP16与1-NPN的结合Binding curves of CsasCSP16 to 1-NPN;C:CsasCSP16与配体的荧光竞争结合fluorescence competition of CsasCSP16 with ligands;D:CsasCSP16对不同气味物质的结合亲和力(以1/Ki表示)的比较Comparison of the binding affinities (indicated by 1/Ki) of CsasCSP16 with different components

深绿色区域内为氨基酸残基的最佳区域,绿色区域内为氨基酸残基较合适的区域,其他区域为氨基酸残基勉强许可区和不合理区
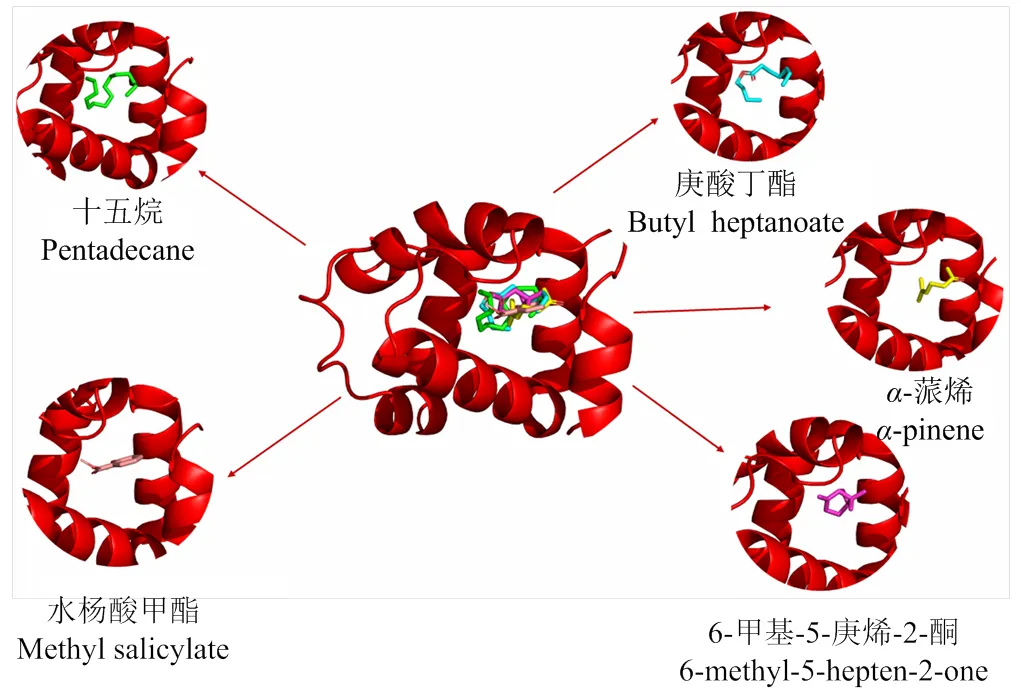
图5 CsasCSP16在预测的结合口袋与不同气味分子结合模式
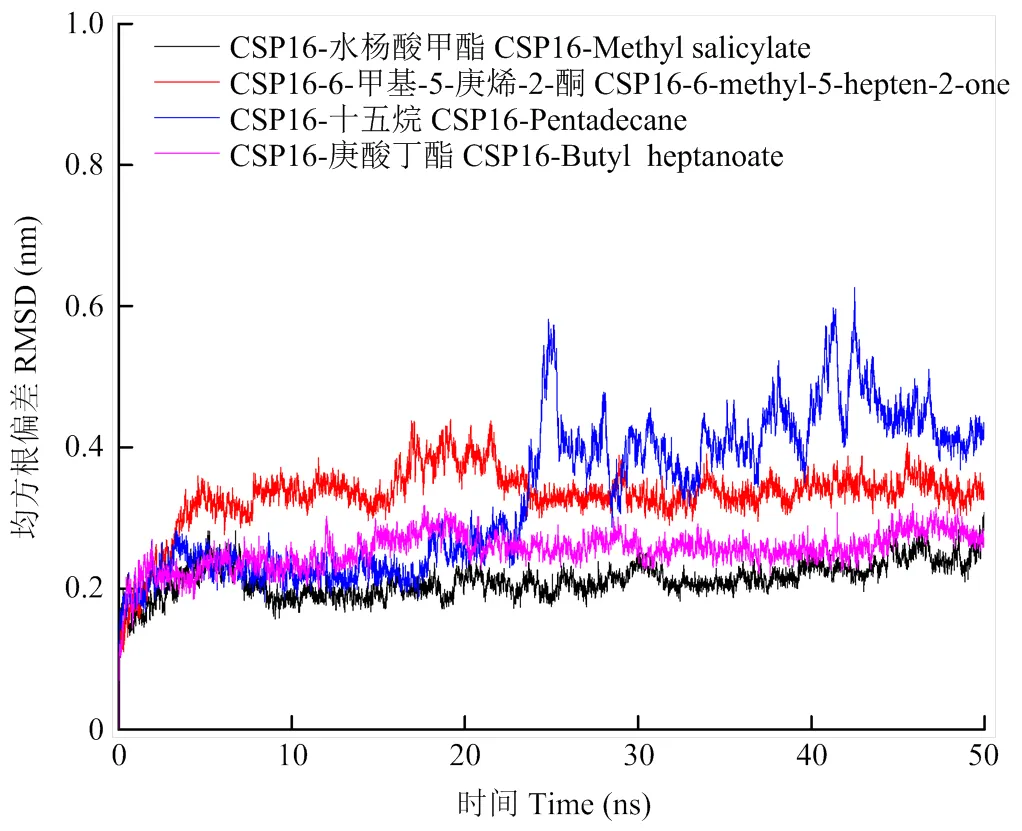
图6 CsasCSP16与配体复合物主链Cα原子RMSD随时间变化曲线
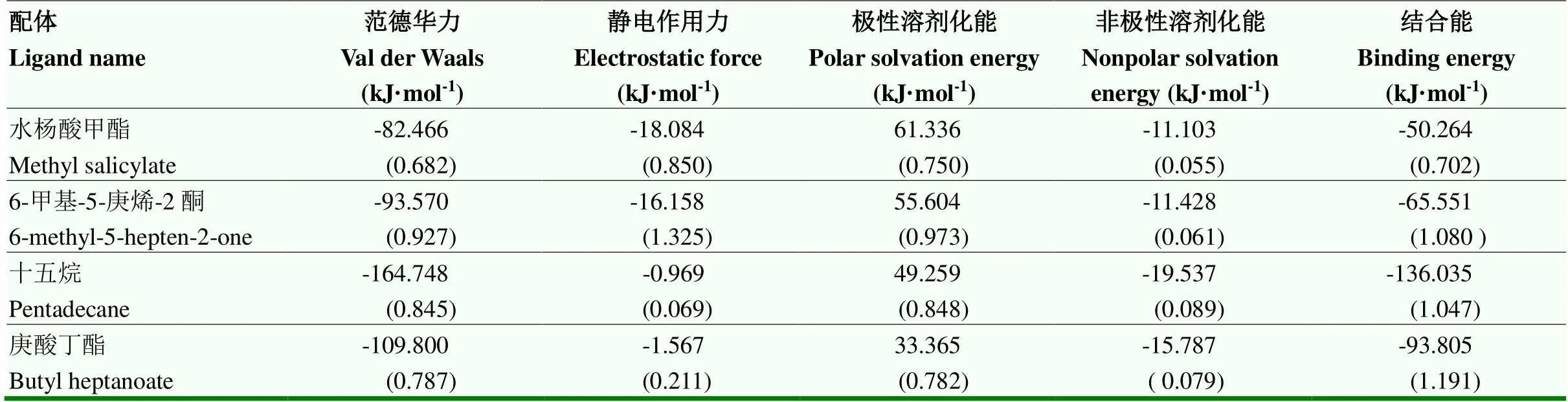
表2 CsasCSP16与配体分子的结合自由能和组分
2.5 结合自由能
采用MM-PBSA的方法计算CsasCSP16与配体的结合自由能[33]。CsasCSP16-水杨酸甲酯、CsasCSP16-6-甲基-5-庚烯-2-酮、CsasCSP16-十五烷和CsasCSP16-庚酸丁酯的结合自由能分别为-50.264、-65.551、-136.035和-93.805 kJ·mol-1,显然CsasCSP16可以与4种配体分子形成稳定的复合物。该结果与荧光竞争结合试验一致。结合自由能可以反映出哪些能量分项影响配体的亲和力。从表2中发现范德华力能量向提供了主要的驱动力,其次为静电作用力和非极性溶剂化能。溶剂化自由能非极性部分能提供负能量,不利于CsasCSP16与配体的结合,这是由于非极性的溶剂与配体分子相互排斥造成的。残基与配体的空间结构和相互作用力的类型可以揭示这些氨基酸残基与配体的关键相互作用。从图7看出,CsasCSP16与配体分子主要是以范德华力连接在一起,并且水杨酸甲酯与苏氨酸68(Thr68)形成氢键。从CsasCSP16与配体气味分子三维结构图(图8)可以看出,气味分子大多被包围在由疏水性氨基酸组成的结合腔。
2.6 能量分解
使用MM-PBSA方法得到单个氨基酸残基水平(per-residue level)的结合自由能贡献值,其数值结果为单个氨基酸残基与配体分子相互作用的总和。单个残基能量贡献详细情况如图9和表3所示,在CsasCSP16与水杨酸甲酯形成的复合物中,有6个氨基酸残基的能量贡献>1 kJ·mol-1。其中异亮氨酸49(Ile49)贡献的能量高达6.2 kJ·mol-1。相反,酪氨酸28(Tyr28)、谷氨酰胺64(Gln64)和苏氨酸68(Thr68)不利于水杨酸甲酯的结合,提供了负面的能量贡献;在CsasCSP16与6-甲基-5-庚烯-2-酮形成的复合物中,有11个氨基酸残基贡献的能量>1 kJ·mol-1,相反,精氨酸50(Arg50)提供负面的能量贡献,这是由于Arg50属于亲水性氨基酸,不利于CsasCSP16与6-甲基-5-庚烯-2-酮形成复合物。CsasCSP16与十五烷形成的复合物中,有14个氨基酸的能量贡献>1 kJ·mol-1,精氨酸91(Asp91)和组氨酸94(His94)提供反向结合的能量。CsasCSP16与庚酸丁酯形成的复合物中有14个氨基酸残基贡献的能量>1 kJ·mol-1,利于CsasCSP16与配体相结合的氨基酸属于疏水性氨基酸,Ile49、缬氨酸71(Val71)、异亮氨酸72(Ile72)和酪氨酸90(Tyr90)贡献的能量>3 kJ·mol-1,并且Ile49参与了CsasCSP16与4种气味分子结合的过程,由此可以推断Ile49、Val71、Ile72和Tyr90可能是CsasCSP16与配体结合的关键氨基酸残基。

图7 CsasCSP16与配体相互作用的关键氨基酸残基(二维结构)
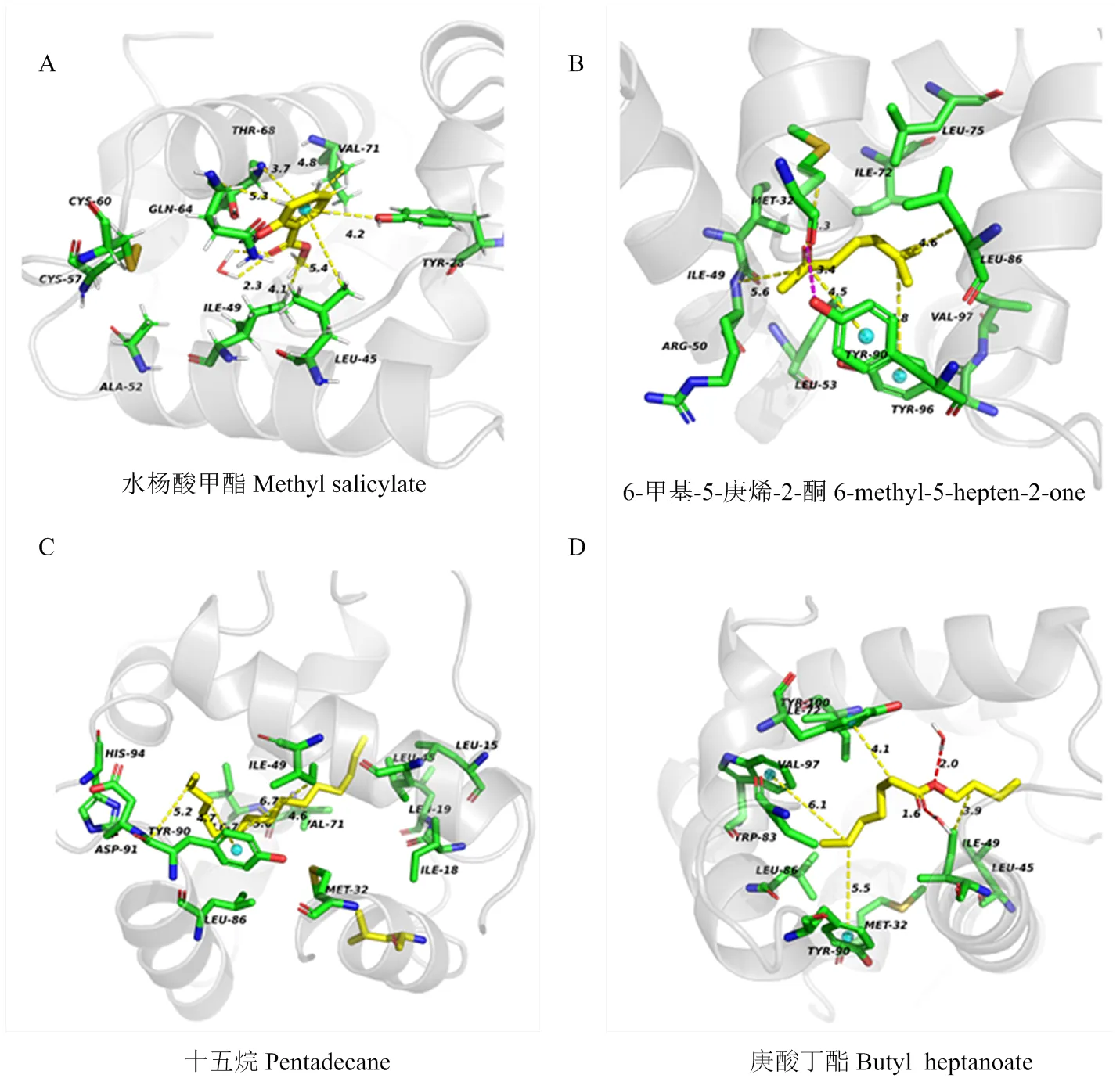
图8 CsasCSP16 与配体相互作用的氨基酸残基(三维结构)
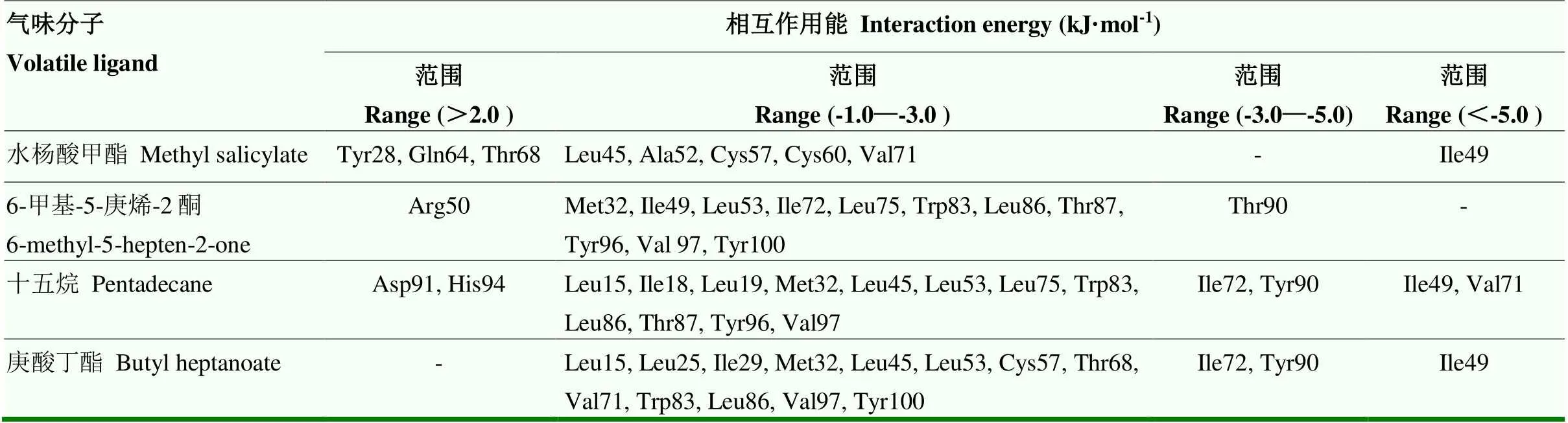
表3 主要残基对结合自由能的贡献

X轴表示CsasCSP16氨基酸残基的序号数,Y轴表示配体与CsasCSP16氨基酸残基之间的相互作用能量大小。重要的残基用相应的文字标记
3 讨论
在昆虫嗅觉感器中,化学感受蛋白结合疏水性的气味分子,穿过亲水性的淋巴液,传递给嗅觉神经元,将化学信号转化为电信号。本研究通过包涵体复性及Ni-NTA技术纯化得到了较高纯度的CsasCSP16蛋白,具有与天然蛋白一致的活性。Zhu等研究发现,含有His标签的融合蛋白与不带His标签的融合蛋白对气味分子的结合特性无影响[17]。结合特性研究结果表明,CsasCSP16与水杨酸甲酯、6-甲基-5庚烯-2-酮、十五烷、庚酸丁酯和-蒎烯显示出较强的结合特性,推测CsasCSP16可能是桃小食心虫识别这些气味分子的靶标基因。在触角中高表达的其他昆虫的CSP与寄主挥发物分子也具有较高的结合活性。如苜蓿盲蝽()AlinCSP4与法尼烯、-法尼醇、-紫罗兰酮有特异性的结合活性,AlinCSP1与3-己烯醇、2-己烯醛和正戊醛具有特异性结合的高亲和活性[35]。CsasCSP16不仅在触角中有表达,在雌、雄成虫翅中的表达量要高于其他组织,这与蜜蜂CSP3的表达谱比较一致,在翅中的功能有待于进一步研究[36]。ZHOU等研究发现,飞蝗()翅上存在许多化学感觉器,推测在翅中高表达的化学感受蛋白与接收寄主挥发物的次生代谢物有关[37]。本研究通过荧光竞争结合试验筛选出5个与CsasCSP16结合的气味分子,为进一步开发高效的引诱剂提供了参考依据。
荧光竞争结合试验可辨别CSP与不同气味分子的结合能力强弱,为了精确地揭示CSP与气味分子结合的具体作用机制,需要准确了解蛋白晶体结构。Tian等通过对苹果蠹蛾()性信息素结合蛋白(CpomPBP2)三维建模、分子对接预测了其与35种气味分子的亲和性,结果发现CpomPBP2与十二烷醇亲和性最高,荧光竞争结合表明CpomPBP2与十二烷醇抑制常数(Ki)值最小[38]。Venthur等用分子对接和分子动力学模拟预测欧洲葡萄蛾()性信息素结合蛋白(LbotPBP1)与11种性信息素分子和6种寄主挥发物的结合机制,发现十二碳烯是LbotPBP1的最好配体[39]。Fan等在研究禾谷缢管蚜()RpadOBP3与EBF相互作用的机制时,在疏水结构域中发现了3个氨基酸残基与EBF结合紧密,分别为色氨酸71(Trp71)、色氨酸68(Trp68)、苯丙氨酸26(Phe26)[40]。这对于进一步研究桃小食心虫CSP与气味物质的作用机制具有指导性意义。
本研究进一步开展了CsasCSP16与荧光竞争结合筛选得到的5种气味分子的分子对接,发现水杨酸甲酯与CsasCSP16的苏氨酸68(Thr68)残基之间形成氢键。这与Sandler等对家蚕()BmorPBP1与蚕蛾醇的研究结果一致,他们认为氢键在BmorPBP1专一性识别性信息素过程中发挥关键作用[41]。因此,CsasCSP16蛋白能够特异性识别水杨酸甲酯。水杨酸甲酯是许多植物的挥发物,由病虫害诱导产生,且可趋避害虫。如经斜纹夜蛾()危害的水稻产生大量的水杨酸甲酯,可显著趋避褐飞虱()[42]。CHEN等研究发现,白背飞虱()CfurCSP5与水稻挥发物水杨酸甲酯也具有高亲和性[43]。CsasCSP16与高亲和性气味分子结合能研究发现,CsasCSP16-水杨酸甲酯结合自由能最高,为-50.264 kJ·mol-1,该结果与荧光竞争结合试验一致;CsasCSP16与气味分子的相互作用力主要是范德华力和疏水作用力,异亮氨酸49(Ile49)、缬氨酸71(Val71)、异亮氨酸72(Ile72)和酪氨酸90(Tyr90)贡献的结合能>3 kJ·mol-1,因此推测这4种氨基酸在CsasCSP16结合气味分子的过程中发挥关键作用。在Chen等对白背飞虱CfurCSP5与水稻挥发物分子对接的研究中发现,CfurCSP5与具有高亲和性的气味分子结合过程中亮氨酸44(Leu44)、异亮氨酸64(Ile64)、丝氨酸98(Trp98)和苯丙氨酸101(Phe101)等疏水性氨基酸均参与其中[43]。同样地,在对小菜蛾CSPMbraA6与配体的研究中发现,亮氨酸(Leu)、异亮氨酸(Ile)和色氨酸(Trp)是与配体结合过程中的关键氨基酸[9]。因此,推测CsasCSP16与水杨酸甲酯结合最关键的氨基酸为异亮氨酸,水杨酸甲酯是否可以用作桃小食心虫寻找寄主的趋避物质有待于进一步探索。
4 结论
桃小食心虫CsasCSP16能与寄主植物的5种气味分子结合,可能参与桃小食心虫对寄主挥发物的识别。分子对接发现异亮氨酸49(Ile49)、缬氨酸71(Val71)、异亮氨酸72(Ile72)和酪氨酸90(Tyr90)可能是CsasCSP16与配体分子结合的关键氨基酸残基。
[1] 孙丽娜, 田志强, 张怀江, 李艳艳, 闫文涛, 岳强, 仇贵生. 氯虫苯甲酰胺干扰桃小食心虫交配的转录组分析. 中国农业科学,2018, 51(15): 2925-2936.
SUN L N, TIAN Z Q, ZHANG H J, LI Y Y, YAN W T, YUE Q, QIU G S. Transcriptome analysis of disruption of mating in the peach fruit moth () by chlorantraniliprole. Scientia Agricultura Sinica, 2018, 51(15): 2925-2936. (in Chinese)
[2] OLSSON S B, The Bengaluru Consortium. New frontiers for chemical ecology: Reaffirming a commitment to the göteborg resolution. Journal of Chemical Ecology, 2017, 43(1): 2-3.
[3] PELOSI P, ZHU J, KNOLL W. Odorant-binding proteins as sensing elements for odour monitoring. Sensors,2018, 18(10): 3248.
[4] 杜立啸, 刘杨, 王桂荣. 昆虫外周嗅觉系统信号转导机制研究进展. 中国科学: 生命科学,2016, 46(5): 573-583.
DU L X, LIU Y, WANG G R. Molecular mechanisms of signal transduction in the peripheral olfactory system of insects. Scientia Sinica Vitae,2016, 46(5): 573-583. (in Chinese)
[5] PELOSI P, IOVINELLA I, ZHU J, WANG G, DANI F R. Beyond chemoreception: diverse tasks of soluble olfactory proteins in insects. Biological Reviews of the Cambridge Philosophical Society, 2018, 93(1): 184-200.
[6] MEI T, FU W B, LI B, HE Z B, CHEN B. Comparative genomics of chemosensory protein genes (CSPs) in twenty-two mosquito species (Diptera: Culicidae): Identification, characterization, and evolution. PLoS ONE, 2018, 13(1): e0190412.
[7] ZHANG Y N, ZHU X Y, ZHANG Q, YIN C Y, DONG Z P, ZUO L H, DENG D G, SUN L, LI X M.assembly and characterization of antennal transcriptome reveal chemosensory system in. Journal of Asia-Pacific Entomology,2016, 19(4): 1077-1087.
[8] ZHANG Y N, KANG K, XU L, ZHU X Y, QIAN J L, ZHANG Z J, HE P, LI X M. Deep sequencing of antennal transcriptome fromto characterize odorant binding protein and chemosensory protein genes. Journal of Stored Products Research,2017, 74: 13-21.
[9] CAMPANACCI V, LARTIGUE A, HÄLLBERG B M, JONES T A, GIUDICI-ORTICONI M T, TEGONI M, CAMBILLAU C. Moth chemosensory protein exhibits drastic conformational changes and cooperativity on ligand binding. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(9): 5069-5074.
[10] LARTIGUE A, CAMPANACCI V, ROUSSEL A, LARSSON A M, JONES T A, TEGONI M, CAMBILLAU C. X-ray structure and ligand binding study of a moth chemosensory protein. The Journal of Biological Chemistry, 2002, 277(35): 32094-32098.
[11] MCKENNA M P, HEKMAT-SCAFE D S, GAINES P, CARLSON J R. Putativepheromone-binding proteins expressed in a subregion of the olfactory system. The Journal of biological chemistry, 1994, 269(23): 16340-16347.
[12] KITABAYASHI A N, ARAI T, KUBO T, NATORI S. Molecular cloning of cDNA for p10, a novel protein that increases in the regenerating legs of(American cockroach). Insect Biochemistry and Molecular Biology, 1998, 28(10): 785-790.
[13] CHENG D, LU Y, ZENG L, LIANG G, HE X.regulates the integument and moulting process of larvae in the red imported fire ant,. Scientific Reports,2015, 5: 9245.
[14] XUAN N, GUO X, XIE H Y, LOU Q N, LU X B, LIU G X, PICIMBON J F. Increased expression of CSP and CYP genes in adult silkworm females exposed to avermectins. Insect Science, 2015, 22(2): 203-219.
[15] BAUTISTA M A, BHANDARY B, WIJERATNE A J, MICHEL A P, HOY C W, MITTAPALLI O. Evidence for trade-offs in detoxification and chemosensation gene signatures in. Pest Management Science, 2015, 71(3): 423-432.
[16] GUO W, WANG X, MA Z, XUE L, HAN J, YU D, KANG L.andgenes modulate the switch between attraction and repulsion during behavioral phase change in the migratory locust. PLoS Genetics,2011, 7(2): e1001291.
[17] ZHU J, IOVINELLA I, DANI F R, LIU Y L, HUANG L Q, LIU Y, WANG C Z, PELOSI P, WANG G. Conserved chemosensory proteins in the proboscis and eyes of Lepidoptera. International Journal of Biological Science, 2016, 12(11): 1394-1404.
[18] DANI F R, MICHELUCCI E, FRANCESE S, MASTROBUONI G, CAPPELLOZZA S, LA MARCA G, NICCOLINI A, FELICIOLI A, MONETI G, PELOSI P. Odorant-binding proteins and chemosensory proteins in pheromone detection and release in the silkmoth. Chemical Senses,2011, 36(4): 335-344.
[19] IOVINELLA I, DANI F R, NICCOLINI A, SAGONA S, MICHELUCCI E, GAZZANO A, TURILLAZZI S, FELICIOLI A, PELOSI P. Differential expression of odorant-binding proteins in the mandibular glands of the honey bee according to caste and age. Journal of Proteome Research, 2011, 10(8): 3439-3449.
[20] JACQUIN-JOLY E, VOGT R G, FRANCOIS M C, NAGNAN-LE MEILLOUR P. Functional and expression pattern analysis of chemosensory proteins expressed in antennae and pheromonal gland of. Chemical Senses,2001, 26(7): 833-844.
[21] WARIS M I, YOUNAS A, ADEEL M M, DUAN S G, QUERSHI S R, KALEEM ULLAH R M, WANG M Q. The role of chemosensory protein 10 in the detection of behaviorally active compounds in brown planthopper,. Insect Science, 2020, 27(3): 531-544.
[22] TIAN Z, QIU G, LI Y, ZHANG H, YAN W, YUE Q, SUN L. Molecular characterization and functional analysis of pheromone binding proteins and general odorant binding proteins fromMatsumura (Lepidoptera: Carposinidae). Pest Management Science, 2019, 75(1): 234-245.
[23] TIAN Z, SUN L, LI Y, QUAN L, ZHANG H, YAN W, YUE Q, QIU G. Antennal transcriptome analysis of the chemosensory gene families in(Lepidoptera: Carposinidae). BMC Genomics,2018, 19(1): 544.
[24] 刘孝贺, 孙丽娜, 张怀江, 闫文涛, 岳强, 仇贵生. 桃小食心虫成虫化学感受蛋白CSPs的基因克隆及表达谱分析. 中国果树, 2020(2): 91-96.
LIU X H, SUN L N, ZHANG H J, YAN W T, YUE Q, QIU G S. Cloning and expression profile ofadults chemosensory protein. China Friuts, 2020(2): 91-96. (in Chinese)
[25] QUAN L F, QIU G S, ZHANG H J, SUN L N, LI Y Y, YAN W T. Sublethal concentration of beta-cypermethrin influences fecundity and mating behavior of(Lepidoptera: Carposinidae) adults. Journal of Economic Entomology, 2016, 109(5): 2196-2204.
[26] 李玲, 谭瑶, 周晓榕, 庞保平. 沙葱萤叶甲气味结合蛋白GdauOBP20的基因克隆、原核表达及其结合特性. 中国农业科学,2019, 52(20): 3705-3712.
LI L, TAN Y, ZHOU X R, PANG B P. Molecular cloning, prokaryotic expression and binding characterization of odorant binding protein GdauOBP20 inScientia Agricultura Sinica, 2019, 52(20): 3705-3712. (in Chinese)
[27] 李都, 牛长缨, 李峰奇, 罗晨. 斑翅果蝇气味结合蛋白OBP56h与小分子化合物的结合特征. 中国农业科学, 2019, 52(15): 2616-2623.
LI D, NIU C Y, LI F Q, LUO C. Binding characterization of odorant binding protein OBP56h inwith small molecular compounds.Scientia Agricultura Sinica, 2019, 52(15): 2616-2623. (in Chinese)
[28] MORRIS G M, HUEY R, LINDSTROM W, SANNER M F, BELEW R K, GOODSELL D S, OLSON A J. AutoDock4 and autodocktools4:automated docking with selective receptor flexibility. Journal of Computational Chemistry, 2009, 30(16): 2785-2791.
[29] LIU Y, GRIMM M, DAI W T, HOU M C, XIAO Z X, CAO Y. CB-Dock: a web server for cavity detection-guided protein-ligand blind docking. Acta Pharmacologica Sinica, 2020, 41(1): 138-144.
[30] PRONK S, PALL S, SCHULZ R, LARSSON P, BJELKMAR P, APOSTOLOV R, SHIRTS M R, SMITH J C, KASSON P M, VAN DER SPOEL D,HESS B, LINDAHL E. GROMACS 4.5: a high- throughput and highly parallel open source molecular simulation toolkit. Bioinformatics, 2013, 29(7): 845-854.
[31] HUANG J, RAUSCHER S, NAWROCKI G, RAN T, FEIG M, DE GROOT B L, GRUBMULLER H, MACKERELL A D, JR. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods,2017, 14(1): 71-73.
[32] 张贺, 卢俊瑞, 穆江蓓, 刘金彪, 杨旭云, 王美君, 张瑞波. 3MBA类FtsZ蛋白抑制剂的分子动力学模拟及抗菌作用机制. 物理化学学报,2015, 31(3): 566-575.
ZHANG H, LU J R, MU J B, LIU J B, YANG X Y, WANG M J, ZHANG R B. Molecular dynamics simulation and antibacterial mechanism of 3MBA derivatives as FtsZ protein inhibitors. Acta Physico-Chimica Sinica, 2015, 31(3): 566-575. (in Chinese)
[33] KUMARI R, KUMAR R, Open Source Drug Discovery Consortium, LYNN A. g_mmpbsa-a GROMACS tool for high-throughput MM- PBSA calculations. Journal of Chemical Information and Modeling,2014, 54(7): 1951-1962.
[34] HOU T, ZHANG W, CASE D A, WANG W. Characterization of domain-peptide interaction interface: a case study on the amphiphysin-1 SH3 domain. Journal of Molecular Biology, 2008, 376(4): 1201-1214.
[35] GU S H, WANG S Y, ZHANG X Y, JI P, LIU J T, WANG G R, WU K M, GUO Y Y, ZHOU J J, ZHANG Y J. Functional characterizations of chemosensory proteins of the alfalfa plant bugindicate their involvement in host recognition. PLoS One,2012, 7(8): e42871.
[36] LI H L, NI C X, TAN J, ZHANG L Y, HU F L. Chemosensory proteins of the eastern honeybee,: Identification, tissue distribution and olfactory related functional characterization.Comparative Biochemistry and Physiology. Part: Biochemistry and Molecular Biology, 2016, 194/195: 11-19.
[37] ZHOU S H, ZHANG J, ZHANG S G, ZHANG L. Expression of chemosensory proteins in hairs on wings of(Orthoptera: Acrididae). Journal of Applied Entomology,2008, 132(6): 439-450.
[38] TIAN Z, LIU J Y, ZHANG Y L. Structural insights intopheromone binding protein 2 mediated prediction of potentially active semiochemicals. Scientific Reports, 2016, 6: 22336.
[39] VENTHUR H, MACHUCA J, GODOY R, PALMA-MILLANAO R, ZHOU J J, LARAMA G, BARDEHLE L, QUIROZ A, CEBALLOS R, MUTIS A. Structural investigation of selective binding dynamics for the pheromone-binding protein 1 of the grapevine moth,. Archives of Insect Biochemistry and Physiology, 2019, 101(3): e21557.
[40] FAN J, XUE W X, DUAN H X, JIANG X, ZHANG Y, YU W J, JIANG S S, SUN J R, CHEN J L. Identification of an intraspecific alarm pheromone and two conserved odorant-binding proteins associated with (E)-beta-farnesene perception in aphid. Journal of Insect Physiology, 2017, 101: 151-160.
[41] SANDLER B H, NIKONOVA L, LEAL W S, CLARDY J. Sexual attraction in the silkworm moth: structure of the pheromone- binding-protein-bombykol complex. Chemistry and Biology,2000, 7(2): 143-151.
[42] 周强, 徐涛, 张古忍, 古德祥, 张文庆. 虫害诱导的水稻挥发物对褐飞虱的驱避作用. 昆虫学报, 2003, 46(6): 739-744.
ZHOU Q, XU T, ZHANG G R, GU D X, ZHANG W Q. Repellent effects of herbivore-induced rice volatiles on the brown planthopper,Stål. Acta Entomologica Sinica, 2003, 46(6): 739-744. (in Chinese)
[43] CHEN G L, PAN Y F, MA Y F, WANG J, HE M, HE P. Binding affinity characterization of an antennae-enriched chemosensory protein from the white-backed planthopper,(Horvath), with host plant volatiles. Pesticide Biochemistry and Physiology,2018, 152: 1-7.
Ligands Binding Characteristics of Chemosensory Protein CsasCSP16 of
LIU XiaoHe1, QIU GuiSheng1, TONG ZhaoGuo2, ZHANG HuaiJiang1, YAN WenTao1, YUE Qiang1, SUN LiNa1
1Research institute of Pomology, Chinese Academy of Agricultural Sciences, Xingcheng 125100, Liaoning;2School of Agriculture Science, Xichang University, Xichang 615013, Sichuan
【】is one of the most destructive pests on orchards in north China. The objectives of this study are to get the recombinant CsasCSP16 protein, characterize the binding profiles of CsasCSP16 with some small molecular volatiles from host plants, and predict the key residue between CsasCSP16 and putative ligands. In overall this research possibly lays a theoretical and practical foundation for the understanding of olfactory mechanism in.【】The recombinant protein CsasCSP16 was induced to express by constructing prokaryotic expression system, and purified by using the Ni-NTA agarose affinity column. Then, the fluorescence competitive assay was applied, and N-phenyl-1-naphthylamine (1-NPN) was selected as the fluorescence probe to measure the binding profiles of CsasCSP16 recombinant with 33 host plant volatiles and 2 sex pheromone compounds. The Modeller software was used to build three-dimensional model. CSPMbraA6 (PDB ID: 1N8U) was used as the template for homology modeling of CsasCSP16 protein structure. The modeled structure of CsasCSP16 was docked with 35 selected ligands by Autodock vina. Then, the complexes from above step were introduced to GROMACS (2019.3) to conform the stability. Moreover, the binding energies of the complexes were calculated by g_mmpbsa and the key residues interaction between CsasCSP16 and host volatiles were predicted.【】The recombinant expression vector was successfully constructed, and the recombinant protein of CsasCSP16 with high purity was obtained by bacterial system. Further, competitive fluorescence binding assays with 35 candidate ligands showed that CsasCSP16 had high binding affinities against methyl salicylate, 6-methyl-5-hepten-2-one, pentadecane, butyl heptanoate and-pinene, and the recorded Ki values were 6.59, 6.25, 3.50, 6.73 and 4.47 µmol·L-1, respectively. Ligand docking results revealed that the Vina Scores of CsasCSP16-methyl salicylate, CsasCSP16-6-methyl-5-hepten-2-one, CsasCSP16-pentadecane, CsasCSP16-butyl heptanoate and CsasCSP16--pinene were -6.1, -5.3, -5.8, -5.2 and -6.6, respectively. Finally, the g_mmpbsa analysis demonstrated that the binding energies of CsasCSP16-methyl salicylate, CsasCSP16-6-methyl-5- hepten-2-one, CsasCSP16-pentadecane and CsasCSP16-butyl heptanoate were -50.264, -65.551, -136.035 and -93.805 kJ·mol-1, respectively. Ile49, Val71, Ile72 and Tyr90 contribute energy more than 3 kJ·mol-1, suggesting their key involvement in the function of CsasCSP16. 【】The CsasCSP16 has a certain binding capacity with several odors from host plant volatiles, suggesting that it may play an important role in the localization of host plants. Ile49, Val71, Ile72, and Tyr90 may be the key residues involved in the function of CsasCSP16.
; chemosensory protein (CSP); prokaryotic expression; competitive binding; molecular docking; dynamic simulation

10.3864/j.issn.0578-1752.2021.05.007
2020-05-11;
2020-06-22
国家重点研发计划(2017YFD0200300)、国家自然科学基金(31601643)
刘孝贺,E-mail:liuxiaoheaye@163.com。通信作者孙丽娜,E-mail:sunlina@caas.cn
(责任编辑 岳梅)

