环境DNA技术在水生入侵生物监测中的应用
林渊源,赵峥,*
1. 昆明滇池(湖泊)污染防治合作研究中心,昆明学院,昆明 650214 2. 昆明市河湖生态健康评估与修复院士工作站,昆明 650214
近年来,经济全球化进程愈快,国家、地区间的贸易交往日益频繁,主动和被动引入的外来生物愈多,为外来物种入侵创造了条件,同时也给全球环境带来了生态风险[1-2]。外来水生生物随着频繁的航运、水产品养殖引种等直接和间接途径引入新的栖息地,在新的生态系统迅速繁殖扩散,与本地物种竞争食物和空间或直接掠杀吞食本地种,导致本地生物群落结构改变、生物多样性水平降低,正严重威胁水生生态系统的健康与稳定,影响着入侵地的经济、社会发展和公众健康[3-4]。比如臭名昭著的外来生物福寿螺,它们取食多种植物幼苗或根茎、危害作物生长,携带传染病原物,不仅带来直接经济损失和危害人类健康,而且其代谢物会改变水体环境、影响水体生物群落结构,影响入侵地生物多样性,加剧了入侵地的生态风险[5]。据不完全统计,外来入侵生物构成的直接经济损失每年逾2 000亿元[6],由水生入侵藻类引发的赤潮爆发造成的经济损失每年高达5亿元[7],由巴西龟入侵造成的间接经济损失已达1 000亿元[8]。
为了有效防控外来入侵有害生物,各国政府每年都要投入大量的人力、物力和财力对外来生物进行严格的检疫、查封和堵截以及对入侵生物的灭杀清除[9-10]。但外来生物一旦构成入侵,再根除就非常困难,甚至不可能,以防为主、防治结合被认为是外来入侵生物防控最有效的策略,因此,外来生物的早期监测和风险预警研究随之兴起。目前外来生物监测主要依赖于形态学观察和分类,不仅工作量大,而且要求监测人员具备相关的形态、分类学经验,难以满足快速大量监测的需求[11]。且外来生物在引入初期通常群体规模极小,可能难以察觉或被忽略,尤其是外来水生生物,其物种繁多,多数形体微小、形态多变,与陆生外来生物相比隐蔽性更强、更易扩散,通常难以及时监测和有效评估预警,进而构成入侵,危害生态环境及人类健康[3, 12]。因此,开发高效、灵敏的新技术应用于外来水生入侵生物监测至关重要。
环境DNA(environmental DNA, eDNA)泛指从水、土壤和沉积物等环境介质中提取出来的总DNA,其源于有机体本身或其生命过程中释放的分泌物、内含物、血液及孢子、花粉或脱落的器官等[13]。自Ficetola等[14]首次将环境DNA条形码分析(eDNA barcoding)引入对入侵种美国牛蛙(Ranacatesbeiana)的检测以来,环境DNA技术(eDNA technologies)为水生入侵生物监测开辟了新路径,在入侵生物的定性、定量及入侵机制探索方面都有广泛应用(图1)。普通聚合酶链式反应(PCR)主要用于物种的定性检测,以eDNA为模板,通过对目标物种或种群的特异性基因扩增、测序,从而实现新种鉴定、外来种和濒危种监测。而定量PCR(qPCR)和微滴式数字PCR技术(ddPCR)则能对eDNA定量,量化表征物种的丰富度和生物量等,在物种种群大小估算、资源调查等方面极具潜力[15]。且随着第二代测序技术(next -generation sequencing, NGS)的发展,eDNA整合宏条形码分析深化发展为环境DNA宏条形码技术(eDNA-metabarcoding),通过提取环境介质中的总DNA进行高通量测序以分析环境中生物物种组成及丰度,可实现生物物种的高效鉴别及多生物群落监测,已应用于低丰度生物物种检测、隐匿种发现、生物多样性调查等方面[16-17]。与耗时费力的传统形态学生物监测、资源调查(比如围网捕捞、电气捕鱼)相比,eDNA技术具有低耗、高效、高灵敏度及对生物体无损伤等特点,优势得天独厚[18],其在外来水生生物监测方面具有十分广阔的应用前景。
本文首先总结了eDNA结合条形码或宏条形码技术在水生入侵生物监测研究中的应用实例;然后从eDNA的获取、条形码区域的PCR扩增和数据分析这3个方面探讨了eDNA技术实施的方案、关键步骤;最后对促进eDNA在水生生物入侵监测应用的方式及前景进行了展望,以期为水生生物入侵防控提供基础理论和技术支撑。
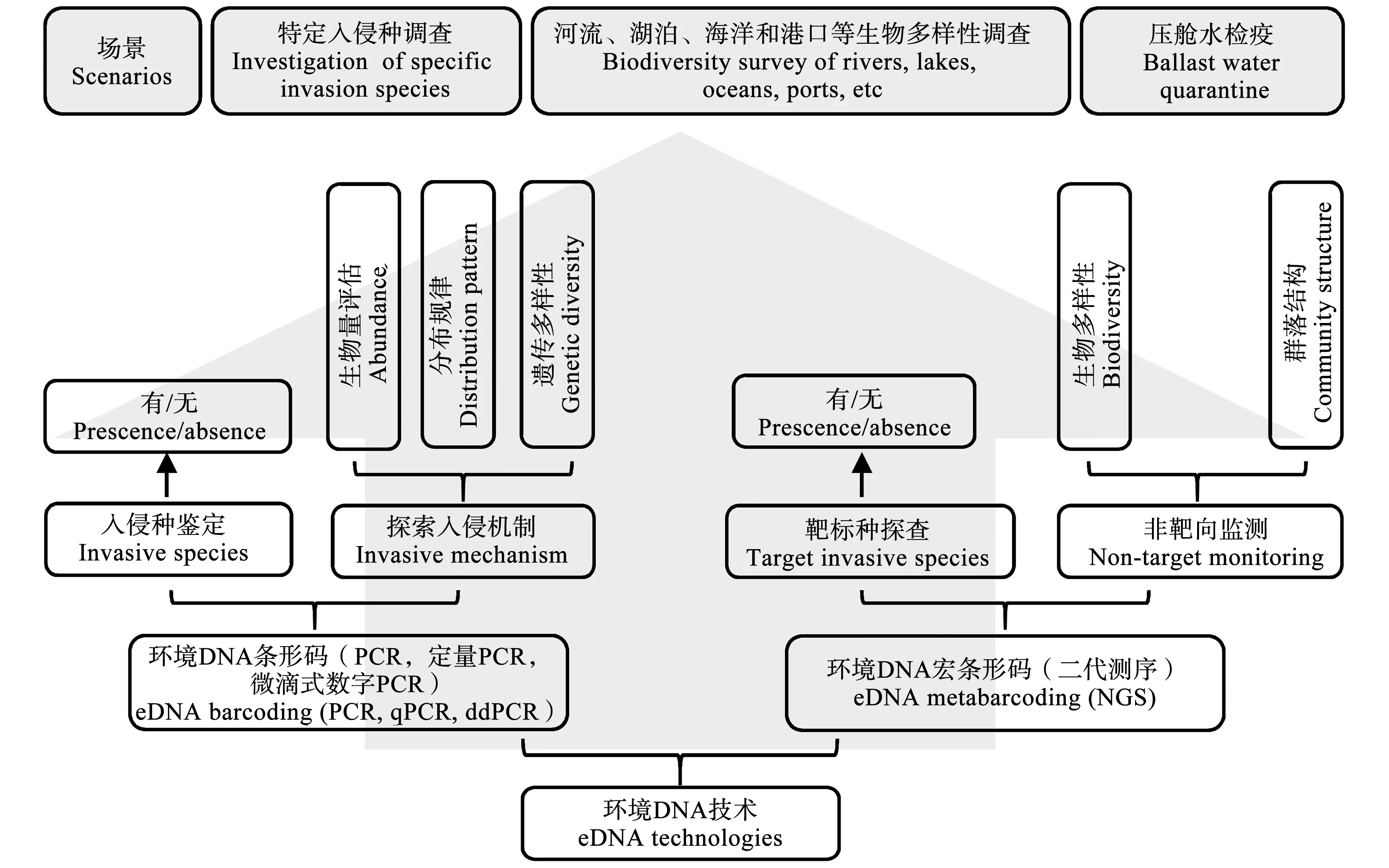
图1 环境DNA(eDNA)技术应用于入侵生物研究注:PCR表示聚合酶链式反应,qPCR表示荧光定量PCR,ddPCR表示微滴式数字PCR,NGS表示二代测序技术。Fig. 1 Environmental DNA (eDNA) technologies applied to the study of invasive speciesNote: PCR represents polymerase chain reaction; qPCR represents quantitative real time PCR; ddPCR represents droplet digital PCR; NGS represents next generation sequencing.
1 eDNA条形码技术应用于水生入侵生物监测及入侵机理研究(Exploring aquatic invasive species and invasion mechanism using eDNA barcoding)
1.1 目标入侵物种探查
对入侵生物采取控制措施的先决条件是迅速而准确地识别具有威胁性的外来物种,通过常规PCR方法对目标生物进行特异性扩增以检测目标物种的存在是入侵生物监测的主要方式之一。2008年,Ficetola等[14]首次将eDNA与条形码分析引入水生生物监测,他们提取法国18个自然水体中的eDNA后利用基因特异性引物进行PCR扩增并测序,检出了传统方法未检出的入侵物种美国牛蛙(Ranacatesbeiana),为水生生物监测引入了新的研究方法,开辟了水生入侵生物早期监测的新视野,并将该技术逐渐拓展到鱼类、爬行动物和软体动物等多类生物监测。Jerde等[19]将eDNA技术用于鳙鱼(Hypophthalmichthysnobilis)和鲢鱼(Hypophthalmichthysmolitrix)的监测,发现连接密西西比河和大湖盆地的芝加哥水道面临着外来鱼类入侵的威胁。Piaggio等[20]开发了适用于检测缅甸蟒蛇(Pythonbivittatus)的eDNA条形码监测方法,并在美国南佛罗里达的5个野外水域里检出了该蛇。Ardura等[21]设计开发了北美楔蛤的物种特异性分子标记及eDNA监测方法,并利用波罗的海和维斯瓦泻湖水样评估了该法的可行性,发现即使在种群稀少且规模较小的情况下,也能从eDNA中检测到北美楔蛤的痕迹。总之,eDNA条形码技术为快速精准探查目标入侵生物的存在提供了便利,从而促进管理部门能够迅速做出反应、指导部署该物种的防控工作。
1.2 入侵机制研究
为了更有效地防控外来生物入侵,研究者们不仅着重于外来物种的早期监测及预警,也越来越重视解析入侵生物的入侵机理。eDNA技术简便高效,便于大规模、大尺度分析,已逐渐应用于探索入侵物种遗传多样性、种群扩散规律和生物量评估等。基于线粒体DNA条形码分析,Serrao等[22]对收集到的121个黑鱼样本和已有GenBank数据进行聚类分析,结果分成了49个分支,显示其丰富的遗传多样性,为黑鱼入侵路径的推断提供了有效依据。Takahara等[23]利用eDNA分析追踪了蓝鳃太阳鱼(Lepomismacrochirus)在日本本土及周边岛屿70个水域的分布情况,有19个水域监测到了此入侵鱼类的存在,主要分布在日本本土,周边岛屿的扩散趋势不明显。通过qPCR或ddPCR法,eDNA技术不仅可以确定目标生物的存在,还能量化表征物种的丰富度和生物量,为入侵物种监管治理提供更有效的基础数据。Goldberg等[24]发现在清除新西兰泥螺(Potamopyrgusantipodarum)21~44 d后还能在水体中检测到其DNA的存在,通过qPCR检测,他们发现此螺在波特纳夫河的分布密度约为每平方米11~144个,说明eDNA调查在帮助早期发现入侵性水生无脊椎动物及其丰度计算、风险预警方面具有很高的潜力。Nathan等[25]利用传统PCR、qPCR、ddPCR方法分别对环境中虾虎鱼DNA在24 h内的存在和丰度进行了检测,结果表明,即使在低目标生物密度,3种方法都能有效检测到其DNA的存在,qPCR和ddPCR估算出了相似的DNA浓度,且ddPCR速度更快,花费更少。Jo等[26]利用多重实时荧光定量PCR技术对3种外来鱼类和3种受威胁的本地鱼类在日本99个池塘的季节分布进行了检测,发现所有鱼类的eDNA检出率在初夏都较高,而且外来鱼类的eDNA更容易在汽车更容易到达的池塘中检测到。这都可为阐明入侵物种的扩散入侵过程,最终针对性地指导制定相关治理政策提供可靠证据。
2 eDNA宏条形码技术应用于水生入侵生物监测(Monitoring aquatic invasive species using eDNA metabarcoding)
随着DNA测序技术的飞速发展,eDNA条形码整合高通量测序技术深化发展为eDNA宏条形码技术,其低耗、高效和高灵敏度的优势极大拓展了其在水生生态系统入侵生物的早期监测与预警中的应用,在靶标和非靶标水生入侵生物监测及压舱水检疫上都有长足应用。
2.1 靶标种监测
针对靶标生物设计特异性引物对eDNA进行扩增和分析以检测靶标物种的存在和丰度是利用eDNA宏条形码技术进行入侵种监测的重要方式,已涉及藻类、甲壳类和鱼类等多类生物的监测。为了解我国辽东湾海域是否存在甲藻(Stoeckeriaalgicida)的入侵风险,宋伦等[27]针对微型/微微型浮游植物设计18SrDNAV4区特异性引物对辽东湾浮游植物进行了检测,发现夏季Stoeckeriaalgicida在辽东湾东、西两岸密度较高,致灾风险较高,必须加强监管。Muha等[28]利用eDNA宏条形码技术对比斯开湾和邻近地区的入侵性海藻(Codiumfragile)进行定期监测,为评估Codiumfragile的潜在扩张及其管理干预提供参考。利用eDNA宏条形码技术检测靶标水生入侵动物的研究较多,已应用于缨鳃虫(Sabellaspallanzanii)[29]、贻贝(Dreissenapolymorpha、Dreissenarostriformisbugensis)[30-31]和克氏原螯虾(Procambarusclarkii)[32]等生物。且随着DNA提取技术、测序技术等的飞速发展,eDNA宏条形码技术在入侵监测的应用也越来越成熟、规范,比如Lin等[33]优化了eDNA提取方法和检测引物,并提出了利用eDNA分析监测美国牛蛙地理分布的标准技术规范。
2.2 非靶标种监测
通过设计高灵敏度通用引物对eDNA进行扩增和高通量测序分析以比较群落物种组成、对非靶标入侵生物进行监测,可广泛全面预警生态系统中各类外来生物的入侵风险。Brown等[34]利用通用引物Uni18S扩增出加拿大海岸线和五大湖主要港口浮游生物的18SrDNAV4区,经系统进化分析,24个物种被鉴定为外来入侵种,其中11个种在该地区首次被检出。Rey等[35]基于COI和18SrDNAV9区设计通用引物,在西班牙毕尔巴鄂港检测到了79个外来种,其中包括了大多数之前生物调查已鉴定出的外来物种及7个从未检出的物种。Westfall等[36]利用宏条形码技术监测了西北太平洋生物的群落组成,识别了12种已知的外来物种以及另外7种无记录的外来物种,为外来物种的早期监测和防控,减轻生物入侵的发生提供基础数据。Chen等[37]同时利用eDNA宏条形码技术和形态学监测对我国青岛海底世界水族馆的非原生物种多样性进行了调查研究,eDNA宏条形码监测鉴定出了24个非本地种及另外4个可能的非本地操作分类单元,而形态学监测则只鉴定到20个非本地种,认为eDNA宏条形码监测较形态学监测更灵敏,但同时为了提高物种的检出率和降低假阳性率,建议利用eDNA宏条形码监测时应进行多方位的采样和严格设置阴性对照。
2.3 压舱水检疫
船舶压舱水及其淤泥被认为是外来生物传播入侵的主要驱动力,压舱水的检疫监管可为外来入侵生物的防控提供基础数据和警示,eDNA宏条形码技术作为一种新型监测手段在船舶压舱水及其淤泥中外来生物的检疫监管中已被广泛应用。利用eDNA宏条形码技术,Rey等[38]对抵达美国切萨皮克湾的11艘船舶的压载水的生物物种组成进行了分析,并在其中发现了外来种长腹剑水蚤属(Oithonadavisae)。Ardura等[39]从7艘抵达西班牙比斯开湾希洪港的船舶的压载水中检测出了72种藻类,其中22.2%为比斯开湾的外来物种,11.1%被归类为有害物种。Lin等[40]利用eDNA宏条形码技术对比了淡水、半咸水压舱水及其氯处理压舱水的浮游动物群落结构组成差异后认为氯处理压舱水不能有效减少入侵风险,并呼吁进行氯处理、紫外灭杀等多方式联用以降低压舱水带来的生物入侵风险。
3 eDNA技术实施流程的关键因素(The key factors of implementation scheme of eDNA technologies)
尽管eDNA技术为入侵生物学研究开辟了一条新的道路,但其应用实施尚处于探索阶段,也还未形成一套广谱通用的操作流程。比如由于不同生境下生物、物理和化学环境参数存在很大差异,这些差异会严重影响eDNA的来源、运输和状态等,并可能最终对eDNA检测结果产生影响[41];而进行eDNA实验操作的各个环节比如标记基因的选择、数据处理参数的设置等也都可能影响最终的结果[17]。因此,我们从eDNA的获取、条形码区域的PCR扩增和数据分析3个方面来系统综述eDNA宏条形码技术实施操作流程中的关键因素,并对各个环节需要注意的细节进行说明和讨论。
3.1 eDNA的获取
目前对于eDNA获取的方法还未有统一的标准流程,而从环境中捕获和提取eDNA的方法可能会强烈影响其最后的结果,现有研究一般从取样方式、采样介质、DNA浓缩和抽提方法等方面进行优化。首先,研究者会根据研究目的、目标物种的生活习性、潜在丰度及综合考虑研究区域的自然地理环境比如水体面积、水源质量等因素来选择取样的方式,且由于生物体DNA在环境中存在与否的随机性较大,为提高检出率、减少误差,生物学重复十分必要,每个点位一般至少收集3个平行样本[42-43]。在溪流和浅水系统检测中可直接采集表层水提取eDNA进行分析而不影响其多样性[44-45];在深水系统(比如湖泊、深海)则需谨慎考虑,因为在生态系统中不同物种可能存在不同的空间分布特征,适当的采样深度可以改善物种检测度[46-47],在防止eDNA分散的屏障下面或周围采集水样,并为每个样本汇集不同层次的水以减少采样引起的偏差不失为一个有效方式[43]。其次,对样品进行有效的浓缩比如异丙醇或乙醇离心沉淀和过滤可提高eDNA的质量,增加检出率。离心沉淀一般只适用于浓缩小体积(~90 mL)水样,而过滤则可大大增加样品体积,且过滤器的保存比水样更简便,通常可选择使用纤维滤膜过滤水样后经Longmire缓冲液、乙醇或超低温存储过滤器处理以富集保存eDNA[43, 48]。最后,据研究目的使用相应DNA提取方法进行提取,除了少数研究中采用十六烷基三甲基溴化铵或苯酚-氯仿-异戊醇抽提法等提取DNA外,目前大多数研究都采用商品化DNA提取试剂盒来抽提样品以获得高质量eDNA[49]。
杨江华[50]研究了采样方法、采样体积对浮游动物宏条形码测序结果的影响,认为直接过滤1 L的水样适于研究轮虫的多样性,用浮游动物网富集5~10 L的水样适于研究枝角类和桡足类多样性,初步实现了浮游动物宏条形码监测技术的标准化。Shu等[49]综合比较了现有利用eDNA检测鱼类的文献后认为,检测鱼类,可采集1 L或2 L的表层水,通过0.7 μm的玻璃纤维滤膜过滤收集eDNA,然后用DNeasy Blood and Tissue Kit或Power Water DNA Isolation Kit进行提取,可以得到高质量的eDNA和可靠的结果。Kumar等[51]总结众多研究中水eDNA样品的收集和处理的方法流程后认为,虽然很难归纳一套适应于所有eDNA宏条形码技术的操作规范,但多数情况下,在合适孔径大小的硝酸纤维素过滤器上浓缩eDNA,并在Longmire缓冲液中存储过滤器,然后用DNeasy Blood and Tissue Kit或类似试剂盒能够获得充足、高质量的eDNA。总之,为尽可能减少eDNA获取操作过程所引起的偏差,应该选择一个比较一致的、可靠的操作流程以便实现监测技术的标准化。
3.2 条形码区域的PCR扩增
eDNA分析得以实施的关键是条形码序列的扩增,选择合适的分子标记基因(即条形码区域)并设计高分辨率的通用引物十分重要[17, 52]。分子标记基因的选择一般遵循2个原则:(1)包含相对保守的区域来设计通用性强的引物以便于PCR扩增;(2)选定条形码区域的种间差异大、种内差异小,能有效鉴别物种,目前已开发出多个适于eDNA分析的标记基因(表1)。
线粒体和核基因区段都被报道可作为标记基因用于eDNA分析,但不同分子标记基因适用范围不同,不同物种对引物的亲和力也不同,其扩增效率也就可能不一致,经扩增后一些物种的丰度可能会被“提高”或“降低”,或根本无法被扩增,进而导致错误结论,应据研究目的综合选择适当的分子标记基因[53-54]。核糖体小亚基16S核糖核酸(16SrRNA)基因既有高度保守性区域,又有多个高度变异性区域,是蓝藻多样性研究应用最广泛的基因[55]。16-23SrRNA基因转录间隔序列(ITS)属内物种间差异显著,基于ITS基因对拟圆孢藻属进行系统发育分析发现其3个物种可各自形成彼此区分的单系类群[56]。线粒体细胞色素C氧化酶亚基I(COI)基因具有进化速度快和功能保守性强的特点,在区分近缘物种或亚种的鉴别中极具优势,是动物条形码研究中的“黄金标志”[57],常用来分析浮游动物[58]、甲壳动物[59]和鱼类[60]等。但也研究表明COI基因较快的进化速度会导致较大的种内多样性,比如萼花臂尾轮虫(Brachionuscalyciflorus)的COI就有多个不同的基因型[61]。核糖体18SrRNA基因序列进化速度较慢,覆盖度高,能覆盖大部分的真核生物群落,适用于研究亲缘关系较远的生物群落结构[62]。Zhan等[63]比较了COI、16SrRNA和18SrRNA等分子标记基因在浮游动物宏条形码研究中的效果,发现其所用的COI引物无法扩增出高质量的PCR产物,并建议使用18SrRNA基因和线粒体16SrRNA基因进行浮游动物条形码研究。杨江华[50]比较了常用COI、18SV9及基于浮游动物16SrRNA基因序列设计的16S引物进行水体宏条形码监测的差别,认为在不考虑物种水平的多样性并需要涵盖尽可能多的生物类群时优先选用18SV9引物,为研究浮游动物多样性时选择COI引物,而关注轮虫群落多样性时使用16S引物更合适。Shu等[49]比较分析了现有利用eDNA检测鱼类的文献后认为,基于细胞色素B基因(Cytb)的特异性引物可有效鉴定目标鱼类及评估其物种丰富度;联合使用12S和16SrRNA的通用引物可使宏条形码评估鱼类生物多样性的准确性与覆盖度增加。
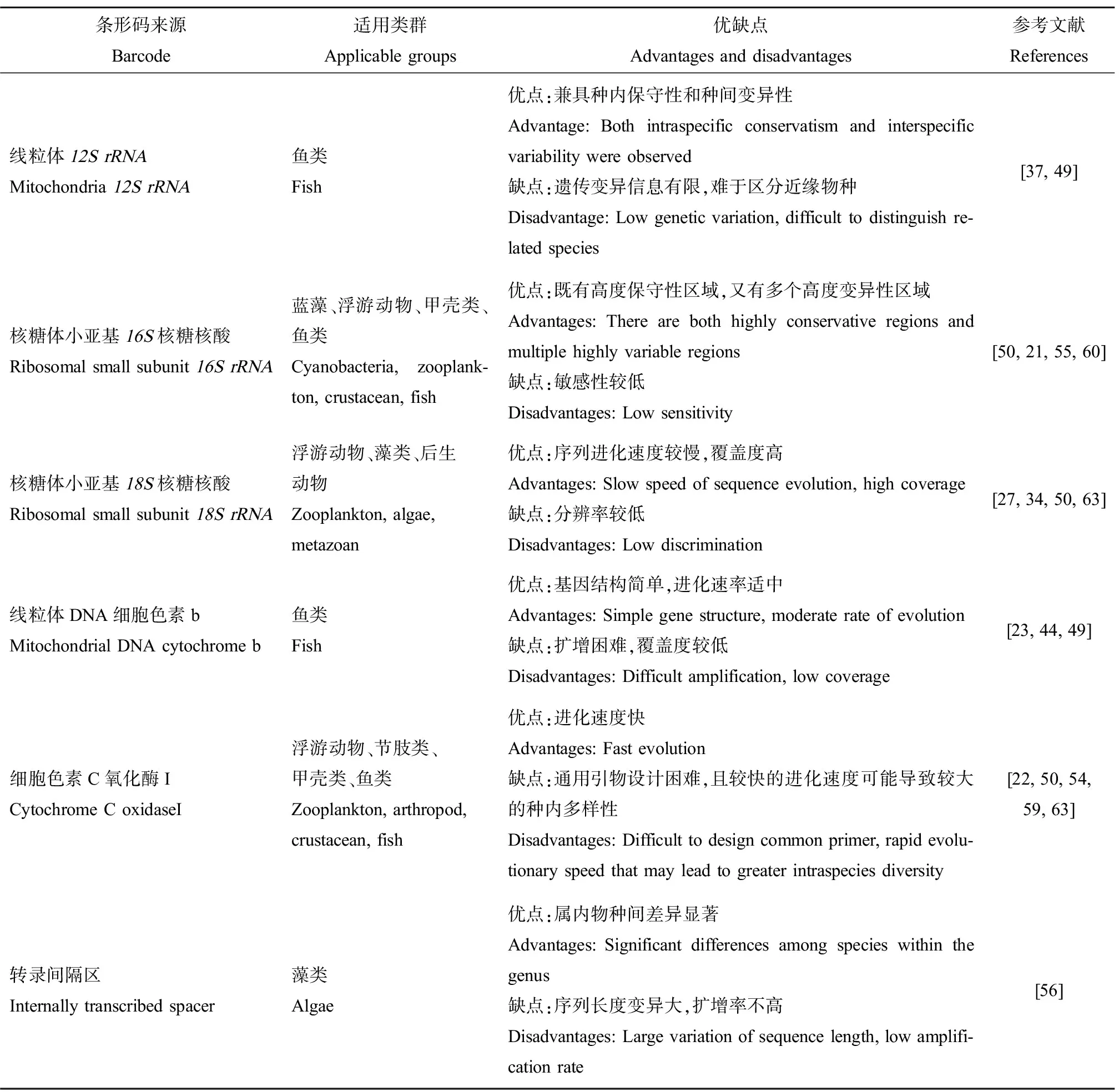
表1 eDNA分析常用标记基因简述Table 1 Description of commonly used marker genes in eDNA studies
针对所选的分子标记基因进行引物设计及之后的PCR扩增过程则相对程序化。目前有一些工具如Primer 3、Primer Express和ecoPrimers,皆可基本满足eDNA研究中引物设计的要求[64-65]。PCR扩增一般分为常规PCR和实时荧光定量PCR。宏条形码分析基于通用引物来扩增多种生物的目标基因,属常规PCR扩增;而为定量评估物种丰度则可利用特异性引物进行qPCR分析[32, 66]。在PCR过程中应严格设置阴性、阳性对照以排除假阴性与假阳性结果,且由于各方面因素的干扰比如eDNA的降解程度、PCR的扩增偏向性、基因的多拷贝等,定量分析评估物种丰度的方法还有待探索完善[67]。
3.3 数据分析
eDNA分析的首要目标是识别环境样品中存在的各类生物。qPCR、ddPCR分析是对特定序列的鉴定和定量,一般针对特定物种。而宏条形码分析中,PCR扩增产物经高通量测序获得大量序列信息,对这些序列进行物种注释及进一步个性化分析极具挑战。数据处理过程主要包括:(1)对原始数据进行预处理以去除低质量的污染序列,比如引物中错配的序列、长度过短的序列及PCR嵌合体等,综合计算平台QIIME、SeqClean和UCHIME等都可以用来去除污染得到高质量优化序列[52, 65]。然而严格的质量修剪虽然可以有效地去除低质量短序列,但过于保守的参数设置可能会删除过多的低丰度序列,从而导致稀有种被丢失、外来种被忽略、生物多样性被低估[52, 68]。(2)序列聚类,即将扩增子序列聚类为操作分类单元(operational taxonomic unit, OTU)。通过距离度量方法计算不同序列之间的相似性,并设定物种间分类阈值,将相似度超过阈值的序列聚类为相同OTU,一个理想的OTU只包含来自一个物种的序列,从而可用OTU来鉴别物种、评价其生物多样性。一般97%的相似性被认为代表了种间和种内之间变异,可被用来区分不同类群,因此,在宏条形码分析中,OTU聚类的分类阈值通常设置为97%[52, 57]。然而分类阈值的设置易使序列信息差异太小的物种被掩盖,导致生物多样性被低估,尤其给序列相似性较高的本地种和外来种鉴别带来干扰[63, 68],在外来入侵生物监测中造成假阴性结果,失去防控的有利时机。近年来,基于熵值在单核苷酸的精度上来区分序列之间差异的处理方法即准确序列变异(exact sequence variant, ESV)被提出,可检出序列高度相似但属于不同种属的物种和生态型,克服物种丢失的弊端,为入侵生物的早期监测和风险预警提供助力[69-71]。(3)物种注释,即通过DNA序列相似性来鉴别环境中存在的物种,其过程主要依靠与参考数据库的比对,参考数据库的大小和质量决定了宏条形码技术分析结果的可靠性[52, 72]。NCBI的Genebank数据库(https://www.ncbi.nlm.nih.gov/genbank/)、生命条形码(BOLD)数据库(http://www.boldsystems.org)和综合席尔瓦(SILVA)数据库(http://www.arb-silva.de/)是目前使用最为广泛、物种序列信息较为完整的参考数据库。然也存在序列注释的物种名称未由分类学家精确鉴定、许多序列未注释物种名称、许多物种无参考序列等问题,导致eDNA宏条形码序列比对注释结果通常存在一定比例未注释的序列。参考数据库的不准确性和不完整性是目前利用eDNA进行生物多样性评估的主要障碍。相信随着研究的深入,参考数据库物种序列的不断丰富和完善,利用宏条形码技术注释物种也将越来越准确。(4)个性化分析。获得物种信息后可以针对研究的具体内容进行个性化分析,比如α、β多样性分析、系统进化分析、物种丰度分析及稀有种、入侵种鉴定等[18, 55, 73]。总之,数据分析的每一步都需要对稀有分类单元的保留或删除、序列数量及计算时间等因素之间进行权衡以确保所得结果的准确性和可靠性。
4 促进eDNA技术应用于水生入侵生物监测的方式(The direction that promotes eDNA technologies application in aquatic invasion species monitoring)
水生入侵生物的监测是有效防控生物入侵的长期性工作,其监测成本、效率至关重要。与传统形态观察和分类法相比,eDNA技术操作流程简化,监测成本大大缩减,且只采集栖息地环境样品,对生物及其生境无损伤,在入侵生物监测研究中具多方面显著优势[18]。但eDNA分析作为一项新兴技术,还存在一些不足,科学家们也正从各个方向努力以弥补缺陷,促进其应用推广。Sepulveda等[74]沿用Daubert标准从5个方面评估了eDNA分析作为一种新兴技术应用于入侵生物监测的可靠性,认为阻碍其发展最重要的因素是eDNA分析本身的不确定性及环境科学家和管理者之间的协调和沟通障碍,研究者和管理者应从eDNA分析流程、管理者行为、风险容忍度评估和公众参与等方面构建决策树以做出正确决策。作为eDNA研究人员,我们可以从以下方面促进其发展与应用。
(1)eDNA获取过程的优化及其标准化操作流程的建立。尽可能完整获取环境中的总DNA对后续全面、真实地反映物种在环境中的分布至关重要,在外来生物的早期监测和预警中亦如此。虽然许多eDNA技术标准已在国际期刊上发表,但是影响eDNA研究的因素很多,比如由于不同生物类群适于栖息的环境不同而导致不同环境介质中留存的DNA种类可能大不相同[35];不同自然地理环境及理化指标的差异导致DNA的降解差异[43]等。为提升eDNA监测结果与实际分布的一致性,更广泛全面地发现生态系统中的各类外来生物,我们可以尝试同时从不同环境介质中提取DNA以获得更加全面的物种分布信息,并应进一步优化样品收集保存技术(比如滤膜材料的更新、保存液的优化)及DNA提取方法;应有效地规避样品间交叉污染、设置阴性对照以提升其准确性和可靠性。(2)不同的数据分析方式也可能产生不同的监测结果,比如分类阈值的不同设置可能会带来假阴性结果,影响外来物种的鉴别检出,应慎重选择和设置生物信息学工具及参数[68],致力于开发性能准确、流程简化的软件工具包以增强不同研究结果的可对比性及准确性。(3)参考数据库的不完整性和不准确性是限制eDNA宏条形码技术进行物种鉴定的主要障碍[72],应齐心协力构建完善的物种DNA条形码参考数据库、建立全球范围的资源信息共享和应用平台以推动该技术的应用与推广。(4)把握技术进步与跨学科合作的机遇。随着测序技术的进一步发展,第三代测序技术即单分子读取技术应运而生[75],不仅进一步降低了测序成本、提高了测序长度和准确性,而且不依赖于PCR扩增,解决了PCR扩增偏向性问题,可极大地提高检出准确度及物种丰度的计算准确性,将大大助力于外来生物的入侵防控;而遥感技术为标准化获得时空尺度的全球性数据提供可能,机器学习可通过数据统计和逻辑推理计算大量数据并科学预测,eDNA宏条形码技术与卫星遥感、机器学习的联合使用可评估生态系统和生物多样性随时间的变化趋势,重构生物网络关系,为其在入侵生物学领域的发展和应用提供更了广阔的空间[73];且随着公众对生态环境保护的日益关注,公民科学团体正越来越多地在生态环境保护中发挥作用,未来可致力于开发商业化的eDNA分析试剂盒并推广于全民参与的生物多样性监测、入侵生物发现检测中,促进生态环境保护。
利用eDNA技术对特定外来生物的入侵机制研究也是未来生物入侵研究的重要方向,其在特定入侵物种的遗传多样性分析、生物量估算、入侵路径以及外来物种在入侵过程中各种影响因素的相互作用等方面极具潜力,可为准确调查入侵生物种群大小、分布状况、扩散模式及对本地生物遗传多样性的影响,制定科学合理的控制策略提供依据。未来可从开发能准确鉴别特定外来物种和相关近缘物种的特异性条形码,探索影响eDNA与生物量线性关系的生物和非生物因素,校正优化eDNA定量方法(比如考虑使用灵敏度较高ddPCR[15])等方面促进eDNA技术在入侵生物治理中的应用。
总之,生物入侵严重威胁入侵地的生态系统平衡,如不及时采取有效措施,越来越多的本地物种将濒临灭绝,生物多样性水平将持续降低。eDNA技术具有低耗、高效、高灵敏度等优势,可高效监测易遭受生物入侵的区域,为入侵生物的预警和防治提供基础数据和技术支持,在水生入侵生物研究中的前景十分广阔。此外,eDNA技术的便捷和可操作性也可为公民科学团体参与入侵生物的快速监测提供可能,将大大提升公众的生物保护兴趣和热情。相信随着分子生物学技术的发展和相关操作流程的标准化,eDNA技术将越来越广泛地运用于入侵生物学、保护生物学等生态学领域当中,为相关监管部门针对具体情况制定相应的防治措施提供参考,助力全球生物多样性保护。

