高效非损伤性扇贝DNA提取方法的建立及效果评价❋
张天琦,刘平平,吕 佳❋❋,马 岑,黄晓文,张嘉俊,王 师,2
(1.中国海洋大学海洋生命学院,海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003;2.青岛海洋科学与技术试点国家实验室,海洋生物学与生物技术功能实验室,山东 青岛 266071)
贝类是海洋生态系统的重要底栖动物类群,同时也是我国海水养殖产业的重要养殖种类。近些年迅速发展起来的分子标记技术,在贝类的遗传学研究以及贝类种质创新和新品种培育等方面发挥了重要作用[1-3]。特别是简化基因组测序技术的出现可高效获得全基因组范围的标记信息[4-7],使实现贝类全基因组选育成为可能[8]。在贝类遗传学研究中,有时需要在保持贝类活体状态的情况下开展,如对珍稀濒危贝类的保护遗传学研究。此外在贝类分子育种实践中,常常需要在不影响扇贝生存或正常生理状态的前提下,对大规模育种群体进行基因组DNA提取,用于后续基因型测定分析,因此开发高效、简便快速的非损伤性DNA提取方法对贝类全基因组选育的开展以及珍稀濒危贝类的遗传学研究具有重要的意义。
贝类基因组DNA提取方法大致可以分为以下几类:第一类是以酚氯仿抽提法[9]或商业试剂盒(如E.Z.N.A. Mollusc DNA Kit (Omega Bio-tek)、TIANamp Marine Animals DNA Kit))为代表的DNA提取方法,由于需要较大的入口组织量(大于10 mg),取材通常是致死性的;第二类是损伤性较低的取样方法,例如剪取少量外套膜[10]、剪取鳃丝[11]、抽取血淋巴[12]等方式,虽然不会导致实验对象死亡,但往往会对其造成不同程度的损伤,比如感染、畸形等[13];第三类是以不损伤活体的前提下取样进行DNA提取,例如过滤养殖环境中的海水[14]、收集粪便或擦拭组织用于DNA提取,前两种方式都需要将个体放置于独立的空间进行采样,操作较为繁琐,处理批量样品时核酸提取效率低。Karlsson等[12]在淡水珍珠贻贝中评价了擦拭内脏团的DNA提取效果,提出擦拭内脏团是一种理想的非损伤性的活体取材方式,但核酸的提取需要利用商业化的微量DNA提取试剂盒完成,对于批量样品的DNA提取费用较高。由此可见,以上几类DNA提取方法在大规模育种群体的基因分型检测中都有其应用的局限性,亟需一种在不损伤个体前提下快速廉价的提取贝类基因组DNA的方法,既能满足育种工作的需求,又能为珍稀濒危贝类的遗传学研究提供有效的方法。
本研究旨在提出一种基于组织擦拭和简单裂解相结合的核酸提取方法,在不损伤扇贝活体的情况下实现简便快速、低成本的DNA提取,适用于大规模扇贝样品的应用。实验以虾夷扇贝(Patinopectenyessoensis)、栉孔扇贝(Chlamysfarreri)和海湾扇贝(Argo-pectenirradians)活体为研究对象探索了两种不同的擦拭材料(棉签、滤纸)对不同组织(鳃丝、内脏团、外套膜)擦拭后的DNA提取效果,从DNA产量、纯度、完整度等方面进行几种方法的比较分析,通过SNP分子标记片段的扩增和微卫星标记的分型验证了该DNA提取方法应用于基因分型的可行性。
1 材料与方法
1.1 实验材料
成体虾夷扇贝、栉孔扇贝、海湾扇贝购买于青岛南山海鲜市场,在海水中暂养10 d后取材,期间每天换水2次。
1.2 扇贝的非损伤性DNA提取方法
1.2.1 DNA材料的获取 采用两种擦拭材料分别对3种扇贝的不同组织部位进行取材,每种扇贝各取7只个体实验。具体的擦拭方法为,待扇贝自然张开壳后,轻轻用手指固定,留有一道5~10 mm缝隙,用竹棒棉签或滤纸条伸进缝隙中,轻轻擦拭目标组织表面3次。然后将取材后的棉签或滤纸放入1.5 mL EP管中。具体的取样分组如下所列:
CG组(Cotton_gill):用一只无菌竹棒棉签伸进缝隙中,轻轻擦拭鳃丝表面3次;
CV组(Cotton_viscera):用一只无菌竹棒棉签伸进缝隙中,轻轻擦拭内脏团表面3次;
CM组(Cotton_mantle):用一只无菌竹棒棉签伸进缝隙中,轻轻擦拭外套膜表面3次;
FG组(Filter paper_gill):用一张滤纸条(5 mm×100 mm)(Whatman,英国)伸进缝隙中,轻轻擦拭鳃丝表面3次;
FV组(Filter paper_viscera):用一张滤纸条伸进缝隙中,轻轻擦拭内脏团表面3次;
FM组(Filter paper_mantle):用一张滤纸条伸进缝隙中,轻轻擦拭外套膜表面3次。
1.2.2 细胞裂解与蛋白质消化 向上述EP管中加入100 μL裂解液,裂解液成分包括100 mmol/L NaCl、10 mmol/L Tris-HCl、1 mmol/L EDTA(pH=8.0)、1.0% SDS(生工,上海)和0.1 mg/mL的蛋白酶K(索莱宝,北京),室温下裂解15 min。
1.2.3 DNA分离 裂解结束后,取出EP管中的棉签或滤纸,吸取DNA溶液直接用于分子生物学下游实验或置于-20 ℃保存。
1.3 DNA质量检测
用1.0%琼脂糖凝胶电泳检测DNA的完整度、纯度,电压100 V,电泳30 min;用Colibri紫外分光光度计(Titertek-Berthold,德国)检测DNA浓度、OD260/OD280的比值。使用RNA酶(赛默飞,美国)对DNA溶液中的RNA进行消化,再用EZ-10柱式DNA纯化试剂盒(生工,上海)对其进行纯化,用紫外分光光度计检测纯化后DNA的浓度和纯度。
1.4 虾夷扇贝DNA分子标记扩增与检测
以虾夷扇贝CG组和FG组提取得到的DNA为模板进行分子标记的扩增。选取已报道的带型清晰、稳定性好的SSR引物[15-16]和SNP引物[17](见表1),引物由生工生物工程(上海)股份有限公司合成。PCR反应体系为:DNA模板25 ng,F/R引物(10 μmol/L)各0.2 μL,2×Taq Plus Master Mix 12.5 μL,补水至25 μL。PCR程序为:95 ℃预变性5 min;后进入30个循环,包括95 ℃变性15 s、退火温度下20 s、72 ℃延伸30 s;最后72 ℃延伸5 min。PCR反应结束后,MS1、MS2产物经由QIAquick PCR纯化试剂盒(Qiagen,德国)纯化后用10%丙烯酰胺凝胶电泳检测;SNP1、SNP2产物用1.5%琼脂糖凝胶电泳检测。

表1 虾夷扇贝分子标记核心序列、长度、引物序列及其退火温度
2 结果与分析
2.1 DNA提取质量检测
通过不同的取材方式获取的个体基因组DNA电泳检测结果见图1。所有泳道都可见清晰明显的基因组DNA条带,阴性对照的泳道内无明显的DNA条带。所有方法均能成功的提取基因组DNA,并且样品之间的条带亮度较为均一稳定,尽管有不同程度的降解,但总体而言基因组DNA条带完整度较高。
通过紫外分光光度计检测扇贝基因组DNA的浓度和纯度见表2。在棉签擦拭组中,虾夷扇贝、栉孔扇贝和海湾扇贝分别测得的平均浓度为97.66、97.22和82.18 ng/μL,滤纸擦拭组中虾夷扇贝、栉孔扇贝和海湾扇贝分别测得的平均浓度70.64、 62.86和53.13 ng/μL。在3种扇贝中,2种擦拭材料均能获取得率较高的DNA,在不同样品之间以及不同扇贝中的应用具有较好的可重复性。判断DNA纯度的常用指标是OD260/OD280,纯净的DNA的OD260/OD280比值约为1.8。所有组中OD260/OD280的比值均小于1.8,所提DNA中含有蛋白质污染。由于是直接吸取的裂解液对溶液内的DNA进行检测,并未进行纯化等操作,必然会有蛋白残留于溶液中。通常,如果蛋白残留严重,点样孔中蛋白与部分DNA的结合会出现电泳孔亮度较高的现象,但图1显示点样孔的亮度适中,泳道内DNA条带亮度比较高,说明本方法有较高的核酸提取效率。推测是由于擦拭法获取的组织量有限使得蛋白污染能够有较好的控制。另外,由于裂解液中含有盐离子以及RNA等杂质,通过吸光度计算得到的浓度可能会存在偏差,因此我们对DNA溶液消化RNA后进行纯化,检测DNA浓度与OD260/OD280(见表2)。其中OD260/OD280的比值均在1.8左右,说明无蛋白质残留,OD260/OD230的比值在2.5左右,说明无糖类、盐离子等污染,测定的DNA浓度数值真实可信。与纯化前相比,各组DNA浓度均有不同程度的损失,但与纯化前DNA浓度呈现出相同的趋势。
经过棉签擦拭和滤纸擦拭的扇贝个体在一个月之后都生存状态良好,存活率可以达到100%,说明2种擦拭方式对3种扇贝的生存状态均未产生明显的影响,表明本方法是一种在扇贝中均适用的非损伤性DNA提取方法。
2.2 不同方法提取DNA的结果比较
通过对采样部位的比较显示,在3种扇贝中,不论是滤纸法还是棉签法取样操作,DNA得率由高到低的组织分别是:鳃丝、内脏团和外套膜,这与传统的酚氯仿提取方法具有类似的结果。在虾夷扇贝、栉孔扇贝和海湾扇贝的实验中,擦拭鳃丝的DNA产量均为最高,纯化前分别达到14.364、15.363和13.834 μg,纯化后达到5.560、6.120和5.885 μg(见图2),因此在DNA需求量较大的情况下适宜选取鳃丝作为贝类DNA提取的擦拭部位。对不同采样材料提取的DNA结果比较可以看出(见图2),在DNA的得率上3种扇贝的所有组织中棉签擦拭的产量都要高于滤纸擦拭的方式,DNA得率的检测结果与电泳检测结果吻合,电泳图上可以看出棉签法提取的虾夷扇贝和海湾扇贝DNA有较为明显的泳道拖尾现象,擦拭鳃丝组和内脏团组更为明显,而在得率最低的外套膜组中泳道背景干净。推测是由于棉签法DNA的上样量较大导致的拖尾的现象。通过滤纸法提取DNA的得率整体上较棉签法低,但是在电泳检测结果中可以看出其DNA提取的完整度和纯度上在不同的扇贝和组织中均有较好的表现。
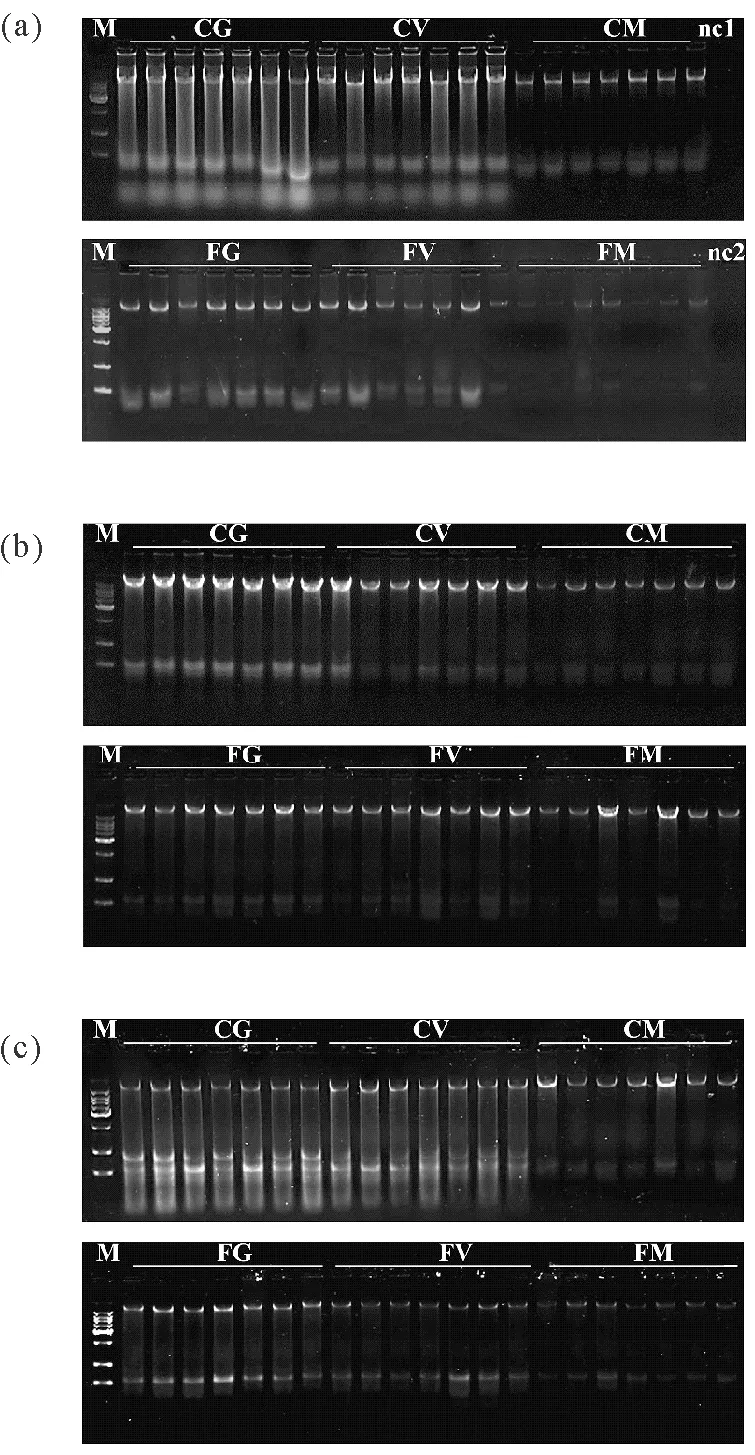
((a) 虾夷扇贝;(b) 栉孔扇贝;(c) 海湾扇贝;M为1 kb Marker;nc1、nc2分别为棉签和滤纸的阴性对照。(a),(b),(c) represented the DNA isolation result of Yesso scallops, Zhikong scallops and Bay scallops respectively. M represented 1 kb DNA Ladder Marker. nc1, nc2 represented the negative control of cotton and filter paper swabbing respectively.)

表2 3种扇贝中不同提取方式的DNA浓度和质量

图2 3种扇贝中不同取材方法获得的DNA纯化后产量
2.3 分子标记PCR产物扩增检测
用棉签擦拭鳃丝(CG)、滤纸擦拭鳃丝(FG)获得的虾夷扇贝DNA直接作为模板,扩增MS1、MS2、SNP1、SNP2共4个DNA分子标记所在的片段。电泳结果显示(见图3),提取的样品DNA均能扩增出条带清晰符合预计片段大小范围的条带。不同个体扩增的微卫星条带表现出一定的多态性,扩增条带长度约为200 bp,符合预期。2个SNP标记引物扩增出100 bp左右的特异性条带,预期PCR产物的大小分别为100和95 bp,电泳结果符合预期目的条带的位置。该片段经Sanger测序验证确认是预先设计的目标区段,并且扩增产物检测无非特异性的杂带,因此该产物可以为HRM高分辨率熔解曲线基因分型系统所用进行SNP位点的筛查和分型。根据琼脂糖凝胶电泳结果可知,两种擦拭方法提取的DNA在扩增的带型以及条带亮度上并无显著差别,在所有个体中都能获得稳定的扩增结果,因此棉签法和滤纸法获取的基因组DNA均可以直接应用于下游基因分型的实验中。本方法获得的DNA溶解于裂解液中,未经纯化等操作,虽含有蛋白酶K、SDS等酶反应抑制剂,但通过稀释的方法制备用于PCR扩增的模板,能够有效地减弱酶抑制剂对下游PCR扩增的影响,获得特异性较好并且稳定的扩增结果。
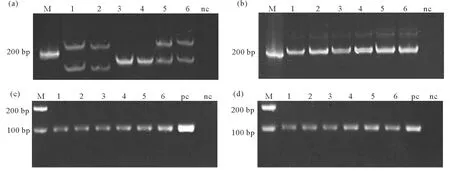
((a) MS1标记;(b) MS2标记;(c) SNP1标记;(d) SNP2标记。M为100 bp Marker;泳道1/3/5和泳道2/4/6分别是以棉签和滤纸擦拭鳃丝方式获取的DNA作为模板的扩增结果;nc为阴性对照。(a),(b),(c),(d) represented the amplified result of Molecular Marker MS1,MS2,SNP1 and SNP2 respectively. M represented 100 bp DNA Ladder Marker. Lane 1/3/5 and lane 2/4/6 was the PCR amplification products using DNA extracted by cotton and filter paper swabbing respectively. nc was negative control using H2O as template.)
2.4 SSR分型结果验证
各选取了6只虾夷扇贝分别验证了棉签擦拭鳃丝法(CG)和滤纸擦拭鳃丝法(FG)提取的DNA用于标记分型的可靠性,以传统的酚氯仿抽提法提取的DNA作为模板检测微卫星标记MS1和MS2的扩增结果作为对照。电泳结果表明,棉签法和滤纸法提取的DNA在扩增效果上并无显著差别,MS1和MS2标记均能获得较好的扩增结果(见图4)。在所有个体中,两种方法的微卫星标记检测结果与对照组具有一致的带型,没有出现基因丢失 (Allelic dropout) 或假等位基因 (False allele)的现象[18]。2种擦拭方法裂解后获得的DNA模板均能够用于基因型检测实验,获得的个体分型结果准确可靠。

((a) CG法扩增标记MS1;(b) FG法扩增标记MS1;(c) CG法扩增标记MS2;(d) FG法扩增标记MS2。M为100 bp Marker;nc为阴性对照;编号相同的泳道为同一个个体的两种方法的检测结果,左侧泳道为擦拭法,右侧泳道为酚氯仿抽提法泳道。(a),(b) represented the amplified result of Molecular Marker MS1 using the DNA extracted by CG and FG method respectively. (c),(d) represented the amplified result of Molecular Marker MS2 using the DNA extracted by CG and FG method respectively. M represented 100 bp DNA Ladder Marker. nc was negative control using H2O as template. Each individual was amplified using the DNA extracted by cotton/filter swabbing (Lane left) and phenol-chloroform (right).)
3 讨论
随着测序技术的发展,越来越多的高效简便、低成本的基因分型技术可供遗传育种研究人员使用,成为贝类分子标记辅助育种和珍稀贝类遗传学研究中应用的重要的技术手段,但这类研究开展的前提是需要在不损伤活体的前提下批量获取样品的基因组DNA。贝类提取DNA最为常见的方法是传统的酚氯仿抽提法[9]或利用商业化试剂盒,但这类方法的取材方式往往对实验对象是致死性的。因此许多研究者围绕贝类非损伤性DNA提取方法进行了探索。例如使用注射器吸取太平洋牡蛎体腔液[19]或过滤牡蛎养殖环境中的海水[14]达到非损伤性取材的目的,但是这类取样方式操作繁琐,大批量样品的取样效率低;张凤梅等[20]研究提出了一种适用于双壳类软体动物壳基因组DNA的提取方法,将贝壳打磨成粉末后裂解后直接沉淀DNA获得,该方法更适用于难以获得活体样本的濒危物种或贝壳标本等;Karlsson等[12]在淡水珍珠贻贝中评价了4种取样方式(抽取血淋巴、刮取足表面黏液、刮取少量外套膜、擦拭内脏团)的DNA提取效果和分型的准确性,结果表明擦拭内脏团的取材方式相比其他方法DNA得率和分型准确性更高,但该研究中利用商品化试剂盒提取DNA,大规模样品的提取成本较高。针对以上局限性,本研究提出一种擦拭法和直接裂解法相结合的贝类DNA提取策略,在不损伤扇贝活体的前提下以极低的成本短时快速的完成样品的核酸提取,为贝类大规模开展分子标记辅助育种研究提供高效率具有实用价值的技术方法。
研究以三种扇贝(虾夷扇贝、栉孔扇贝、海湾扇贝)为实验对象,比较了两种擦拭材料(棉签和滤纸)擦拭鳃丝、内脏团、外套膜3种组织的DNA提取效果,从DNA浓度、纯度、分子标记的扩增及标记分型的准确性等方面进行了比较分析。采用两种擦拭材料均能成功提取主带清晰完整的基因组DNA,棉签法擦拭整体DNA的得率要高于滤纸法。原因可能是棉签的易操作性使得通过接触获得的组织细胞量更多,而通过滤纸接触的力度相对于棉签要小很多,获得组织细胞量较少。但棉签法由于DNA上样量大的缘故,泳道内拖尾现象较为明显。这一点在虾夷扇贝和海湾扇贝中较为明显,但在栉孔扇贝中,两种采样材料提取的效果相仿。因此后续使用棉签擦拭法时需要考虑减少擦拭的次数。由于滤纸具有易于吸附结合核酸的特性,可以进一步将其放置于清洗液中洗脱,去除蛋白酶K、SDS等酶抑制剂[21]达到纯化的目的。高通量基因分型技术的首要步骤是利用限制性内切酶处理基因组DNA,对DNA纯度的要求较高,因此滤纸法在这类技术的应用中更具优势。另外,滤纸更易于记录编号,方便保存,是现场或者野外检测携带的理想的材料。就不同组织而言,擦拭鳃丝提取的DNA得率最高,然后依次为内脏团和外套膜。其中棉签擦拭鳃丝(CG组)的DNA产量最高,可以用于500次以上的PCR反应,棉签擦拭可以满足DNA需求量较大的分子实验需求。棉签法和滤纸法得到的DNA完全可以满足分子标记的扩增要求,所有个体的扩增结果均一稳定,分型结果准确可靠,均可以应用于基因分型检测。两种擦拭材料各有优势,研究者可以结合下游实验的需求来选择合适的采样材料,如对DNA的需求量大时首选棉签法作为采样方式;如对DNA的质量要求较高或者野外现场采样时,优先选择滤纸擦拭的方法。DNA测试比较结果在虾夷扇贝、栉孔扇贝和海湾扇贝实验中呈现基本一致的规律,表明该方法是一种在扇贝中应用稳定并且具有普适性的方法,在贝类亲子鉴定、分子标记辅助育种以及濒危贝类的遗传学分析中具有广阔的应用前景。
本研究中所建立方法的主要创新之处在于将擦拭组织的棉签或者滤纸直接放置于裂解液中,通过优化裂解液的配方和裂解时间使擦拭获得的组织细胞得到充分裂解。针对下游实验的应用,以稀释的方法获得可用于分子标记扩增的基因组DNA模板,通过稀释倍数的调节可以有效地控制SDS等酶抑制剂对下游PCR反应的抑制影响。所有的实验步骤简便易操作,可以短时内完成一次性上百个样本的组织采集和核酸提取,特别适用于大规模贝类样品的分子标记检测和高通量基因分型。本研究的报道的DNA提取方法与已有的贝类基因组DNA提取方法相比具有以下优势:(1)对研究对象基本没有损伤,满足育种亲贝选育和珍稀贝类遗传学研究的需求;(2)操作简便快速,擦拭组织后只需要一步裂解过程即可完成整个流程,用时不超过20 min,不需要水浴锅、高速离心机、隔离水箱等专门仪器设备,适用于实际生产车间内或野外环境的现场DNA提取;(3)实验成本低,每个样品的提取成本仅需0.17元,远低于酚氯仿抽提法(2.10元)和商业试剂盒(8.19~18.08元),适于大规模样品的DNA提取;(4)实验安全性更高,规避了传统方法中酚、氯仿[22]等有毒有害试剂给实验者带来的吸入性安全隐患,大大提高了DNA提取实验的安全性。
4 结语
本研究报道了一种快速、廉价、非损伤性的贝类核酸提取方法,可以获得产量和纯度较高的DNA,是比较理想的非损伤性的贝类DNA 提取技术,特别适用于大量样本或野外环境下活体扇贝的核酸提取,在贝类亲子鉴定、遗传育种、珍稀濒危贝类遗传资源鉴定及保护等方面具有较大的应用潜力。后续可以探索该方法与高通量基因分型技术的联用将有助于提高大规模育种群体的基因分型效率,为贝类全基因组育种实践工作的开展提供有力的技术支撑。

