PacBio测序分析开菲尔发酵过程中细菌演替
史大伟,邢军,马龙,李安,于红,古丽加马力·艾萨,张瑞
(1.新疆师范大学生命科学学院乌鲁木齐830054;2.新疆大学生命科学与技术学院乌鲁木齐830046)
0 引 言
开菲尔是一种传统的发酵乳饮料,可缓解乳糖不耐受症、胃肠道疾病和高血压等[1-5]。富含的微生物能发酵产生有机酸、抗生素和多种香气,其中细菌数量占比较大,起到较重要的作用,对开菲尔的发酵有较大影响[6-13]。
单分子实时测序技术(Single Mo lecu le R eal-Tim e sequencing,SMR T)是目前正在使用的新的测序技术之一,该新技术不仅能获得更长、更准确的DNA序列,而且与传统的方法一样可靠和准确,能快速、准确地在物种水平上识别微生物[14-15]。为了解开菲尔在发酵前后细菌群落的结构和种群变化动态,本研究在0~96 h发酵时间段采集了15份牧民手工制作的发酵样品中的细菌,为分离鉴定开菲尔功能细菌的研究奠定基础,同时,为开菲尔发酵过程中细菌菌落的结构和种群动态变化提供数据。
1 实 验
1.1 材料
样品取自新疆喀什地区的牧民家中,置于冰盒冷藏待用。发酵牛乳乌鲁木齐市超市购买。
1.2 方法
1.2.1 鲜乳杀菌处理
乌鲁木齐市超市购买西域春牌牛奶作为开菲尔发酵基质,采用85℃热处理20 min,备用。
1.2.2 开菲尔粒制备与活化
本实验室于2018年制备获得的开菲尔粒固态发酵剂(原料采自喀什山区牧民家庭制作奶疙瘩,后经过冰盒冷冻保存至实验室)→粉碎→过40目筛→温水30℃泡洗3次→按质量比1∶5加入30℃水打浆→均质→按1∶10比例接种在25℃已消毒牛乳中→25℃培养12 h→过滤获得开菲尔粒→-4℃冷藏备用。
1.2.3 接种开菲尔粒进行发酵培养
活化后获得的开菲尔粒,分别接种于3个装有灭菌鲜乳培养基的三角瓶中,37℃恒温培养箱密封培养,分别在0、24、48、72、96、120 h从3个发酵瓶中取样三份。标记后-80℃冰箱冷冻保存待测。
1.2.4 pH值和滴定酸度的测定
使用精密pH计测量样品的pH值,每样品3个平行。准确称取5 g开菲尔,加40 m L的蒸馏水,3滴酚酞指示剂,混匀,用标准0.1m o l/L N aOH溶液滴定[16]。
1.2.5 样品总DNA提取
通过PCRBIO Taq DNA聚合酶提取样品总DNA。
引物为:27F(5’-gagagtttgatcctggctcag-3’)
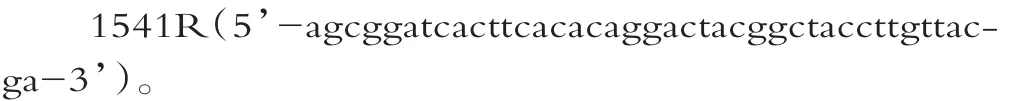
引物包含一组16个核苷酸的条形码。最终反应混合物的体积为50μL。每个样品包含1×PCRBIO反应缓冲液,10 m g模板DN A,每个引物10 m o l和1μg Taq DNA聚合酶。反应条件如下:95℃5 min,然后在95℃30 s,58℃ 45 s和52℃ 1 min进行30个循环,最终延伸52℃5 min。使用Agilent DNA 1000试剂盒和Agilent 2100生物分析仪检查PCR产品的质量。纯化的PCR产物用于通过模板制备试剂盒2.0(Pacific Biosciences Tem p late Prep K it 2.0)构建DNA文库。
1.2.6 样品PacBio SMRT上机测序
建库测序:提取样品总DN A后,根据16S全长引物27F和1541R,合成带有Barcode的特异引物,进行PCR扩增并对其产物进行纯化、定量和均一化形成测序文库(SMRT Bell),建好的文库先进行文库质检,质检合格的文库用PacBio Sequel进行测序。PacBio Sequel下机数据为bam格式,通过sm rtlink分析软件导出CCS文件,根据Barcode序列识别不同样品的数据并转化为fastq格式数据。
1.2.7 序列处理与生物信息学分析
原始数据是根据RS R eadsO finsert.1生成的。Q IIME软件包用于获取高质量序列。为了提取高质量序列,将最小全通量,最小预测准确性,最小插入片段读取长度和最大读取长度分别设置为5、90、1 400和1 800 bp。去除条形码和引物序列以创建数据集。以选择读取长度超过425 bp的序列。然后,将PyNAST和UCLUST用于在序列同一性为100%聚类下比对序列,以获得代表性序列。之后通过UCLUST将序列分类为可操作的分类单位(OTU),其阈值为95%。核糖体数据库用于每个O TU代表序列的分类分配80%的置信度阈值。使用FastT ree中设置的代表性OTU进行分类用于下游分析。α多样性由Shannon-Wiener,Sim pson指数,Chao1指数和稀释性曲线计量评估。加权和非加权计算都应用于主成分分析(principal co-ordinates analysis)以分析微生物群落。使用R软件包3.1.2版和O rigin软件8.5版生成图形。
2 结果与讨论
2.1 开菲尔细菌多样性分析
2.1.1 测序深度评估
为了更全面地揭示开菲尔微生物群落组成及多样性信息,采用了Pac Bio测序技术对不同发酵时间开菲尔样品的微生物群落进行了解析(如图1)。

图1 开菲尔发酵样品稀疏曲线和香农曲线
由图1可知,对于细菌群落,共获得高质量序列61 682条,平均每个样本含有4 072条;根据97%的序列相似度对序列进行OTU划分,共获得1 062个OTU,总体而言,每个开菲尔样品的覆盖率(coverage)均在99%以上,表明测序的深度可以准确反映开菲尔发酵过程细菌群落的真实情况。各发酵样品的稀疏曲线均未进入平台期,因此随着测序量的增加有可能发现新的细菌,另一张图显示随着测序深度的增加香农曲线已进入平台期,说明在该测序深度下,发酵样品中的细菌的多样性可以被充分展示。因此本研究中细菌测序量满足后续生物信息学分析的要求。
2.1.2 开菲尔细菌多样性
细菌群落的Chao1指数在发酵0~96 h呈现先下降后上升再下降后上升的态势,且在0 h达到最大值54.68±0.95。辛普森指数呈现先上升后下降再上升后下降的态势,且在0 h达到最值0.09±0.03。说明开菲尔发酵过程中细菌群落组成前期大于发酵后期,后期发酵细菌多样性较小,见表1。
2.1.3 开菲尔细菌菌群构成
在采样的开菲尔中鉴定出2个细菌门,主要门为厚壁菌门(Firmicutes)变形菌门(Proteobacteria)。在发酵过程中厚壁菌门呈现逐渐增加的趋势。以下为0~96 h相对丰度数据(4.37%、62.80%、59.01%、74.58%、85.69%),在发酵0~96 h之间96 h达到最值。变形菌门呈现逐渐下降的趋势,为0~96 h相对丰度数据(91.74%、37.20%、40.96%、25.42%、4.30%),在发酵0 h处于相对最大值。

表1 开菲尔发酵样品多样性指数表
开菲尔发酵样品中鉴定了30个属,相对丰度大于1%属(图2),在发酵0 h相对丰度大于1%的菌属为:肠杆菌属(Enterobacter)8%,蛭弧菌属(Bdellovibrio)15%,嗜甲基菌属(Methylophilus)14%,鞘氨醇单胞菌属(Sphingomonas)12%,罗尔斯顿菌属(Ralstonia)9%,弯曲杆菌属(Curvibacter)7%,食酸菌属(Acidovora x)7%,苯细菌属(Phenylobacterium)5%。在发酵24 h仅有两种菌属芽孢杆菌属(Bacillus)61%和肠杆菌属(Enterobacter)37%。在发酵48 h有4种菌属气单胞菌属(Aeromonas)40%,嗜热杆菌属(Exiguobacterium)42%,链球菌属(Streptococcus)14%。72 h有4种菌属芽孢杆菌属(Bacillus)73%,肠杆菌属(Enterobacter)7%,气单胞菌属(Aeromonas)18%。96 h有3种芽孢杆菌属(Bacillus)64%,肠杆菌属(Enterobacter)8%,链球菌属(Streptococcus)22%。

图2 发酵样品细菌属组成的柱状
图2中,KSX 001-003为喀什开菲尔发酵0 h样品;KSX 241-243为喀什开菲尔发酵24 h样品;KSX 481-483为喀什开菲尔发酵48 h样品;KSX 721-723为喀什开菲尔发酵72 h样品;KSX 961-963为喀什开菲尔发酵96 h样品。
在发酵过程中芽孢杆菌属呈现先逐渐增加的趋势,在72 h相对丰度达到最大值73%(0~96 h发酵过程中的变化趋势为3%,61%,0%,73%,64%)、肠杆菌属呈现先增加后减少再增加的趋势,在24 h达到最大值(0~96 h发酵过程中的变化趋势为8%,37%,1%,7%,8%)气单胞菌呈现先增后减的趋势,在48 h达到最大值(0~96 h发酵过程中的变化趋势为1%,0%,40%,18%,0%)、链球菌属呈现先增加后减少再增加的趋势,在96 h达到最大值(0~96 h发酵过程中的变化趋势为0%,1%,14%,2%,22%)。
图3中,0 h为喀什开菲尔发酵0 h样品;24 h为喀什开菲尔发酵24 h样品;48 h为喀什开菲尔发酵48 h样品;72 h为喀什开菲尔发酵72 h样品;96 h为喀什开菲尔发酵96 h样品。由图3可知,在0 h鉴定到6种菌种:炭疽芽孢杆菌(Bacillus.anthracis)2%、肠杆菌(Enterobacter sp)8%、嗜水气单胞菌(Aeromonas.diversa)1%、嗜甲基菌(Methylophilus.sp)14%、鞘氨醇单胞(Sphingomonas.leidyi)12%、细菌(bacterium)9%。在发酵24 h鉴定了3种:炭疽芽孢杆菌(Bacillus.anthracis)61%、肠杆菌(Enterobacter.sp)37%、唾液链球菌(S.oralis.subsp)1%。在发酵48 h鉴定到了4种:肠杆菌(Enterobacter.sp)1%、嗜水气单胞菌(Aeromonas.diversa)40%、微小杆菌(Exiguobacterium.sp)42%、唾液链球菌(S.oralis.subsp)14%。在发酵72 h鉴定到了4种:炭疽芽孢杆菌(Bacillus.anthracis)73%、肠杆菌(Enterobacter.sp)7%、嗜水气单胞菌(Aeromonas.diversa)18%、嗜热链球菌(S.thermophilus)2%。在发酵96 h鉴定到4种炭疽芽孢杆菌(Bacillus.anthracis)64%、肠杆菌(Enterobacter.sp)8%、唾液链球菌(S.oralis.subsp)7%、嗜热链球菌(S.thermophilus)15%。炭疽芽孢杆菌在发酵过程中0~24h呈现增加的趋势,在发酵48 h相对丰度为0,随着发酵的进行逐渐增加,相对丰度在发酵72 h达到最大值。肠杆菌在发酵过程中呈现先增后减的趋势,在0~24 h逐渐增加,肠杆菌在48 h相对丰度也为0,在72~96 h逐渐增加。唾液链球菌呈现先增后减的趋势,在48 h相对丰度达到最大值。嗜热链球菌在发酵过程中呈现增加趋势,在0~48 h为检测到嗜热链球菌,在72~96 h嗜热链球菌逐渐增加,96 h达到最大值。

图3 开菲尔发酵样品0~96 h细菌种组成的柱状
2.2 开菲尔细菌差异性
2.2.1 开菲尔细菌差异性分析
图4为不同发酵时间开菲尔样品细菌群落的PCoA分析,结果显示了开菲尔样品在发酵过程中细菌群落的相似性及差异性。
开菲尔发酵样品分布于3个象限,其中发酵0 h(刚开始发酵的开菲尔)的样品单独位于第2象限,表明0 h样品中细菌群落与24~96 h样品细菌群落差异较大。发酵24、48 h的样品位于第4象限。发酵72、96 h的开菲尔样品同时分布于第4象限。不同发酵时间样品之间存在差异性,分布结果同多样性指数相符合。由此可以分为4个阶段:(1)发酵0 h,细菌多样性最高,(2)发酵24 h,细菌多样性开始下降,细菌结构迅速调整;(3)发酵48 h细菌多样性增加。(4)发酵72~96 h,细菌多样性变化较小,群落结构趋于稳定。使用PCoA分析了细菌群落差异性,结果显示不同发酵时间开菲尔样品之间细菌群落组成有明显差异。

图4 发酵样品PCoA图
2.2.2 开菲尔细菌与环境因子相关性
在开菲尔发酵过程中,物种与环境因子相关性可为两类,一类是pH有关,与发酵过程中大多数物种呈正相关关系;另外一类Lactic(滴定酸度),与发酵过程中大多数物种呈负相关关系,如图5。

图5 发酵样品细菌与环境因子关系
由图5可知,其中嗜热杆菌属(Exiguobacterium)、棒杆菌属(Caulobacter)、气单胞菌属(Aeromonas)、葡萄球菌属(Streptococcus)、杆菌属(Thermomonas)与pH呈正相关;芽孢杆菌属(Bacillus)、链球菌属(Streptococcus)、肠杆菌属(Enterobacter)与pH呈负相关。气单胞菌属(Aeromonas)、细菌灭弧菌属(Bacteriovorax)与滴定酸度(Lactic)呈正相关,鞘氨醇属(Sphingobium)、罗尔斯顿菌属(Ralstonia)、弯曲杆菌属(Curvibacter)、嗜甲基菌属(Methylophilus)与滴定酸度(Lactic)呈负相关。
3 结果与讨论
开菲尔发酵过程中细菌群落对于开菲尔制品的质量具有重要意义。目前许多生物学方法已被用来表征开菲尔发酵过程中的细菌群落[17-18]。在本研究中,采用PacBio SMRT测序共检测到2个门、30个属、8个种的细菌,其中优势菌门为厚壁菌门和变形菌门。其中优势菌属为:芽孢杆菌属、肠杆菌属、气单胞菌、链球菌属。鉴定的种为:炭疽芽孢杆菌(Bacillus.anthracis)、嗜甲基菌(Methylophilus.sp)、鞘氨醇单胞菌(Sphingomonas.leidyi)肠杆菌(Enterobacter.sp)、嗜水气单胞菌(Aeromonas.diversa)、微小杆菌(Exiguobacterium.sp)唾液链球菌(S.oralis.subsp)、嗜热链球菌(S.thermophilus)。与其他的测序分析开菲尔相比揭示了新的种[19]。
Gesudu等[20]通过单分子实时测序技术分析库米斯(一种发酵乳制品)细菌群落中演替的动态过程表明,瑞士乳杆菌从0到9 h丰度增加,在9 h达到高峰,然后下降。相比之下,粪肠球菌,杜兰肠球菌和卡塞尔肠球菌在整个发酵过程中逐渐增加,并在24 h后达到最大值。刘文俊等[21]通过PacBio三代测序技术对传统酸马奶进行研究,鉴定到种为:瑞士乳杆菌、乳酸乳球菌、副乳链球菌、棉子糖乳球菌、肠膜阴串珠球菌和产马乳酒乳杆菌等,与本研究有差异。开菲尔在发酵前期存在大量的细菌,随着发酵的进行细菌种类逐渐减少并趋于稳定。Bourrie BC等[22]研究发酵开菲尔中的微生物发现:主要由厚壁菌门和变形菌门组成,属水平则主要由乳酸菌属和醋酸杆菌属组成,其次是芽孢杆菌属和链球菌属,种水平除了比较常见的嗜酸乳杆菌、乳酸乳球菌、德氏乳杆菌、植物乳杆菌、嗜热链球菌、乳酪链球菌、双歧杆菌、醋酸酐菌、酿酒酵母菌、白色念珠菌、干酪乳杆菌和卷曲乳杆菌外,还有特异的开菲尔乳杆菌、马乳酒样乳杆菌、开菲尔念珠菌等。贾宇声等[23]鉴定出的主要细菌是副氏乳杆菌,其次是醋酸杆菌,开菲乳杆菌和乳酸乳球菌,与本研究有差异。有研究报道,发酵剂培养类型、贮藏期和哺乳动物物种以及温度和pH的变化对发酵乳中微生物都有显著影响[24-25]。

