IRF4、ELMO1、CLIP4和MSC启动子甲基化水平在胃癌早期筛查中的应用价值分析
谭玉娥 刘鑫
胃癌是我国最常见的恶性肿瘤之一,严重威胁人类健康。在早期发现胃癌通常是可治愈的,其5年生存率>90%,而晚期胃癌的预后仍然较差[1]。胃癌筛查是发现早期胃癌的主要手段,其中内镜检查,由于具有较高的准确性,被广泛应用于胃癌的早期诊断;此外放大内窥镜加上窄带成像(NBI)在早期胃癌的诊断中的应用,进一步提高了诊断的特异性、敏感性和准确性[2]。但内镜检查需依赖内镜医师的观察技能,具有一定局限性,同时内镜检查存在5%~19%的假阴性率,也容易发生胃癌的漏诊[3]。目前一些血清生物标志物被用于早期胃癌的筛选,例如糖类抗原CA19-9 和癌胚抗原CEA,然而,这些肿瘤标志物的敏感度和特异性仍然较低[4,5]。因此,迫切需要鉴定新的生物标志物以进行更为准确的胃癌的早期诊断。已有文献报道指出,一些肿瘤抑制基因的启动子甲基化过高与胃癌易感性相关[6],与胃癌患者的正常组织相比,许多基因在癌变组织中甲基化[7,8]。目前结合全基因组、表观基因组和基因特异性DNA甲基化分析其在胃癌早期诊断中的应用仍鲜有报道。因此本研究结合总体、表观基因组和基因特异性DNA甲基化分析进行第一阶段生物标记物开发研究,以检验基因特异性启动子甲基化生物标记物以及总体DNA甲基化指数(GDMI)是否能够在内镜活检中将胃癌患者与对照组区别开来,为胃癌早期筛查提供新的思路。
1 资料与方法
1.1 一般资料 研究选取2014至2018年我院358例高风险胃癌患者,所有患者接受了内镜诊断和活检。胃炎对照组的入选标准为患者出现十二指肠胃炎症状,入选标准为根据胃癌的临床诊断,这2个标准均由病理学家确定。其中,282例患者的DNA样本可以进行进一步分析。将纳入研究的参与者随机分为3组:发现组1用于GDMI分析;发现组2用于表观全基因组和基因特异性DNA甲基化分析;验证组用于GDMI和基因特异性启动子DNA甲基化分析。研究中所有患者获得知情同意书,且该研究通过本院伦理委员会审批通过。共有282例患者,符合入选标准,纳入本研究,平均年龄(62.5±14.8)岁。发现组和验证组患者年龄、性别比、临床表现均差异无统计学意义(P>0.05)。150例胃炎患者纳入对照组,其中男92例,女52例;平均年龄(53.4±12.3)岁;幽门螺杆菌阳性81例,发生化生34例。见表1。

表1 患者基线临床特征
1.2 方法
1.2.1 组织样本和DNA提取:活检组织从患者的癌变处和对照组的胃窦处获得,保存于-70℃。胃黏膜组织采用10%甲醛缓冲液固定,石蜡包埋,显微镜下进行组织学检查。使用Sydney系统对苏木精和伊红染色的组织切片进行评分(Lash,2013年),采用PAS染色显示肠化生的活检。用沃辛星银染色法鉴别幽门螺杆菌病变。本研究所用肿瘤组织经组织病理学分类为胃腺癌组织。使用qAMP DNA迷你试剂盒(德国QIAGEN)从冷冻组织样本中提取DNA,保存于-20℃。
1.2.2 总体DNA甲基化分析:使用MDQ1,Imprint®甲基化DNA定量试剂盒(Sigma,美国),通过酶联免疫吸附测定法测定总体DNA甲基化水平。并使用450 nm(A450)处吸光度读数的平均值进行计算。每个样本的GDMI按照以下公式计算:(A450av样本-A450av空白)/(A450av对照-A450av空白)×100%。
1.2.3 表观基因组DNA甲基化分析:采用人甲基化450K DNA 微珠芯片阵列进行无偏差表观全基因组DNA甲基化分析。亚硫酸氢盐外延试剂盒(德国QIAGEN)进行亚硫酸氢盐修饰基因组DNA(2 μg)。将亚硫酸氢盐转化的DNA杂交到人甲基化450K DNA微珠芯片阵列,以识别样本中的不同甲基化区域(DMRs)。为了验证这些结果,我们进行了一个无偏差的表观全基因组DNA甲基化分析,以确定胃癌样本(n=298)中的DMRs,这些样本来自TCGA和胃炎对照组(n=23)。用Illuminaio包将数据导入R。对于数据归一化,使用minfi包应用noob背景减法和染色偏差校正,然后进行归一化和使用minfi中的bump-hunting方法识别病例和对照组间的DMRs。
1.2.4 基因特异性DNA甲基化分析:从表观全基因组分析得到的DMR列表(P<0.05)中选择了4个在DMR窗口中变异较大且CPGs数量最多的基因:interfer调节因子4(IRF4);编码吞噬和细胞运动蛋白1(ELMO1);含帽-甘氨酸结构域的连接蛋白家族成员4(CLIP4);以及编码肌蛋白(MSC)。根据前面描述的方法[9],设计了引物和探针,用荧光定量甲基化特异性PCR(QMSP)定量这4个基因的启动子甲基化。
1.2.5 生物标志物开发流程:采用总体DNA甲基化分析法建立总体DNA甲基化指数(GDMI)。表观全基因组阵列被用来鉴定癌症中不同甲基化的基因特异性启动子区域,可以用甲基化特异性PCR定量。
1.3 统计学分析 应用Stata13(美国得克萨斯州Statacorp)进行分析和管理,采用t检验或方差进行数据对比分析,P<0.05为差异有统计学意义。
2 结果
2.1 总体DNA甲基化和胃癌
2.1.1 在没有发生化生的胃炎患者(平均值=5.69)、发生化生的胃炎患者(平均值=4.68)和胃癌患者(平均值=3.36)中,总体DNA甲基化与胃癌呈负相关(P<0.01)。比较没有化生的胃炎患者和胃癌患者的总体DNA甲基化(P<0.01)。见图1。
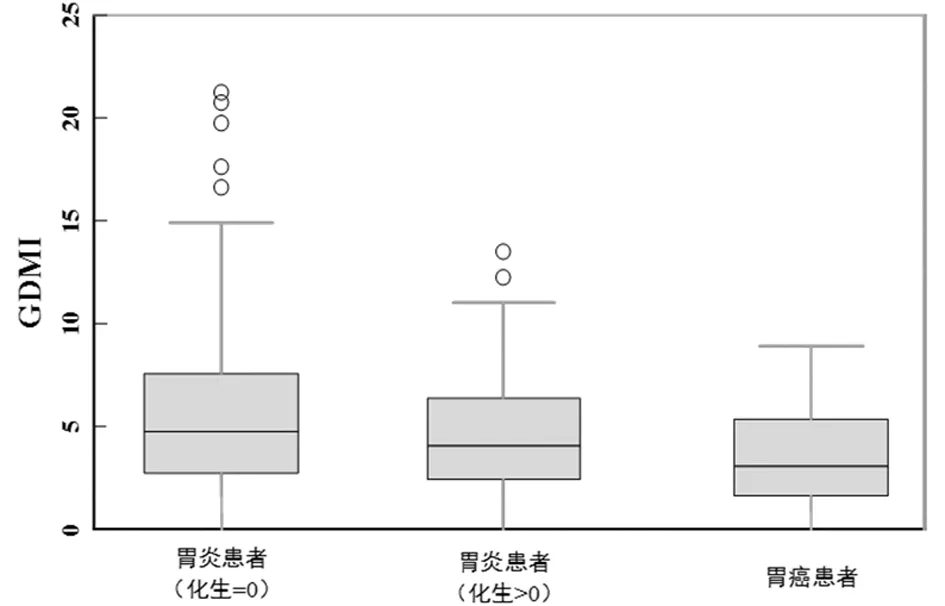
图1 非化生胃炎患者、化生胃炎患者和胃腺癌患者中的GDMI结果
2.1.2 在发现组(n=60)的随机患者中确定了与胃炎相关的总甲基化≥6.5,以评估高风险胃癌临床中的GDMI。在验证组(n=135)中使用该标准来确定胃癌病例,敏感性为84.65%,特异性为31.75%,阴性预测值(NPV)为85.86%,阳性预测值(PPV)为31.12%(P=0.021)。用同样的GDMI标准(临界值)来确定化生 ≥ 10%的病例时,敏感性为86.76%,特异性为35.43%,阴性预测值(NPV)为95.65%,阳性预测值(PPV)为15.86%(P=0.089)。当GDMI阈值提高至9时,对胃癌病例灵敏度和隐形预测值可达到100%。见表2。
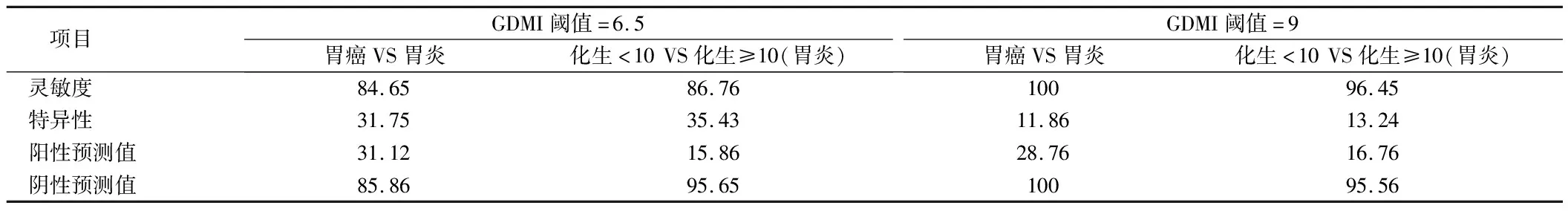
表2 GDMI在内镜活检中对胃炎和胃癌患者检测结果 %
2.2 表观基因组DNA甲基化分析
2.2.1 胃癌和胃炎患者表观基因组甲基化对比分析:对胃炎患者(n=23)和胃癌患者(n=11)进行比较,通过表观全基因组DNA甲基化分析发现了250个具有统计学意义的DMRs(P<0.05)。DMR主要分布在6号染色体(11%),其次是1号染色体(10%)、19号染色体(8%)、2号染色体(7%)、7号染色体(6%)和5号染色体(6%)。其他染色体的DMRs均未超过5%。发现的DRMs主要分布于基因组的以下区域:基因内部215个DMRs(42%);启动子区域91个 DRMs(19%);重叠区域83个DMRs(16%);TSS下游56个DMRs(11%);TSS上游56个DMRs(11%)。每个DMR的单个CpG数量在1~18。大部分(98%)DMR的CpG数低于10。超过10个GpC的DRMs分布于第1外显子的下游区域(15 DMRs)、外显子的内部区域(6 DMRs)、外显子的覆盖区域(3 DMRs)、第1外显子的上游区域(2 DMR)、内含子的内部区域(1 DMR)和2个外显子的重叠区域(1 DMR)中。
2.2.2 在TCGA样本中使用bump-hunting的表观基因组DNA甲基化分析:对TCGA样本中的正常样本(n=12)和癌症样本(n=298)进行表观全基因组DNA甲基化分析,共发现350个具有统计学意义的DMRs(P<0.05)。大多数DMR位于1号染色体(11%),其次是6号染色体(9%)和2号染色体(8%)。DMRs存在于基因组的以下区域:在基因内部67 DMRs(53%);启动子区域30 DMRs(23%);重叠5’区域15DMRs(12%);TSS下游10 DMRs(7%);TSS上游6 DMRs(4%)。每个DMR的单个CpG数量在1~10。大多数低于4(95%)。超过4个CpG的DMR位于第1个外显子的下游区域重叠(9 DMRs),外显子内(5 DMRs),内含子内(3 DMRs),覆盖外显子(1 DMR)。
2.3 与胃癌相关的DMR基因特异性DNA甲基化和TCGA数据集 用定量甲基化特异PCR(quantitative methylation specific PCR,qMSP)确定了135个样本中及TCGA样本集共有的具有统计学意义的DMRs(P<0.05)中有4个启动子甲基化:IRF4、ELMO1、CLIP4和MSC。在散点图的X轴上看到的基因组坐标几乎没有差异,这些散点图通过对我院患者样本和TCGA样本的两个单独的表观全基因组分析说明ELMO1和MSC具有显著性。从无化生胃炎到化生胃炎,再到胃腺癌的组织学进程中,IRF4(P<0.01)、ELMO1(P<0.01)、CLIP4(P<0.01)和MSC(P<0.01)的启动子甲基化与疾病的加重程度密切相关。这些结果表明IRF4、ELMO1、CLIP4和MSC的启动子甲基化可能是胃癌CpG岛甲基化表型(CIMP)的一部分。见图2、3。
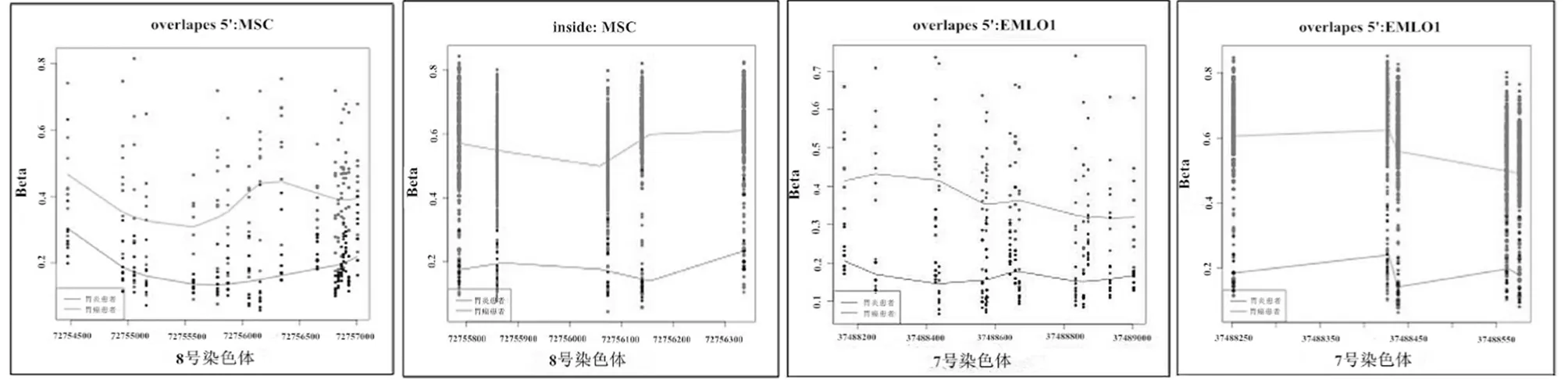
图2 不同样本DMRs分布结果
2.4 具有基因特异性甲基化的预测模型 我们在发现组(n=87)的数据集中使用与胃癌相关的基因特异性甲基化来估计使用IRF4、ELMO1、CLP4或MSC的单独筛选模型来分类验证集中被确定为具有高胃癌基础风险的参与者。在GDMI < 6.5时,正确分类率达到73%。见表3。
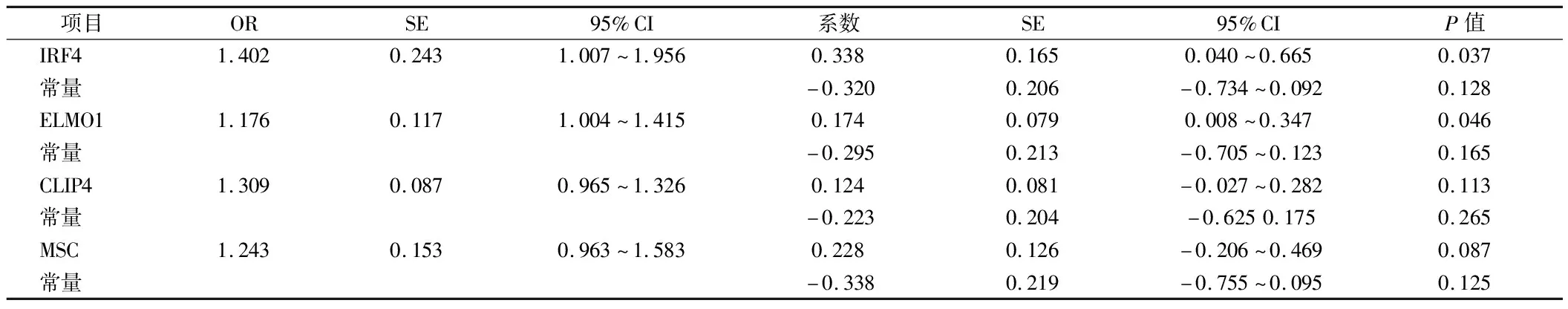
表3 使用IRF4、ELMO1、CLIP4或MSC预测GC风险的模型的优势比和系数
2.5 内镜误诊样本GDMI分析 胃癌患者的总体DNA甲基化水平明显低于胃炎对照组(P=0.002)。对照组平均GDMI为5.7(95% CI,4.93~6.41),胃癌患者平均GDMI为3.7(95% CI,2.99~4.39)。深度炎症与总体DNA低甲基化有关,这是癌症的1个标志。GDMI能够根据炎症深度识别胃炎患者(P=0.01)。患有浅表炎症的胃炎患者的平均GDMI为6.52(95% CI,5.4~7.6),而患有深度炎症的患者的GDMI为4.75(95%CI,3.84~5.67)。此外,在患有深度炎症的胃炎患者中,当比较有肠化生或无肠化生的患者时,我们发现GDMI与此有显著相关性(P=0.03)。与未发生肠化生的胃炎患者(平均值=5.4,95% CI,3.88~6.83)相比,患有深度炎症和肠化生的胃炎患者的总体DNA甲基化水平显著降低(P=0.03)(平均值=3.6,95%CI,2.65~4.63)。见图4。

图3 基因特异性启动子甲基化在不同组患者中的箱线图结果

图4 不同类型患者GDMI对比结果
3 讨论
本研究结合胃癌总体DNA、表观基因组DNA和基因特异性DNA甲基化分析研究内镜活检后的胃癌高风险患者。结果表明,CLIP4、IRF4、ELMO1和MSC的启动子甲基化并且GDMI > 4,是内镜活检中胃癌风险分级中有效分子指标。
CLIP4是T细胞泛素配体家族的成员,抑制T细胞信号传导,CLIP4可以促进T细胞中生长因子的提取诱导的细胞凋亡,并促进T细胞受体、表皮生长因子(EGF)受体和血小板衍生生长因子β受体等各种活化的靶受体的积累[10]。有研究表明胃癌中的CLIP4是甲基化的。IRF属于转录调节因子家族,在各种基因(如干扰素、白介素、MHC Ⅰ/Ⅱ类)、凋亡和分化/成熟的调节中起到重要作用[11]。ELMO1在恶性胶质瘤中起到促进癌细胞迁移和侵袭的作用,ELMO1启动子甲基化作用在人类结直肠癌、肾病和类风湿性关节炎中也有报道[12]。MSC基因编码的肌蛋白作为转录抑制因子,在体内也和E2A蛋白形成异二聚体,抑制E2A蛋白E47在哺乳动物细胞中的激活能力[13]。
本研究探究了GDMI作为胃癌发生的生物标志物的作用。胃癌活检中观察到的低GDMI水平,胃癌上皮发育不良和黏膜内癌组织的LINE-1甲基化水平明显低于相邻的正常胃癌组织,与之前研究结果[14]一致。LINE-1是总体DNA甲基化的一个替代标记,它测量基因组中一组重复元件的甲基化。GDMI比LINE-1更能反映总体DNA甲基化水平,因为它能量化整个人类基因组中的DNA甲基化。本研究进一步表明,GDMI可以鉴别不同程度炎症和化生的胃炎患者。这一有趣的分子差异表明,仅根据胃黏膜上皮细胞的总体DNA甲基化水平,就有可能区分不同程度的胃炎严重程度、肠化生及胃腺癌。
GDMI可以区分炎症的深度,因为患有深度炎症的胃炎患者的GDMI比患有浅表炎症的患者低,这表明在分子水平上,这些患者可能是胃癌最容易发展的患者。我们还发现,当对比肠化生<或>10%的患者,肠化生>10%的患者GDMI较低(P=0.013)。对胃炎患者最初被内镜检查者诊断为癌症,其GDMI值低于胃炎患者,这类患者被内镜检查者和病理学家归类为胃炎患者。此外,这些胃炎患者误诊为癌症患者的内镜下有类似的总体DNA甲基化概况腺癌患者。这些数据支持这样一种观点,即误诊病例具有潜在的致癌分子过程,最有可能恶化为癌症。
目前只有少数研究本身检查了胃癌的总体DNA低甲基化,理由是低甲基化是最早的表观基因组事件,意味着从正常表型向恶性表型的转变。虽然导致癌症甲基化模式整体缺失的确切机制仍有待阐明,但很明显,在胃癌发生的早期阶段,总体DNA低甲基化已经存在,并可能在生物学上发挥各种作用。胃癌病变进展中,全基因组的低甲基化和区域高甲基化已经被证明发生在1个扩大的折叠胃炎,也可能有助于扩散型胃癌的肿瘤发生[15]。总之,这些研究表明,GDMI可能是胃癌发生和发展中重要早期细胞事件的表观基因组反映。我们后续应进一步分析与信号通路中过度及欠表达相关的特定基因位点的甲基化差异,以及这些基因如何与其他类型的癌症相匹配,这些都可能成为精确医学时代新的诊断工具。

