龙眼DlZAT10基因克隆与植物超表达载体构建
李琳 丁峰 潘介春 彭宏祥 张树伟 何新华 徐炯志 黄幸 王金英 王颖 李浩然


摘 要:以龙眼成熟叶片为材料,通过转录组测序及生物信息学分析得到龙眼ZAT10基因全长序列,命名为DlZAT10(GenBank登陆号:MT117769),进一步设计特异引物对其开放阅读框(ORF)全长序列进行克隆和生物信息学分析。DlZAT10基因属于C2H2锌指蛋白家族的C1-2i亚家族,ORF长度为738 bp,编码245个氨基酸,无内含子,有2个典型的C2H2结合位点。该蛋白属于非跨膜亲水蛋白,预测定位于细胞核,存在40个氨基酸磷酸化位点。其二级结构主要由无规则卷曲,以及α-螺旋和β-折叠构成,二级结构与三级结构预测结果高度一致。同源基因进化树分析表明,该基因与柑橘和阿月浑子亲缘关系最近。同时成功构建了植物超表达载体PBI121- DlZAT10,为DlZAT10基因功能的深入研究奠定了基础。
关键词:龙眼;DlZAT10;基因克隆;胁迫
中图分类号:S667.2 文献标识码:A
Abstract: Using the mature leaves of longan as the materials, the full-length sequence of the ZAT10, named DlZAT10 (GenBankID: MT117769) in the longan transcriptome was obtained by transcriptome sequencing and bioinformatics analysis. Furthermore, specific primers were designed for cloning and bioinformatics analysis of the full-length sequence of its open reading frame (ORF). DlZAT10 belonged to the C1-2i subfamily of the C2H2 zinc finger protein family. The ORF sequence was 738 bp in length, encoded 245 amino acids with two typical C2H2 binding sites of C2H2. This protein was non-transmembrane hydrophilic and predicted to be located in the nucleus with 40 amino acid phosphorylation sites. Its secondary structure was mainly composed of random coils, as well as α-helix and β-sheet. The predicted results of the secondary structure and tertiary structure were highly consistent. Phylogenetic tree analysis of the homologous gene indicated that the gene was most closely related to citrus and pistachio. At the same time, the plant overexpression vector pBI121-DlZAT10 was successfully constructed, which would provide basis for the in-depth study of the function of DlZAT10 gene.
Keywords: longan; DlZAT10; gene cloning; stress
DOI: 10.3969/j.issn.1000-2561.2021.01.005
龍眼(Dimocarpus longan Lour.)属于无患子科龙眼属,是我国重要亚热带木本果树之一,中国龙眼种植面积约占世界的59%,李时珍《本草纲目》中有“食品以荔枝为贵,而资益则龙眼为良”,其果实具有益气、安神、滋肤之功效。但生产上龙眼常有“大小年”或隔年开花结果的现象[1],严重影响其产量与经济效益。以往生产经验表明非生物胁迫(如干旱、低温等)能促进龙眼成花,而目前关于龙眼非生物胁迫耐受和诱导成花的分子机制尚未阐明。
锌指蛋白(zinc-finger protein, ZFP)是转录因子的一种,因其具有指状结构且能结合锌而得名[2]。根据锌指蛋白在指状二级结构中与锌离子结合的半胱氨酸和组氨酸残基的数量及顺序的不同,可将其分为九大类:C2H2、C8、C6、C3HC4、C2HC、C2HC5、C4、C4HC3和CCCH(C代表半胱氨酸,H代表组氨酸),其中C2H2型(Cys2 His2)是真核生物基因组中含量最多的锌指蛋白,也是目前研究较为广泛的一类。Takatsuji等[3]在矮牵牛中发现了第一个植物特有的C2H2锌指蛋白,该转录因子在拟南芥中有176个成员,在水稻中有189个成员[4],在烟草中有118个成员[5],植物C2H2型锌指蛋白具有2个独特的结构:一是相邻2个锌指之间的氨基酸序列数目较多且种类变化大;二是与DNA结合处存在一段高度保守的氨基酸序列QALGGH,该序列为植物C2H2锌指蛋白特有[6]。
自然界的植物在生长发育过程中,常遭受干旱、盐碱、高温、霜冻等各种非生物胁迫,其中干旱对植物的影响在所有非生物胁迫中占据首位。转录调节被认为是植物响应和适应胁迫条件最重要的方法之一,在此过程中,转录因子起着关键作用,可通过调控下游胁迫响应基因使植株体内其它相关基因被诱导或抑制,从而对逆境作出反应[7]。其中,C2H2锌指蛋白可通过与DNA、RNA或与蛋白质相互作用以及自身结合在植物形态发生、生长发育、转录激活和逆境胁迫等方面发挥重要作用[8]。如C2H2 型锌指蛋白的C1亚家族在拟南芥不同发育阶段的防御及胁迫响应中起着重要的作用。本研究基于转录组数据并通过RT-PCR法从龙眼中克隆到一个C2H2型锌指蛋白家族基因,命名为DlZAT10,以此转录因子为研究对象,采用生物信息学方法对其进化关系、基因结构、保守结构域、三维结构等进行分析,为今后龙眼ZAT10基因的功能研究打下一定基础,也为非生物胁迫促进龙眼成花的分子调控机制研究做一定的铺垫。
1 材料与方法
1.1 材料
取样地址为广西大学农学院基地龙眼园,土壤为粘性赤红壤土,pH 4.45~5.02,取样树体为长势良好并在成花期进行干旱处理的8年生龙眼树,选择生长良好末次梢顶端下第三张复叶的中部成熟叶作为样品,采后用纸擦去叶片表面灰尘,迅速装袋标记放入液氮内保存。
主要试剂:多糖多酚植物总RNA 提取试剂盒、反转录试剂盒、胶回收试剂盒、质粒提取试剂盒、PrimeSTAR? HS DNA Polymerase、ExTaq?、rTaq?、dNTPs、DNA Marker(2000)、SolutionI、克隆载体PMD18-T、5X In-Fusion HD Enzyme Premix等均购于TaKaRa;大肠杆菌感受态细胞DH5α购于广西南宁国拓生物科技有限公司;植物双元表达载体PBI121由本实验室保存;限制性内切酶XbaI和SmaI购自宝日医生物技术(北京)有限公司;氨苄青霉素(Ampicillin)和卡那霉素(Kanamycin)购自广州生物工程有限公司;其他常规化学试剂均为国产分析纯。在生工生物工程(上海)股份有限公司进行PCR引物合成,在武汉奥科鼎盛生物科技有限公司进行测序,通过基因转录组三代测序以及对比龙眼基因组数据库通过生物信息学分析得到龙眼转录组DlZAT10基因序列全长。
1.2 方法
1.2.1 龙眼叶片总RNA提取及PCR扩增 对研钵、研磨棒及称量勺进行充分消毒灭菌,取50~100 g新鲜样品叶片加液氮充分研磨,提取龙眼叶片中的总RNA,用紫外分光光度计检测所提RNA的浓度和纯度,记录OD260/280、OD260/230的比值和浓度,用琼脂糖凝胶电泳检测其完整性。利用反转录试剂盒合成cDNA,检测正确后?80 ℃保存备用。利用Primer Premier 5.0软件根据前期得到的龙眼转录组DlZAT10基因序列全长设计该基因的上下游引物(表1 c-DlZAT10),长度一般为20~25 bp,选择无发卡结构、无引物二聚体结构、无错配的引物,得到引物后以cDNA为模板通过巢式PCR技术扩增目的基因全长。
1.2.2 DlZAT10基因的克隆与测序 DlZAT10基因PCR产物经 1.2% 的琼脂糖凝胶电泳检测条带清晰,用胶回收试剂盒回收目的基因片段,回收检测后将目的片段与载体PMD18-T在金属浴内恒温16 ℃连接12~24 h,稀释连接液并转化感受态细胞DH5α,摇床扩繁后吸取菌液120 μL涂于含有氨苄青霉素(100 mg/L)的LB(Luria- Bertani)固体培养基上,37 ℃培养15~24 h后挑选阳性克隆菌落,扩大培养后的菌液用通用引物MF47和MR48进行PCR检测,初步检测正确的菌液送生物公司进行测序。
1.2.3 DlZAT10基因的生物信息学分析 使用NCBI網站的BLAST程序结合数据库对DlZAT10进行序列比对,使用在线软件预测蛋白质相对分子量、等电点、亲疏水性、信号肽、跨膜结构、亚细胞定位、结构域、磷酸化位点、二级结构和三级结构等;使用Mega 4.1软件内置的NJ法构建系统进化树,设置Bootstrap值为1000;使用DNAMAN软件进行多序列比对。
1.2.4 DlZAT10基因超表达载体构建 对克隆测序结果正确的基因,利用In-Fusion在线网站设计其超表达引物,分别命名为Oe-DlZAT10-F,Oe-DlZAT10-R(表1中Oe-DlZAT10),通过PCR扩增得到超表达的DlZAT10基因目的片段,利用胶回收试剂盒回收DlZAT10基因目的片段,用5X In-Fusion HD Enzyme Premix把目的片段连接入双酶切质粒PBI121-GFP中(具体方法参见说明书),连接产物转化大肠杆菌感受态细胞DH5α,经扩繁后吸取150 μL涂于添加卡那霉素(100 mg/L)的LB平板上37 ℃倒置培养1~2 d,随后挑选阳性克隆菌落进行扩大培养,对其菌液进行PCR检测。构建的表达载体命名为PBI121-DlZAT10。
2 结果与分析
2.1 DlZAT10基因ORF的克隆
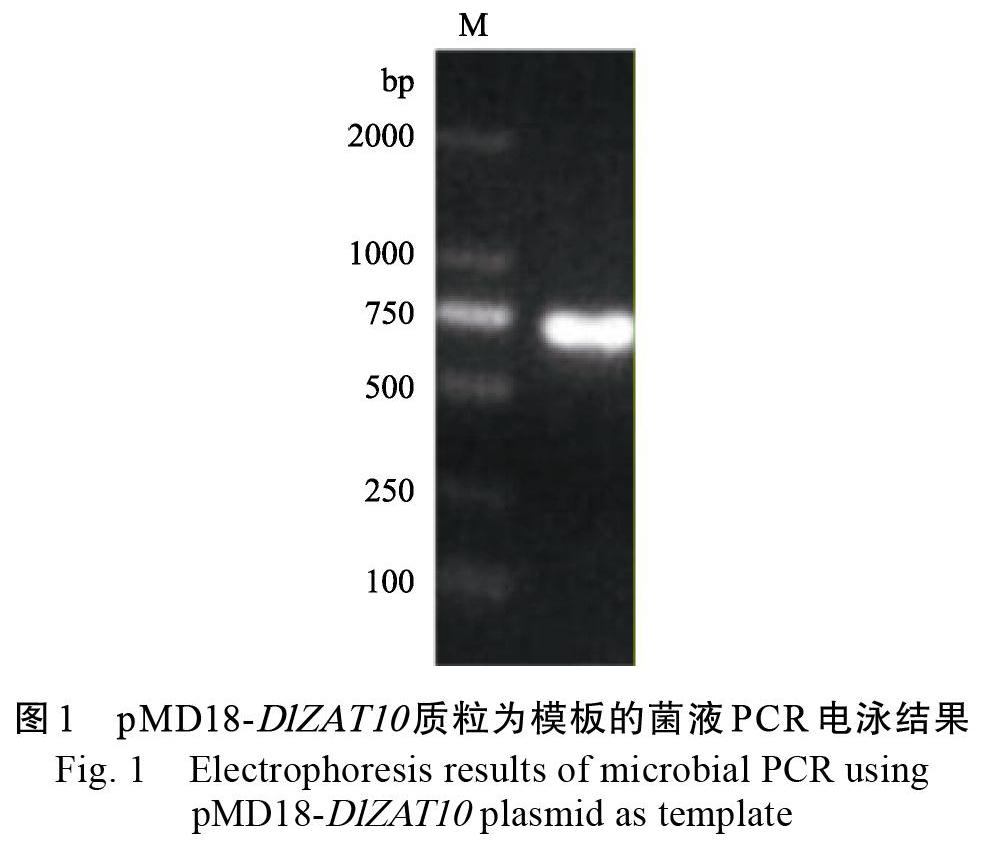
以龙眼cDNA为模板,用设计好的巢式克隆引物进行PCR扩增,经琼脂糖凝胶电泳检测,得到750 bp左右的扩增片段,回收目的片段并与PMD18-T载体连接,菌液PCR检测后得到750 bp左右的目的基因片段(图1),通过对阳性克隆菌落进行扩繁和测序,结果显示DlZAT10基因ORF克隆序列与转录组ORF序列完全一致,该基因ORF全长738 bp,共编码245个氨基酸。
2.2 DlZAT10基因生物信息学分析
2.2.1 DlZAT10编码的蛋白质理化性质预测与分析 利用在线软件ProtParam(https://web.expasy. org/protparam/)预测和分析DlZAT10基因编码的蛋白质理化性质[9],结果显示:该蛋白质分子式为C1144H1778N338O362S11,原子总数为3633,分子量为26411.41 Da;由245个氨基酸组成,带正电荷的残基(Arg+Lys)总数为26,带负电荷的(Asp+Glu)氨基酸残基总数为22,理论等电点pI值为8.62,脂肪指数46.65,总亲水性平均值(GRAVY)为?0.731,属于亲水性蛋白,不稳定指数(II)为76.90,属于不稳定蛋白。
2.2.2 DlZAT10蛋白保守结构域预测与分析 通过在线软件NCBI-CDS(https://www.ncbi.nlm.nih. gov/Structure/cdd/wrpsb.cgi)对DlZAT10蛋白保守结构域预测分析,表明该蛋白属于C2H2超家族,在多肽链的中央附近存在2个结合位点(图2)。通过NCBI-BLAST工具与其他物种氨基酸序列进行比对分析,与预测结果相同,为C2H2型锌指蛋白。
2.2.3 DlZAT10蛋白质信号肽、跨膜结构及亚细胞定位的预测 利用在线分析工具SignalSignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP- 4.1/)对DlZAT10蛋白进行信号肽分析,未发现信号肽,表明该蛋白属于非分泌型蛋白,不经过修饰及转运,直接在细胞内起作用。利用在线软件TM- HMM(http://www.cbs.dtu.dk/services/TMHMM-2.0/)预测DlZAT10蛋白的跨膜螺旋区,参照结果未发现跨膜螺旋区,表明该蛋白为非跨膜蛋白,推测蛋白合成后不经其他途径转运,在细胞内行使催化功能。用植物亚细胞定位在线分析工具PSORT II Prediction(https://psort.hgc. jp/form2.html)进行亚细胞定位分析,结果显示该蛋白定位于细胞核的概率为82.6%,定位于线粒体的概率为8.7%,定位于细胞骨架的概率为4.3%,定位于细胞质的概率为4.3%,由结果推断,DlZAT10蛋白定位于细胞核。
2.2.4 DlZAT10蛋白质磷酸化位点预测 利用在线预测工具NetPhos3.1Server(http://www.cbs.dtu.dk/ services/NetPhos/)分析DlZAT10蛋白的磷酸化位点,结果显示:该蛋白由40个氨基酸磷酸化位点(阈值>0.5)构成,包含16个苏氨酸(Threonine)、22个丝氨酸(Serine)和2个酪氨酸(Tyrosine)磷酸化位点(图3)。推测DlZAT10蛋白活性的调控可能与磷酸化作用有关,且在DlZAT10蛋白中发生磷酸化修饰时以丝氨酸位点磷酸化为主。
2.2.5 DlZAT10蛋白质二级结构与三级结構预测分析 利用在线分析工具PredictProtein(https:// www.predictprotein.org/)对DlZAT10蛋白进行二级结构预测,结果表明该蛋白主要由无规则卷曲构成,共占87.35%,此外α-螺旋占6.94%,β-折叠占5.71%。暴露在蛋白表面的残基占73.06%,其他残基占26.94%。利用SWISS-MODEL Workspace(https://swissmodel.expasy.org/interactive)在线工具预测DlZAT10蛋白三级结构,结果表明,该蛋白主要由无规则卷曲和α-螺旋构成。
2.2.6 DlZAT10蛋白质进化树构建和多重序列比对 将DlZAT10氨基酸序列与NCBI中一致性较高的12种植物序列进行系统进化分析,结果发现,DlZAT10蛋白与CrZAT10(柑橘Citrus reticulata)蛋白和PvZAT10-like(阿月浑子Pistacia vera)蛋白的亲缘关系较近,且和CrZAT10(柑橘Citrus reticulata)、PvZAT10(阿月浑子Pistacia vera)、ZjZAT10(枣Ziziphus jujuba)、CsZAT10(山茶Camellia sinensis)、DzZAT10(榴莲Durio zibethinus)、HbZAT10(巴西橡胶Hevea brasiliensis)、MeZAT10(木薯Manihot esculenta)聚为一类,推测可能在功能上也存在相似性,与AtZAT9-like(拟南芥Arabidopsis thaliana)、JrZAT10-like(核桃Juglans regia)、JcZAT10(麻枫树Jatropha curcas)、RcZAT10(蓖麻Ricinus communis)亲缘关系较远(图4)。
利用DnaMan V6软件进行同源基因的多重序列比对,结果显示该基因含有一个植物锌指蛋白所特有且高度保守的氨基酸序列QALGGH,位于第109~114个氨基酸位置(图5A),属于C2H2家族转录因子,且包含一个大部分锌指蛋白均具有的EAR基序(L/FDLNL/F(X)P),该基序位于蛋白质的C端末(图5B),具有活跃的抑制活性。
2.3 DlZAT10基因的超表达载体构建分析
通过克隆测序鉴定,在目的基因两端引入酶切位点XbaI和SmaI,导入PMD18-T载体,摇菌扩繁提取质粒,然后用XbaI和SmaI双酶切质粒pMD18-T DlZAT10和表达载体质粒PBI121,分别回收目的基因和PBI121大片段,获得具有XbaI和SmaI酶切位点的目的基因和pBI121-GFP载体,采用5X In-Fusion HD Enzyme Premix把目的基因片段连入到PBI121-GFP载体中。连接产物采用菌液PCR鉴定,所用2对引物分别为Npt II-F和Npt II-R以及35S和Oe-DlZAT10-R(NptII为pBI121质粒抗性基因,35S为PBI121质粒启动子)(表1)。35S和Oe-DlZAT10-R的菌液PCR扩增检测获得750 bp左右的目的片段(图6A),Npt II-F和Npt II-R引物的菌液PCR扩增检测获得了750 bp左右的抗性基因片段(图6B),证明目的基因已经插入双元表达载体PBI121-GFP中,超表达载体构建成功。
3 讨论
本研究利用转录组测序及生物信息学分析得到龙眼DlZAT10基因全长序列,通过筛选并结合克隆技术,成功地获得了龙眼DlZAT10基因的ORF全长和超表达载体,并对其序列进行相关生物信息学预测和分析。ZAT10蛋白属于C2H2锌指蛋白家族的C1-2i亚家族,也是该亚家族中研究较为广泛的一类蛋白,相关研究表明,ZAT10在模式植物拟南芥不同发育阶段的防御及胁迫响应中起着重要的作用[10]。
ZAT10最初被鉴定为盐和冷响应蛋白[11],同时在渗透胁迫耐受性中也起着调控作用[12]。Lee等[13]通过荧光筛选法进一步证明了ZAT10在拟南芥植物的冷响应机制中发挥重要的调控作用。通过对比龙眼ZAT10和拟南芥ZAT10蛋白三级结构发现,拟南芥ZAT10蛋白三级结构相对于龙眼ZAT10三级结构较为复杂,含有更多的无规则卷曲和α-螺旋。通过NCBI数据库对比发现:DlZAT10蛋白序列与PvZAT10和CrZAT10的相似度较高,分别为78.23%和72.94%,推测在功能上可能也存在相似性。
ZAT10基因的过表达可提高转基因植物拟南芥对盐、高温、干旱胁迫、渗透和外源H2O2胁迫的耐受性[14-15],研究表明龙眼花芽形成时期适当的低温、干旱以及氧化等胁迫可以诱导龙眼成花[16]。在逆境条件下,锌指蛋白可以作为关键的转录阻遏物,参与植物对不同环境下胁迫的防御和适应反应[17]。ZAT10是ZAT家族中受到广泛研究的成员之一,已被证明可由干旱、盐碱、渗透、氧化胁迫和ABA处理等诱导[18-19]。进一步推测龙眼ZAT10基因可能与龙眼抗逆及成花存在关联。C2H2是一类重要的植物锌指蛋白转录因子,本研究已经完成了对DlZAT10基因的克隆、序列和蛋白的分析以及超表达载体pBI121-DlZAT10的构建,该表达载体构建为DlZAT10基因在拟南芥或龙眼植物的遗传转化进而构建转基因株系提供载体基础,为该基因在龙眼和其他植物中的表达及该基因功能的进一步研究提供参考依据。
参考文献
陈文训. 龙眼生物学特性的研究[J]. 园艺学报, 1963(1): 9-22.
Berg J M, Shi Y. The galvanization of biology: A growing appreciation for the roles of zinc[J]. Science, 1996, 271(5252): 1081-1085.
Takatsuji H, Mori M, Benfey P N, et al. Characterization of a zinc finger DNA-binding protein expressed specifically in Petunia petals and seedlings[J]. The EMBO Journal, 1992, 11(1): 241-249.
Zhang H, Liu Y, Wen F, et al. A novel rice C2H2-type zinc finger protein, ZFP36, is a key player involved in abscisic acid-induced antioxidant defence and oxidative stress tolerance in rice[J]. Journal of Experimental Botany, 2014, 65(20): 5795-5809.
楊明磊, 晁江涛, 王大伟, 等. 烟草C2H2锌指蛋白转录因子家族成员的鉴定与表达分析[J]. 遗传, 2016, 38(4): 337-349.
宋 冰, 洪 洋, 王丕武, 等. 植物C2H2型锌指蛋白的研究进展[J]. 基因组学与应用生物学, 2010, 29(6): 1133-1141.
李 菲, 柳展基, 王立国, 等. 一个新的陆地棉抗逆锌指蛋白基因GhCHP的克隆与分析[J]. 分子植物育种, 2014, 12(6): 1163-1168.
Laity J H, Lee B M, Wright P E. Zinc finger proteins: New insights into structural and functional diversity[J]. Current Opinion in Structural Biology, 2001, 11(1): 39-46.
张树伟, 彭宏祥, 潘介春, 等. ‘禾荔优质晚熟突变体‘GLL-1类黄酮糖基转移酶基因LcUFGT的克隆与表达分析[J]. 广西植物, 2018(11): 46-49.
Mittler R, Kim Y S, Song L, et al. Gain‐and loss‐of‐function mutations in Zat10 enhance the tolerance of plants to abiotic stress[J]. FEBS Letters, 2006, 580(28-29): 6537-6542.
Sakamoto H, Araki T, Meshi T, et al. Expression of a subset of the Arabidopsis Cys2/His2-type zinc-finger protein gene family under water stress[J]. Gene, 2000, 248(1/2): 23-32.
Nguyen X C, Kim S H, Lee K, et al. Identification of a C2H2-type zinc finger transcription factor (ZAT10) from Arabidopsis as a substrate of MAP kinase[J]. Plant Cell Reports, 2012, 31(4): 737-745.
Lee H, Edu G A, Guo Y, et al.. LOS2, a genetic locus required for cold-responsive gene transcription encodes a bi-functional enolase[J]. EMBO (European Molecular Biology Organization) Journal, 2002, 21(11): 2692-2702.
Xu D Q, Huang J, Guo S Q, et al. Overexpression of a TFIIIA‐type zinc finger protein gene ZFP252 enhances drought and salt tolerance in rice (Oryza sativa L.)[J]. FEBS Letters, 2008, 582(7): 1037-1043.
Sakamoto H, Maruyama K, Sakuma Y, et al. Arabidopsis Cys2/His2-type zinc-finger proteins function as transcription repressors under drought, cold, and high-salinity stress conditions[J]. Plant Physiology, 2004, 136(1): 2734-2746.
杨子琴. 龙眼成花与氧化损伤的关系探讨[J]. 热带作物学报, 2015, 36(1): 120-124.
Ciftci-Yilmaz S, Mittler R. The zinc finger network of plants[J]. Cellular and Molecular Life Sciences, 2008, 65(7-8): 1150-1160.
Zhang H, Ni L, Liu Y, et al. The C2H2‐type zinc finger protein ZFP182 is involved in abscisic acid-induced antioxidant defense in rice F[J]. Journal of Integrative Plant Biology, 2012, 54(7): 500-510.
Xie Y, Mao Y, Lai D, et al. H2 enhances Arabidopsis salt tolerance by manipulating ZAT10/12-mediated antioxidant defence and controlling sodium exclusion[J]. PLoS One, 2012, 7(11): e49800.
責任编辑:白 净

