诱变酒酒球菌中抗酸候选基因ATPase和FtsH的表达差异分析及功能验证
文向圆,原雨欣,余东亮,2,刘树文,3,4*
(1 西北农林科技大学葡萄酒学院 陕西杨凌712100 2 秦皇岛金士国际葡萄酒有限公司 河北昌黎066600 3 陕西省葡萄与葡萄酒工程技术研究中心 陕西杨凌712100 4 陕西省合阳葡萄试验示范站 陕西渭南715300)
酒酒球菌(Oenococcus oeni,O.oeni)是从葡萄酒中分离出的革兰氏阳性菌[1-3],通常在酒精发酵后大量繁殖,可以通过启动苹果酸-乳酸发酵(Malolactic fermentation,MLF) 将L-苹果酸转化成L-乳酸[4-5]。MLF 可以降低葡萄酒的酸度,柔和口感,增加葡萄酒的稳定性和香气[6-8]。在葡萄酒苛刻的环境条件下(高酒精度,低pH 值和营养匮乏)[9-11],酒酒球菌因具有良好的抗逆性而成为MLF 的主要乳酸菌种[12-14]。酒酒球菌的生长最适pH 值为4.8,而葡萄酒中的pH 值通常为3.2~3.5,葡萄酒中的低pH 值是限制细菌生长的一个主要因素[15],因此,研究酒酒球菌中的抗酸基因具有十分重要的意义。
在细菌中,FtsH 是一种细胞质膜上的ATP 依赖蛋白酶[16-17]。FtsH 可使膜上的蛋白质脱离,利用ATP 展开这些脱离蛋白并逐步降解它们[18]。在胁迫条件下敲除乳酸菌中FtsH 基因会降低细胞活力[19-21]。FtsH 的沉默使植物乳杆菌(Lactobacillus plantarum,L.plantarum)WSCF1 对高温和高盐的耐受能力及形成细胞膜的能力下降[22]。本研究中的ATPase 基因隶属于FGGY 亚家族的L-核酮糖激酶基因。L-核酮糖激酶主要参与碳水化合物的代谢,在许多细菌的碳源和能量的获得中发挥着重要作用[23-24]。根据这一特性推测其可能与细菌的抗性存在关联。
目前,酒酒球菌中基因操作缺乏有效的分子技术,导致酒酒球菌基因功能的研究一直进展缓慢[25]。有研究成功地将质粒电转化到酒酒球菌中[26];Assad-García 等[27]使用酒精作为膜介质成功实现了外源质粒的导入;Darsonval 等[28]利用反义RNA 技术实现了酒酒球菌中hsp 基因的沉默,然而这样的报道不太多。由于酒酒球菌转化十分困难[7],因此本研究在植物乳杆菌中验证来源于酒酒球菌基因的功能。
本实验室前期通过诱变得到3 对抗酸和酸敏酒酒球菌,并通过重测序比对3 对抗酸和酸敏酒酒球菌中的SNP 突变位点,共定位到181 个可能与抗酸相关的基因[29]。依据文献报道挑取41 个基因进行实时荧光定量PCR(Quantitative real-time PCR,RT-qPCR),将结果分为2 类,一类为抗酸与酸敏菌株表达趋势一致,另一类为抗酸与酸敏菌株表达趋势不一致,从中挑选2 种典型的具有不同表达趋势的基因进行分析,分别为ATPase 基因和FtsH 基因。本研究对ATPase 基因和FtsH 基因进行了表达量差异分析、生物信息学分析及功能验证。分别在最适条件(pH 4.8)和胁迫条件(pH 3.0)的ATB 培养基中提取诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 的RNA,反转录得到cDNA,进行RT-qPCR 分析,得到2 个基因的表达趋势。接着提取2 株诱变菌株的DNA,克隆ATPase 基因和FtsH 基因,进行生物信息分析。最后将两对基因在植物乳杆菌XJ25 中进行功能验证,在胁迫条件(pH 3.2 的MRS 培养基)下测定试验菌株和对照菌株(含空载体)生长能力的差异。
1 材料与方法
1.1 菌株与质粒
诱变酒酒球菌抗酸菌株b1,诱变酒酒球菌酸敏菌株b2[29]和从葡萄酒中分离的植物乳杆菌XJ25 均由本实验室保藏,甲基化缺陷型大肠杆菌(Escherichia coli,E.coli)HST04,Takara-宝日医生物技术(北京)有限公司。
质粒pMG36e 为江南大学惠赠(组成型质粒,3 612 bp,红霉素抗性,启动子为P32)。
1.2 相关试剂与培养基
相关试剂:培养细胞/细菌总RNA 提取试剂盒(DP430)、Universal DNA 纯化回收试剂盒(DP214)、质粒小提试剂盒(DP103),天根生化科技有限公司;PrimeScriptTMRT reagent Kit with gDNA Eraser、TB Green Premix Ex Taq II(Tli RNaseH Plus),Takara-宝日医生物技术有限公司;Phanta Max Super-Fidelity DNA Polymerase,诺唯赞生物科技有限公司;NovoRecRPlus PCR 一步定向克隆试剂盒,近岸蛋白质科技有限公司;XbaI 限制性内切酶、HindIII 限制性内切酶,西安热默尔生物科技有限公司。
培养基:酸性番茄汁培养基(Acid tomato juice broth,ATB)[30]、LB(Luria-Bertani)培养基和MRS 培养基[31]。
1.3 仪器与设备
QuantStudioTM6 Flex System PCR,赛默飞世尔科技有限公司;紫外可见分光光度计,Agilent Technologies;凝胶成像仪,北京赛智科技有限公司;电泳仪,北京君意东方电泳设备有限公司。
1.4 试验方法
1.4.1 诱变酒酒球菌总RNA 提取及RT-qPCR在pH 4.8(最适条件) 和pH 3.0(胁迫条件)的ATB 培养基中,分别培养诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 至指数生长中期,取1 mL 菌液离心收集菌体。根据培养细胞/细菌总RNA 提取试剂盒(DP430)说明书,分别提取4 组处理的总RNA。调整4 组RNA 浓度一致,利用Prime-ScriptTMRT reagent Kit with gDNA Eraser,反转录4 组RNA 为cDNA,存于-20 ℃待用。
为研究pH 4.8 和pH 3.0 条件下,ATPase 和FtsH 在诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2中表达趋势的差异,以ldhD(Gen Bank accession No.AJ831540.1)和gyrA(Gen Bank accession No.AJ831541.1)作为内参基因[32],以上述4 组cDNA为 模板,利用TB Green Premix Ex Taq II(Tli RNaseH Plus)试剂盒,进行RT-qPCR 扩增,反应体系见表1,反应程序为95 ℃30 s,95 ℃5 s,60℃34 s,40 个循环;95 ℃15 s,60 ℃60 s,95 ℃15 s,1 个循环。引物由Primer-Blast 在线设计,本文所有引物均由北京奥科鼎盛生物科技有限公司合成,序列如表2所示。按照2-△ct法分析结果。
1.4.2 基因克隆
1.4.2.1 质粒与目的基因的连接 参照分子克隆实验指南第4 版中的碱裂解法(大量制备),提取大肠杆菌中的质粒pMG36e,使用限制性内切酶XbaI 和HindIII 进行双酶切,得到线性质粒。参照重测序结果,设计扩增引物(表3),从抗酸菌株b1中 扩 增ATPase 和FtsH,命 名 为ATPase1 和FtsH1,从酸敏菌株b2 中扩增ATPase 和FtsH,命名为ATPase2 和FtsH2。ATPase PCR 反应程序为95 ℃3 min,95 ℃30 s,55 ℃30 s,72 ℃2 min,35 个循环;72 ℃,10 min;FtsH PCR 反应程序为:95 ℃3 min,95 ℃30 s,55 ℃0 s,72 ℃2.5 min,35 个循环;72 ℃,10 min。
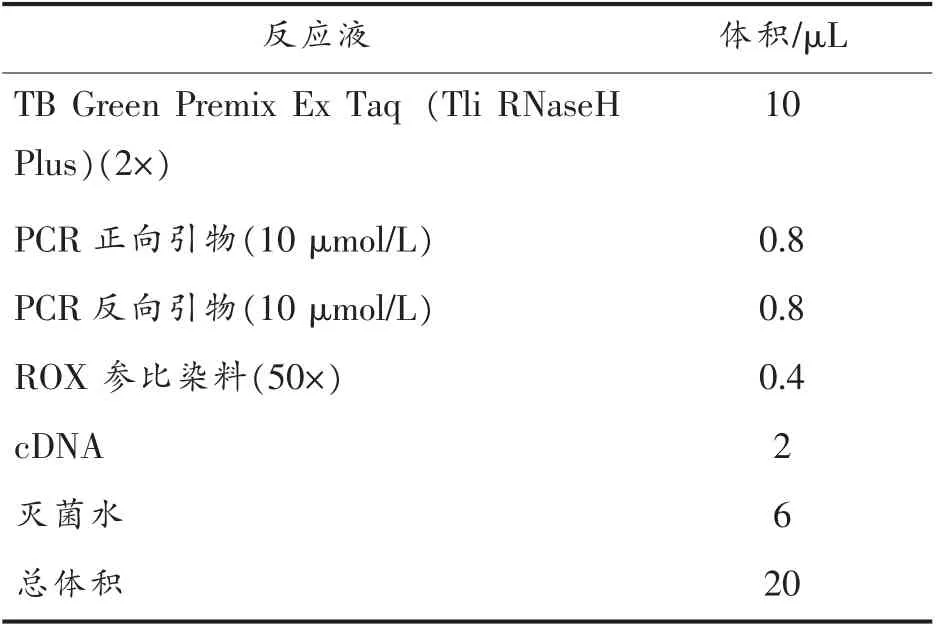
表1 RT-qPCR 反应体系Table 1 RT-qPCR reaction system

表2 RT-qPCR 基因引物序列Table 2 RT-qPCR primer sequence of genes
分别纯化线性质粒和目的基因,利用NovoRec®Plus PCR 一步定向克隆试剂盒,将线性载体和目的基因进行同源重组,同源臂为20 bp左右。

表3 同源重组基因引物序列Table 3 Gene primer sequences of homologous recombination
1.4.2.2 大肠杆菌的转化 有研究证实以甲基化缺陷大肠杆菌HST04 为构建重组质粒的载体菌株,可显著提高重组质粒转入植物乳杆菌的效率[33],因此,本研究以甲基化缺陷型大肠杆菌HST04 为构建重组质粒的载体菌株。
利用CaCl2法制备大肠杆菌HST04 的感受态细胞。100 μL 感受态细胞中加入10 μL 连接产物,利用热激法进行转化。涂布于固体LB 培养基(含红霉素200 μg/mL),37 ℃培养24 h,挑取单菌落进行菌落PCR 鉴定。挑取大肠杆菌HST04 的阳性转化子,分别提取重组质粒pMG36e-ATPase1,pMG36e-ATPase2,pMG36e-FtsH1 和pMG36e-FtsH2。
1.4.3 生物信息学分析 将提取的4 个重组质粒测序,引物为载体上的通用引物,如表4所示。分析目的基因的氨基酸序列。用ClustalX 2 进行序列比对,由EsPript 3.0 作图;在NCBI 和UniProt上查找序列的基本信息;使用TMHMM Server v.2.0 在线预测蛋白质的跨膜结构域;应用MEGA 5.1,以邻接法构建系统发育树,Bootstrap 为1 000。
1.4.4 重组植物乳杆菌的构建及功能验证 根据田雨[34]的方法制作植物乳杆菌XJ25 感受态细胞,将pMG36e-ATPase1,pMG36e-ATPase2,pMG36e-FtsH1,pMG36e-FtsH2 和空载体pMG36e 分别电转到植物乳杆菌XJ25 感受态细胞中,挑单菌落进行PCR 鉴定,挑选阳性转化子保存于-80 ℃。
将保存于-80 ℃的重组植物乳杆菌活化2 次,取指数生长中期菌液,按2%的比例接种于pH 3.2的MRS 培养基中,每隔3 h 测定菌液在波长600 nm 处的吸光度,绘制生长曲线。
1.5 数据统计分析
采用SPSS 分析软件对RT-qPCR 结果进行配对t 检验,分析其显著性。
2 结果与分析
2.1 诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2中ATPase 和FtsH 的相对表达量
在诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2中,为研究ATPase 和FtsH 的表达趋势,在pH 4.8和pH 3.0 的ATB 培养基中,分别测定抗酸菌株b1 和酸敏菌株b2 指数生长中期ATPase 和FtsH的相对表达量。如图1a 所示,以pH 4.8 为对照,胁迫条件下ATPase 的表达量在b1 和b2 中均显著上调(P<0.05);在b1 中上调约13 倍,在b2 中约上调5 倍;如图1b 所示,以pH 4.8 为对照,胁迫条件下FtsH 的表达量在b1 中无显著变化,在b2 中显著上调(P<0.05)。由此可知,胁迫条件下,诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 中ATPase 和FtsH 具有不同的表达趋势。
2.2 诱变酒酒球菌ATPase 和FtsH 基因克隆及生物信息学分析
2.2.1 基因基本信息 为充分了解2 个基因的基本信息,在NCBI 和UniProt 中分别查找这2 个基因的信息,如表5所示。
图1 诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 中ATPase 和FtsH 的相对表达量Fig.1 Relative expression levels of ATPase and FtsH in the mutagenized acid-tolerant O.oeni b1 and acid-sensitive O.oeni b2
2.2.2 基因克隆 从诱变酒酒球菌抗酸菌株b1中扩增ATPase1 和FtsH1,从酸敏菌株b2 中扩增ATPase2 和FtsH2,分别与质粒pMG36e 重组,构建质粒 pMG36e-ATPase1,pMG36e-ATPase2,pMG36e-FtsH1 和pMG36e-FtsH2。使用载体上的通用引物pMG36eseq-F 和pMG36eseq-R 进行扩增,结果如图2所示。空载体的理论扩增长度为406 bp,ATPase 的理论扩增长度为1 988 bp,FtsH的理论扩增长度为2 594 bp。由图2可知,空载体和2 个基因的PCR 条带大小正确。结果初步表明,重组质粒pMG36e-ATPase1,pMG36e-ATPase2,pMG36e-FtsH1 和pMG36e-FtsH2 构建成功。

表5 ATPase 和FtsH 的基本信息Table 5 Basic information of ATPase and FtsH
2.2.3 氨基酸序列比对 为研究ATPase1 和ATPase2 及FtsH1 和FtsH2 编码蛋白的序列差异,将重组质粒测序,比较目的基因的氨基酸序列,应用ClustaL X 2.0 进行序列比对,EsPript 3.0 作图,如图3所示。ATPase 共发生5 处非同义突变,突变位点分别为57(D-V),123(V-E),173(K-E),248(D-G) 和521(I-V);FtsH 共发生2 处非同义突变,分别为150(A-V)和680(D-N)。
2.2.4 功能结构域及跨膜结构域预测 用Pfam在线预测功能结构域,TMHMM Server v.2.0 在线预测跨膜结构域。
ATPase 有2 个功能结构域,分别为:12-241,FGGY-N;274-473,FGGY-C。综合序列比对结果可知,ATPase 在功能结构域内共发生3 处突变,分别为57(D-V)和123(V-E)位于功能结构域FGGY-N 中,173(K-E)位于功能结构域FGGY-C中。由图4a 可知ATPase 无跨膜结构域。
FtsH 有4 个功能结构域,分别为:12-130,FtsH ext;229-362,AAA;384-428,AAA+;442-630,肽键家族M41。综合序列比对结果可知,FtsH在功能结构域内无突变。由图4b 可知FtsH 有2个跨膜结构域,位点分别为13-30 和134-156。综合序列比对结果可知,FtsH 的134-156 跨膜结构域中发生一个突变,突变位点是150(A-V)。
2.2.5 系统发育树构建 为研究ATPase 和FtsH的进化关系,应用MEGA 5.1,以模式菌株大肠杆菌和酿酒酵母(Saccharomyces cerevisiae)中对应的氨基酸序列及与酒酒球菌亲缘关系较近的乳酸菌,构建系统发育树。由图5a 可知,在近源物种中,酒酒球菌中的ATPase 与乳酸乳球菌(Lactococcus lactis)的进化关系最近,其次是肠系膜明串珠菌(Leuconostoc mesenteroides) 和植物乳杆菌,与植物乳杆菌中的序列同源性为55%,其中植物乳杆菌为本研究中功能验证所用的宿主菌。有趣的是,酒酒球菌中的ATPase 与非近源物种鸟肠球菌(Entercoccus avium)的进化关系较近。由图5b 可知酒酒球菌中的FtsH 与酒球菌属(Oenococcus)的菌亲缘关系最近,与植物乳杆菌的序列同源性为60%。由此可知,酒酒球菌中ATPase 和FtsH 序列与植物乳杆菌中的序列存在一定的差异。
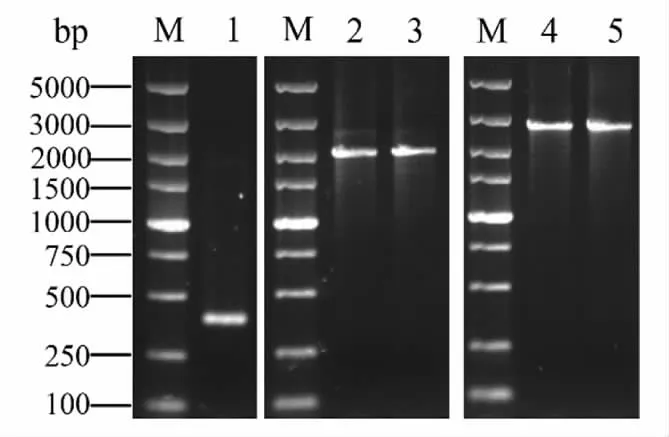
图2 重组质粒的PCR 条带Fig.2 PCR band of recombinant plasmid

图3 ATPase 和FtsH 的氨基酸序列比对结果Fig.3 Amino acid sequence alignment of ATPase and FtsH
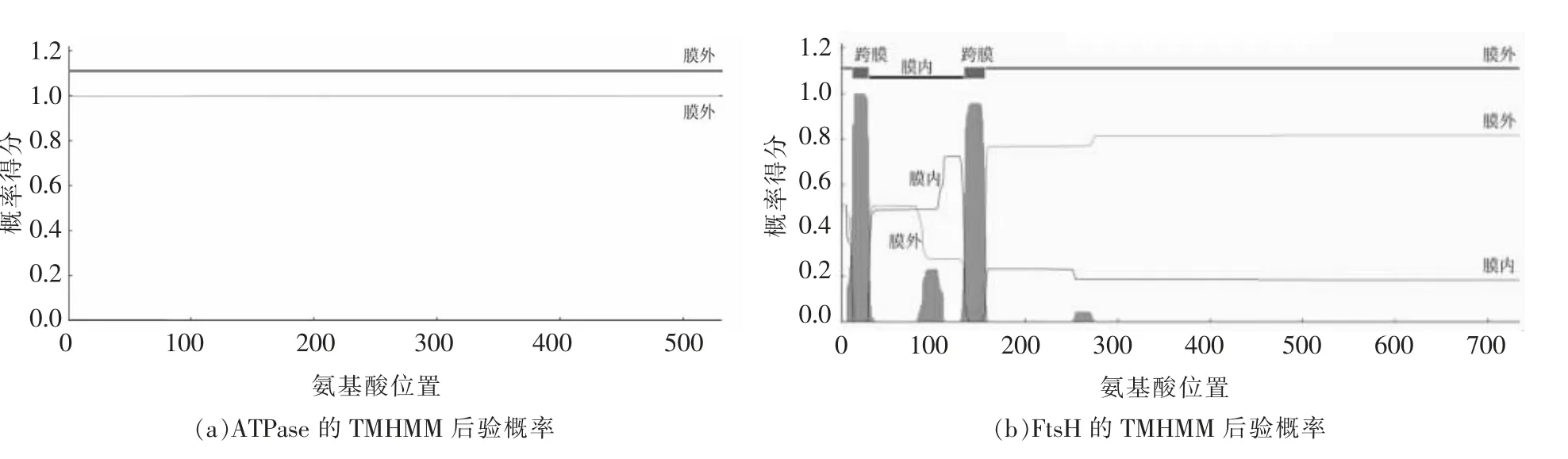
图4 ATPase 和FtsH 的蛋白跨膜结构域预测Fig.4 Protein transmembrane domain prediction of ATPase and FtsH

图5 ATPase 和FtsH 的系统发育树Fig.5 Phylogenetic tree of ATPase and FtsH
2.3 重组植物乳杆菌的构建
为鉴定重组植物乳杆菌是否构建成功,挑取转化子进行菌落PCR 鉴定。如图6所示,对照组pMG36e-XJ25(以下简称con),ATPase1-pMG36e-XJ25 和ATPase2-pMG36e-XJ25(以下简称a1 和a2) 及FstH1-pMG36e-XJ25 和FstH2-pMG36e-XJ25(以下简称f1 和f2)的PCR 扩增条带大小正确,表明5 个重组植物乳杆菌构建成功。
2.4 重组植物乳杆菌胁迫条件下的生长曲线
为研究胁迫条件下a1 和a2 及f1 和f2 与对照(con)生长能力间的差异,在pH 3.2 的MRS 培养基中接种重组植物乳杆菌con,a1,a2,f1 和f2,使其初始OD 值约为0.08,测定其生长曲线,如图7所示。在胁迫条件下,a1 和a2 的生长能力均强于con,a1 与a2 生长能力之间无明显差异;f1 生长能力强于con,而f2 与con 生长能力无明显差异。结果表明,ATPase1 和ATPase2 均可以提高菌株的抗酸性;FtsH1 可以提高菌株的抗酸性,而FtsH2 不能提高菌株的抗酸性。
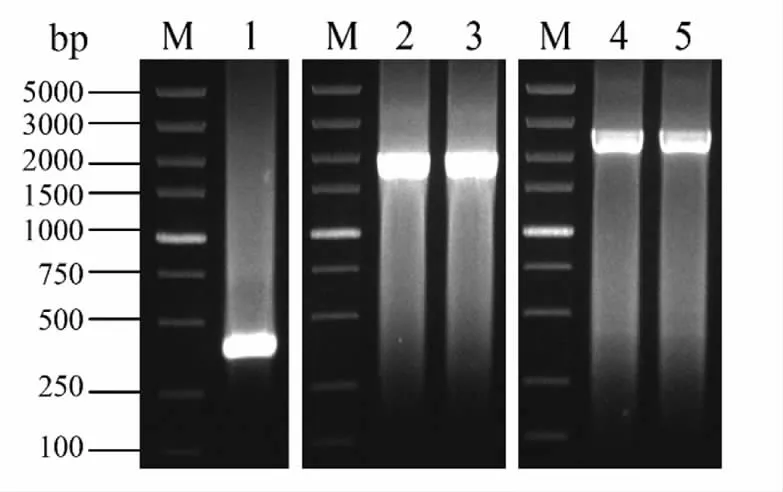
图6 重组植物乳杆菌的鉴定Fig.6 Identification of recombinant L.plantarum

图7 重组植物乳杆菌在pH 3.2 的MRS 培养基中的生长曲线Fig.7 Growth curve of recombinant L.plantarum in MRS medium at pH 3.2
3 结论与讨论
1) 以pH 4.8 为对照,在pH 3.0 的ATB 培养基中,诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 中的ATPase 表达趋势一致,均显著上调。由功能验证结果可知,ATPase 可提高菌株抗酸性,且5个突变位点57(D-V),123(V-E),173(K-E),248(D-G)和521(I-V)未影响该蛋白抗酸性。
ATPase 在UniProt 中被推测为L-核酮糖激酶,属于FGGY 家族,具有磷酸转移酶活性,主要参与碳水化合物代谢过程,而碳水化合物的代谢是细胞获取能量的重要途径。结合ATPase 在b1和b2 中的表达量差异和ATPase 的功能来看,胁迫条件下抗酸菌株b1 获取的能量可能大于酸敏菌株b2,这可能是导致诱变酒酒球菌b1 和b2 生长能力差异的一个原因。由重组植物乳杆菌a1 和a2 生长能力无明显差异推测,ATPase 中的5 处非同义突变不影响该蛋白抗酸性。
2) 以pH 4.8 为对照,在pH 3.0 的ATB 培养基中,诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 中的FtsH 表达趋势不一致,在b1 中无显著变化,在b2 中显著上调。FtsH 可提高菌株抗酸性;突变位点150(A-V)和680(D-N)影响了该蛋白的抗酸性。
FtsH 是一种细胞质膜上的ATP 依赖性蛋白酶[17-18]。由FtsH 在b1 和b2 中的表达量差异及表型差异推测,位于跨膜结构域的150(A-V)突变位点影响了FtsH2 的功能,使其不能正常发挥作用,因此导致重组植物乳杆菌f1 和f2 在胁迫MRS 培养基中的生长差异。同时也正是由于FtsH2 功能的丧失,使得酸敏菌株b2 中相关调节通路得不到反馈,从而试图通过升高FtsH 的表达量来弥补这种不足。
3) 综合2 个基因的表达情况和重组植物乳杆菌的表型差异,在筛选诱变酒酒球菌的抗酸基因时,可以将关注点放在诱变酒酒球菌抗酸菌株b1 和酸敏菌株b2 在胁迫条件下,相对表达量差异较大或表达趋势不同的基因上,这也为后续抗酸基因的筛选奠定了基础。

