植物胼胝质合成酶基因的鉴定和家族成员扩增方式分析
戴明丹 冯珂 张晔 张智研 孙俊玲 张一名


【摘 要】 通过进化生物学方法对胼胝质合成酶(CalS)进行分析,对于获取的基因家族成员应用不同的系统进化方法分析。在该基因家族中获得了六个比较稳定的进化分支,并探究其进化关系和拷贝扩增方式。最后从33个植物物种内鉴定得到了317个CalS成员。分析发现,仅有高等植物(维管植物,Vascular plants)具有完整的CalS基因,其中苔藓植物和蕨类植物均有三种类型的CalS成员,被子植物主要有六种类型的CalS成员。CalS基因的扩增方式以片段重复为主,但也存在串联重复。
【关键词】 胼胝质合成酶;基因家族;进化;拷贝积累形式
Identification of CalloseSynthase Gene and Analysis of Amplification Pattern of Its Family Members in Plants
Dai Mingdan,Feng Ke, Zhang Ye,Zhang Zhiyan,Sun Junling,Zhang Yiming*
(Langfang Normal University, Langfang 065000 ,China)
【Abstract】 Evolutionary biology method is used to analyze the obtained gene family members, and different phylogenetic analysis methods are applied. Finally, six stable evolutionary branches of the gene family were obtained, and their evolutionary relationship and copy amplification mode were explored. In this study, 317 CalS members were identified from 33 plant species. It was found that only higher plants (vascular plants) had complete CalS genes. Moss and ferns had three types of CalS members, and angiosperms had six types of CalS members. The amplification of CalS gene copys was mainly in the form of heavy fragment, but there were also tandem repeats.
【Keywords】CalS;gene family;evolution;amplification of copies
〔中圖分类号〕 S666 〔文献标识码〕 A 〔文章编号〕 1674
0 引言
胼胝质合成酶(Callose Synthase,CalS)是以UDP-G(二磷酸尿苷葡糖)为底物催化合成胼胝质反应中一类重要的酶[1],而胼胝质是广泛存在于细胞中的多糖,其主要成分是β-1,3-葡聚糖,胼胝质的合成和降解涉及植物发育的各种过程、应对多种胁迫[2]和入侵病原的防御等[3],因此,胼胝质合成酶在保障植物进行正常生命活动中具有重要意义。
CalS属于葡聚糖合成酶(Glucan Synthase)家族[4],它是一种分子量较大的蛋白,具有多个跨膜结构域,含有3个保守的功能域Vta1(Vps twenty associated 1)、FKS1(FK506 sensitivity 1)和Glucan Synthase。许多植物都含有这一类基因,这些基因在细胞板形成、胞间连丝调节、筛孔及纤维形成、花粉发育、配子发育、韧皮部运输、气孔形成及抗逆境等过程中均有不同的调节功能。[5]而且CalS在胼胝质壁、胼胝质塞和花粉管壁的合成中起主导作用。[6]
CalS蛋白参与孢子发育是陆生植物具有的一个共同特征,但各个成员之间的进化关系少有报道[7]。目前,关于植物CalS的研究多集中于模式植物拟南芥(Arabidopsis thaliana)且其基因分为两大类,一类有助于细胞分裂和生殖,一类有助于加强结构细胞壁[4]。在葡萄(Vitisvinifera) [3]等植物细胞中,CalS通过合成胼胝质增强了植物对外界的抵抗能力,并且促进了其自身的生长发育以及植物营养物质的转移等多种代谢过程,CalS基因表达升高,胼胝质局部沉积能够明显提升植物应对病原体的攻击能力。[8]
现如今,随着越来越多植物全基因组数据的陆续公布以及新版本基因组的不断完善,我们有机会针对多种植物类群,从更宏观的视角上对CalS基因家族进行研究。本文通过选取不同分类关系的33个植物物种,对其CalS基因家族成员在全基因组水平上进行大规模鉴定和结构研究,并在前人工作的基础上,进一步梳理植物CalS基因家族不同成员的演化关系,并且通过应用系统发生分析、假基因筛选以及基于同义突变速率的分化时间推测等方法对其进化历程进行初步探索,从而为进一步研究植物CalS基因不同亚型的潜在功能提供理论基础。
1 材料与方法
1.1 数据来源
目前各大数据库中,部分类群缺少可靠注释的基因组,因此本研究根据植物分类学进化关系,以及被子植物APG分类系统[9],选取基因组注释较完整,且在分类体系中具有代表性的33个植物物种进行分析。其中所涉基因组数据来源于NCBI Genome(https://www.ncbi.nlm.nih.gov/genome)、Plants ensemble(https://plants.ensembl.org)以及植物JGI数据库phytozome v12.1(https://phytozome.jgi.doe.gov/pz/portal.html)(表1)。
1.2 CalS基因的鉴定
首先,在Pfam[10]数据库(http://pfam.xfam.org)下载CalS蛋白特征结构域的隐马尔可夫模型(HMM, hidden markov model)文件(号码:PF02364)。随后应用HMMER v 3.1[11]在物种全蛋白序列中搜索候选基因。由于CalS基因家族蛋白结构域较大(模型长度819个氨基酸),因此选择连续结构域,序列覆盖率大于80%,并且e值小于1e-200;或者结构域分离,但间隔不超过50个氨基酸,各部分序列覆盖率之和大于80%,且每个部分的e值小于1e-200的蛋白为候选成员。随后,选取每个基因的第一個拷贝提交候选成员到SMART(Simple Modular Architecture Research Tool: http://smart.embl-heidelberg.de)进行验证。所得结果再与背景种子树中建树确认后,作为CalS基因家族成员进入下游分析环节。
1.3 建立系统发生关系
用MEGA7[12]软件构建CalS基因家族成员的系统发生关系,采用蛋白全长序列,使用ClustalW[13]和Muscle[14]两种方法进行比对,以Maximum Likelihood方法(ML)[15]和Neighbor-Joining法(NJ)[16]建树,Bootstrap均为1000。构建系统发生关系通常选择重要分支Bootstrap值更大的方法,对比发现Muscle比对NJ法所得系统发生关系中主要分支的Bootstrap值更大,因此后续均选择此法,并且将所得进化树提交至在线工具itol[17](https://itol.embl.de;v 4.3.2)实现可视化。
1.4 家族成员扩增分析
本研究主要对片段重复(segmental duplication)和串联重复(tandem duplication)[18]两种复制机制进行研究。将基因间隔不超过2000bp [19]的CalS基因家族成员,识别为串联重复基因;对于片段重复,本研究专门针对成员拷贝数在10个以上的物种在Plant Genome Duplication Database (http://chibba.agtec.uga.edu/duplication/)数据库中进行重复片段的搜索,并得到相应的重复片段以及碱基同义替换率(Ks),并按公式“T=Ks/2λ”估算相应基因之间的分离时间[20]。同时用CodeML[21]计算物种内成员间的Ks值。
2 结果与分析
2.1 植物CalS基因家族成员的跨物种分布
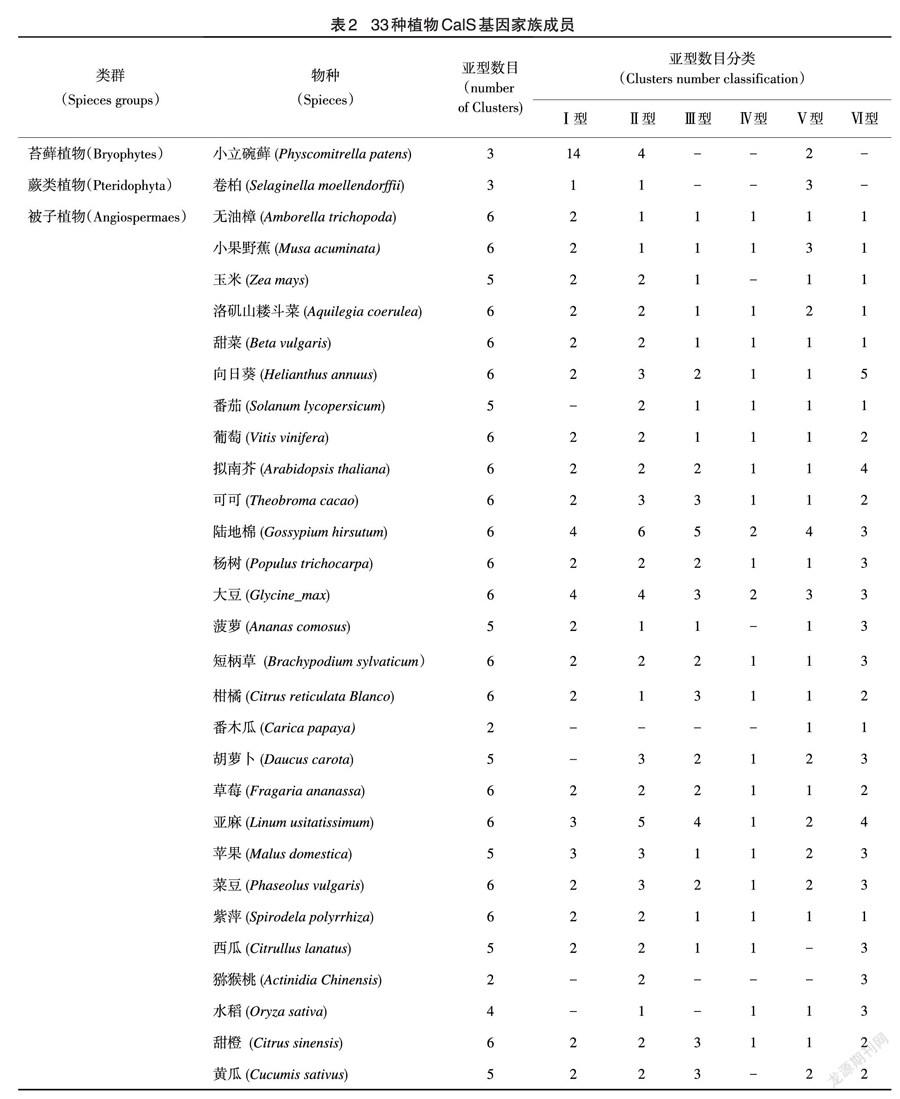
经过对植物界33个物种的全基因组进行搜索和过滤,最终发现了317个较为可靠的CalS基因家族成员,分布于30个植物物种内,藻类植物(红藻:Cyanidioschyzonmerolae;团藻:Volvoxcarteri;衣藻:Chlamydomonasreinhardtii)中未搜索到CalS基因家族成员。从筛选结果中可知,本研究所涉及的所有维管植物中均含有至少1个CalS基因家族成员,但不同类群的成员数量存在明显差异。其中,蕨类植物CalS基因拷贝数比较少,不超过5个。而CalS基因家族成员数量最多的物种则属于被子植物,其中双子叶植物——陆地棉(Gossypiumhirsutum),成员数高达24个,是本研究中CalS基因家族成员最多的物种。在双子叶植物中,大豆(Glycine max)含有19个CalS基因,位居第二。番木瓜(Carica Papaya)仅有两个CalS基因,是本研究中含CalS基因最少的高等植物种类(表2)。
通过对比高等植物的CalS蛋白的亚型分布,最原始的植物无油樟已经含有了六个亚型,而且多个被子植物类群中的个别物种虽有某个亚型成员的缺失,但是CalS不同亚型在物种类群中总体上分布较为平均,说明被子植物的早期就有CalS蛋白六个亚型的分化。一共有19个物种完全具有六个亚型,11个物种不完全具有六个亚型。缺失个别亚型成员的11个物种包括,小立碗藓(Physcomitrella patens)、卷柏(Selaginellamoellendorffii)、番木瓜(Carica papaya)、猕猴桃(ActinidiaChinensis)、玉米(Zea mays)、黄瓜(Cucumissativus)、番茄(Solanumlycopersicum)、胡萝卜(Daucuscarota)、菠萝(Ananascomosus)、水稻(Oryza sativa)、西瓜(Citrullulanatus)。
2.2 植物CalS基因家族成员的结构和系统发育分析
研究发现,不同方法所构建的系统进化树都包括六个稳定的主要分支,但是在不同的系统发育分析中,不同亚型之间的进化关系并不稳定(见图1、2),虽然不能确定各个亚型在进化过程中的从属关系,但结合前人结果可将317个成员分为六个亚型[4]。Ⅱ型CalS基因数量最多,占比为21.5%,Ⅰ型CalS基因数量次之,为21.2%,Ⅵ型CalS基因数量第三,Ⅵ型为20.8%,Ⅲ型为14.8%,Ⅳ型为7.9%,Ⅴ型为13.9%,尤其是被子植物类群部分物种含有较多的Ⅰ型和Ⅱ型CalS基因。从基因结构分析中发现,各成员间mRNA序列长度差异较大,从794(西瓜Citrullus lanatus,Gene id: ClCG03G013930.1,属于Ⅰ型)到2112(菠萝Ananascomosus,Gene id: Aco025277.1,属于Ⅵ型),但是聚类相近的家族成员,具有相似的基因结构。植物CalS基因家族的氨基酸序列等电点差异较小,其数值主要在7.5-9之间,平均等电点为8.97。其中Ⅰ型平均8.51,Ⅱ型平均9.27,Ⅲ型平均8.66,Ⅳ型平均8.65,Ⅴ型平均9.12,Ⅵ型平均9.36。由此发现不同亚型的CalS基因成员等电点略有起伏,但是并未发现其是否与亚型分化存在关联。
2.3 植物CalS基因家族成员的复制
在对30个物种的分析中,发现在亚麻(Linumusitatissimum)、苹果(Malusdomestica)、可可(Theobromacacaov)中存在串联重复基因(表3)。针对片段重复,我们在PGDD数据库中搜索了本研究所涉及到的CalS基因拷贝数较多的物种,小立碗藓(Physcomitrella patens)拷贝数大于10个,向日葵(Helianthus annuus)、拟南芥(Arabidopsis thaliana)、可可(Theobroma cacao)、陆地棉(Gossypiumhirsutum)、杨树(Populustrichocarpa)、大豆(Glycinemax)、短柄草(Brachypodiumsylvaticum)、胡萝卜(Daucuscarota)、亚麻(Linumusitatissimum)、苹果(Malusdomestica)、菜豆(Phaseolus vulgaris)、甜橙(Citrus sinensis)。最终在拟南芥(Arabidopsis thaliana)、可可(Theobroma cacao)、杨树(Populustrichocarpa)、大豆(Glycinemax)、短柄草(Brachypodiumsylvaticum)搜索到8对片段重复,其中三对为Ⅵ型,两对为Ⅱ型,两对为Ⅲ型,还有一对为不同亚型之间的片段重复,为Ⅰ型、Ⅵ型基因。另外,未发现包含Ⅳ型、Ⅴ型的片段重复,可能是成员数量较少的原因。根据片段重复的有效Ks值估算分离时间,发现这些片段重复的分离时间与所在物种的基因组加倍时间相似(表4)。
为进一步研究CalS基因家族的复制事件,本研究以成员数量最多的陆地棉(Gossypiumhirsutum)为模型。对陆地棉(Gossypiumhirsutum)中所有CalS成员之间的Ks值进行分析,发现同型成员之间Ks值主要是在2~4之间,一般在3左右,说明陆地棉Ks值除无效值之外其余的趋近于饱和。同型之间Ks值基本相近,然后按棉属植物平均同义替换率为每109年2.6个碱基(λ=2.6×10-9)[22]估算陆地棉(Gossypiumhirsutum)CalS成员的分离时期。结果发现,同型成员之间的分歧事件与前人预测的三次陆地棉(Gossypiumhirsutum)基因加倍时间相似,一是被子植物中共有的基因组加倍事件(γ事件)[23],二是57-70 mya的棉属植物基因组加倍[24],三是在5-10 mya之间的四倍体棉花A、D异源染色体祖先的分离[25]。计算出的陆地棉(Gossypiumhirsutum)Ks值在各个亚型间其数值差异不大,本研究所涉及的其他成员数较多的物种内也发现了此类现象(陆地棉Ks值数据见图2)。
综上,结合PGDD数据以及对陆地棉(Gossypiumhirsutum)CalS基因的分析,本研究认为高等植物的基因组加倍事件(whole-genome duplication, WGD)在物种CalS基因家族成员的积累过程中,发挥了最主要的作用。
3 讨论
3.1 植物CalS基因的遗传分析
通过对不同类群植物中317个CalS基因进行聚类分析发现,植物CalS基因家族至少包含了六个起源于共同祖先的不同类型,这六个亚型共同从一个大的分支分化而来,说明这六个亚型可能是同源的,并且在绿色植物中逐渐分化,该六个亚型极有可能是被子植物从原始的苔藓植物、蕨类植物继承和演化而来。根据图1发现Ⅰ型、Ⅱ型共同处于一个分支,这两个亚型之间亲缘关系可能较近,而Ⅲ型、 Ⅳ型、Ⅵ型处于同一分支,这三个亚型间的亲缘关系可能比较近。
结合前人对于拟南芥(Arabidopsis thaliana)中12个CalS基因家族成员的研究,我们发现,不同亚型的基因成员具有不同的功能。其中Ⅰ型、Ⅱ型、Ⅴ型在有丝分裂周期参加花粉发育[4]。如在Ⅰ亚型中,AT3G07160对小孢子进入有丝分裂及不对称分裂非常重要,AT2G36850参与花粉发育,是细胞板上胼胝质生物合成所需,同时也参与调节胞间连丝的气孔形态和沉积[26-28];在Ⅱ亚型中,AT4G04970、AT4G03550这两个基因在四分体内分离小孢子的胼胝质壁的合成、防止小孢子发育早期胼胝质壁降解以及孢子体组织细胞板形成中都发挥了重要作用,并且AT4G03550在外壁形成和花粉壁形态的形成中也具有一定作用[29-32];Ⅴ亚型中AT2G13680对于萌发花粉管壁和胼胝质塞中的胼胝质沉积、小孢子发生过程中外壁形成和花粉生活力的需要以及防止花粉在发育早期退化都具有重要作用[33-34]。此外,我们还发现Ⅲ型、 Ⅳ型、Ⅵ型主要在应对胁迫状况时发挥作用,例如属于Ⅲ亚型的AT1G06490是筛板处胼胝质沉积所需[35],Ⅳ亚型中AT3G14570在病原菌感染和机械胁迫下调节胞间连丝通透性[36],Ⅵ亚型AT1G05570与UDP葡萄糖转移酶形成复合物,在胞质分裂时定位于细胞板[37],AT5G13000参与胞间连丝中胼胝质的积累[38],AT5G36870在分枝近轴芽的原基中表达,但其确切作用未知[39]。这种功能上的相似性似乎也印证了系统发生分析中,Ⅲ型、 Ⅳ型、Ⅵ型这三个亚型之间,以及Ⅰ型、Ⅱ型之间的亲缘关系。
3.2 被子植物CalS基因的亚型分布
根据每种植物在各个亚型之间的分布,除番木瓜(Carica papaya)外,其他物种中均有Ⅱ型CalS基因。由图1推断Ⅰ型、Ⅱ型CalS基因可能是原始亚型,此外,三个亚型在相对原始的植物(小立碗藓Physcomitrella patens和卷柏Selaginellamoellendorffii)就已存在,但其基因成员在Ⅰ型、Ⅱ型数目较多,Ⅴ型数目较少,因此,V亚型或许也是最原始的胼胝质合成酶之一。在本研究所涉及的所有被子植物中均含有六个亚型的CalS基因或相应的假基因遗迹。说明可能被子植物之前就已经形成了六个亚型的分化。而在部分物种内,其部分亚型成员功能的退化或缺失,似乎暗示了不同亚型之间存在一定的功能替代作用。虽然番木瓜(Carica papaya)只有Ⅴ型、Ⅵ型CalS基因,但是经假基因筛选发现其存在Ⅰ型、Ⅱ型、Ⅲ型、 Ⅳ型的假基因,原因可能是在進化过程中被淘汰掉了。而对于西瓜(Citrullulanatus)和菠萝(Ananascomosus)存在的几个假基因,其序列长度与其他序列对比太长,存在一定的可疑性,本次研究不予考虑。猜测其长度过长可能是终止子未识别等情况。
3.3 植物CalS基因的擴增方式分析
对于本研究中搜索到的片段重复,杨树(Populustrichocarpa)CalS基因家族成员中存在相同亚型及不同亚型成员间的片段重复,同型成员中估算的分离时间与前人预测的基因组加倍时间相似,一是被子植物的形成时间,为300 mya[23],二是被子植物中共有的基因组加倍事件(γ event),加倍时间为170~235 mya[23],三为其自身的基因组加倍时间(表4)。而对于不同亚型间的片段重复(Potri.001G230000,Potri.001G011900),其CalS基因成员的估算分离时间为578 mya,明显高于被子植物共有的基因组加倍事件(γ event)的时间,此外,在本研究中,这一对片段重复为孤立事件,且此对片段重复成员所属亚型为Ⅵ型和Ⅰ型,为两个聚类关系比较远的亚型(图1),说明虽然这种亚型间的分化可能发生在被子植物形成之前,但六个亚型间的总体差异较小。
4 结论
在本研究涉及的33个植物物种中,除红藻、团藻和衣藻不含有CalS基因外,剩余的30个物种均含有CalS基因,共识别到317个不同类型的CalS基因家族成员,这些成员主要可归为六大类;不同物种的CalS基因拷贝数差异明显,并且拷贝数较多的物种集中于被子植物类群。在本研究中所有的高等植物,除番木瓜外,均含有Ⅱ型基因。而对于扩增方式的分析发现,在CalS基因家族成员在适应性进化规程中,苹果、亚麻、可可这三个物种中各有一对成员属于串联重复,其余成员的扩增方式均为片段重复,这说明片段重复在植物CalS基因拷贝积累过程中发挥了主要作用。以上研究结果,使我们增进了对植物CalS基因家族的进化历程的理解,为进一步探究该基因家族的进化以及其功能结构进化打下了基础。
参考文献:
[1] Kelly R, Register E, Hsu M J, et al. Isolation of a gene involved in 1,3-beta-glucan synthesis in Aspergillusnidulans and purification of the corresponding protein[J]. Journal of Bacteriology,1996, 178(15):4381-4391.
[2] PⅠr?elová B, Matu?íková Ⅰ. Callose: the plant cell wall polysaccharide with multiple biological functions[J]. ActaPhysiologiaePlantarum, 2013, 35(3):635-644.
[3] Yu Y , Jiao L , Fu S , et al. Callose Synthase Family Genes Involved in the Grapevine Defense Response to Downy Mildew Disease[J]. Phytopathology, 2016, 106(1):56.
[4] ZáveskáDrábková L, Honys D. Evolutionary history of callose synthases in terrestrial plants with emphasis on proteins involved in male gametophyte development[J]. PLoS One, 2017,12(11):187331-187355.
[5] 彭蕴,范海芳,雷天刚, 等.柑橘胼胝质合成酶基因家族的表达分析[J].园艺学报,2019,46(2):330-336.
[6] Abercrombie JM, O'Meara BC, MoffattAR, et al. Developmental evoluion of flowering plant pollen tube cell walls:callose synthase (CalS) gene expression patterns[J].Evodevo, 2011,2(1):14.
[7] Shi X, Han X, Lu TG.Callose synthesis during reproductive development in monocotyledonous and dicotyledonous plants[J]. Plant Signal Behav, 2016, 11(2):1062196-1062201.
[8] Barratt DH, K?lling K, Graf A,etal.Callose synthase GSL7 is necessary for normal phloem transport and inflorescence growth in Arabidopsis[J]. Plant Physiol, 2011, 155(1):328-341.
[9] ChaseMW, ChristenhuszMJM, FayMF, et al.An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG IV[J]. Botanical Journal of the Linnean Society, 2016, 18 (1): 1-20.
[10] Bateman A, Coin L, Durbin R, et al. The Pfam protein families database[J]. Nucleic Acids Res, 2004, 32(Database issue):D138-D141.

