基于COI和Cytb基因的黄腰响蜜系统发育分析和分歧时间估算
元青苗 段玉宝*
(1.西南林业大学,云南省高校极小种群野生动物保育重点实验室,昆明,650224;2.西南林业大学生物多样性保护学院,昆明,650224)
随着分子生物学的发展,更多的鸟类(Aves)mtDNA基因全序列被测定[1],线粒体基因组作为一种研究手段在揭示鸟类的起源、系统发生[2-3]、分歧时间的研究中发挥着重要作用[4-5]。在mtDNA的13个蛋白编码基因中,COI和Cytb基因常用于动物类群的分类和系统发生关系的研究中[6-7],具有良好的解析作用。
1 材料与方法
1.1 材料数据来源
用于研究的样品采集于云南省西北部的泸水县高黎贡山,在此区域中获得1只黄腰响蜜个体;取0.5 g左右的肌肉组织,采用无水乙醇进行浸泡,保存于西南林业大学生物多样性保护学院。其余19个物种的COI和Cytb基因序列均来自于NCBI(National Center for Biotechnology Information)数据库。其中,将犀鸟目(Bucerotiformes)作为外群[11],共下载2条序列。每条COI和Cytb序列的长度分别大于650 bp和861 bp。物种信息详见表1。
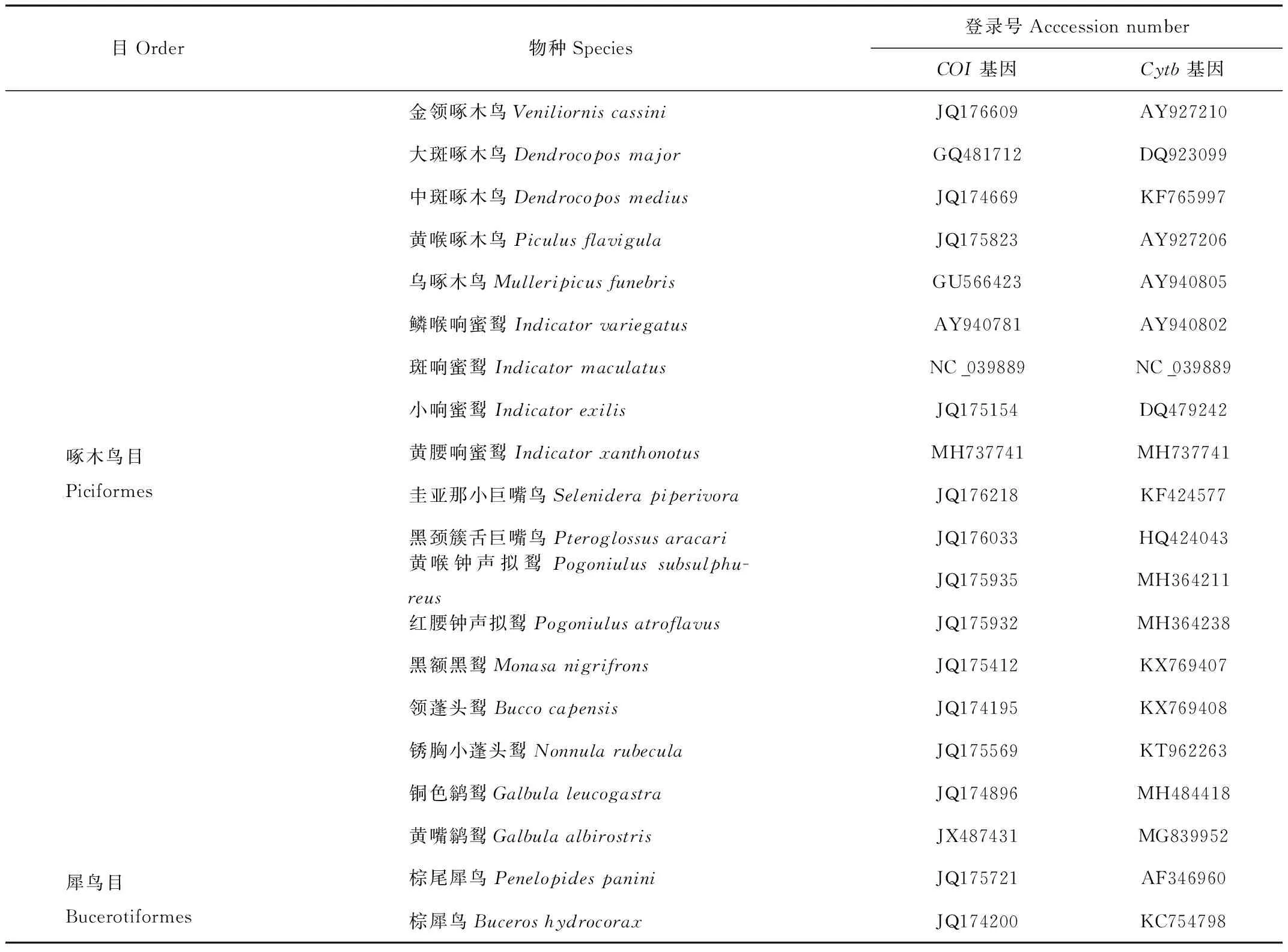
表1 本研究所需的物种基因序列信息Tab.1 List and gene information of species in this study
1.2 DNA的提取、扩增及测序
从采集的肌肉组织样品中提取总线粒体DNA,DNA提取完成后加入100 μL ddH2O进行溶解,保存于-20℃的冰箱中。
线粒体基因组的引物和反应体系参照Sorenson等[12]的研究。DNA提取和PCR初步扩增完成后,经过1%的琼脂糖凝胶电泳检测,在紫外灯下观察,然后将扩增成功的产物送往上海派森诺生物科技有限公司进行测序。黄腰响蜜的线粒体全基因组信息也已提交NCBI数据库,登录号为MH737741。
2 数据处理
2.1 序列处理
在MEGA 7软件[13]中导入目标物种的序列,20条序列进行多重比对,比对好后对序列进行剪切,每条COI基因序列的最终长度均为650 bp,Cytb序列为861 bp。然后将比对剪切好的COI和Cytb序列分别整理成Fasta和Mega格式进行保存。
2.2 线粒体COI和Cytb基因的饱和性检验
运用DAMBE 5.2.63软件[14]分析所有物种的序列饱和性。如果Iss与Iss.c满足Iss 在MEGA7中,选用Kimura 2-parameter模型[15]计算响蜜科响蜜属4种鸟类的种间遗传距离。 使用贝叶斯法(bayesian inference,BI)[16]和最大似然法(maximum likelihood,ML)[16]建树。在ClustalX 1.83软件中转换整理好的序列文件的格式,保留为Nexus和Phylip格式。 构建树算法可以采用马尔可夫链蒙特卡罗(MCMC)算法提供所有树的验证频率。最高的后验概率树是最优解,也是最优选择的树[17]。在jModelTest 0.1.1软件[18]中算出核苷酸替代类型数(nst)、位点间速率分布(rates)等参数以及分析出研究系统发育关系最优的模型为GTR+I+G。MrBayes软件是构建BI树的常用软件[19],该软件只支持贝叶斯法建树,所以本文运用MrBayes v3.2.1构建BI树。建树时使用4个马尔可夫链,外群则选择棕犀鸟(Buceroshydrocorax)。运行后,共进行2次run,二者相互独立,将起始的25%作为burn-in删除。运行20万代后,给出的标准误差(sd)小于0.01,则运算结束。最后,在Figtree v1.2.2中查看生成的文件。 最大似然法建树则是通过概率的计算进行的,ML系统发育树可以利用MEGA7进行构建。 使用BEAST v2.5.2软件[20-21]估算黄腰响蜜的分歧时间,此方法根据Jarvis等[11]依据鸟类化石记录对多个目的分歧时间估算的结果作为校正点,啄木鸟目和佛法僧目(Coraciiformes)的分化时间约为43 Ma,犀鸟目的分化时间发生约为58 Ma。将 “*.nxs”格式的文件用于分析,采用松弛的分子钟(relaxed clock log normal)模型,树的先验值计算选择Yule Model模型,运行1 000万代。使用Tracer v1.5评估结果的稳定性,之后再进一步分析,最终得到一个含有物种分歧时间的树文件。可在Figtree v1.2.2中查看含有黄腰响蜜分化时间的文件。 通过DAMBE 5.2.63软件分析20个物种的核苷酸序列饱和性,导入Fasta格式的序列文件,COI序列分析结果显示Iss=0.191 3 此外,为了更加直观地看出核苷酸序列的饱和程度,做饱和度分析的散点图(图1A和图1B),横坐标为F84遗传距离;纵坐标为转换Ts和颠换Tv数。图1A显示,当F84距离为0.330 8时,Ts与Tv并未相等,未出现饱和现象,则本研究所获得的COI序列可以构建系统发育树。同样地,当F84距离为0.441 8时,Ts与Tv未相等,未出现饱和现象(图1B),则本研究所获得的Cytb序列可以构建系统发育树。 表2 基于COI基因的响蜜科鸟类遗传距离Tab.2 Genetic distance between species of Indicatoridae based on COI 表2 基于COI基因的响蜜科鸟类遗传距离Tab.2 Genetic distance between species of Indicatoridae based on COI 序号No.种名Species12341鳞喉响蜜Indicator variegatus2斑响蜜Indicator maculatus0.0213小响蜜Indicator exilis0.1120.0934黄腰响蜜Indicator xanthono-tus0.1200.1200.154 表3 基于Cytb基因的响蜜科鸟类遗传距离Tab.3 Genetic distance between species of Indicatoridae based on Cytb 表3 基于Cytb基因的响蜜科鸟类遗传距离Tab.3 Genetic distance between species of Indicatoridae based on Cytb 序号No.种名Species12341鳞喉响蜜Indicator variegatus2斑响蜜Indicator maculatus0.0323小响蜜Indicator exilis0.1320.1264黄腰响蜜Indicator xanthono-tus0.1770.1780.194 利用COI(图2A)和Cytb(图2B)基因重建的BI和ML系统发育树结构显示出两种基因构建的系统树拓扑结构基本一致,具有相似的系统发育关系,以BI树的拓扑结构作为展示。据图显示,黄腰响蜜与响蜜科响蜜属聚为一大支(A:PP=0.99,BP=82;B:PP=1.00,BP=100),与啄木鸟科互为姐妹群,并有较高的支持率(A:PP=0.97,BP=75;B:PP=0.96,BP=74)。在响蜜科内部,黄腰响蜜构成单系;在4种鸟类中,最早分化出来的类群是黄腰响蜜,其次是小响蜜(Indicatorexilis)。鳞喉响蜜(Indicatorvariegatus)与斑响蜜(Indicatormaculatus)聚为一支(A:PP=0.98,BP=91;B:PP=1.00,BP=100)。 Tracer v1.5软件中显示Ess值大于200,说明在BEAUti中设置的参数是合理的,最后得到含有物种分歧时间的树文件。响蜜科鸟类的分化时间详见图3,可知响蜜科鸟类起源于新第三纪中新世时期,距今约20.44(15.88,25.77)Ma,而黄腰响蜜也在此时间段内分化出来(20.44 Ma)。此外,在渐新世和中新世阶段,啄木鸟目鸟类的多样性不断扩大,物种多样性越来越丰富。 COI和Cytb基因片段序列被广泛地应用于解决鸟类的分子系统发育问题[22]。本文的结果显示,黄腰响蜜聚在响蜜科响蜜属物种所在分支里,并且有着很高的支持率。黄腰响蜜所在的响蜜科与啄木鸟科有着姐妹群的关系,这与Prum等[23]的研究结果一致。而在响蜜科内部,黄腰响蜜是最早分化出来的物种,其次是小响蜜;且黄腰响蜜无姐妹群。 本研究分子定时的结果显示啄木鸟科分化于14.95(14.02,15.89)Ma,与Prum等[23]的研究结果(Prum等研究表明啄木鸟科的分化时间约为15 Ma)相近,表明了分歧时间推断的合理性和准确性,进而为评估黄腰响蜜的分歧时间提供支持。响蜜科鸟类的分化可追溯到新生代新第三纪中,黄腰响蜜在新第三纪中新世阶段分化,时间节点为20.44(15.88,25.77)Ma。在这一时期,青藏高原发生了广泛的隆起(22—20 Ma),并随着多个隆升事件[24-25],直到3.4 Ma到1.6 Ma,高原抬升4 000 m以上,这被认为显著影响了其形貌和鸟类物种的多样化[26]。所以黄腰响蜜的形成可能与青藏高原隆起有关,其历史和地理模式受到了青藏高原及周边区域隆升的强烈影响。2.3 响蜜科鸟类种间遗传距离计算
2.4 系统发育树的构建
2.5 分歧年代评估
3 结果与分析
3.1 基因序列饱和性分析
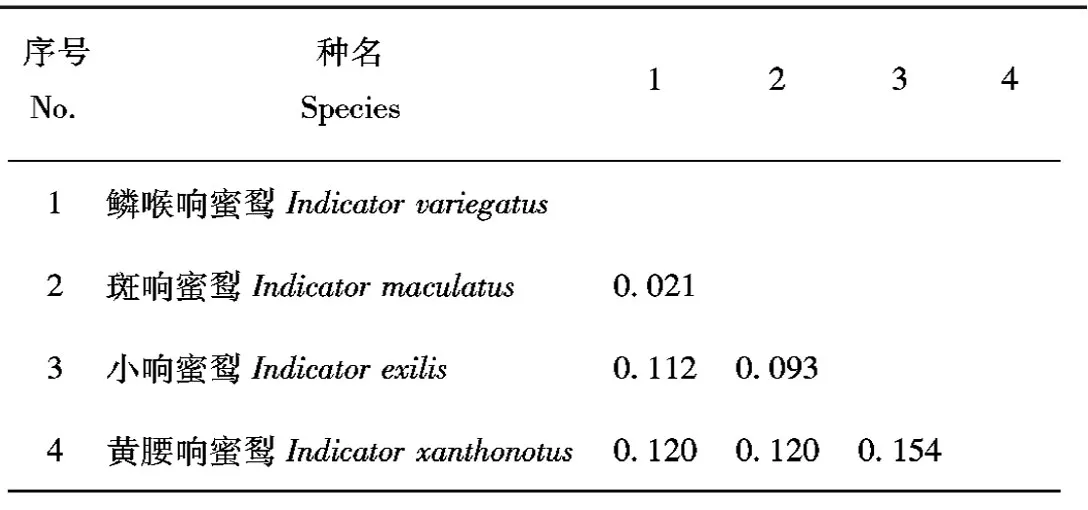
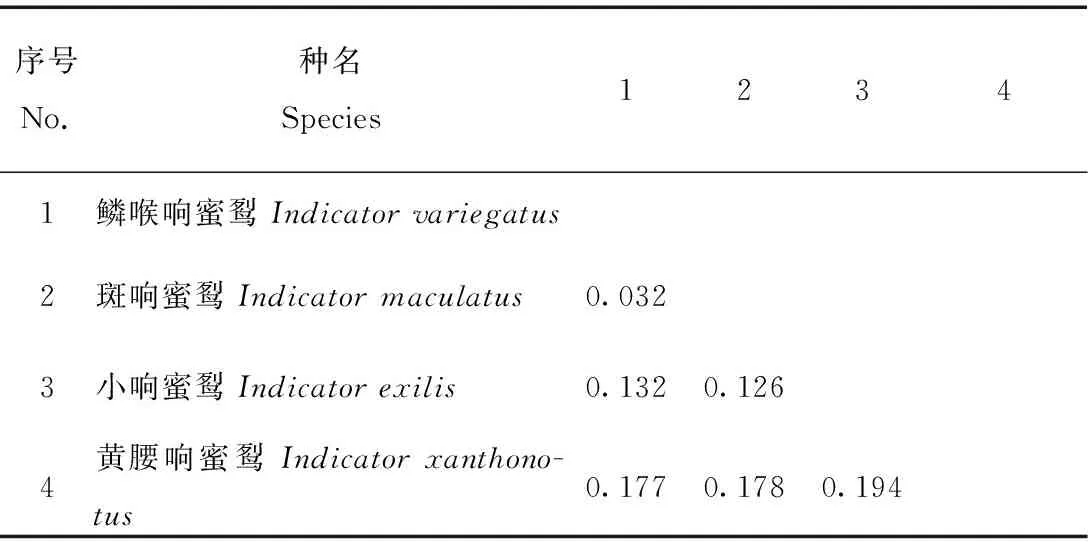
3.2 响蜜科鸟类种间遗传距离分析
3.3 系统发育树分析
3.4 分歧时间分析
4 讨论
4.1 黄腰响蜜的系统发育分析
4.2 黄腰响蜜的起源分化

