基于全波段高光谱的冬小麦生长参数估算方法比较
纪景纯 刘建立 牛玉洁 宣可凡 蒋一飞 邓皓东 ,3 李晓鹏
(1中国科学院南京土壤研究所,210008,江苏南京;2中国科学院大学,100049,北京;3河海大学水文水资源学院,210008,江苏南京)
利用高光谱遥感进行作物生长状态的监测具有无损、高效和灵敏的特点,前人对基于高光谱数据的生长参数估算开展了大量的研究。Li等[1]根据可变重要性值(variable importance in projection,VIP)对光谱反射率、光谱一阶导数、反射/吸收位置和植被指数进行筛选,最终利用筛选出的14个特征建立了偏最小二乘回归(partial least squares regression,PLSR)反演叶面积指数(leaf area index,LAI)模型,决定系数(R2)达到0.88,均方根误差(RMSE)为0.943,通过特征筛选保留了高光谱的有效信息,在减少运算量同时保证了反演精度。Jia等[2]使用连续投影算法(successive projections algorithm,SPA)结合协同区间偏最小二乘法(synergy interval partial least squares,SIPLS)处理高光谱数据,筛选出8个特征波段,建立叶片生物量的反演方程,建模集R2达0.79,验证集RMSE为0.059kg/m2,较其他的特征选择技术提供了更少的不相关、非共线性的光谱变量,降低了模型整体的复杂度,缩短了模型运行时间。Duan等[3]考虑了氮的垂直分布特征,基于绿度指数(GI)、修正归一化差值(mND705)和归一化差分植被指数(NDVI)分别建立了冬小麦叶片总氮浓度线性模型,提出了不同生育期遥感探测的有效冠层,该方法建立的反演模型R2和归一化均方根误差(nRMSE)分别为0.61和8.84%、0.59和8.89%、0.53和9.37%,显著提高了对叶片氮含量的反演精度和模型的稳定性。Huang等[4]则通过相关性分析,利用450~488nm和500~540nm范围的一阶导数之和构建了小麦赤霉病指数(wheat scab index,WSI),利用新指数与其他传统植被指数构建的多重逐步回归模型预测病情等级,发现WSI较常规指数与病情等级具有更显著的线性关系,针对性的新指数提高了对病情的判断水平。前人反演作物参数多侧重于数据降维,提取高光谱的有效信息,针对不同的监测指标筛选出了不同的特征并建立了相应的参数反演模型,回归方法以PLSR为主。特征筛选和建模过程需要一定的先验知识,且降维过程本身比较复杂,在实际应用过程中,往往需要对同一时期的多种生长参数进行快速监测。
机器学习模型以其强大的数据处理能力和不需要先验知识的特点在作物参数反演中有很大的优势。Bao等[5]在使用成像高光谱数据进行小麦品种分类研究中,使用线性判别分析(linear discriminant analysis,LDA)、支持向量机(support vector machine,SVM)和极限学习机(extreme learning machine,ELM)分别对全波段和使用主成分分析(principal component analysis,PCA)、连续投影算法(SPA)和随机蛙跳(random frog,RF)筛选的特征波段建立分类模型,基于全波段光谱反射率建立的ELM模型分类效果最优。机器学习模型可以用于全波段的高光谱数据处理,不进行特征筛选的降维过程,极大地简化了高光谱数据的应用流程。为了简化作物参数从建模到估算的整体过程,找出可以直接用于批量处理冠层高光谱数据,实时监测作物生长状态的方法,本研究选择广泛使用的PLSR和不依赖于先验知识的机器学习方法支持向量回归(SVR)和前馈神经网络(FNN)进行了利用全波段高光谱数据估算生长参数的方法比较[6-7]。
1 材料与方法
1.1 试验设计
试验地点位于河南省封丘县潘店乡,该地区年均气温13.9℃,无霜期220d左右,全年日照时数为2 300~2 500h,土壤类型主要为黄河沉积物发育的轻壤质潮土,主要的作物栽培模式为冬小麦-夏玉米轮作制度。
试验于2018-2019年冬小麦生长季进行,供试品种为百农矮抗58。根据当地农户的常规施氮量190kg/hm2,设置 N0、N1、N2、N3、N45 个氮肥梯度,单季小麦施氮量分别为0、150、190、230和270kg/hm2,应用尿素作为单一氮源,60%作为基肥,40%作为追肥。磷肥用量为130 P2O5kg/hm2,钾肥用量为120 K2O kg/hm2,不进行差异处理。N0、N1、N3和N4处理重复9次,N2处理重复12次,共计48个小区。种植前翻耕1次,基肥深施,追肥撒施。试验地的单元面积为48m2(8m×6m),试验单元四周埋设80cm深的混凝土板防止土壤溶液侧向交换。该试验地的连续氮肥梯度处理超过10年,不同氮肥处理的冬小麦生长差异显著,为建立生长参数估算模型提供了各类型样本。

图1 氮肥梯度试验布置Fig.1 Experimental design of nitrogen rate test
1.2 数据采集
根据冬小麦追肥和产量形成的关键时期,试验于返青拔节期(3月25日)、孕穗期(4月16日)、扬花初期(4月28日)和乳熟初期(5月18日)进行地面生长参数和冠层高光谱数据的采集。
1.2.1 地面生长参数的测定 监测的生长参数包括地上部生物量(aboveground biomass,AGB)、叶面积指数(leaf area index,LAI)、全氮含量(total nitrogen content,N)和叶绿素浓度(chlorophyll concentration,CC)。除全氮含量外,在每个小区内按照五点取样法进行数据采集,总样本容量为240株。
AGB为单株小麦烘干重均值与种植密度的乘积:小麦烘干重均值在每个采样点取5株,105℃杀青30min,再将温度调至80℃烘干至恒重;种植密度为1m2内小麦种植的行数与单行内实测的小麦株数的乘积。使用北京澳作生态仪器有限公司的SunScan冠层分析仪,选择晴朗的天气于10∶00-14∶00测定LAI;将每小区5个取样点的样品混合粉碎后按照凯氏定氮法测定全氮含量;使用美国Apogee公司生产的MC-100植物叶绿素仪测定叶绿素浓度,每个取样点选取1株分别测定顶部功能叶和植株中部叶片的叶基、叶中和叶尖,共6个点,计算均值代表该取样点叶绿素浓度。
1.2.2 冠层光谱反射率的采集 根据前人的研究,与作物各项生长参数相关的光谱范围主要在可见~近红外区间(叶绿素400~700nm,植株氮素含量450~520、515~585、625~695、730和930nm,地上部干生物量650~700、500~550和900~940nm),总体而言,400~1 000nm的光谱范围能够反映作物的生长状况[8-10]。为了减少输入特征总量,提高运算效率,使用ASD Field Spec 4便携式光谱仪中VNIR探测器所监测的光谱范围(350~1 000nm),剔除噪声干扰,最终选取400~1 000nm的光谱反射率。光谱采样间隔为1.4nm,光谱分辨率为3nm。该波段范围也是目前广泛使用的无人机载高光谱仪器的监测范围,利用该范围高光谱数据进行的建模比较,对样本量大的无人机载数据处理具有参考价值[6,11]。选择晴朗的天气于10∶00-14∶00进行测定,探测器距冠层20cm,监测点与取样点位置相同。
1.3 数据建模方法
对PLSR、SVR和FNN 3种建模方法进行比较。其中PLSR是目前广泛使用的高光谱数据处理方法;SVR模型对高维度、小样本数据有较好的处理能力;FNN是研究较多、成熟且广泛使用的机器学习方法。在每个处理内按照7∶3的比例随机划分建模集与验证集,随后合并为整体的建模集与验证集。
1.3.1 PLSR PLSR是一种多自变量对多因变量的线性回归建模方法,可以同时实现提取变量特征、分析变量间相关性和回归建模,适合多元数据的统计分析。当自变量个数多、存在多重相关性且观测数据样本量较小时,PLSR具有传统的经典回归分析方法所没有的优势。使用PLSR建模的过程:假定p个自变量{X1,...,Xp}和q个因变量{Y1,…,Yq},在X与Y中分别提取出成分t1和u1,使t1和u1尽可能大地携带他们各自数据表中的变异信息,且t1和u1的相关程度最大。第一对成分t1和u1被提取后,分别实施X对t1以及Y对u1的回归。若回归方程此时交叉有效性小于0.0975,则成分确定;否则将利用X被t1以及Y被u1解释后的残余信息进行第二轮的成分t2和u2提取,继续实施X和Y对t2和u2的回归,对上述过程进行迭代,直到满足精度要求为止[1,12]。
1.3.2 SVR SVR是Vapnik在长期系统的统计学理论研究基础上提出的机器学习算法[13],其基本思想是寻找最优的超平面,使所有样本点距离超平面的总体偏差最小,即所有样本的类内方差最小。对于不可分的非线性数据,SVR使用核函数将低维数据映射到高维空间,在高维空间找到线性可分的超平面,能够很好地解决高维特征的回归问题。当样本不是海量数据时,使用SVR进行回归的准确率高且泛化能力强[14],适用于自变量维度大于样本量且样本总量较小的全波段光谱估算生长参数问题。根据前人的研究,推荐在缺乏先验知识的条件下使用径向基核函数(RBF),核函数系数(g)和惩罚系数(C)是SVR模型的重要参数,前者影响模型的复杂程度和泛化能力,后者表征模型对离群点的重视程度。g和C系数通过6折交叉验证确定,误差阈值设为10-7。
1.3.3 FNN FNN是一种模仿生物神经网络结构和功能的计算模型,用于估算函数。FNN包括输入层、隐含层和输出层,每一层的神经元接受前一层的输出,处理后传输给下一层,数据单向流动。神经网络的每一层与下一层之间都存在一个参数矩阵,首先随机初始化每一层的参数矩阵,然后从输入层开始,依次计算下一层每个神经元的激活值,直到计算出输出层神经元的激活值。对于维度较高的线性不可分数据,与线性回归和SVR不同,神经网络通过线性分类器的叠加组合完成分类,同时避免增加无效维度。对于高光谱数据处理分析同样适用。隐层节点数是重要的模型参数,节点数的设置对模型的拟合效果和泛化能力影响巨大。根据国内外的应用实例,隐层节点数的经验公式为,N为隐层节点数,α为1~10之间的整数,m为输入神经元数,n为输出神经元数[15]。
本试验中隐层节点数通过3折交叉验证进行选择。输入特征为400~1 000nm光谱反射率,输入层节点数601个,输出值为相应的生长参数,输出层节点数为1。根据隐层节点数经验公式得到本试验的推荐隐层节点数范围25~35。设置隐层节点数5、15、20、25、30、35进行建模比较。根据预试验建模结果的均值,选择满足模型拟合优度R2大于0.90,均方根误差RMSE与最小值相差较小的节点数设置;在不影响估算精度和拟合优度的条件下,结合运算效率,优先选择较小的节点数。最终的设置结果见表1。
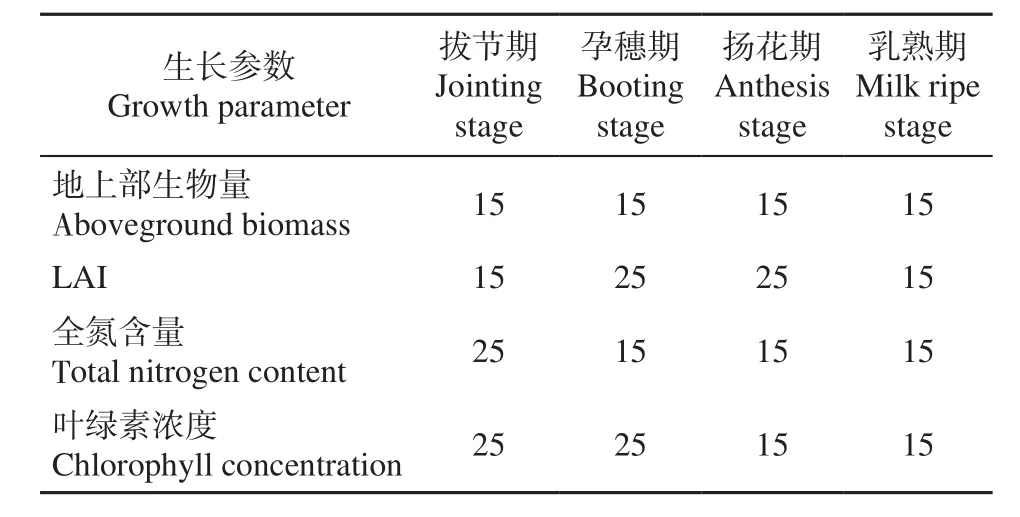
表1 FNN隐层节点数设置Table 1 Number of nodes in the hidden layer of FNN
1.4 模型评价指标
模型评价包括对建模效果和估算效果的评价,主要选用以下参数:
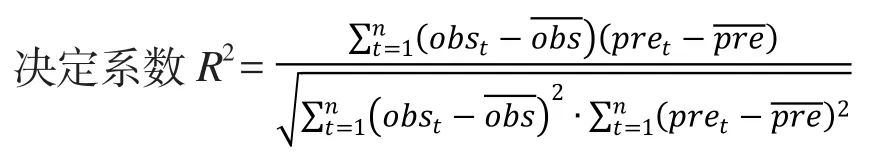
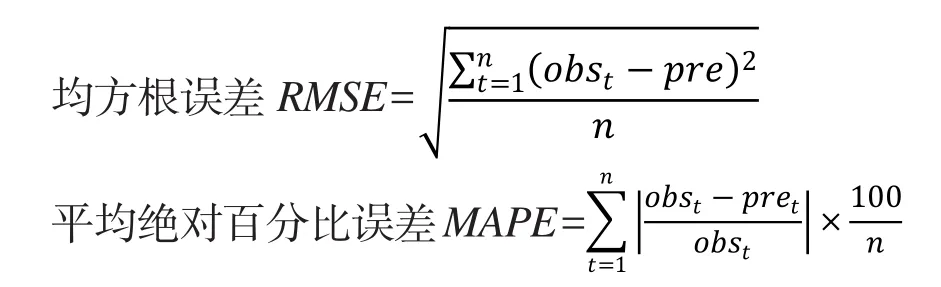
3个公式中obs为生长参数实测值,pre为生长参数的预测值。建模集和验证集的R2越大,RMSE和MAPE越小,模型的建模及估算效果越好[16]。所有评价指标均计算10折交叉验证的均值。
2 结果与分析
2.1 不同氮肥梯度下冬小麦生长参数及冠层光谱

图2 不同氮肥梯度下冬小麦生长参数Fig.2 Growth parameters of winter wheat under different nitrogen fertilizer gradients
由图2可知,拔节期和孕穗期,AGB随施氮量增加而上升,不施氮(N0)、缺氮(N1)、常规施氮(N2、N3)与过量施氮(N4)间差异显著。扬花期,不缺氮处理(N2、N3、N4)间差异不显著,与缺氮处理差异显著,N2、N3、N4较N0高730~742g/m2,较N1高98~111g/m2。乳熟期,N4显著高于其他处理,N0显著低于其他处理。5个处理中N0LAI全生育期最低(0.40~1.16),N1次之(1.49~4.05),且二者与其他处理差异显著。拔节期和孕穗期不缺氮处理(N2、N3和N4)间差距小。扬花期和乳熟期,不施氮、缺氮、常规施氮与过量施氮间差异显著。拔节期N3全氮含量最高达到2.21%,且5个氮肥梯度间差异显著。其余生育期N4最高,但与N3差异不显著,仅在乳熟期显著高于其他处理,与N3差值为0.18%。N0和N1与其他处理差异显著。总体趋势表现为随着施氮量的增加全氮含量也相应增加,且所有处理在乳熟初期全氮含量均有所下降。在5个氮肥梯度中N0叶绿素浓度全生育期均最低(117.6~221.4μmol/m2),且在乳熟期下降了 103.8 μmol/m2;N1略高(421.9~458.3μmol/m2),但全生育期波动小;N4叶绿素浓度最高,除拔节期与N3差异不显著外,其余生育期均显著高于其他处理。常规施氮处理,叶绿素浓度全生育期波动较小,N2为 453.4~526.8μmol/m2,N3为 465.9~542.9μmol/m2。
由图3可知,N0处理冠层光谱反射率在绿峰处反射率较其他处理高,全生育期差值0.04%~0.15%;在红谷处反射率较其他处理高0.02%~0.20%。总体趋势上,随着氮肥施用量的增加,红边~近红处的反射率上升。拔节期N1、N2、N3和N4处理该处反射率与N0差值分别为0.03%、0.05%、0.10%和0.10%,孕穗期N1与N0差值为0.20%,其余处理与N0差值为0.25%,扬花期N1与N0差值为0.23%,其余处理与N0差值为0.25%,乳熟初期N1与N0差值为0.08%,其余处理与N0差值为0.12%。除拔节期外,不缺氮处理间(N2、N3和N4)的冠层光谱差异主要表现为绿峰反射值不同。长期不施氮处理N0整体长势及光谱反射率均与其他处理差距较大,其余处理间的差异远小于较不施氮处理的差异,数据分布呈两簇分布。

图3 不同氮肥梯度下冬小麦冠层光谱反射率Fig.3 Canopy hyperspectral reflectance of winter wheat under different nitrogen fertilizer gradients
2.2 PLSR、SVR和FNN的建模效果比较
在1.3中确定的参数设置下,分别采用PLSR、SVR和FNN对拔节-乳熟期各生长参数进行建模,图4为建模集数据的估算散点图,表2为建模评价指标的10折验证的均值。3种方法的地上部生物量在拔节期和孕穗期拟合优度均低于扬花期和乳熟期,其中孕穗期PLSR的R2最低,仅有0.87,而乳熟期SVR和FNN的R2则高达0.95。两种机器学习模型在扬花期、乳熟期R2为0.94~0.95,MAPE为5.25%~5.59%,优于PLSR,且SVR的精度稍高于FNN。不同生育期3种方法LAI的建模效果均稳定,PLSR的R2稍低于SVR和FNN,MAPE分别高2.20%~4.67%和1.20%~3.40%。SVR和FNN全氮含量的MAPE分别较PLSR低29.28%~65.73%和22.07%~57.06%,R2分别高3.19%~8.89%和2.20%~7.78%,SVR和FNN之间建模效果相近。3种方法中,SVR对叶绿素浓度的建模效果最优,全生育期MAPE为4.70%~6.49%,R2为0.89~0.96。
PLSR全生育期对建模集的反演精度均低于两种机器学习方法,MAPE在6.65%~8.40%之间。SVR对地上部生物量、LAI、全氮含量和叶绿素浓度建模的全生育期R2均值分别为0.93、0.95、0.97和0.93,较PLSR分别提高了3.33%、1.60%、5.18% 和2.76%;FNN较PLSR分别提高了3.89%、2.13%、4.36%和3.59%。总体而言,SVR的建模效果最优。

图4 PLSR、SVR和FNN模型对建模集样本的估算结果Fig.4 Fitted results for calibration samples of PLSR, SVR and FNN model

表2 PLSR、SVR和FNN模型的建模评价指标Table 2 The evaluation indicators of PLSR, SVR and FNN model in calibration set
2.3 PLSR、SVR和FNN的估算效果比较
将2.2中构建的模型用于对应的验证集数据,比较3种方法的估算效果。图5为验证集估算参数散点图,表3为验证集的评价指标均值。全生育期SVR的地上部生物量拟合优度和估算精度均高于PLSR和FNN,尤其是估算精度,MAPE较PLSR和FNN分别降低了5.73%~22.78%和10.74%~17.40%,RMSE分别降低了4.04%~18.51%和6.85%~12.06%。各生育期SVR和PLSR的LAI拟合优度相近,但SVR的估算精度更高,MAPE较之降低了10.63%~35.07%。SVR拟合优度R2比FNN高1.09%~4.35%,估算精度也更高,MAPE比FNN低17.10%~28.63%。除扬花期外,SVR全氮含量的估算效果均优于PLSR和FNN,R2分别提高了1.06%~5.56%、1.06%~2.20%,MAPE分别降低了9.23%~33.33%、14.47%~18.18%。扬花期3种模型估算性能相当,PLSR精度略高。全生育期SVR叶绿素浓度的估算效果均优于PLSR和FNN。除扬花期外,SVR的R2较PLSR提高了1.06%~1.10%,MAPE降低了9.15%~20.14%;SVR的R2较FNN提高了1.06%~4.55%,MAPE降低了19.80%~21.33%。只有扬花期SVR的优势不明显,拟合优度与PLSR和FNN相同,RMSE较PLSR高1.17%,但MAPE较之低2.41%,较FNN低6.89%。

图5 PLSR、SVR和FNN模型对验证集样本的估算结果Fig.5 Predicting result of growth parameters of PLSR, SVR and FNN model
SVR对地上部生物量、LAI、全氮含量和叶绿素浓度估算的全生育期MAPE均值分别为7.46%、7.05%、4.04%和6.85%,较PLSR的均值分别低1.25%、2.15%、0.88%和0.82%;较FNN的均值分别低1.30%、2.36%、0.56%和1.39%;估算的R2全生育期均值分别为0.90、0.94、0.92和0.91,对所有生长参数均高于PLSR和FNN;对4种生长参数所估算的RMSE全生育期均值在3种模型中也最低。SVR在3种模型中对验证集数据生长参数变异性解释力最强,估算精度最高。

表3 PLSR、SVR和FNN模型的估算评价指标Table 3 The evaluation indicators of PLSR, SVR and FNN model in validation set
在8核16G的配置下,从建模到结果估算耗时比较,PLSR仅需几十秒,SVR耗时在10min以内,FNN耗时需要数小时。
3 讨论
本研究选取冬小麦冠层光谱400~1 000nm波段反射率,采用PLSR、SVR和FNN对冬小麦全生育期地上部生物量、LAI、全氮含量和叶绿素浓度进行建模估算,发现3种模型对生长参数变异情况的解释性都较高,R2均在0.86以上。3种建模方法中,SVR模型全生育期MAPE均值分别为7.46%、7.05%、4.04%和6.85%,R2均值分别为0.90、0.94、0.92和0.91,总体估算效果在3种参与比较的建模方法中最优,与梁栋等[17]LAI发现的最优估算方法的结果一致,说明SVR在多种生长参数的建模估算方面均优于PLSR,更适用于作物监测。本研究通过对不同生育期参数分时期建模,取得了较好的估测结果。与贺佳等[18]利用高光谱植被指数分时期监测冬小麦地上部生物量的结果相近,R2稍高于贾学勤等[19]利用多种植被指数结合PLSR的生物量监测结果,说明分时期进行生长参数监测能够有效改善高光谱遥感监测效果。通过综合比较多种生长参数的最优估算时期,发现地上部生物量最佳监测时期为扬花初期;LAI全生育期估算结果波动较小;全氮含量在返青拔节-孕穗期估算精度稍高于扬花-乳熟期;叶绿素浓度不适于在扬花初期进行估算。
与传统的特征筛选、降维再建模的监测方法相比,本研究通过使用全波段高光谱反射率直接参与建模,虽然没用显著提高监测精度,但是以较少的运算量简化了数据处理的流程,有利于高光谱数据在实际农业监测中的应用推广。
4 结论
PLSR、SVR和FNN 3种方法均能实现全波段高光谱反演作物生长参数的目标。就建模过程而言,PLSR最为简单,只需要设置交叉验证的阈值;SVR需要根据推荐选择核函数,交叉验证设置核函数系数;FNN则需要通过预试验进行人为选择隐层节点数,相对复杂。对于建模集的预测性能,FNN和SVR两种机器学习方法均优于PLSR,对参数的解释性和精度均较高。对于独立的验证数据,SVR的估算性能最优,PLSR次之,FNN由于需要大量的样本进行训练,同时存在陷入局部最优解的问题,对于验证数据的估算表现不佳。
传统的高光谱数据建模处理采用先降维后建模方法,一定程度上提高了运算速率,但面向多元监测指标时,建模反演整体过程复杂。随着运算力的不断提升和对高维数据的处理能力不断提高,特征数量对数据处理与分析的制约越来越小。本研究使用原始冬小麦冠层光谱反射率进行建模,简化了特征筛选的过程,达到了理想的估算结果,为高光谱数据的处理提供了新的思路。在利用全波段高光谱估算冬小麦生长参数的试验中,SVR在估测方法中表现最优,同时运算速率适中,可以满足大面积作物生长状况实时监测的需求。

