三链核酸分子探针的稳定性及其生物传感应用
王焕翔,潘松兰,杨 华,薛 倩,邓祥熙,孙芸琳,赵聪慧,邹 振
(长沙理工大学化学与食品工程学院,细胞化学湖南省重点实验室,湖南长沙410114)
0 引言
核酸分子探针是一类建立在生物分子相互作用(如DNA/RNA杂交、DNA/蛋白质作用等)基础上,可特异性识别互补序列的核酸、蛋白质或小分子物质的功能寡聚核苷酸[1]。它具有设计简便、易合成、易修饰、特异性高、自身结构可预测等优点,已被广泛用于生物传感领域[2-4]。1957年,Feisenfeld和Davies等[5]发现某些基因中特定的同聚嘌呤或者同聚嘧啶序列,在一定的条件下由第三条单链寡核苷酸 (triplex-forming oligonucleotides,TFOs)插入到双链的大沟之中,通过Watson-Crick与Hoogsteen氢键作用形成三螺旋核酸结构。三螺旋核酸结构碱基的构成具有专一性,其基本构型由C·G-C、G·G-C、和T·A-T形成的平行三重链结构以及C·G-C、G·G-C、T·A-T、和A·A-T形成的反向平行三链结构组成[6]。这为三链核酸分子探针的研究以及实际的应用奠定了理论基础。以三螺旋结构为基础的三链核酸分子探针也随之被开发应用于生化传感等领域。该文主要从影响三链核酸稳定性的因素以及三链核酸在生化传感中的功能应用进行阐述。
1 影响三链核酸稳定性的因素
1.1 长度的影响
寡聚脱氧核苷酸(ODN)的长度在三链结构形成的过程中是一个重要影响因素。ODN长度太短Hoogesteen氢键作用力太弱,不足以形成三链结构,ODN长度太长容易形成核酸二级结构,不利于三链结构的形成。Brown课题组报道,三链核酸第三条核酸链的长度影响三链结构的形成。当双链长度为30个碱基,第三条链长度为22个碱基时,能形成稳定的三链核酸结构。ODN低于或高于该22个碱基时,三链核酸结构难形成[7]。
1.2 碱基序列的影响
三链核酸结构各组成单元碱基序列高度匹配,碱基的错配、缺失会明显的降低三链结构的形成。三链核酸结构的某一结合位点碱基发生改变,会在一定程度上导致第三条链的扭曲,无法形成三链结构。在相同的条件下,由G、A构成的ODN具有更高的亲和力,能快速形成三链结构。而由T、C构成的ODN形成较为稳定的三链核酸结构所需的条件更为苛刻[8]。由此可见,序列的选择对于三链的形成是不容忽视的。
1.3 碱基修饰的影响
近些年来对三链核酸的组成链进行了大量的化学修饰,以满足对分析检测对象的需求。其中甲基化是碱基修饰的手段之一,一般在胞嘧啶的5位点进行甲基化。通过碱基修饰扩大三链核酸存在的酸度范围,而且在一定程度上提高三链自身的(Tm)解链温度和亲和常数[9]。提高三链自身稳定性是甲基化所引起的疏水作用,形成过程中的熵变能证实这一观点。不是所有的取代基团都能增强三链的稳定性。比如溴或者丙基在C碱基的5位点进行修饰反而会降低三链的稳定性,相同情况下在RNA中U的5位点进行修饰,却会进一步的促进三链的形成[10]。造成这种差异的可能是取代反应过程中成键的电荷密度发生改变。溴电负性强,不仅会增强嘧啶链3位点电荷密度使氢键供体能力得到大大的提升,也会进一步降低1位点电荷密度使受体能力减弱。当这两种效应不对等便会削弱三链核酸的稳定性。对磷酸基团进行甲基化修饰形成的三链结构稳定性与ODN长度密切相关[11]。
1.4 温度和pH的影响
在酸性条件下,胞嘧啶(C)质子化,增加了C-G·C碱基对的稳定性,促使三链的形成;而在中性条件下胸腺嘧啶(T)去质子化,增加了T-A·T碱基对的稳定,使三链结构能稳定存在。酸性环境中,三链的稳定性取决于第三条链中C碱基的比例[12]。一般根据其C-G·C/T-A·T比例的不同,可以设计不同类型的可切换三链核酸分子开关。从解链温度(Tm)对酸度的依赖性和形成的焓变来看,质子在三链解离的过程中被释放,但其数量要少于第三条链中胞嘧啶的数目。这表明单链中胞嘧啶被部分质子化[13]。
1.5 离子浓度的影响
由于分析检测的多样性,不同的离子在溶液中既能降低双链和寡聚核苷酸链之间的排斥力,又能使核酸链之间的亲和性发生显著的改变。Mg2+嵌到核酸链中间降低局部过高的负电荷,促进了三链核酸的形成[14]。Volker等[15]通过升高钠离子浓度明显提高了三链核酸结构的稳定,当ODN中富含T碱基时,形成的三链结构比相应的富含C所形成的三链对于钠离子的浓度表现出更高的依赖性。这可能是由于电荷的不同所导致的,前者具有较高的负电荷,而后者由于C的质子化作用使负电荷密度下降所造成的。除了钠离子和镁离子等阳离子外,多胺质子化在一定程度上影响三链的形成。溶液中存在精胺时,有效降低局部负电荷促进了三链的形成。同时Ag+通过C-G·C碱基对的络合促进三链的形成[16]。
2 三链核酸分子探针在生化传感中的应用
由于核酸序列的可设计性,三链结构在核酸探针信号转导方面具有巨大的优势。三链核酸探针能够进行荧光、拉曼、电化学生化传感平台的设计,用于复杂体系中生物标志物的检测,具有普适性、灵敏度高,特异性高的特点。
2.1 三链分子探针荧光生化传感应用
荧光传感具有响应快,检测限低等优点,是核酸探针常用的信号报告方式。传统核酸探针MB(分子信标)在5’和3’修饰荧光团和猝灭团。遇到靶序列后,两者分离恢复荧光信号,实现对靶序列的检测。基于此原理,Lu等设计了三链核酸分子开关(TMS)结构[17],它是由5’、3’分别标记了FAM、BHQ 1的ptMB链和外加的sfO链组装形成。环部设计为miRNA的互补序列,茎部设计为可形成三链结构的T·A-T、C·G-C碱基对,通过Watson-Crick氢键和Hoogsteen氢键组成。当遇到靶标miRNA,TMS打开荧光信号恢复。与传统分子信标相比,TMS检测限更低,显著提高信号背景比(图1)。
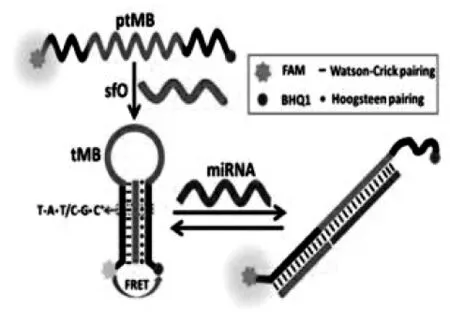
图1 三链分子开关用miRNA检测[17]Fig.1 Classical triplex molecular beacons for microRNA-21 detection[17]
Yang等首次提出三链分子探针具有分子识别单元和信号报告单元分离的优势。他们设计了一系列通用型三链核酸分子开关(TMS)平台,用于目标分子的高灵敏检测和细胞成像。如图2所示,Yang等设计了一种基于环茎部型三链生物传感器平台。Aptamer/待测物的特异性识别作用与环状部分露出的脚趾进行碱基配对,使三链结构被拆卸,该探针开关被打开,识别单元与信号单元实现分离,裸露出来的第三条链两端的配对碱基重新组合,形成发夹结构使得两端被标记的芘分子彼此之间靠近,芘分子荧光强度升高,从而实现了三磷酸腺苷(adenosine triphosphate,ATP)、α-凝血酶(α-thrombin,Tmb)与L-精氨酰胺(Largininamide,L-Arm)的检测[17]。借助该三链核酸分子开关的思路,后续科研工作者也进行了一系列的参考与改进,用于miRNA的成像等[18]。
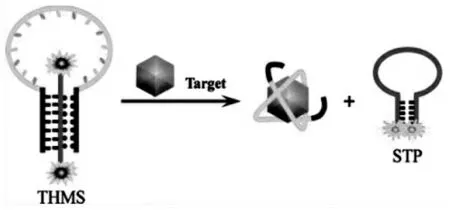
图2 基于适配体三螺旋分子开关的传感平台设计[18]Fig.2 Design of sensing platform based on aptamer triple helix molecular switch[18]
基于上述三链分子探针分子识别与信号报告单元分离的优势,Zheng等将该策略用于细胞内生物标志物分析,他们设计了一种金纳米粒子(AuNPs)集成可编程三螺旋分子开关(THMS),实现从同质溶液到活细胞的多个目标的生物传感[19]。三链分子探针在没有遇到mRNA时,标记的荧光被金颗粒猝灭。遇到靶mRNA时,标记荧光团的核酸链掉落显示荧光。结果表明,这种提出的可编程THMS可成功用于对活细胞中的多信使RNA (mRNA)进行成像,并显着扩展了THMS传感平台的范围(图3)。
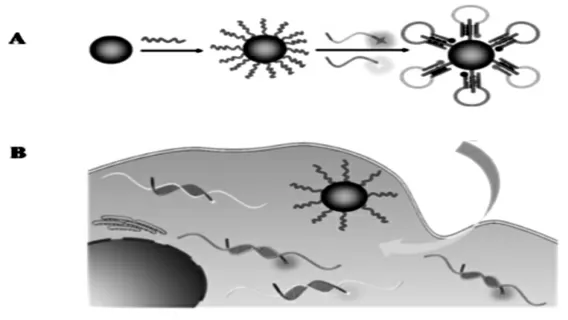
图3 可编程DNA三螺旋分子开关在生物传感中的应用[19]Fig.3 Application of programmable DNA triple helix molecular switch in biosensing[19]
三链分子探针包含三链核酸结构,三链核酸结构的稳定性受pH值调控。基于此,Zheng等[20]设计了一种基于三链核酸结构的DNA四面体纳米探针。与传统的四面体的DNA纳米探针略有不同,三链核酸结构被用作重要的构象转换元件。由于肿瘤细胞中较低的胞外pH值,将靶向mRNA的H1和H2通过Hoogsteen氢键稳定地组装在DNA四面体的延伸短发夹形探针上,形成DNA三链结构。当细胞内pH值和靶mRNA的同时满足时,H1和H2之间触发了杂交链反应(HCR),它们从DNA三链体的解离中释放出来,通过激活生成的长双链DNA上的荧光共振能量转移(FRET)的信号,实现信号的放大,同时指示了目标mRNA的表达,从而达到监测活细胞中的pH和mRNA(图4)。
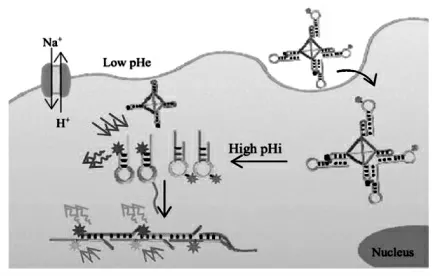
图4 基于三链功能化四面体纳米探针用于细胞内pH和肿瘤相关mRNA的成像[20]Fig.4 Based on triple-stranded functionalized tetrahedral nanoprobe for imaging of intracellular pH and tumorrelated mRNA[20]
基于三链核酸结构受pH调控的原理,Cui等将pH调控策略用于DNAzyme,他们设计了一种酸可切换DNAzyme纳米装置来控制活细胞中的DNAzyme活性,并在原位实现Zn2+、Pb2+同时可视化[21]。这种纳米装置建立在DNAzyme前体(DPs)和酸转换三链DNA(SW-DNA)结构上,在4.5~7.0的pH范围内精确响应。进入细胞之前,DPs的杂交状态成功地使DNAzymes失活。纳米装置进入活细胞中,SW-DNA将在溶酶体强酸环境中,从单链变为三链结构,然后与SW-DNA杂交的链被释放,随后与DPs反应形成活性DNAzyme,可进一步实现细胞内Zn2+、Pb2+同时成像。此外,该策略作为构建各种酸可切换纳米器件的强大平台具有广阔的前景(图5)。
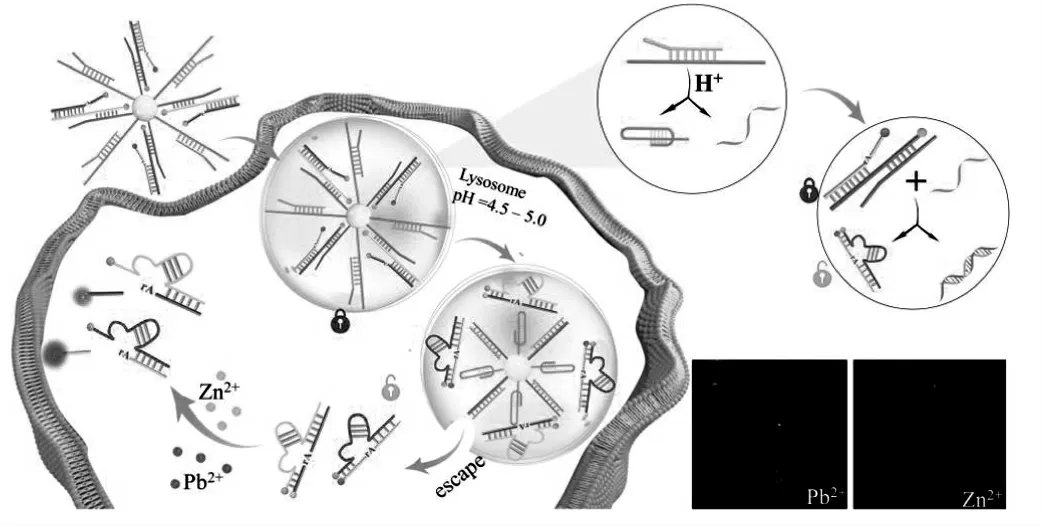
图5 用于对活细胞中的多种金属离子进行成像的酸可切换DNAzyme纳米装置[21]Fig.5 Acid-switchable DNAzyme nanodevice for imaging multiple metal ions in living cells[21]
除了受pH影响,三链核酸结构具备催化功能,Cu2+依赖的DNAzyme需要形成三链核酸结构才具备切割功能。基于此,Kuang课题组[22]构建了一种依赖于手性的圆偏振光(CPL)活化纳米组件,该组件由DNAzyme响应的核酸分子探针和金纳米棒改性的尖晶状铂上转换修饰形成复合型纳米颗粒(UCNPs)修饰而成,该纳米结构不仅具有良好的稳定性,并且能同时定量分析活细胞中的多种二价金属离子。细胞内Zn2+,Mg2+通过DNAzyme形成的双链结构进行响应,而Cu2+通过第三条单链的配对形成三链结构进行响应,从而实现了对细胞内低丰度的三种金属离子的高灵敏检测分析。该检测策略的联合使用不仅拓展了纳米技术的药物递送以及成像研究,而且进一步拓展了三链核酸分子探针与其他技术联用的思路(图6)。
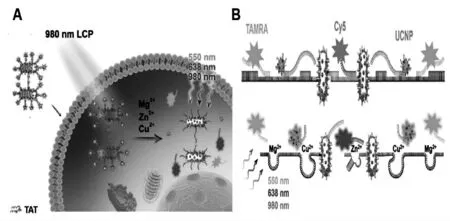
图6 用于细胞内三重离子检测的AuNR@Pt二聚体-UCNP纳米平台[22]Fig.6 AuNR@Pt dimer for intracellular triple ion detection-UCNP nano platform[22]
基于三链核酸的催化功能,Yang等开发设计了一种基于DNA四面体的三维3D捕获器,用于活细胞中长非编码RNA MEG3成像[23],通过三链结构的引入大大加速反应进程。其中3D捕获器由形成三螺旋的dsDNA,能够结合长非编码RNA MEG3的5'端富含GA的结构域和四个针对MEG3上连续结构域的发夹形反义序列组成。一旦被细胞摄取,3D捕获器形成DNA-RNADNA三螺旋结构并触发捕获器的基于杂交的字符串分解来快速捕获长非编码RNA MEG3。由于三螺旋诱导的“多米诺效应”,分解反应时间比无法形成三螺旋的反应要短得多。3D捕捉器可检测长达129个核苷酸的长链靶标,检测限为0.36 nmol/L,为监测内源性RNA提供了可靠的策略(图7)。

图7 三链核酸驱动的3d纳米捕获器用于细胞内长链非编码RNA MEG3的成像[23]Fig.7 Visualization of long noncoding RNA MEG3 in living cells by a triple-helix-powered 3d catcher[23]
2.2 三链分子探针拉曼生化传感应用
C.V.Raman和他的学生发现,单色光照射到物质上发生散射,散射光频率会发生改变的现象[24],其中非弹性散射叫拉曼散射。Jeanmaire等发现分子数量相同时,电极表面的拉曼信号比溶液中的拉曼信号增强了6个数量级,他们指出了增强效应与粗糙的表面有关,称为表明增强拉曼效应[25]。经典的表明增强拉曼平台包括贵金属基底、拉曼报告基团、保护层、目标分子捕获。拉曼信号作为信号输出方式最大的特点就是高灵敏度、对样品无影响。
结合三链分子探针识别单元与信号报告单元分离的优势,利用DNA开关设计用于制造可逆和可再生的拉曼活性底物,出现了以拉曼信号为报告信号的三链分子探针生化传感。如图8所示,基底由Au膜和发夹状DNA链(热点生成探针,HSGP)组成,并用染料官能化的银纳米颗粒(AgNPs)标记。识别特定触发的另一个ss-DNA用作天线。HSGP固定在Au膜上,将染料官能化的AgNP吸引到Au表面附近,并产生强的拉曼信号(SERS)。利用目标物与天线环状区域的特异性亲和,诱导DNA三链结构变化。当HSGP与天线的两个臂段的杂交形成三链核酸结构,染料官能化的AgNP从Au表面分离出来,实现SERS信号分子在Au膜表面的距离发生改变,产生SERS信号传感响应。与其触发器的相互作用将天线从三链结构中释放出来,实现识别单元与信号单元的分离,并恢复HSGP的发夹结构,创建了有效的“关-开”拉曼信号可切换开关[26]。该设计表现出出色的可逆性,以及优良的重现性和表面增强拉曼散射(SERS)效果的可控性,进一步拓展了三链核酸探针在SERS传感方面的实际应用。
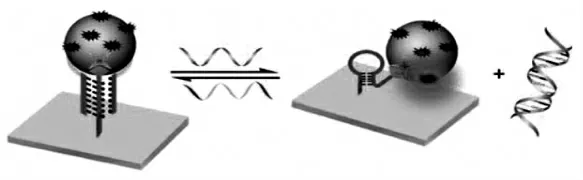
图8 DNA纳米机器导向可逆SERS活性基板的工作原理[26]Fig.8 Working principle of the DNA nanomachinedirected reversible SERS-active substrate[26]
Zheng等利用表面增强拉曼散射现象设计了一种基于适体通用放大信号检测多个目标分析物的方法。该方法使用ss-DNA作为通用触发(UT)来诱导SERS活性热点的形成[27]。SERS活性热点是通过4个氨基苯硫醇(4-ABT)编码的金纳米粒子与DNA聚合物通过特定的Au-S键结合而形成的。适体探针与其靶标之间的相互作用激活自组装从而形成DNA聚合物,三个短DNA探针通过杂交链反应(HCR)的方式干扰三螺旋适体/UT结构。从而实现了对输出信号的放大,同时构建了对包括凝血酶,腺苷和CEM癌细胞的检测成像相应的三链核酸分子探针,该模型的构建及响应的模式进一步激发了SERS的新设计和在生物传感等领域中的应用(图9)。
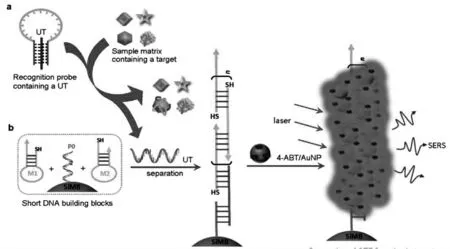
图9 用于检测多个目标分析物的具有放大信号的通用SERS检测器[27]Fig.9 Universal SERS detector with amplified signal for detecting multiple target analytes[27]
2.3 三链分子探针电化学生化传感应用
电化学生物传感器是快速地感应目标物质并将这些物质的浓度与电流/电压关联起来的传感器。具有成本低,灵敏度高,操作简单等优点。在过去几十年里,构建了许多种分析平台实现目标物的检测。传统的电化学传感器使用单一电流信号输出,易受到背景干扰。为避免干扰,发展了比率型电化学传感器。Xiong等开发了电化学EDNA生化传感器用于目标DNA(T-DNA)检测[28]。用亚甲基蓝(MB-DNA)标记的发夹DNA捕获探针通过Au-S键锚定在金电极上。接着用两端标记了二茂铁(Fc-DNA)的单链DNA形成三螺旋构象。当T-DNA存在时,Fc-DNA与T-DNA杂交,三螺旋结构解开并使MB-DNA恢复到其发夹结构。Fc标记物从GE电极表面扩散开,而MB标记物仍然贴在它附近,导致Fc峰值电流降低和MB峰值电流增加。IMB/IFc值与T-DNA在0.5至80 pmol/L呈线性关系,检测限低至0.12 pmol/L。该生化传感器在电化学检测多种分析物方面具有巨大潜力,可作为早期临床诊断和生物医学研究的生物传感平台(图10)。
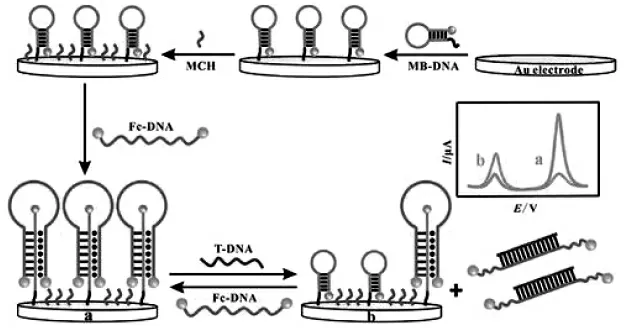
图10 用于核酸超灵敏检测的三螺旋分子开关电化学比例生物传感器[28]Fig.10 Triple-helix molecular switch Electrochemical Ratiometric Biosensor for Ultrasensitive[28]
相比于传统的电化学传感策略,Yang等结合三链分子探针分子识别单元和信号报告单元分离的优势,利用纳米孔技术设计了三链分子探针(TMS)用于阿尔兹海默症生物标志物分析[29]。实验中设计并合成了中间链长度不同的三个钳形TMS,三个TMS环部序列分别为靶标检测物Tau 381、AAT、BACE 1的核酸适配体序列,茎部设计为三链核酸序列。在没有遇到靶标检测物时,TMS体积较大,不能穿过纳米孔。遇到靶标检测物TMS解开,释放中间的单链DNA。单链DNA体积小可以穿过纳米孔,当不同穿过纳米孔的单链DNA序列长度不一致时,产生不同的阻断电流用来分析靶标物。利用三链核酸分子开关分子识别单元与信号报告单元分离的优势,实现了Tau 381、AAT、BACE1三种物质的同步正交分析(图11)。
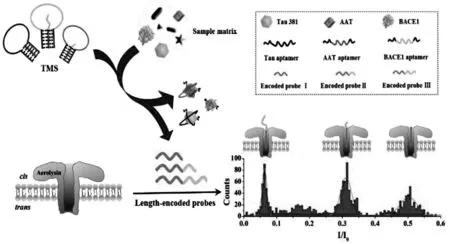
图11 基于长度编码的三链分子开关纳米孔的阿尔兹海默症生物标志物同步分析[29]Fig.11 Synchronous analysis of alzheimer's disease biomarkers based on length-encoded triple-stranded molecular switch nanopores[29]
3 结论与展望
与传统的核酸探针相比,三链核酸探针具有特殊的稳定性。三链核酸探针自身稳定性受第三条链长度、碱基序列、碱基修饰、温度和pH值、离子浓度的影响。三链核酸探针具有探针识别单元与信号报告单元分离、受pH调控、催化功能等优势。结构可调控,有效避免了实际体系中出现的假信号。在实际体系疾病相关标志物检测中,三链核酸探针提供新的生物传感方法。近年来诊疗一体化的理念被提出来,国内外研究人员构建了用于生物标志物成像、治疗的核酸分子探针。但在生物体系应用过程中,核酸分子探针被核酸酶切割出现假信号,出现了一系列核酸分子探针稳定性增强策略。三链分子探针具有超螺旋结构,相比于普通分子探针可能具有更好的生物稳定性。随着研究的深入,三链核酸探针将成为生物传感领域不可缺少的一部分。

