基于PCR-DGGE分析宣威火腿的细菌群落结构
邹颖玲,刘姝韵,王桂瑛,*,普岳红,葛长荣,廖国周,*
(1.云南农业大学食品科学技术学院,云南昆明650201;2.云南农业大学云南省畜产品加工工程技术研究中心,云南昆明650201)
宣威火腿是云南省传统名特优产品,其以“身穿绿袍,色香味美,咸淡相宜,风味独特,食而不腻”等特点名扬海内外,距今已有两百七十多年的历史[1],它是由原产地域内饲养的含有乌金猪血统的鲜猪后腿在原产地经过腌制、发酵、成熟制作而成[2]。宣威火腿名气取决于其特定的品质,而火腿特定品质的形成又取决于宣威独特的地理气候环境[3]。境内独特的天然环境,促进火腿微生物的大量繁殖,从而保证火腿的持续发酵[4]。微生物在肉制品的风味形成和安全性方面发挥着各自独特的作用,在发酵过程中,发酵微生物会引起原料中的蛋白质、脂肪等主要组分发生微生物及生物化学变化,进而影响发酵肉制品的品质,促进其产生芳香化合物,提升肉制品的风味。并且能通过抑制有害微生物的生长繁殖减少盐用量、降低亚硝酸盐残留和生物胺含量[5]。
聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophorsis,PCR-DGGE)是一种分析微生物区系的分子指纹技术,能够快速、高效、全面地分析微生物的组成及群落结构,是细菌分类和鉴定的有效工具[6-9]。与传统培养方法相比,PCR-DGGE技术不需培养,而是直接从样品中提取总DNA,不仅能检测到难培养或不能培养的微生物,而且能同时检测多种微生物,耗时短。该技术虽然在肉制品微生物方面起步较晚,但近年来广泛应用于原料肉及各种肉制品微生物群落结构的分析[10-11]。目前,运用免培养的分子生物学技术研究宣威火腿微生物区系鲜有报道,基于此,本研究旨在通过PCRDGGE技术分析不同加工年份(1年、2年和3年)的宣威火腿细菌群落结构及多样性,为进一步探讨宣威火腿品质奠定基础。
1 材料与方法
1.1 材料
1.1.1 样品来源
宣威火腿样品:云南省宣威市某公司提供,并分别在加工1年、2年和3年火腿的表面和内部(距肉表面 5 cm)取约 10 g肉样品,标记为:A1、A2(1 年腿的表面和内部),B1、B2(2年腿的表面和内部),C1、C2(3年腿的表面和内部),将6个样品真空包装,置于-20℃保存以待分析。
1.1.2 试剂及仪器
乙醇、冰醋酸、硝酸银、37%甲醛、氢氧化钠:国药集团化学试剂有限公司;十六烷基三甲基溴化铵环境微生物DNA提取试剂盒、1×TAE缓冲液、ddH2O:北京博友顺有限公司;DNA纯化试剂盒、DH 5α感受态细胞:北京天根生化科技公司;dNTP、rTaq、pMD18-T Cloning Kit:日本Takara公司;40%甲叉双丙烯酰胺:美国 Bio-Rad公司;甲酰胺(去离子)、Urea、APS:美国Amresco公司;四甲基乙二胺:美国Sigma公司;DNA纯化试剂盒:美国OMEGA公司。
Centrifuge 5415D离心机:德国Eppendorf公司;T-gradient PCR仪:德国Biometra公司;SDC-6恒温槽:宁波新芝有限公司;Gel-Doc2000凝胶成像仪、Dcode变形梯度凝胶电泳仪:美国伯乐Bio-Rad公司;JYSPFT十六烷基三甲基溴化铵:北京军意东方仪器有限公司。
1.2 方法
1.2.1 样品中总DNA的提取
具体操作步骤按照十六烷基三甲基溴化铵环境微生物DNA提取试剂盒进行,所得DNA保存于-20℃待分析。
1.2.2 细菌16S rDNA片段的PCR扩增
参考赵乾宇[12]的方法,以样品基因组DNA为模板,采用细菌通用引物GC-338F和518R扩增样品16S rDNA高变区序列。所用引物序列如表1所示。

表1 引物信息Table 1 Primer information
PCR扩增体系(50μL)为:10×PCR缓冲液5μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;GC-338F(20 mmol/L)1 μL;518R(20 mmol/L)1 μL;模板 DNA 50 ng;用 ddH2O 补至 50 μL。
PCR扩增程序为:94℃预变性5 min;94℃变性1 min,55℃复性 45 s,72℃延伸 1 min,30个循环;最终72℃延伸10 min。
PCR产物采用DNA纯化试剂盒纯化回收。
1.2.3 PCR产物的DGGE分析
参考汤玲娟等[13]的方法,并适当调整。取10 μL PCR的产物进行变形梯度凝胶电泳(DGGE)分析。采用变形梯度为35%~55%、浓度为8%的聚丙烯酰胺凝胶(化学变性剂为100%尿素7 mol/L和40%的丙烯酰胺)在1×TAE缓冲液中150 V 60℃下电泳5 h。
1.2.4 DGGE图谱中优势条带的回收与测序
参考郑艳等[14]的方法,用灭菌的手术刀切下待回收DGGE条带,采用DNA纯化试剂盒回收目的条带。以2 μL回收产物为模板,338F/518R为引物进行PCR扩增。PCR扩增体系(50 μL)为:10×PCR缓冲液 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq (5 U/μL)0.4 μL;338F(20 mmol/L)1 μL;518R(20 mmol/L)1 μL;模板 DNA 2 μL;补ddH2O至50 μL。PCR扩增程序为:94℃预变性 4 min;94℃变性 30 s,55℃复性 30 s,72℃延伸30 s,30个循环;最后,72℃延伸10 min。将重新扩增的DNA片段切胶回收、纯化后,连接到Pmd18-T载体上,并转化至DH5α感受态细胞中,筛选阳性克隆,菌液由华大基因对插入的细菌16S rDNA片段进行序列测定。
1.2.5 细菌多样性指数计算
细菌多样性指数是研究群落物种数和个体数以及均匀度的综合指标。根据电泳图谱中样品条带数目及每个条带的强度(灰度),对各样品中细菌多样性指数、均匀度和丰富度等指标进行分析。DGGE图谱采用Quantity one软件对每个样品的电泳条带数目、条带密度进行数字化分析,香农指数(H)、丰度(S)和均衡指数(E)等指标被用来比较不同样品的多样性情况[15]。参考郑艳等[14]的方法进行计算,计算公式如下所示。

式中:pi为样品中单一条带的强度在该样品所有条带总强度中所占的比率;N为DGGE图谱单一泳道上所有条带的丰度;Ni为第i条带的丰度;S是某样品中所有条带数目总和。
2 结果与分析
2.1 宣威火腿细菌DNA提取结果
宣威火腿样品细菌DNA电泳图见图1。
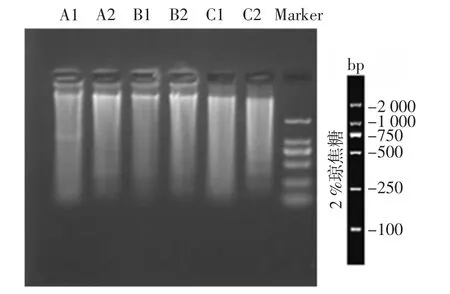
图1 宣威火腿样品细菌DNA电泳图Fig.1 Electrophoresis of bacterial DNA from Xuanwei ham samples
由图1可以看出,从不同加工年份宣威火腿表面和内部提取出的细菌DNA片段完整性较好,有利于后续的PCR扩增。
2.2 细菌16S rDNA的PCR扩增
宣威火腿样品细菌组16S rDNA的PCR扩增结果见图2。
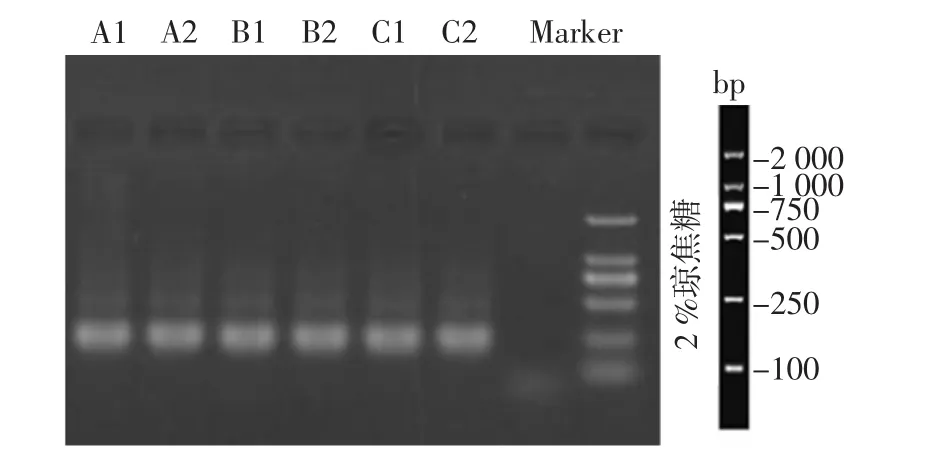
图2 宣威火腿样品细菌组16S rDNA的PCR扩增结果Fig.2 PCR amplification results of 16S rDNA of bacterial group of Xuanwei ham sample
以GC-338F和518R为引物,从不同加工年份宣威火腿的表面和内部6个样品中提取的总DNA为模板进行16S rDNA片段扩增,得到200 bp的DNA片段(图2),所有样品条带清晰,符合DGGE分析。
2.3 DGGE指纹图谱及分析结果
宣威火腿样品的DGGE图谱见图3,宣威火腿样品的DGGE条带强度示意图见图4。
用DGGE图谱中条带数来反映细菌多样性,泳道中各条带均代表一种不同的细菌,条带亮度用来反映细菌的丰富度[16]。由图3和图4可以看出,明显分离的条带有40条,显示出宣威火腿中丰富的细菌种类,其中1年腿表面有16个主要条带,内部有16个;2年腿表面有23个,内部有15个;3年腿表面有29个,内部有22个,多数条带为每个泳道共有,说明有共有的细菌类型存在于不同加工年份宣威火腿表面和内部。另外共有条带的亮度也存在一定的差异,说明其中细菌群落数量存在差异。

图3 宣威火腿样品的DGGE图谱Fig.3 DGGE map of Xuanwei ham samples

图4 宣威火腿样品的DGGE条带强度示意图Fig.4 Schematic diagram of DGGE band intensity of Xuanwei ham samples
2.4 主要电泳条带的测序比对结果分析
主要条带的序列测定结果见表2。

表2 主要条带的序列测定结果Table 2 The sequencing results of the main bands

续表2 主要条带的序列测定结果Continue table 2 The sequencing results of the main bands
将宣威火腿样品主要的40条电泳条带进行克隆与测序,测序结果与GenBank中的序列进行比对,从中获取各个同源性信息。由表2可知,有1条条带未测出结果,测出的39条回收带有37条的同源性较高,在95%以上。
由DGGE图谱可知,宣威火腿中主要的细菌有39种,较为丰富。加工1年宣威火腿表面最亮的条带为37和39,其中条带37为Chromohalobacter,条带39为Chromohalobacter marismortui。加工1年宣威火腿内部最亮的条带为 14、17、21、26、37 和 39,其中条带 14 为Staphylococcus equorum(马胃葡萄球菌)、条带17为Staphylococcus xylosus(木糖葡萄球菌)、条带21为Acinetobacter junii(琼氏不动杆菌)、条带 26为Tetragenococcus halophilus(嗜盐四联球菌)。加工2年宣威火腿表面最亮的条带为14、16和26,其中条带16为Staphylococcus warneri(沃氏葡萄球菌)。2年宣威火腿内部最亮的条带为14、17、19、26和39,其中条带19为Staphylococcus epidermidis(表皮葡萄球菌)。加工3年宣威火腿表面最亮的条带为 3、4、6、7、8、9、12、14、26和27,其中条带3为未培养细菌、条带4为Staphylococcus carnosus(肉葡萄球菌)、条带 6为Staphylococcus saprophyticus(腐生葡萄球菌)、条带7为Staphylococcus pentosaceus(戊糖葡萄球菌)、条带8为Staphylococcus arlettae(阿尔莱特葡萄球菌)、条带9为Staphylococcus simulans(模仿葡萄球菌)、条带12为Chromohalobacter japonicus、条带 27为 Lentibacillus salarius。加工3年宣威火腿内部最亮的条带为19、21、25和26,其中条带25为未培养细菌。由DGGE图谱可知,不同加工年份宣威火腿的表面及内部均检测出Tetragenococcus halophilus(嗜盐四联球菌),由此可推断,Tetragenococcus halophilus(嗜盐四联球菌)是宣威火腿的优势菌种。不同加工年份宣威火腿的表面及内部检测出了大量的马胃葡萄球菌、木糖葡萄球菌、表皮葡萄球菌、戊糖葡萄球菌等葡萄球菌属,由此可看出,葡萄球菌属是宣威火腿的优势菌属。另外,在宣威火腿中还检测出了3种未培养细菌,其在宣威火腿中的作用有待进一步研究。
2.5 样品间细菌群落结构相似性指数与聚类分析
戴斯系数比较PCR-DGGE图谱的相似性结果见表3。
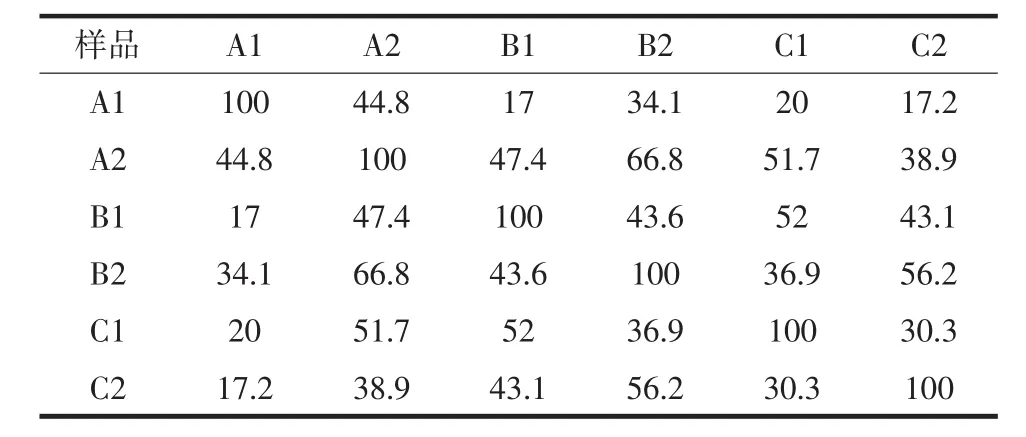
表3 戴斯系数比较PCR-DGGE图谱的相似性Table 3 Comparison of the similarity of PCR-DGGE spectra by the Dyce coefficient %
DGGE图谱的相似性是通过Quantity One软件进行分析的,通过分析得到不同加工年份宣威火腿细菌群落相似性指数。由表3可知,加工1年宣威火腿内部与加工2年内部的细菌群落结构相似性最高,为66.8%,加工1年表面与加工2年表面的细菌群落结构相似性最低,为17%。从宣威火腿表面看,加工2年的与加工3年表面细菌群落结构相似性为52%,分别比加工1年与加工2年表面细菌群落结构相似性和加工1年与加工3年表面细菌群落结构相似性高35%、32%。从宣威火腿内部看,加工1年与加工2年内部细菌群落结构相似性分别比加工1年与加工3年内部细菌结构相似性和加工2年与加工3年内部细菌结构相似性高27.9%、10.6%。图5为不同加工年份宣威火腿表面及内部的条带相似性聚类分析图。

图5 宣威火腿样本间的聚类图Fig.5 Cluster diagram between Xuanwei ham samples
由图5可知,加工1年、2年和3年宣威火腿表面及内部的细菌群落按相似程度分为3类,加工1年宣威火腿表面的细菌群落为一类,加工2年和加工3年表面的细菌群落聚成一类,3个不同加工年份宣威火腿内部的细菌群落聚为一类,说明加工时间对宣威火腿表面及内部的细菌群落有一定的影响。
2.6 宣威火腿样品的细菌群落多样性
宣威火腿各样品间的细菌群落多样性分析见表4。
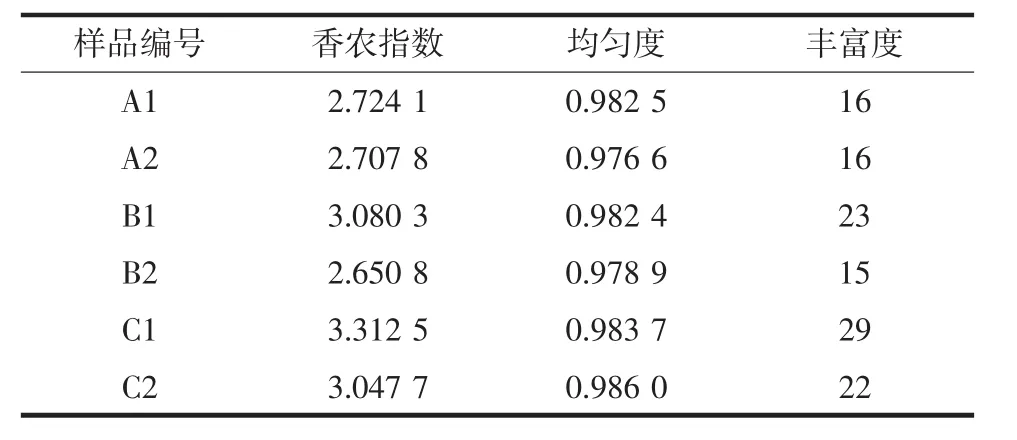
表4 宣威火腿各样品间的细菌群落多样性分析Table 4 Analysis of bacterial community diversity among different samples of Xuanwei ham
微生物多样性指数用于评估微生物群落的丰富度。香农指数(H)用于评估微生物群落的多样性,H越大,说明微生物群落多样性越高;均匀度指数(E)用于评估微生物分布的均匀程度,E值越大,说明微生物群落分布越均匀;丰富度指数(S)是指一个群落或生境中物种数目的多寡[17]。由表4可看出,不同加工年份宣威火腿表面的香农指数、均匀度指数和丰富度指数均高于内部的数值,说明不同加工年份宣威火腿表面及内部细菌群落多样性有差异。加工3年宣威火腿表面的香农指数、均匀度指数和丰富度指数最大,说明其样品的细菌群落种类和数量最多,且较均匀。
3 讨论
干腌火腿作为传统发酵食品,微生物是其品质特征形成的一个重要影响因素。大量研究表明,微生物与火腿发酵有着密切的关系。参与火腿发酵的微生物群体主要包括细菌、霉菌、酵母,不同的菌群对火腿的发酵起着至关重要的作用,如细菌中葡萄球菌和微球菌等。细菌在发酵肉制品中起着重要的作用[18],球菌在发酵肉制品中充当还原亚硝酸盐和形成过氧化氢酶的角色,利于发色及分解过氧化物,改善产品色泽及延缓酸败,此外球菌可以分解蛋白质和脂肪分泌脂肪酶和蛋白酶,改善产品风味[19]。Essid等[20]研究发现,从Tunwasian传统腌肉中分离出30株木糖葡萄球菌均具有过氧化氢酶活性,能使硝酸盐、亚硝酸盐含量减少。李平兰等[21]研究发现宣威火腿中的优势菌群为葡萄球菌和微球菌、霉菌。耐盐性的葡萄球菌和微球菌的代谢活动以及火腿表面大量霉菌的生长是宣威火腿独特风味形成的基础。
本研究运用PCR-DGGE分析不同加工年份宣威火腿表面和内部的细菌群落结构,研究结果表明,葡萄球菌属和Tetragenococcus halophilus(嗜盐四联球菌)分别是宣威火腿的优势菌属与优势菌种,这与黄艾祥等[22]的研究结果相似。Hinrichsen L等[23]研究意大利干腌火腿加工过程中微生物区系的变化发现,表皮和肌肉中不同的发酵阶段会有不同的优势菌群,但最终成熟的火腿表面和内部都是葡萄球菌为优势菌,这与试验的结果相一致。葡萄球菌可将NO3-转化为NO,进一步与肌红蛋白结合,形成亚硝基肌红蛋白,使肉品呈现其特有的色泽[24]。葡萄球菌还能去除肉制品中乳酸代谢产生的H2O2,促进芳香类风味的形成[25]。另外,葡萄球菌具有耐盐性,且有盐的环境有利于其生长,这就造成了葡萄球菌在火腿表面是优势菌[26]。另外,本研究还检测出了未培养细菌,表明PCR-DGGE技术相比于传统培养法更精确,同时该技术会受环境因素、PCR扩增引物的偏好性等因素的影响,从而影响该技术的准确性。因此还需结合环境因素对宣威火腿细菌群落结构及多样性研究进行深入、全面的分析。
4 结论
从不同加工年份宣威火腿中共检测到了39种细菌,存在于宣威火腿中的细菌主要为马胃葡萄球菌、木糖葡萄球菌、表皮葡萄球菌、戊糖葡萄球菌等葡萄球菌属以及Tetragenococcus halophilus(嗜盐四联球菌)、Microccccus varians(变异微球菌)等,其中葡萄球菌属和Tetragenococcus halophilus(嗜盐四联球菌)分别为宣威火腿中的优势菌属与优势菌种;此外,在宣威火腿中还检测出了未培养细菌。多样性分析表明,加工3年的宣威火腿表面细菌群落多样性最高,加工1年火腿表面细菌群落多样性最低。加工3年火腿内部细菌群落多样性最高,加工2年火腿内部细菌群落多样性最低。相较于传统的分离培养法,采用PCRDGGE技术分析宣威火腿的细菌菌落结构,结果更全面、更直观,这也为进一步研究宣威火腿独特风味的形成提供了技术支持。