昌化江河谷隔离对海南岛特有植物盾叶苣苔遗传多样性的影响
李歌 凌少军 陈伟芳 任明迅 唐亮

摘 要: 为评估盾叶苣苔的遗传多样性与遗传分化格局,探索影响盾叶苣苔遗传变异地理分布的因素,该研究采集盾叶苣苔(Metapetrocosmea peltata)11个种群172份材料,通过PCR扩增和测序分析核糖体转录间隔区(ITS)序列的变异式样。结果表明:(1)盾叶苣苔物种水平的遗传多样性很高(HT=0.998, π=0.023 5),种群间基因流很弱(Nm=0.04)且存在强烈的遗传分化(GST=0.375)。(2)单倍型分析显示,盾叶苣苔的单倍型大多是种群特异的,仅白马岭与南茂岭种群有共享单倍型。(3)Mantel test表明,遗传距离和地理距离存在一定相关性(相关系数r=0.322,P=0.010)。(4)Structure聚类分析将盾叶苣苔划分为6种遗传成分,其地理分布与昌化江河谷导致的隔离样式基本一致,基于Nei遗传距离的种群聚类分析支持這一结果,显示盾叶苣苔遗传多样性的分布受到昌化江河谷的隔离作用。(5)AMOVA分析确定67%的变异来自地区间,表明地理隔离是盾叶苣苔种群分化的重要因素。这表明昌化江及其支流所引起的海南岛山地内部隔离是盾叶苣苔种群发生强烈遗传分化的重要原因,从而导致盾叶苣苔在物种水平具有较高的遗传多样性。上述研究结果为海南特有苦苣苔资源盾叶苣苔的保护和可持续利用提供了理论指导,将有助于理解海南岛特有植物和其他海岛植物遗传变异的地理分布及其影响因素。
关键词: 盾叶苣苔, 遗传多样性, 遗传结构, 地理隔离, 岛屿特有种
中图分类号: Q948.15
文献标识码: A
文章编号: 1000-3142(2020)10-1505-09
Effects of geographic isolation caused by Changhua River on genetic diversity of Hainan-endemic Metapetrocosmea peltata (Gesneriaceae)
LI Ge1, LING Shaojun1, CHEN Weifang1, REN Mingxun1, TANG Liang1,2*
( 1. Research Center for Terrestrial Biodiversity of South China Sea, Institute of Tropical Agriculture and Forestry, Hainan University, Haikou 570228, China; 2. Institute of Tropical Rain Forest National Park, Hainan University, Haikou 570228, China )
Abstract: Metapetrocosmea peltata (Gesneriaceae) is an endemic species distributed in the middle and southern mountainous area of Hainan Island. In this study, we collected 172 M. peltata individuals from 11 natural populations, and assessed the genetic diversity and differentiation of this species based on sequence variation of intergenic transcribed spacer (ITS) region. In addition, the factors that may impact the level and distribution of genetic diversity were explored. The results were as follows: (1) M. peltata had a high level of genetic diversity at its species level (HT=0.998, π=0.023 5), however, the gene flow between populations was very weak (Nm=0.04), whereas the genetic differentiation was quite strong (GST=0.375). (2) Most haplotypes of M. peltata were population specific, only except for BM and NM populations, which shared a few haplotypes. (3) Mantel test showed that the genetic distances between haplotypes were correlated with their geographic distances (correlation index r=0.322, P=0.010). (4) Structure analysis clustered individuals of M. peltata into six genetic groups, whose geographic distributions were largely concordant with the isolation pattern of Hainan mountainous area caused by Changhua River. The geographic distribution of genetic clusters was further supported by population cluster analysis based on Neis genetic distance. This study manifests the isolation effect of Changhua River on the distribution of genetic diversity of M. peltata. (5) AMOVA analysis showed that 67% genetic variation was partitioned among populations, suggesting genetic differentiation of this species was mainly caused by geographic isolation. In conclusion, geographic isolation of Hainan mountainous area caused by Changhua River and its tributaries should be the driving force for the strong genetic differentiation among M. peltata populations, and also may lead to the high genetic diversity detected at the species level of M. peltata. This study not only provides recommendation for efficient conservation and utilization of M. peltata, a potential ornamental plant species, but also facilitates the understanding of geographic distribution of genetic variation for Hainan Island endemic and other plant species.
Key words: Metapetrocosmea peltata, genetic diversity, genetic constructure, geographic isolation, island endemic species
岛屿物种的分布区通常比较狭小,种群规模小,基因流受岛屿复杂地形的影响而减弱或阻断,是研究种群遗传分化的理想材料(王中生等, 2004)。岛屿的气候、地形等因素会影响种群的大小与分布,进而影响其遗传多样性与遗传结构(Stuessy et al., 2014)。海南岛具有明显的地形特征,五指山和鹦哥岭为海南最高峰,以其为中心向外围海拔逐渐降低(邢福武, 2012)。昌化江为海南第二大河,它发源于琼中空禾岭,横贯海南岛的中西部,自东北向西南流经琼中五指山,在乐东县转向西北,经东方市和昌江县向西汇入北部湾(周祖光, 2015)。昌化江支流众多,将海南岛中南部山地分割为不连续的地理区域,这种地形特点对海南特有植物遗传多样性和遗传分化的影响目前并没有得到深入研究。
苦苣苔科(Gesneriaceae)植物大多具有独特的花形与花色和良好的耐荫性,观赏价值极高,因而受到广泛关注(温放, 2008; 代正福和罗冠勇, 2009; 史佑海等, 2011)。中国苦苣苔科植物均属于苦苣苔亚科,海南岛及其附近地区是苦苣苔亚科的多样性热点地区之一(李振宇和王印政, 2005; 韦毅刚等, 2010; 许为斌等, 2017)。海南岛共有24种苦苣苔亚科植物,包括2个特有属和8 个特有种(含1变种)(韦毅刚等, 2010; 邢福武, 2012)。昌化江及其支流是导致海南岛中南部山地彼此隔离的重要因素,海南苦苣苔科植物的起源与进化很可能受到昌化江河谷以及岛屿地质历史的影响(王策等, 2015; 凌少军等, 2017)。
盾叶苣苔(Metapetrocosmea peltata)隶属于苦苣苔科盾叶苣苔属,为单种属,是海南特有种,通常生长在海拔400~800 m的湿润岩壁上或石壁夹缝中,种群呈斑块分布,对生境要求严格。近年来,由于气候变化、局部地区干旱加剧、溪流干涸,导致盾叶苣苔部分种群缩减甚至灭绝,现已被列入易危物种名单(韦毅刚等, 2010)。盾叶苣苔现存种群的遗传变异是否充足,昌化江引起的海南岛中南部山地隔离是否影响了盾叶苣苔遗传变异的地理分布,如何有效地保护盾叶苣苔的遗传多样性,这些问题尚未得到解答。核糖体转录间隔区(ITS)替换速率快、测序难度小,常用于近缘类群的系统发育与种群遗传学研究(段义忠等, 2010; 王晓雄等, 2011)。本研究选用ITS标记分析海南特有种盾叶苣苔的种群遗传多样性与遗传结构,探究海南岛地理格局对盾叶苣苔种群遗传变异水平与空间分布的影响,为海南特有种的起源、进化和保育提供理论基础。
1 材料与方法
1.1 材料
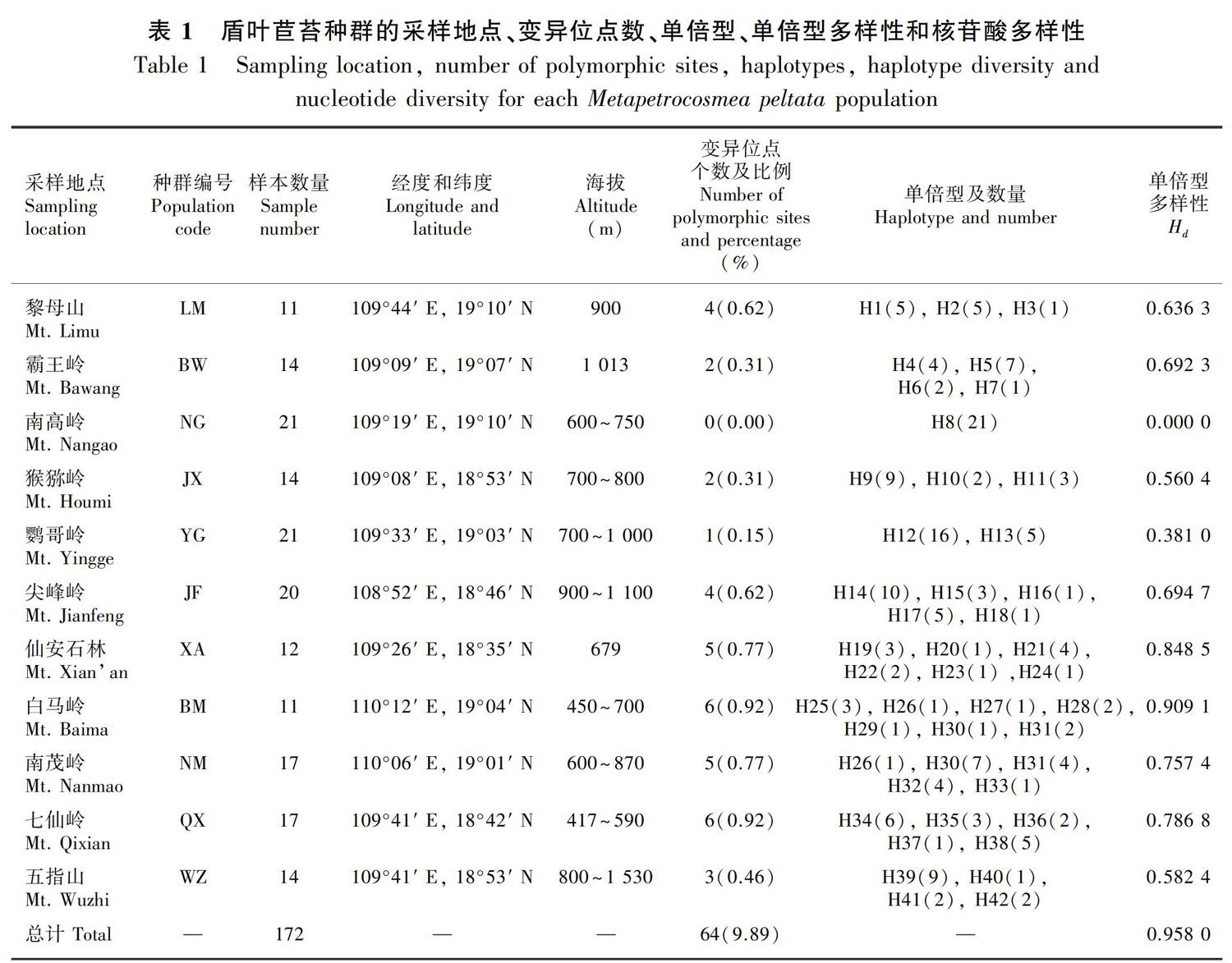
本研究采集了盾叶苣苔11个种群共172份样本,采样点覆盖该种在海南岛的主要分布地点(韦毅刚, 2010)。野外采集生长良好、无病斑的幼叶,用硅胶快速干燥后带回实验室,每种群采集11~21个体(表1)。昌化江及其支流将这11个种群分割在3块不连续的区域中,其中猕猴岭(JX)、鹦哥岭(YG)、霸王岭(BW)、南高岭(NG)与黎母山(LM)种群位于昌化江以北侧,尖峰岭(JF)与仙安石林(XA)种群大体位于昌化江的西南,其余种群分布在昌化江的东南。同侧种群被沟谷与河流进一步分割(图1)。
1.2 DNA提取和PCR扩增、测序
采用改良的CTAB法 (Doyle, 1990),从硅胶干燥的叶片中提取总DNA。由通用引物ITS4(5′ TCCTCCGCTTATTGATATGC 3′)和ITS 5HP(5′ GGAAGGAGAAGTCGTAACAAGG 3′)(White et al., 1990)扩增盾叶苣苔的ITS片段。
PCR扩增体系为25 μL,包括17 μL去离子水,2.5 μL 10× 缓冲液,0.5 μL 10 mM dNTps,5 μmol·L-1引物各0.5 μL,1 μL DNA模板和0.5 μL 5 U·μL-1 Taq酶。ITS片段扩增程序:94 ℃预变性4 min;94 ℃变性1 min,55 ℃退火40 s,72 ℃延伸1 min,循环36次,最后72 ℃延伸10 min。PCR产物经琼脂糖凝胶电泳检测后送广州天一辉远生物科技有限公司测序。
1.3 数据统计分析
先用ContigExpress 2014对测序结果手工校正,然后通过MEGA 6.0 (Kumar et al., 2008)程序构建序列比对。用DnaSP 5.1 (Librado & Rozas, 2009)确定单倍型,并计算单倍型多样性(Hd)、核苷酸多样性(π)、种群间平均基因流(Nm)和居群间遗传分化系数(FST)。采用PERMUT (Pons & Petit, 1996)软件计算种群内平均遗传多样性
(HS)、物种水平总遗传变异(HT),以及遗传分化系数(GST和NST)。GST只考虑单倍型的频率,NST考慮了单倍型的频率和序列差异(Raymond & Rousset, 1995)。NST大于GST通常表明种群存在谱系地理结构,其显著性通过1 000次置换进行检验。使用GenAlEx 6.5 (Peakall & Smouse, 2012)进行AMOVA (Analysis of Molecular Variance)分析,将总遗传变异在种群内、种群间与地区间三个层次上进行剖分。用Mantel检验(Mantel, 1967)比较盾叶苣苔种群遗传距离与地理距离之间的相关性,通过10 000次随机重复检验显著性。采用Network 5.0 (Bandelt, 1999)的中点连接法(Median-joining Networks)对盾叶苣苔11个种群构建单倍型网络图。种群遗传结构用Structure 2.3.4分析,MCMC链起始的105世代不计入似然值计算,随后重复抽样106世代。K代表遗传成分的种数,其取值范围设置为1~11,对于每个K重复10次聚类分析,将10次运算的平均似然值作为每个K的似然值。聚类模型假定种群可以混合,且等位基因频率是相关的(Pritchard et al., 2000)。使用Structure Harvester进行DeltaK作图分析,以确定最优K值,即最可能的遗传成分的数目(Evanno et al., 2005)。将Structure输出结果上传至CLUMPAK server绘制遗传结构图(Jakobsson & Rosenberg, 2007)。基于Nei遗传距离使用MEGA 6.0中的邻接法构建盾叶苣苔地理种群的聚类关系图(Tamura et al., 2013)。
2 结果与分析
2.1 序列变异
经比对,ITS序列的长度为647 bp,一共检测到64个变异位点,包括7个碱基插入缺失突变。种群BM和QX的变异位点比例最高,为0.92%;种群NG的ITS序列没有变异;其余种群的变异位点比例在0.15%~0.77%之间(表1)。
2.2 单倍型多样性与地理分布
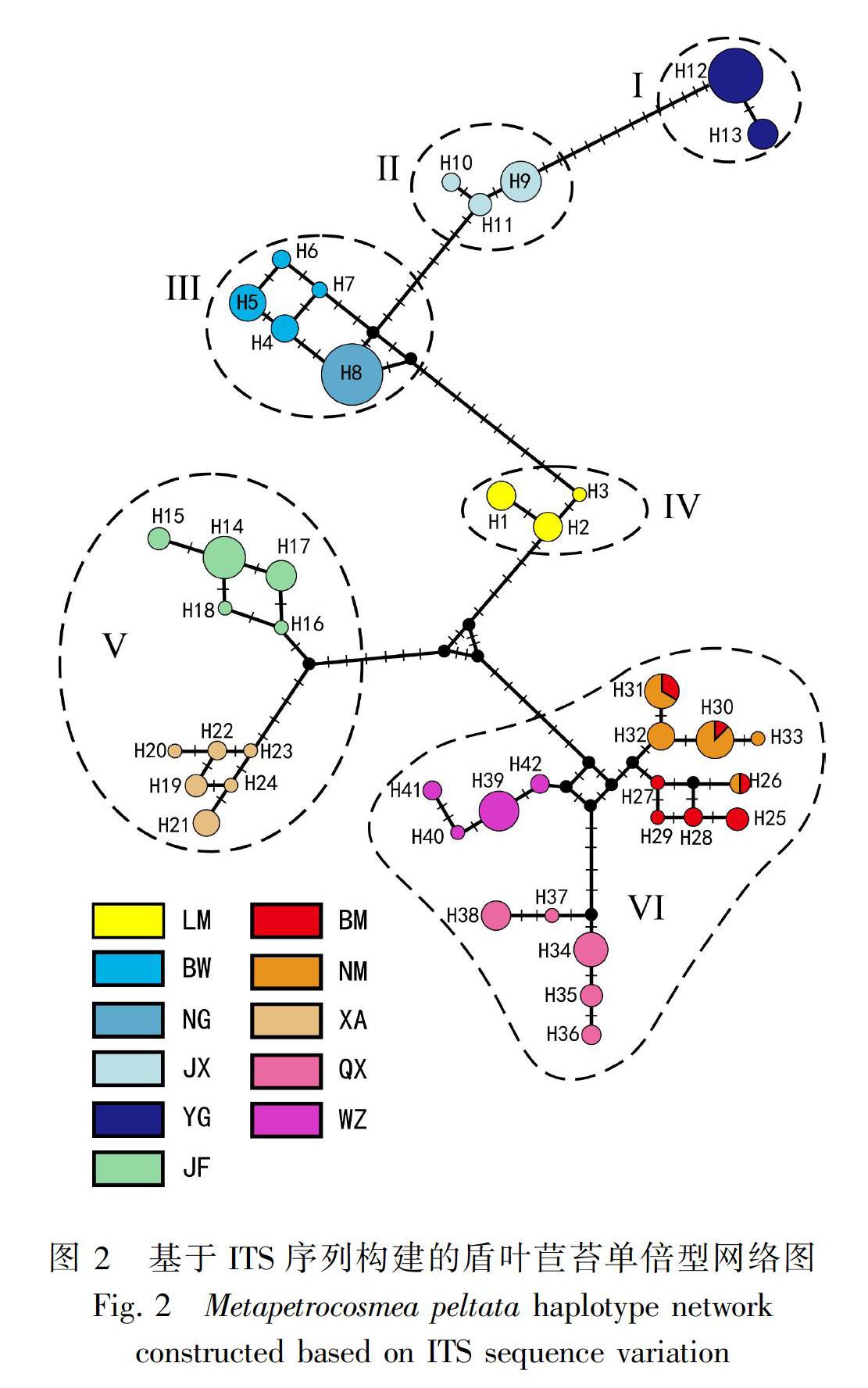
基于ITS序列共发现42个单倍型,盾叶苣苔种群的单倍型个数从1~7不等(表1)。大部分种群拥有独特的单倍型,只有地理距离较近的种群BM和NM共享了单倍型H28、H30和H31。盾叶苣苔种群的单倍型多样性较高,除种群NG仅有1种单倍型外,其余种群均有2种以上的单倍型,单倍型多样性在0.381 0~0.909 1之间。
盾叶苣苔的单倍型网络具有清晰的地理结构。不同种群的单倍型在网络图上处于相对独立的位置,地理距离近的种群其单倍型的遗传距离也较近(图2)。根据单倍型网络可以大致将盾叶苣苔的11个采样种群划分为6组:Ⅰ: YG;Ⅱ: JX;Ⅲ: NG与BW;Ⅳ: LM;Ⅴ: JF与XA;Ⅵ: BM、NM、QX与WZ(图2)。组内单倍型的遗传距离(0.004±0.003)远小于组间单倍型的遗传距离(0.026±0.006)。
2.3 种群遗传结构与遗传分化
DeltaK作图分析发现K=6时DeltaK取得最大值(图3:A)。盾叶苣苔6种遗传成分的地理分布与基于单倍型网络划分的6组种群完全对应(图3:B)。位于昌化江以北的5个种群,除BW与NG共享同一成分(Ⅲ),其余种群均由独特遗传成分构成。位于昌化江西侧及南侧的JF与XA由成分V构成,位于昌化江东南的4个种群(QX、WZ、NM与BM)由成分VI构成(图3:B)。这6种遗传成分的地理分布与海南岛中南部山地的隔离样式基本吻合(图1)。盾叶苣苔种群聚类分析结果与单倍型网络图和Structure分析结果一致。相同遗传成分的种群首先聚为单系进化支。昌化江东南侧4个种群构成的进化支与其余种群的遗传距离较远,分布在昌化江北侧和西南的种群则构成另一进化支(图4)。种群YG和西南部的JF、XA聚在一起,而位于昌化江以北的种群JX、BW、NG与LM构成一单系,其单倍型彼此遗传距离较小(图2)。
将上述6种遗传成分所在种群的地理分布区作为地区层次进行AMOVA分析。表2结果显示,地区间、种群间与种群内的遗传变异分别占总变异量的67%、27%和6%,揭示盾叶苣苔的遗传变异主要来源于不同地区种群的遗传差异。DnaSP估计种群间的平均基因流Nm=0.04,种群间遗传分化系数FST=0.8。Mantel test显示地理距离和遗传距离存在一定相关性(相关性系数r=0.322,P=0.010)。基于单倍型计算的盾叶苣苔总遗传多样性HT=0.998 (±0.012 1),种群内平均遗传多样性HS=0.624 (±0.076 8),而基于DNA序列计算的盾叶苣苔物种水平核苷酸多样性π=0.023 5,单种群核苷酸多样性在0~0.004 6之间,远低于物种水平的遗传多样性。度量种群遗传分化的指数GST和NST分别为0.375±0.078 1和0.933±0.015 2,NST显著大于GST (P<0.001)。
3 讨论与结论
3.1 海南岛地理隔离对盾叶苣苔遗传变异空间分布的影响
本研究中,昌化江河谷将海南岛中南部山地分割,形成山地内部彼此隔离的地理格局。隔离将减弱甚至阻断基因交流,是种群遗传分化的主要原因(Slatkin, 1987; Ouborg, 1999; Manel et al., 2003)。通常GST > 0.25,表明种群间有很强的遗传分化(Buso et al., 1998)。盾叶苣苔的GST= 0.375,显示不同地理种群存在强烈的遗传分化。基因流可以降低种群分化,如果基因流较弱(Nm<1),就不能抵挡由隔离引起的种群趋异(Slatkin, 1985)。盾叶苣苔生长在林下、溪边及石缝中,植株矮小,其花粉和种子传播的距离很有限。加之昌化江河谷的隔离作用,不同地理种群发生基因流的可能性较小。本研究估计盾叶苣苔的基因流Nm=0.04,远小于1,显示种群间基因交流很有限,这与盾叶苣苔种群存在强烈遗传分化的结果一致。Structure分析确定了6种遗传成分,其中4种(Ⅰ、Ⅱ、Ⅲ、Ⅳ)分布在昌化江以北,成分Ⅴ和Ⅵ分布在昌化江的西南與东南,同侧种群因山谷与昌化江支流的隔离作用而进一步分化为不同的遗传成分。单倍型网络和种群聚类分析揭示,分布在昌化江东南的盾叶苣苔与其余种群有较大的遗传分化。昌化江河谷阻断了东南种群与北部种群的基因交流,而五指山与仙安石林间的山谷导致东南与西南两地种群隔离,形成昌化江以北、东南和西南三地彼此隔离的地理格局。盾叶苣苔6种遗 传成分的地理分布, 以及地理种群的聚类关系与海南岛中南部山地的隔离样式大致吻合,进一步证实海南岛昌化江河谷造成的地理隔离是导致盾叶苣苔遗传分化的重要原因。邢婀娜等(2018)利用ITS序列变异研究海南特有种毛花马铃苣苔(Oreocharis dasyantha)遗传变异的水平与地理分布,结果发现毛花马铃苣苔在昌化江河谷两侧有较明显的分化,推测隔离是河谷两侧种群遗传趋异的主要原因。苦苣苔科植物受到地理隔离的影响容易发生明显的遗传分化,如报春苣苔属中国特有种牛耳朵(Primulina eburnea)生长在中国西南部的石灰岩地区,有东西两个主要分布区,间隔距离大约200 km。Gao et al. (2015)发现西部与东部的牛耳朵种群存在明显分化(FST=0.665),认为地理隔离是导致东西两地种群出现遗传分化的重要因素。综上所述,本研究揭示盾叶苣苔遗传变异的地理分布与海南山地的隔离样式基本一致,表明地理隔离是促使盾叶苣苔遗传分化格局形成的重要因素。
3.2 盾叶苣苔的遗传多样性
一般認为特有种、狭域种或濒危种的遗传多样性较低(Ellstrand & Elam, 1993; Hamrick & Godt, 1996),也有研究显示这些物种仍能维持较高水平的遗传变异。这可能是由于种群间发生了剧烈遗传分化,虽然单个种群的多样性不高,但整个物种却具有较高水平的遗传变异(Dixo et al., 2009)。例如,青藏高原濒危植物唐古特大黄(Rheum tanguticum)的种群稀少,但总遗传多样性较高(HT=0.632) (王爱兰和李维卫, 2017)。唐古特大黄经历了漫长的种群分化,不同地理种群的遗传分歧很大,导致物种水平的遗传多样性较高,同时唐古特大黄异交的繁育系统也使得遗传变异能长期维持在种群中。使用ITS序列估计盾叶苣苔物种水平的遗传多样性(HT=0.998,π=0.023 5)远高于单个种群的变异(种群内平均遗传变异HS=0.624,π=0~0.004 6)。海南岛中南部山地被昌化江河谷及山谷分割,导致盾叶苣苔种群显著分化,在物种水平上具有较丰富的遗传变异。Song & Thomas (2007)研究多年生草本植物Boechera fecunda发现,该种遗传结构受地理隔离影响,基因流被阻断,种群显著分化(FST=0.57),导致物种整体维持较高水平的遗传变异(HT=0.46)。与盾叶苣苔地理分布和繁育系统类似的Elymus glaucus,岛屿特有多年生草本植物Lithophragma maximum,以及海南岛特有植物毛花马铃苣苔,均因地理隔离引起种群强烈分化,使得物种的遗传多态性水平较高(Furches et al., 2008; Hufford et al., 2013; 邢婀娜等, 2018)。物种维持高水平遗传变异的另一原因是祖先多态性没有发生显著丧失(Frankham & Briscoe, 2005)。苦苣苔科报春苣苔(Primulina tabacum)是我国华南石灰岩地区的特有种,仅分布在湖南和广东交界的狭窄地区,但物种水平的遗传多样性较高(PPB=85.6%,HE=0.339,I=0.495)(Ni et al., 2006)。Ni et al. (2006)认为报春苣苔在冰期通过避难所保留了大部分祖先多态性,该种目前的遗传变异仍然比较丰富。盾叶苣苔分布在低纬度热带地区,第四纪冰期的扰动对其影响甚微,遗传变异能够稳定的维持在种群中(颜家安, 2006; 陈冬梅等, 2011)。因此,其盾叶苣苔种群间遗传分化和祖先多态性维持,可能是其物种水平具有较高遗传多样性的主要原因。
3.3 盾叶苣苔的保护措施
一般岛屿特有种的种群规模小,其生境脆弱且分布范围狭窄,比大陆种群或物种更易濒危或灭绝(Ortega et al., 2000)。盾叶苣苔为海南特有种,对生境要求严格,只生长在湿润崖壁上或石壁夹缝中,目前因生境退化种群持续缩减(韦毅刚等, 2010)。本研究揭示盾叶苣苔67%的遗传变异存在于地区间,需要尽可能多地保护不同地理种群,以维持盾叶苣苔物种水平的遗传多样性。11个种群中,NG(π=0.000 0, Hd=0.000 0)在ITS序列上无变异,YG (π=0.000 6, Hd=0.381 0)的变异水平很低,应作为重点种群优先保护,同时引入不同基因型个体以增加NG与YG的遗传变异。考虑到盾叶苣苔具有6种遗传成分,因此每种成分都应予以保护;昌化江以北的YG (I)、JX ( Ⅱ)与LM (IV)各自具有独特遗传成分且只存在于单个种群,因此这些种群也应优先保护。
参考文献:
BA NDELT HJ, FORSTER P, ROHL A, 1999. Median-joining networks for inferring intraspecific phylogenies [J]. Mol Biol Evol, 16(1): 37-48.
BUSO GSC, RANGEL PH, FERREIRA ME, 1998. Analysis of genetic variability of South American wild rice populations (Oryza glumaepatula) with isozymes and RAPD markers [J]. Mol Ecol, 7(1): 107-117.
CHEN DM, KANG HZ, LIU CJ, 2011. An overview on the potential quaternary glacial refugia of plants in China Mainland [J]. Bull Bot Res, 31(5): 623-632. [陈冬梅, 康宏樟, 刘春江, 2011. 中国大陆第四纪冰期潜在植物避难所研究进展 [J]. 植物研究, 31(5): 623-632.]
DAI ZF, LUO GY, 2009. Resources of wild gesneriaceae ornamental plant in tropical region of China [J]. Guizhou Acta Sci, 37(1): 13-15. [代正福, 罗冠勇, 2009. 中国热带特有野生苦苣苔科观赏植物资源 [J]. 贵州农业科学, 37(1): 13-15.]
DIXO M, METZGER JP, MORGANTE JS, et al., 2009. Habitat fragmentation reduces genetic diversity and connectivity among toad populations in the Brazilian Atlantic Coastal Forest [J]. Biol Conserv, 142(8): 1560-1569.
DOYLE J, 1990. Isolation of plant DNA from fresh tissue [J]. Focus, 12(1): 13-15.
DUAN YZ, ZHANG DJ, GAO QB, et al., 2010. Characteristic of molecular evolution of Sibiraea angustata based on nrDNA ITS and cpDNA trnL-F squnece analysis [J]. Bull Bot Res, 30(2): 146-151. [段义忠, 张得钧, 高庆波, 等, 2010. 窄叶鲜卑花(Sibiraea angustata) nrDNA ITS和cpDNA trnL-F序列分子进化特点的分析 [J]. 植物研究, 30(2): 146-151.]
ELLSTRAND NC, ELAM DR, 1993. Population genetic consequences of small population size: Implications for plant conservation [J]. Ann Rev Ecol Syst, 24(24): 217-242.
EVANNO G, REGNAUT S, GOUDET J, 2005. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study [J]. Mol Ecol, 14(8): 2611-2620.
FRANKHAM R, BRISCOE DA, 2005. Introduction to conservation genetics [M]. Beijing: Science Press. [FRANKHAM R, BRISCOE DA, 2005. 保育遗传学导论 [M]. 北京: 科学出版社.]
FURCHES MS, WALLACE, LE, HELENURM K, 2009. High genetic divergence characterizes populations of the endemic plant Lithophragma maximum(Saxifragaceae) on San Clemente Island [J]. Conserv Genet, 10(1): 115-126.
GAO Y, AI B, KONG H, et al., 2015. Geographical pattern of isolation and diversification in karst habitat islands: A case study in the Primulina eburnea complex [J]. J Biogeogr, 42(11): 2131-2144.
HAMRICK JL, GODT MJW, 1996. Effects of life history traits on genetic diversity in plant species [J]. Phil Trans Roy Soc B-Biol Sci, 351(1345): 1291-1298.
HUFFORD KM, MAZER SJ, HODGES SA, 2013. Genetic variation among mainland and island populations of a native perennial grass used in restoration [J]. AoB Plants, 6(3): 623-626.
JAKOBSSON M, ROSENBERG NA, 2007. CLUMPP: A cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure [J]. Bioinformatics, 23(14): 1801-1806.
KUMAR S, NEI M, DUDLEY J, et al., 2008. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences [J]. Brief Bioinform, 9(4): 299-306.
LI ZY, WANG YZ, 2005. Plants of Gesneriaceae in China [M]. Zhengzhou: Henan Science and Technology Publishing House. [李振宇, 王印政, 2005. 中國苦苣苔科植物 [M]. 郑州: 河南科学技术出版社.]
LIBRADO P, ROZAS J, 2009. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 25(11): 1451-1452.
LING SJ, MENG QW, TANG L, et al., 2017. Gesneriaceae on Hainan Island: Distribution patterns and phylogenetic relationships [J]. Biodivers Sci, 25(8): 807-815. [凌少军, 孟千万, 唐亮, 等, 2017. 海南岛苦苣苔科植物的地理分布格局与系统发育关系 [J]. 生物多样性, 25(8): 807-815.]
MANEL S, SCHWARTZ MK, LUIKART G, et al., 2003. Landscape genetics: Combining landscape ecology and population genetics [J]. Trend Ecol Evol, 18(4): 189-197.
MANTEL N, 1967. The detection of disease clustering and a generalized regression approach [J]. Appl Microbiol Biotechnol, 59(2-3): 209-220.
NI X, HUANG Y, WU L, et al., 2006. Genetic diversity of the endangered Chinese endemic herb Primulina tabacum (Gesneriaceae) revealed by amplified fragment length polymorphism (AFLP) [J]. Genetica, 127(1-3): 177-183.
ORTEGA JF, GUERRA AS, KIM SC,et al., 2000. Plant genetic diversity in the Canary Islands: A conservation perspective [J]. Am J Bot, 87(7): 909-919.
OUBORG NJ, PIQUOT Y, GROENENDAEL JMV, 1999. Population genetics, molecular markers and the study of dispersal in plants [J]. J Ecol, 87(4): 551-568.
PEAKALL R, SMOUSE PE, 2012. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research — an update [J]. Bioinformatics, 28(12): 2537-2539.
PONS O, PETIT RJ, 1996. Measuring and testing genetic differentiation with ordered versus unordered alleles [J]. Genetics, 144(3): 1237-1245.
PRITCHARD JK, STEPHENS M, DONNELLY P, 2000. Infe-rence of population structure using multilocus genotype data [J]. Genetics, 155(2): 945-959.
RAYMOND M, ROUSSET F, 1995. An exact test for population differentiation [J]. Evolution, 49(6): 1280-1283.
SHI YH, XU SS, HUANG JW, 2011. Evaluation of wild plant resources and their ornamental characteristics of Hainan chicory [J]. N Hortic, (11): 79-82. [史佑海, 徐世松, 黃觉武, 2011. 海南苦苣苔科野生植物资源及其观赏特性评价 [J]. 北方园艺, (11): 79-82.]
SLATKIN M, 1985. Gene flow in natural populations [J]. Ann Rev Ecol Syst, 16(1): 393-430.
SLATKIN M, 1987. Gene flow and the geographic structure of natural populations [J]. Science, 236(4803): 787-792.
SONG BH, THOMAS MO, 2007. High genetic diversity and population differentiation in Boechera fecunda, a rare relative of Arabidopsis [J]. Mol Ecol, 16(19): 4079-4088.
STUESSY TF, TAKAYAMA K, LPEZSEPLVEDA P, et al., 2014. Interpretation of patterns of genetic variation in endemic plant species of oceanic islands [J]. Bot J Linn Soc, 174(3): 276-288.
TAMURA K, STECHER G, PETERSON D,et al., 2013. MEGA6:Molecular evolutionary genetics analysis version 6.0. [J]. Mol Biol Evol, 30(12): 2725-2729.
WANG AL, LI WW, 2017. Genetic diversity of an endangered species, Rheum tanguticum (Polygonaceae), on the Qinghai-Tibetan Plateau [J]. Acta Ecol Sin, 37(21): 7251-7257. [王爱兰, 李维卫, 2017. 青藏高原濒危植物唐古特大黄的遗传多样性 [J]. 生态学报, 37(21): 7251-7257.]
WANG C, LIANG XQ, ZHOU Y, et al., 2015. Construction ofage frequencies of provenances on the eastern side of the Yinggehai Basin: Studies of LA-ICP-MS U-PB ages of detrital zircons from six modern rivers, western Hainan, China [J]. Earth Sci Front, 22(4): 277-289. [王策, 梁新权, 周云, 等, 2015. 莺歌海盆地东侧物源年龄标志的建立:来自琼西6条主要河流碎屑锆石LA-ICP-MS U-PB年龄的研究 [J]. 地学前缘, 22(4): 277-289.]
WANG XX, LE JP, SUN H, et al., 2011. Phylogeographical study on Eriophyton wallichii (Labiatae) from alpine scree of Qinghai-Tibetan Plateau [J]. Plant Divers Resour, 33: 605-614. [王晓雄, 乐霁培, 孙航, 等, 2011. 青藏高原高山流石滩特有植物绵参的谱系地理学研究 [J]. 植物分类与资源学报, 33: 605-614.]
WANG ZS, AN SQ, LENG X, et al., 2004. Population genetic diversity of the insular plant Neolitsea sericea based on random amplified polymorphic DNA (RAPD) [J]. Acta Ecol Sin, 24(3): 414-422. [王中生,安树青,冷欣, 等, 2004. 岛屿植物舟山新木姜子居群遗传多样性的RAPD分析 [J]. 生态学报, 24(3): 414-422.]
WEI YG, WEN F, MOLLER M, et al., 2010. Gesneriaceae of South China [M]. Nanning: Guangxi Science & Technology Publishing House. [韦毅刚, 温放, MOLLER M, 等, 2010. 华南苦苣苔科植物 [M]. 南宁:广西科学技术出版社.]
WEN F, 2008. Studies on investigation and introduction of wild ornamental resources of Gesneriaceae in Guangxi [D]. Beijing: Beijing Forestry University. [温放, 2008. 广西苦苣苔科观赏植物资源调查与引种研究 [D]. 北京: 北京林业大学.]
WHITE TJ, BRUNS T, LEE S, et al., 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics [M]//PCR Protocols: A guide to methods and applications. New York: Academic Press: 315-322.
XING EN, XU ST, REN MX, 2018. Age structure and gene flows of fine-scale populations of Oreocharis dasyantha (Gesneriaceae), an alpine herb endemic to Hainan Island [J]. J Trop Biol, 1(9): 37-46. [邢婀娜, 徐诗涛, 任明迅, 2018. 海南特有毛花马铃苣苔小尺度局域种群的年龄结构与基因流 [J]. 热带生物学报, 1(9): 37-46.]
XING FW, 2012. Inventory of plant species diversity of Hainan [M]. Wuhan: Huazhong University of Science and Technology Press. [邢福武, 2012. 海南植物物种多样性编目 [M]. 武汉: 华中科技大学出版社.]
XING GY, 2004. Starting from the initial establishment of Hainan Island [J]. Hainan Arch,3: 43-47. [邢关英, 2004. 从海南岛最初设治说起 [J]. 海南档案, 3: 43-47.]
XU WB, GUO J, PAN B, et al., 2017. Diversity and distribution of Gesneriaceae in China [J]. Guihaia, 37(10): 1219-1226. [許为斌, 郭婧, 盘波, 等, 2017. 中国苦苣苔科植物的多样性与地理分布 [J]. 广西植物, 37(10): 1219-1226.]
YAN JA, 2006. Paleontology and ecologic environmental evolution of the quaternary in Hainan Island [J]. J Palegeogr, 8(1): 103-115. [颜家安, 2006. 海南岛第四纪古生物及生态环境演变 [J]. 古地理学报, 8(1): 103-115.]
YU ZY, 2012. Regulation mechanism of tourist ecological effect must be established to build hainan into international tourism island of Hainan [J]. Ecol Environ, 1: 166-170. [余中元, 2012. 国际旅游岛建设旅游用地生态效应调控机制研究 [J]. 生态经济, 1: 166-170.]
ZHOU ZG, 2015. Analysis on the ecological compensation mechanism of the three major rivers in Hainan Island [C]. Changchun: The Chinese Lake BBS. [周祖光, 2015. 海南岛三大河流生态补偿机制探析 [C]. 长春: 中国湖泊论坛.]
( 责任编辑 蒋巧媛 )

