基于粗提物的水稻条纹病毒体外“抓帽”体系
林文忠,吴然,金晶,丘萍,张洁,吴祖建,杜振国
基于粗提物的水稻条纹病毒体外“抓帽”体系
林文忠,吴然,金晶,丘萍,张洁,吴祖建,杜振国
(福建农林大学植物病毒研究所/闽台作物有害生物生态防控国家重点实验室,福州 350002)
布尼亚病毒目()和正黏病毒科()病毒从帽子结构下游10—20个碱基处切割寄主mRNA,以5′端切割产物(帽子序列)作为引物起始自身基因组的转录,生成含一段异源帽子序列的病毒mRNA,这一过程称为“抓帽”。水稻条纹病毒(rice stripe tenuivirus, RSV)是一种布尼亚病毒,其“抓帽”机制还不甚清楚。【】研究RSV粗提物能否从溶液中的外源mRNA“抓帽”,以建立一种便捷的体外体系,用于解析RSV“抓帽”和转录机制。以PEG沉淀和超速离心法粗提RSV,利用聚丙烯酰胺凝胶电泳(SDS-PAGE)和质谱技术分析粗提物成分。然后取少量粗提液,加入富含珠蛋白()mRNA的兔网织红细胞裂解液(rabbit reticulocyte lysate,RCL)和合适浓度的金属离子配制体外“抓帽”体系。待体外反应结束,提取体系中总RNA,以巢式RT-PCR扩增含珠蛋白-mRNA帽子序列的RSV mRNA,并对其进行克隆,通过序列分析比较RSV体外“抓帽”与体内“抓帽”的异同。从100 g感染RSV的水稻叶片获得RSV粗提液2 mL。除RSV病毒粒子外,粗提液中还含核酮糖-1,5-二磷酸羧化酶等多种叶绿体蛋白。取2 μL粗提液,加入4 mmol·L-1MgCl2、2 mmol·L-1NTP、0.8 U·μL-1RNA酶抑制剂和8 μL RCL,配成20 μL反应体系,30℃孵育1.5 h后,巢式RT-PCR扩增到了目的条带,表明RSV粗提物能切割溶液中的珠蛋白-mRNA,并利用其帽子序列合成自身mRNA。在反应体系中加入帽子结构类似物m7G(5′)ppp(5′)G后进行相同反应,目的条带变淡,且变淡程度与所加入m7G(5′)ppp(5′)G的浓度成正比,表明识别帽子结构是RSV从溶液中切割利用珠蛋白-mRNA的前提。对巢式RT-PCR产物克隆和测序后分析发现,与在体内的情况类似,RSV从帽子结构下游的A或C处切割珠蛋白-mRNA,得到的帽子序列与病毒模板链3′端的U或G配对后引发转录。在利用珠蛋白-mRNA帽子序列进行转录的过程中,RSV频繁使用引发与重配,且在合成核蛋白基因时比在合成主要非核蛋白基因时使用该机制的频率更高。RSV粗提物能从溶液中的mRNA“抓帽”,且在切取和利用帽子序列的方式上,其表现与细胞内的RSV无明显差别。因而,粗提物可以用来解析RSV的“抓帽”机制,甚至用于研究RSV的转录。
水稻条纹病毒;抓帽机制;体外转录
0 引言
【研究意义】水稻条纹病毒(rice stripe tenuivirus,RSV)是我国最重要的粮食作物病毒之一,深入认识RSV侵染机理,实现RSV的可持续防控,是关系到我国粮食安全的重大需求[1-2]。作为基因表达的第一步,“抓帽”(cap-snatching)是RSV侵染周期中最关键的环节之一[3]。建立一种便捷的体外体系,有助于深入研究RSV的“抓帽”机制。【前人研究进展】真核生物信使RNA(mRNA)5′端有一个帽子结构,能保护mRNA免受5′到3′外切酶降解的同时,在mRNA的翻译起始等过程中也起重要作用[4]。除非有一些替代机制,病毒也需要给自己的mRNA添加帽子结构。病毒以多种不同的机制实现这一目的,其中布尼亚病毒目()和正黏病毒科()病毒普遍采用“抓帽”。在此过程中,它们从帽子结构下游10—20个碱基处切割寄主mRNA,然后利用5′端切割产物(帽子序列)作为病毒模板链的转录引物,使每条病毒mRNA都含一小段来源于寄主mRNA的帽子序列[5]。“抓帽”现象最早发现于流感病毒(正黏病毒科),迄今为止研究最多的也是该病毒[6-7]。相应地,“抓帽”已成为抗流感病毒药物开发的一个重要靶标[8]。RSV是布尼亚病毒目白纤病毒科()纤细病毒属()的代表种,自然条件下由灰飞虱()以持久增殖型方式传播,主要侵染水稻和小麦等禾本科作物[9-10],实验室条件下,也可以通过机械接种或昆虫传毒方式侵染模式植物本氏烟和拟南芥[11-12]。RSV基因组由4条单链RNA(ssRNA)组成,按分子量大小分别称为RNA1—RNA4。RNA1—RNA4在5′和3′末端有一段保守的反向互补序列,分别为5′-A1C2A3C4AAAGUC-和3′-U1G2U3G4UUUCAG-。RNA1采用负义编码,而RNA2—RNA4采用双义编码,即它们的病毒链(vRNA)和互补链(vcRNA)都可以作为转录的模板[9-10]。Shimizu等[13]最先克隆了RSV的mRNA,发现它们除拷贝自基因组的序列(下称拷贝序列),还含一段异源帽子序列,表明RSV使用“抓帽”机制;Yao等[14]以RSV与黄瓜花叶病毒(cucumber mosaic virus,CMV)共侵染本氏烟,发现RSV可从CMV抓取帽子序列。对RSV从CMV“抓帽”后合成的mRNA进行克隆和分析发现,大部分RSV mRNA在帽子序列和拷贝序列之间含一个或多个AC重复,表明RSV在利用从CMV抓取的帽子序列进行转录时,频繁使用引发与重配(prime- and-realign)机制[14-15]。RSV使用引发与重配机制的频率与帽子序列的长短有关,帽子序列越短,使用该机制的频率越高。本实验室首次对RSV mRNA进行了5′端的高通量测序,并根据所得数据,提出了RSV“抓帽”的基本模型[16]。在RSV侵染的本氏烟叶片浸润含pCHF3载体的农杆菌,RSV会从pCHF3表达的mRNA“抓帽”。基于此,近期建立了一种向RSV呈递特定mRNA(供其“抓帽”)的实验体系,并应用该体系初步验证了RSV“抓帽”的基本模型[17]。【本研究切入点】目前,对RSV“抓帽”机制的很多细节还不清楚。体外“抓帽”体系在解析流感病毒“抓帽”机制的过程中发挥了重要作用,在一些布尼亚病毒上也有成功的应用[18-21]。利用这一体系,研究者可以定量地向病毒提供特定mRNA,甚至特定的帽子序列,以研究病毒如何选择和利用帽子序列[22-26]。显然,这样的体系对RSV“抓帽”和转录机制研究也具有重要意义。【拟解决的关键问题】确定RSV粗提物能否从外源mRNA“抓帽”,并研究RSV体外“抓帽”与体内“抓帽”在方式上是否存在差异。
1 材料与方法
试验于2019年6月至2020年5月间在福建农林大学植物病毒研究所完成。
1.1 试验材料
1.1.1 供试毒株与植物 携带RSV的灰飞虱由江苏省农业科学院季英华研究员提供,引入本实验室后,在水稻上继代培养。带毒灰飞虱取食过的水稻幼苗种植于日光温室,出现典型RSV感染症状后用于RSV的提取。
1.1.2 主要试剂 主要试剂及其来源见表1,其他常规化学试剂均为国产分析纯。
1.2 试验方法
1.2.1 RSV的粗提和聚丙烯酰胺凝胶电泳检测 取感染RSV的新鲜水稻叶片约100 g,剪碎后加入研钵,以液氮浸没并迅速研磨至粉末状。随后参照Wang等[27]描述的步骤提取RSV,以0.01 mol·L-1PBS缓冲液(0.27 g·L-1KH2PO4,1.42 g·L-1Na2HPO4,8 g·L-1NaCl,0.2 g·L-1KCl,pH 7.4)代替了文献[27]中的PB缓冲液。最终提取物以2 mL PBS溶解,分装后置于-80℃冰箱备用。
为了解RSV粗提物的蛋白组分,取粗提液80 μL,加入5×蛋白上样缓冲液20 μL,混合均匀后,沸水浴5 min,于恒压120 V下进行聚丙烯酰胺凝胶电泳(SDS-PAGE,5%浓缩胶,12%分离胶)。每孔上样20 μL,以6 μL Direct-loadTMColor Prestained Protein Marker(GenStar)作为分子量标准。电泳完毕,凝胶经考马斯亮蓝染色和脱色液脱色后用相机进行拍照,并割取主要蛋白条带,送上海启研生物科技有限公司进行质谱鉴定。

表1 本研究所用的主要试剂和试剂盒
1.2.2 体外“抓帽”反应体系 RSV体外“抓帽”体系参照Van Knippenberg等[28]的报道略有改动,含2 μL RSV粗提液,8 μL兔网织红细胞裂解液(rabbit reticulocyte lysate,RCL),4 mmol·L-1MgCl2,2 mmol·L-1NTP及0.8 U·μL-1RNA酶抑制剂,总体积20 μL,30℃下恒温孵育1.5 h。
1.2.3 体外“抓帽”所产生的RSV mRNA的巢式RT-PCR检测和克隆测序 上述反应完成后,以RNA纯化试剂盒对总RNA进行纯化,具体步骤参照说明书进行,纯化产物以15 μL RNAase-free去离子水溶解。RCL中含大量珠蛋白-和珠蛋白-mRNA,本研究仅扩增了RSV从珠蛋白-抓帽后的转录产物。所用巢式RT-PCR基本原理如图1所示,以primer 1合成第一链,然后以primer 2和3扩增cDNA。Primer 3的3′末端10个碱基与珠蛋白-mRNA最5′端10个碱基相同,5′端15个碱基(下划线部分)主要作用是增加引物长度。RSV共转录7种不同的mRNA,本文仅扩增了其vRNA3的转录产物和vcRNA4的转录产物[29-30]。扩增和所用的primer 1和2如图1所示。反转录体系:RNA 2 µL(约0.6 µg),primer 1 0.25 µmol·L-1,HiScript® II Enzyme Mix 2 µL,10×RT Mix 2 µL,总体积20 µL。反转录程序参照试剂盒说明书进行。PCR体系:cDNA 2 µL,primer 2和3各0.5 µmol·L-1,2×ExTaq Mix 12.5 µL,总体积25 µL。PCR反应程序:94℃预变性3 min后,按照94℃ 30 s,55℃ 30 s,72℃ 30 s,进行31次循环,最后于72℃延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳后,于凝胶成像仪下观察目的条带。需要时切割目的条带,以HiPure Gel Pure DNA Mini Kit进行DNA回收,回收产物连接pMD19-T后转化大肠杆菌,并挑取20—30个单菌落进行测序。作为对照,除以图1所示的巢式RT-PCR扩增RSV mRNA,还扩增了RSV的vcRNA3和vRNA4。其基本流程与巢式RT-PCR相同,但以病毒自身序列特异性引物取代了图1所示的primer 3 (vcRNA3:5′-GCTACAATGGGCACCAACAAGC-3′;vRNA4:5′-AGATGCAAGACGTACAAAGGAC-3′)。

图1 利用巢式RT-PCR扩增RSV从珠蛋白-α“抓帽”后合成的NP(转录自vRNA3)和NCP(转录自vcRNA4)mRNA
2 结果
2.1 RSV粗提物的获得
从100 g感染RSV的水稻叶片获得RSV粗提液2 mL。取少量粗提液进行SDS-PAGE,考马斯亮蓝染色后可观察到4条主要的蛋白条带,分子量依次约250、50、30和11 kD(图2)。质谱鉴定结果表明,条带1主要为光系统II相关蛋白,条带2主要为核酮糖1,5-二磷酸羧化酶(RuBisCO)大亚基和RuBisCO活化酶,条带3主要为RSV的NP,条带4主要为RuBisCO小亚基。由这些结果可知,RSV粗提物含大量杂质,而叶绿体是杂质成分的重要来源。RNA1编码的RdRp也是RSV的结构蛋白[31],但可能因为含量太低,未观察到该蛋白对应的条带。
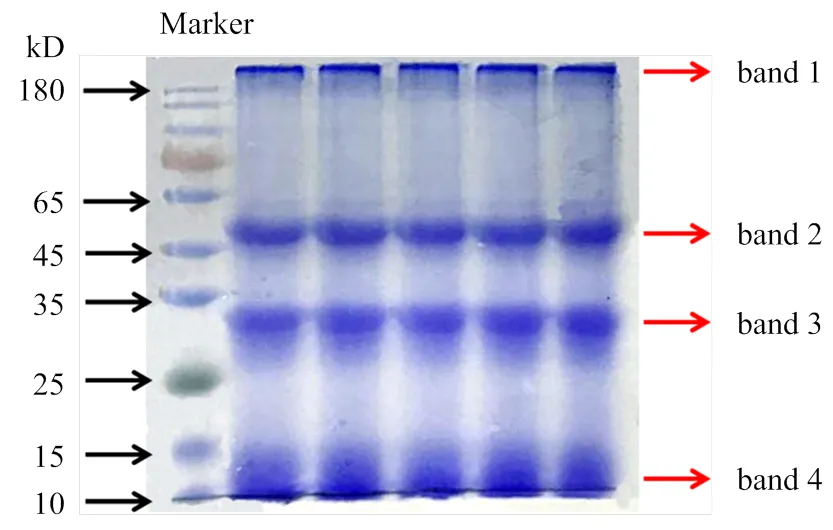
Band 1—4:4个进行质谱鉴定的主要蛋白条带The 4 protein bands subjected to mass spectrometry
2.2 RSV粗提物“抓帽”产物的检测
取体外“抓帽”反应产物,用巢式RT-PCR扩增含珠蛋白-mRNA帽子序列的RSV和mRNA,均能得到目的条带。与病毒粒子中含更多的vRNA相符,(vRNA转录产物)条带明显亮于(vcRNA转录产物)条带(图3-A)。作为对照,以95℃热失活2 min的RSV粗提液配制反应体系,反应结束后进行同样的检测,未观察到目的条带。无论是否对RSV粗提液进行热失活,反应结束后均能检测到RSV基因组RNA(图3-A)。上述结果表明粗提液中的RSV能利用溶液中的mRNA作为帽子序列供体合成自身mRNA,且这种能力需要有活性的RSV蛋白来实现。
为验证RSV需要识别帽子结构才能利用溶液中的mRNA作为帽子序列供体,在反应体系中加入了不同浓度的帽子结构类似物m7G (5′) ppp (5′) G。如图3-B所示,m7G (5′) ppp (5′) G明显减少含珠蛋白-mRNA帽子序列的的生成。当m7G (5′) ppp (5′) G浓度为1.5 mmol·L-1时,目的条带基本消失。相反,m7G (5′) ppp (5′) G对反应后的RSV基因组RNA的检出性没有影响,表明m7G (5′) ppp (5′) G不会造成反应体系中RNA的降解。

M:DNA marker。A:以正常或热失活RSV粗提物进行体外“抓帽”,然后利用巢式RT-PCR扩增含珠蛋白-α帽子序列的RSV NCP(孔1和3)或NP(孔2和4) mRNA,以RT-PCR扩增vcRNA4上NCP对应区域(孔6和8)或vRNA3上NP对应区域(孔5和7)Untreated or heat inactivated RSV crude preparations were used in the in vitro cap-snatching assay. Nested RT-PCR was used to detect RSV NCP (lanes 1 and 3) or NP (lanes 2 and 4) mRNAs with capped-RNA leaders derived from globin-α. RT-PCR was used to detect a NP-corresponding region on RSV vRNA3 (lanes 5 and 7) or a NCP-corresponding region on RSV vcRNA4 (lanes 6 and 8);B:在0—1.5 mmol·L-1帽子类似物m7G (5′) ppp (5′) G存在下进行体外“抓帽”,然后以巢式RT-PCR扩增含珠蛋白-α帽子序列的RSV NCP(孔1—4),以RT-PCR扩增vcRNA4上NCP对应区域(孔5—8)The cap analog m7G (5′) ppp (5′) G was added at a final concentration of 0.5, 1 or 1.5 mmol·L-1. Nested RT-PCR was used to detect RSV NCP with capped-RNA leaders derived from globin-α (lanes 1 to 4) and RT-PCR was used to detect a NCP-corresponding region on RSV vcRNA4
2.3 RSV粗提物“抓帽”产物分析
为进一步验证RSV粗提物能从外源mRNA“抓帽”,并分析这种情况下产生的RSV mRNA,对图3-A中Lane 1和2的目的条带进行了割胶回收。经连接、转化和挑取单克隆测序后,得到RSVmRNA序列19条,含非冗余单一序列7条(表2),mRNA序列28条,含非冗余单一序列6条(表3)。
分析发现,这些序列都可用之前通过体内研究提出的模型解释[16-17]:(1)RSV从C12或C18切割珠蛋白-mRNA,得到末端为C的帽子序列mG---C。该帽子序列与RSV基因组3′端G2配对后从U3引发转录,生成m7G---CAC。m7G---CAC可以继续按照模板延伸,直至生成完整的RSV mRNAm7G---(表3,第一行,下划线部分为能Map到病毒基因组的碱基),也可以与模板链脱离,以新延伸出的AC与RSV基因组3′端的U1G2配对,重新从U3引发转录,即发生引发与重配(图4-A)。引发与重配可发生多次,其结果是RSV mRNA在帽子序列与所谓的拷贝序列之间出现1或多个AC重复(表2第2、4和5行及表3的第3行);(2)除C12或C18外,RSV还可以在A14切割珠蛋白-mRNA,得到末端为A的帽子序列m7G---C13A14。该帽子序列可以利用A14与RSV基因组3′端的U1配对,或利用C13A14与RSV基因组3′端的G2U3配对(图4-B、4-C)。同样,在转录起始之后,RSV可以使用0到多次引发与重配,生成多种不同形式的RSV mRNA。按照这种解释,RSV利用珠蛋白-mRNA帽子序列生成(58%)时比生成(33%)时更频繁使用引发与重配,这与之前的发现也完全相符[16]。
阴影部分表示对应珠蛋白-mRNA的序列,下划线部分表示能Map到RSV基因组(3′-U1G2U3G4UUUCAG-)的碱基;加黑字体表示因引发与重配而生成的额外碱基;“+”表示有发生引发与重配;“-”表示没有发生引发与重配。表3同
RSV mRNA nucleotides that can be mapped to the 5′-terminus of globin-mRNA are dashed, and those can be mapped to the genome of RSV (3′-U1G2U3G4UUUCAG-) are underlined. Nucleotides produced by priming and realignment are in bold. The symbol “+” indicates that the sequence can be explained by at least one round of priming and realignment, whereas “-” indicates that the sequence might be produced without priming and realignment. The same as Table 3

表3 RSV从珠蛋白-α mRNA“抓帽”后生成的NCP mRNA

A:帽子序列与RSV基因组G2配对capped-RNA leaders base pair with the G2 of RSV template RNA;B:帽子序列与RSV基因组U1配对capped-RNA leaders base pair with the U1 of RSV template RNA;C:帽子序列与RSV基因组G2U3配对capped-RNA leaders base pair with the G2U3 of RSV template RNA
3 讨论
体外体系已被广泛应用于研究流感和一些布尼亚病毒的“抓帽”及与“抓帽”紧密伴随的转录[18-26]。然而,这些研究都使用纯度较高的病毒粒子,需要繁杂的预备实验。为获得一种便捷的实验体系,本文研究了RSV粗提物的“抓帽”活性。
叶绿体是RSV粗提物中杂质成分的重要来源(图2)。根据条带亮度判断,叶绿体蛋白在粗提物总蛋白中的占比可能超过2/3。推测其原因是叶绿体在离心过程中与RSV病毒粒子发生了共沉淀。虽然它们在后续过程中被破坏,但本研究未以密度梯度离心将它们释放出的蛋白与RSV进行分离。按照这种推测,RSV粗提物中应该还含有来源于线粒体及核糖体等细胞器或大分子的蛋白,只是相较于叶绿体蛋白来讲,这些蛋白的占比较低。
尽管含有大量杂质,本研究结果表明RSV粗提物具有体外“抓帽”活性,且在切割和利用帽子序列的方式上,其表现与细胞内的RSV没有明显差别。主要证据有以下两点:(1)以RSV粗提物与RCL配制反应体系,短时间的孵育后,可以检测到含珠蛋白-mRNA帽子序列的RSV mRNA,而帽子类似物可以降低这类mRNA的合成(图3),表明RSV能利用溶液中的mRNA作为帽子序列供体来合成自身mRNA,且同在体内一样,这一过程需要帽子结构的识别;(2)对体外合成的mRNA进行分析发现,RSV粗提物在A或C处切割珠蛋白-mRNA,得到能与自身模板链3′端U或G配对的帽子序列。在以这些帽子序列进行mRNA合成时,RSV频繁使用引发与重配(图4)。这些现象都完全符合之前通过体内研究提出的模型。值得指出的是,虽然纤细病毒的体外转录活性已有报道[31-32],但还未有研究者对体外转录产物进行测序验证。从这方面讲,本文是首次明确表明纤细病毒可在体外从外源mRNA“抓帽”。
布尼亚病毒和正黏病毒在“抓帽”后的转录过程中普遍使用引发与重配机制[3,33]。笔者前期发现,RSV也使用引发与重配,而且RSV在转录vRNA时比在转录vcRNA时更频繁使用该机制[16]。起初,笔者推测这可能是RSV与寄主互作的结果,或者反映了RSV vRNA和vcRNA在表达上的时空差异。但后来发现,这种现象在RSV建立侵染的极早期就已出现[34]。因而,推测RSV对引发与重配机制的差异化使用可能是其模板链的固有差异造成的,与其他因素无关。本研究发现RSV在体外合成(转录自vRNA)时也比合成(转录自vcRNA)时更频繁使用引发与重配,这进一步支持了第二种推测。
RSV可与多种双生病毒共侵染本氏烟。在共侵染植株中,RSV大量从双生病毒抓取帽子序列。通过高通量测序技术获得RSV mRNA的帽子序列后,将它们Map到双生病毒基因组,可以得到一个精细的双生病毒转录起始位点(transcriptional start site,TSS)图谱。基于此,本实验室提出“抓帽”可被利用于病毒甚至寄主植物TSS的鉴定[35]。然而,该方法有其明显缺点,即需要病毒的共侵染。一方面,这在很多情况下是无法实现的;另一方面,RSV有可能会影响双生病毒TSS的选择。RSV体外“抓帽”体系的建立为解决这一缺点提供了可能。因而,除解析“抓帽”和转录机制外,本实验建立的体系还有潜在的应用价值。
4 结论
RSV粗提物能从外源mRNA“抓帽”,并利用抓取的帽子序列转录自身模板链。在切割外源mRNA和利用其帽子序列的方式上,粗提物的表现与细胞内的RSV无明显差别,因而,基于粗提物的体外体系可以用于解析RSV的“抓帽”机制,甚至研究该病毒“抓帽”之后的转录机制。
[1] 谢联辉, 林奇英, 魏太云, 吴祖建. 水稻病毒. 北京: 科学出版社, 2016.
XIE L H, LIN Q Y, WEI T Y, WU Z J.. Beijing: Science Press, 2016. (in Chinese)
[2] 周益军. 水稻条纹叶枯病. 南京: 江苏科学技术出版社, 2010.
ZHOU Y J.. Nanjing: Jiangsu Science and Technology Press, 2010. (in Chinese)
[3] OLSCHEWSKI S, CUSACK S, ROSENTHAL M. The cap-snatching mechanism of Bunyaviruses., 2020, 28(4): 293-303.
[4] RAMANATHAN A, ROBB G B, CHAN S H. mRNA capping: biological functions and applications., 2016, 44(16): 7511-7526.
[5] DECROLY E, FERRON F, LESCAR J, CANARD B. Conventional and unconventional mechanisms for capping viral mRNA., 2012, 10(1): 51-65.
[6] DE VLUGT C, SIKORA D, PELCHAT M. Insight into influenza: A virus cap-snatching., 2018, 10(11): 641.
[7] WALKER A P, FODOR E. Interplay between influenza virus and the host RNA polymerase II transcriptional machinery., 2019, 27(5): 398-407.
[8] GARCÍA-SASTRE A. Snatch-and-grab inhibitors to fight the flu., 2019, 177(6): 1367.
[9] FALK B W, TSAI J H. Biology and molecular biology of viruses in the genus., 1998, 36: 139-163.
[10] 张恒木, 孙焕然, 王华弟, 陈剑平. 水稻条纹病毒分子生物学研究进展. 植物保护学报, 2007, 34(4): 436-440.
ZHANG H M, SUN H R, WANG H D, CHEN J P. Advances in the studies of molecular biology of rice stripe virus., 2007, 34(4): 436-440. (in Chinese)
[11] XIONG R Y, WU J X, ZHOU Y J, ZHOU X P. Identification of a movement protein of thetenuivirus rice stripe virus., 2008, 82(24): 12304-12311.
[12] SUN F, YUAN X, ZHOU T, FAN Y J, ZHOU Y J.is susceptible to rice stripe virus infections., 2011, 159(11/12): 767-772.
[13] SHIMIZU T, TORIYAMA S, TAKAHASHI M, AKUTSU K, YONEYAMA K. Non-viral sequences at the 5′ termini of mRNAs derived from virus-sense and virus complementary sequences of the ambisense RNA segments of rice stripe tenuivirus., 1996, 77(3): 541-546.
[14] YAO M, ZHANG T Q, ZHOU T, ZHOU Y J, ZHOU X P, TAO X R. Repetitive prime-and-realign mechanism converts short capped RNA leaders into longer ones that may be more suitable for elongation during rice stripe virus transcription initiation., 2012, 93(1): 194-202.
[15] GARCIN D, LEZZI M, DOBBS M, ELLIOTT R M, SCHMALJOHN C, KANG C Y, KOLAKOFSKY D. The 5′ ends of Hantaan virus () RNAs suggest a prime-and-realign mechanism for the initiation of RNA synthesis., 1995, 69(9): 5754-5762.
[16] LIU X J, JIN J, QIU P, GAO F L, LIN W Z, XIE G H, HE S M, LIU S M, DU Z G, WU Z J. Rice stripe tenuivirus has a greater tendency to use the prime-and-realign mechanism in transcription of genomic than in transcription of antigenomic template RNAs., 2018, 92(1): e01414-17.
[17] LIN W Z, WU R, QIU P, JIN J, YANG Y Y, WANG J L, LIN Z L, ZHANG J, WU Z J, DU Z G. A convenientcap donor delivery system to investigate the cap snatching of plant bunyaviruses., 2020, 539: 114-120.
[18] RAO P, YUAN W, KRUG R M. Crucial role of CA cleavage sites in the cap-snatching mechanism for initiating viral mRNA synthesis., 2003, 22(5): 1188-1198.
[19] NOSHI T, KITANO M, TANIGUCHI K, YAMAMOTO A, OMOTO S, BABA K, HASHIMOTO T, ISHIDA K, KUSHIMA Y, HATTORI K, KAWAI M, YOSHIDA R, KOBAYASHI M, YOSHINAGA T, SATO A, OKAMATSU M, SAKODA Y, KIDA H, SHISHIDO T, NAITO A.characterization of baloxavir acid, a first-in-class cap-dependent endonuclease inhibitor of the influenza virus polymerase PA subunit., 2018, 160: 109-117.
[20] KOMODA K, ISHIBASHI K, KAWAMURA-NAGAYA K, ISHIKAWA M. Possible involvement of eEF1A in tomato spotted wilt virus RNA synthesis., 2014, 468/470: 81-87.
[21] LEAHY M B, DESSENS J T, PRITLOVE D C, NUTTALL P A. The Thogoto orthomyxovirus cRNA promoter functions as a panhandle but does not stimulate cap snatching., 1998, 79(3): 457-460.
[22] VAN KNIPPENBERG I, LAMINE M, GOLDBACH R, KORMELINK R. Tomato spotted wilt virus transcriptasedisplays a preference for cap donors with multiple base complementarity to the viral template., 2005, 335(1): 122-130.
[23] GEERTS-DIMITRIADOU C, ZWART M P, GOLDBACH R, KORMELINK R. Base-pairing promotes leader selection to primeinfluenza genome transcription., 2011, 409(1): 17-26.
[24] TE VELTHUIS A J W, OYMANS J. Initiation, elongation and realignment during influenza virus mRNA synthesis., 2018, 92(3): e01775-17.
[25] BARR J N. Bunyavirus mRNA synthesis is coupled to translation to prevent premature transcription termination., 2007, 13(5): 731-736.
[26] VAN KNIPPENBERG I, GOLDBACH R, KORMELINK R.transcription of tomato spotted wilt virus is independent of translation., 2004, 85(5): 1335-1338.
[27] WANG Q, LIU Y Q, HE J, ZHENG X M, HU J L, LIU Y L, DAI H M, ZHANG Y X, WANG B X, WU W X,.encodes a sulphotransferase and confers durable resistance to rice stripe virus., 2014, 5: 4768.
[28] VAN KNIPPENBERG I, GOLDBACH R W, KORMELINK R. Purified tomato spotted wilt virus particles support both genome replication and transcription., 2002, 303(2): 278-286.
[29] KAKUTANI T, HAYANO Y, HAYASHI T, MINOBE Y. Ambisense segment 3 of rice stripe virus: the first instance of a virus containing two ambisense segments., 1991, 72(2): 465-468.
[30] KAKUTANI T, HAYANO Y, HAYASHI T, MINOBE Y. Ambisense segment 4 of rice stripe virus: possible evolutionary relationship with phleboviruses and uukuviruses ()., 1990, 71(7): 1427-1432.
[31] TORIYAMA S. An RNA-dependent RNA polymerase associated with the filamentous nucleoproteins of rice stripe virus., 1986, 67(7): 1247-1255.
[32] NGUYEN M, RAMIREZ B C, GOLDBACH R, HAENNI A L. Characterization of theactivity of the RNA-dependent RNA polymerase associated with the ribonucleoproteins of rice hoja blanca tenuivirus., 1997, 71(4): 2621-2627.
[33] KOPPSTEIN D, ASHOUR J, BARTEL D P. Sequencing the cap- snatching repertoire of H1N1 influenza provides insight into the mechanism of viral transcription initiation., 2015, 43(10): 5052-5064.
[34] 和思淼. 水稻条纹病毒mRNA 5′端“额外AC”的来源研究[D]. 福州: 福建农林大学, 2019.
HE S M. Understanding the origin of the extra dinucleotide AC at the 5′ termini of rice stripe tenuivirus mRNAs[D]. Fuzhou: Fujian agriculture and forestry university, 2019. (in Chinese)
[35] LIN W Z, QIU P, JIN J, LIU S M, ISLAM S U, YANG J G, ZHANG J, KORMELINK R, DU Z G, WU Z J. The cap snatching of segmented negative sense RNA viruses as a tool to map the transcription start sites of heterologous co-infecting viruses., 2017, 8: 2519.
AnCap-snatching System of Rice Stripe Tenuivirus Based on Crude Virion Preparations
LIN WenZhong, WU Ran, JIN Jing, QIU Ping, ZHANG Jie, WU ZuJian, DU ZhenGuo
(Plant virus research institute, Fujian Agriculture and Forestry University/State Key Laboratory of Ecological Pest Control for Fujian and Taiwan Crops, Fuzhou 350002)
【】Viruses of the orderand the familycleave host cellular mRNAs 10-20 nucleotides downstream of the cap structure and use the 5′ terminal cleavage product as a primer to initiate the transcription of their template RNAs. This process is called cap-snatching, the result of which is that all viral mRNAs contain a host-derived capped-RNA leader (CRL). Rice stripe tenuivirus (RSV) is a plant-infecting bunyavirus for which the mechanism of cap-snatching remains poorly characterized.【】The objective of this study is to establish a convenientsystem to dissect the cap-snatching mechanisms of RSV, this study is designed to test whether crude preparations of RSV snatch CRLs from exogenously supplied mRNAs.【】Crude preparations of RSV were obtained by PEG precipitation combined with differential centrifugation. The protein components of the preparations were analyzed by SDS-PAGE and mass spectrometry. After adding rabbit reticulocyte lysate (RCL) rich inmRNAs, the preparations were used to establish ancap-snatching reaction mix. Total RNA was extracted from the reaction mix at the end of the reaction. RSV mRNAs with CRLs obtained frommRNAs were detected using nested RT-PCR. The products of the nested RT-PCR were cloned, and the sequences were analyzed.【】Two mL of crude RSV preparations was obtained from 100 g of virus-infected rice. The preparations contain chloroplast proteins including ribulose-1,5-bisphosphate carboxylase/oxygenase in addition to RSV virions. A 20 μL reaction system containing 2 μL RSV crude preparations, 8 μL RCL, 4 mmol·L-1MgCl2, 2 mmol·L-1of each NTP and 0.8 U·μL-1RNase inhibitor was prepared and incubated at 30℃ for 1.5 h. Clearly discernable bands with expected sizes were obtained in the nested RT-PCR, indicating that RSV crude preparations can cleave-mRNAs and use their CRLs. After adding the cap analogue m7G (5′) ppp (5′) G, the brightness of the bands was decreased and the extent of the decrease correlated with the concentration of the cap analogue, indicating that cap binding is essential for the cleavage. PCR bands with expected sizes were recovered and sequenced after cloning. Sequence analysis revealed a scenario reminiscent of that seen: RSV cleaves-mRNAs after A or C to obtain CRLs capable of base-pairing with its template RNAs. In initiating transcription with these CRLs, RSV uses the prime-and-realign mechanism frequently and has a greater tendency to use this mechanism in synthesizing nucleoprotein gene () than in synthesizing non-capsid protein gene () mRNAs.【】The crude preparations of RSV can snatch CRLs from exogenously added mRNAs and use the CRLs to prime transcription in a way just like it does. Therefore, RSV crude preparations can be used to dissect the cap-snatching or the transcription of RSV.
rice stripe tenuivirus (RSV); cap-snatching;transcription

10.3864/j.issn.0578-1752.2020.21.012
2020-06-15;
2020-07-13
国家自然科学基金(31672005,31870150)、福建省自然科学基金(2019J01656,2016J05071)
林文忠,E-mail:linwenzhong1991@qq.com。通信作者吴祖建,E-mail:wuzujian@126.com。通信作者杜振国,E-mail:duzhenguo1228@163.com
(责任编辑 岳梅)

