不同生活环境天鹅肠道菌群的多样性分析
曹乐天 王璐阳 张艺凡 张凯慧 董睿龙 王悦欣 李丰波 张龙现*
(1.河南农业大学牧医工程学院,郑州,450046;2.河南黄河湿地国家级自然保护区三门峡管理处,三门峡,472000)
天鹅(Cygnus)是一种大型游禽,为国家Ⅱ级保护动物。天鹅属于脊索动物门(Chordata),鸟纲(Aves),雁形目(Anseriformes),鸭科(Anatidae),天鹅属,有大天鹅(C.cygnus)、小天鹅(C.bewickii)、黑天鹅(C.atratus)等许多种,是鸭科中个体最大的类群。天鹅多群栖在多草的湖泊、水塘、河流等地带,主要以水生植物的叶、根、茎和农作物种子幼苗等为食,也吃水生昆虫、螺类和软体动物[1-2]。天鹅在我国分布较广,国内许多省份均有大天鹅的繁殖地和越冬地,例如河南三门峡、山东荣成等[3];原产于澳洲的黑天鹅在国内主要为人工饲养[4],作为观赏动物;而我国的长江中下游流域则是小天鹅的重要越冬地[5]等。
由于不同地点的天鹅饮食营养和栖息地环境不一样,因此其体内的肠道菌群也有所差异。许多研究表明,肠道菌群在动物健康中起着重要的作用,包括防止病毒、细菌、真菌等病原体的感染,促进免疫系统的发育和成熟,修复受损的上皮组织等[6]。肠道菌群还能够增加宿主从饮食中获取能量的潜力。目前国内还没有对天鹅肠道菌群的研究报道,本研究对3个不同地点生活的天鹅进行采样,分析其体内肠道菌群的差异,希望对天鹅的进一步研究和更好地保护提供科学依据。
1 材料与方法
1.1 样本来源
于2018年12月分别在三门峡市天鹅湖、郑州市动物园和河南农业大学采集天鹅粪便样品,所有粪便样品均为新鲜粪便样品,每份粪便样品去除外层部分采集10—15 g分别收集到单独的采样袋中,并标记采样地点、品种等信息,低温运输至河南农业大学兽医寄生虫学实验室,并于6 h内置于-80 ℃超低温冰箱中保存。
样品分为4组:A组为从河南农业大学一个地点采集3份样品,天鹅为人工饲养黑天鹅,命名为A1—A3;B组样品从郑州市动物园一个地点采集,采集4份,天鹅为人工饲养黑天鹅,命名为B1—B4;C组和D组样品从三门峡的两个地点采集样本,每个地点4份,2个地点的天鹅均为大天鹅迁徙至三门峡越冬,命名为C1—C4,D1—D4。
1.2 粪便DNA提取
将采集的粪便样品严格按照粪便DNA提取试剂盒(EZNA D4015-02)的操作说明进行DNA提取,提取后的DNA样品置于-80 ℃超低温冰箱中保存,然后送至华大基因公司进行测序分析。
1.3 样品处理
DNA样品被接收后,对样品构建文库,用合格的文库进行cluster制备和测序,测序平台为Illumina平台(HiSeq 2 500/4 000),对15份样品进行16S rRNA基因的V4—V5区进行扩增。用下机得到的数据进行相应的生物信息分析。
1.4 数据分析
下机数据经过数据过滤,滤除低质量的reads,剩余高质量的clean data方可用于后期分析;通过reads之间的overlap关系将reads拼接成tags。在97%的序列相似度为阈值进行OTU(operational taxonomic units)聚类,得到每个样品的OTU个数,使用Venn图展示多样品共有和各自特有OTU数目,直观展示样品间OTU的重叠情况。对样品进行PCA分析(principal component analysis),通过分析不同样品OTU(97%相似性)组成反映样品的差异和距离。分析样品alpha多样性(alpha diversity),对单个样品中物种多样性的分析,包括Chao指数、ACE指数以及Shannon指数等。分析样品beta多样性(beta diversity),用来比较1对样品在物种多样性方面存在的差异大小。坐标分析(principal co-ordinates analysis,PCoA)是研究数据相似性或差异性的可视化方法,选择 PC1、PC2、PC3三个特征值,作为距离矩阵中最主要的坐标,展示各个样品间的差异大小。然后通过OTU与Greengene数据库比对,进行物种注释,置信度阈值设置为0.6。使用UPGMA(unweighted pair group method with arithmetic mean)方法进行聚类分析制作聚类树。本实验还通过PICRUSt[7]利用16S rRNA 基因预测其宏基因组,与KEGG数据库(Kyoto Encyclopedia of Genes and Genomes)进行比对,寻找同源基因,对可能存在的二级KEGG通路及丰度值进行预测。
1.5 统计学分析
采用SPSS 26软件(https://www.ibm.com/cn-zh/analytics/spss-statistics-software)统计分析软件中的单因素ANOVA分析各组数据组间差异的显著性,通过Duncan’s新复极差检验法进行各组间的多重比较,结果用“平均数±标准误”表示。
2 结果
2.1 测序结果
去除引物后所有样品一共剩余920 967条tags,平均每个样品61 397条,平均长度376 bp。在97%相似度下将其聚类为用于物种分类的OTU,15个样品共产生2 338个OTU。
2.2 OTU分析
2.2.1 OTU Venn图分析
图1为OTU Venn图,图中不同颜色图形代表不同样品或者不同组别,不同颜色图形之间交叠部分数字为2个样品或2个组别之间共有的OTU个数,多个颜色图形之间交叠部分数字为多个样品或组别之间共有OTU个数。从图中可以看出,A组独有的OTU个数较多,为1 193个,4组共有的OTU个数为174个。

图1 OTU Venn图Fig.1 OTU Venn diagram
2.2.2 OTU PCA分析
基于OUT丰度的PCA显示C组和D组的主成分相似度较高,而A组、B组和CD两组分别在PC1和PC2方向上存在分离,A组和B组的差异主要来自PC2。总体来看,总方差的61.28%来自两个特征值(PC1和PC2),它们是稳定可靠的,结果表明4组的主成分存在较大差异(图2)。横坐标为PC1,反映了最大方差值的特征值,括号中的百分比表示该特征值对样品差异的贡献值;纵坐标为PC2,反映了第二大方差值的特征值。图中点分别表示各个样品,不同颜色代表样品属于不同的分组。
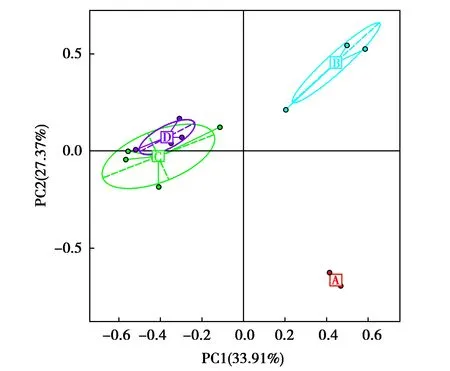
图2 基于OTU丰度的PCA(按description分组)Fig.2 PCA based on OTU abundance(grouped by description)
2.3 Alpha多样性分析
从图3和表1看出,A组的Chao1指数和ACE指数显著高于B、C、D 3组(P<0.05),B、C、D 3组的Chao1指数和ACE指数没有显著差异(P>0.05)。4组的Shannon指数中,C组和D组差异不显著(P>0.05),而A组和B组均与其他组有显著差异(P<0.05)。其中,Chao指数和ACE指数反映样品中群落的丰富度,指数越大,说明样品中的物种越丰富。而Shannon指数反映群落的多样性,Shannon指数越大,说明样品中物种多样性越大。说明A组的菌群物种丰度和多样性显著高于其他组。
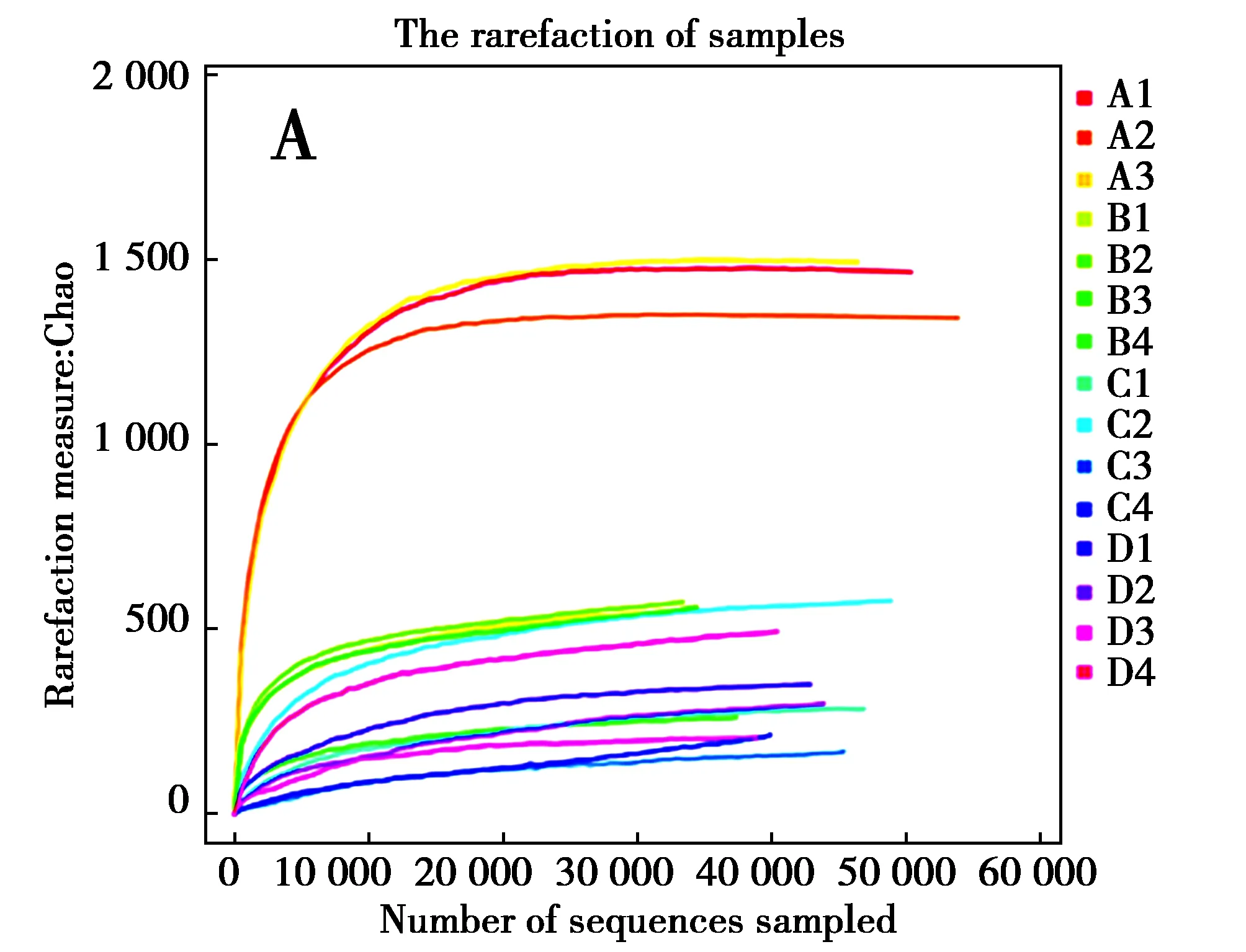
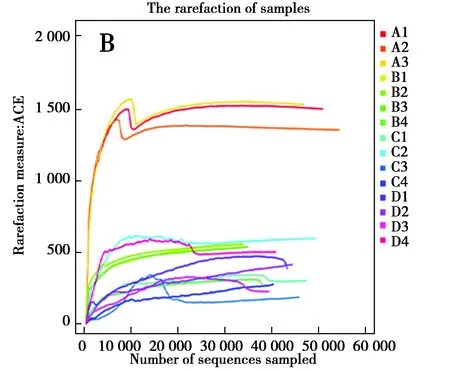
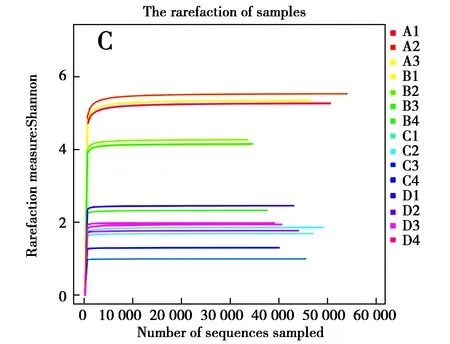
图3 Alpha多样性不同指数的稀释曲线Fig.3 The rarefaction curve of different indices of alpha diversity 注:A:Chao1 指数稀释曲线;B:ACE指数稀释曲线;C:Shannon指数稀释曲线 Note:A:the rarefaction curve of Chao1 index.B:the rarefaction curve of ACE index.C:the rarefaction curve of Shannon index

表1 Alpha多样性不同指数数据分析
2.4 Beta多样性分析
2.4.1 Beta多样性热图
通过图形将beta多样性数据进行可视化,并对样品进行聚类,具有相似beta多样性的样品聚类在一起,反映了样品间的相似性。图4显示,C组和D组的差异较小,物种多样性相似度较高;而A组和B组与其他组的差异较大,物种多样相似度较低。
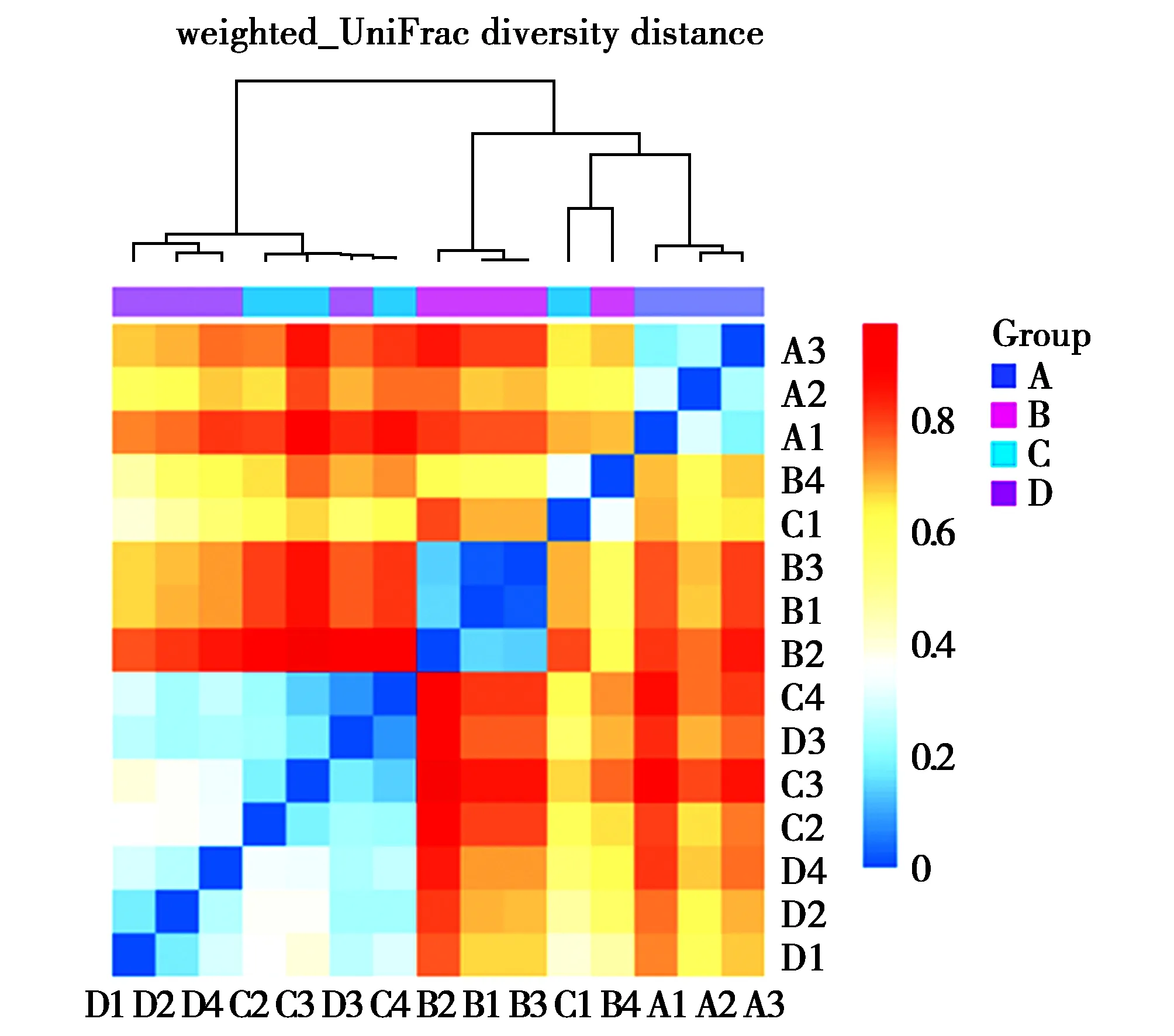
图4 基于weighted UniFrac的beta多样性热图Fig.4 Beta diversity heatmap based on weighted UniFrac
2.4.2 主坐标分析
图5所示为基于 weighted UniFrac的PCoA分析结果。从图中看出,C组和D组聚拢在一起,在3个方向上均聚集在一起,结构差异不显著;CD组与其他两组相比在PC1和PC2方向上分布零散,说明物种组成差异显著。在PC1和PC3方向上,A组和B组聚拢趋势显著,在PC2方向上有一定的差异。如果2个样品距离较近,则表示这2个样品的物种组成较相似。说明4组的组间物种组成差异明显;而组内样本差异较小,具有较好的生物学重复性。
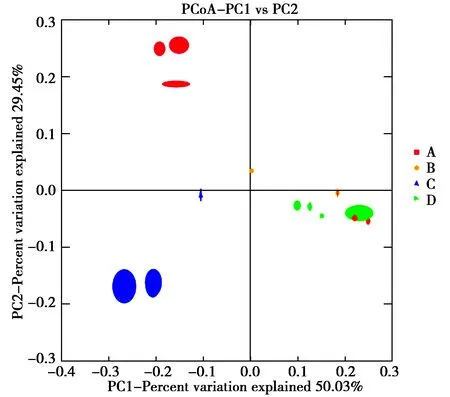


图5 基于weighted UniFrac的PCoA分析Fig.5 The PCoA score plot of gut microbiota based on weighted UniFrac
2.5 样品间物种组成聚类分析
聚类图显示,A组与其他3组处于不同的分支,说明与其他组的物种组成相似度低。B组与其他组处于不同分支但有一个样品和C组一样品归类于一分支。而C组和D组样品较为靠近,枝长较短,说明2个组的物种组成较为相似(图6)。
2.6 肠道菌群相对丰度差异分析
物种柱状图可以直观地展示各样本物种组成及比例,反映样本间物种的变化情况。横坐标是样本名称,纵坐标是注释到的物种相对丰度。该分类水平未注释到的合并为Unclassified,丰度在所有样品均低于0.5%的物种全部合并成Others。
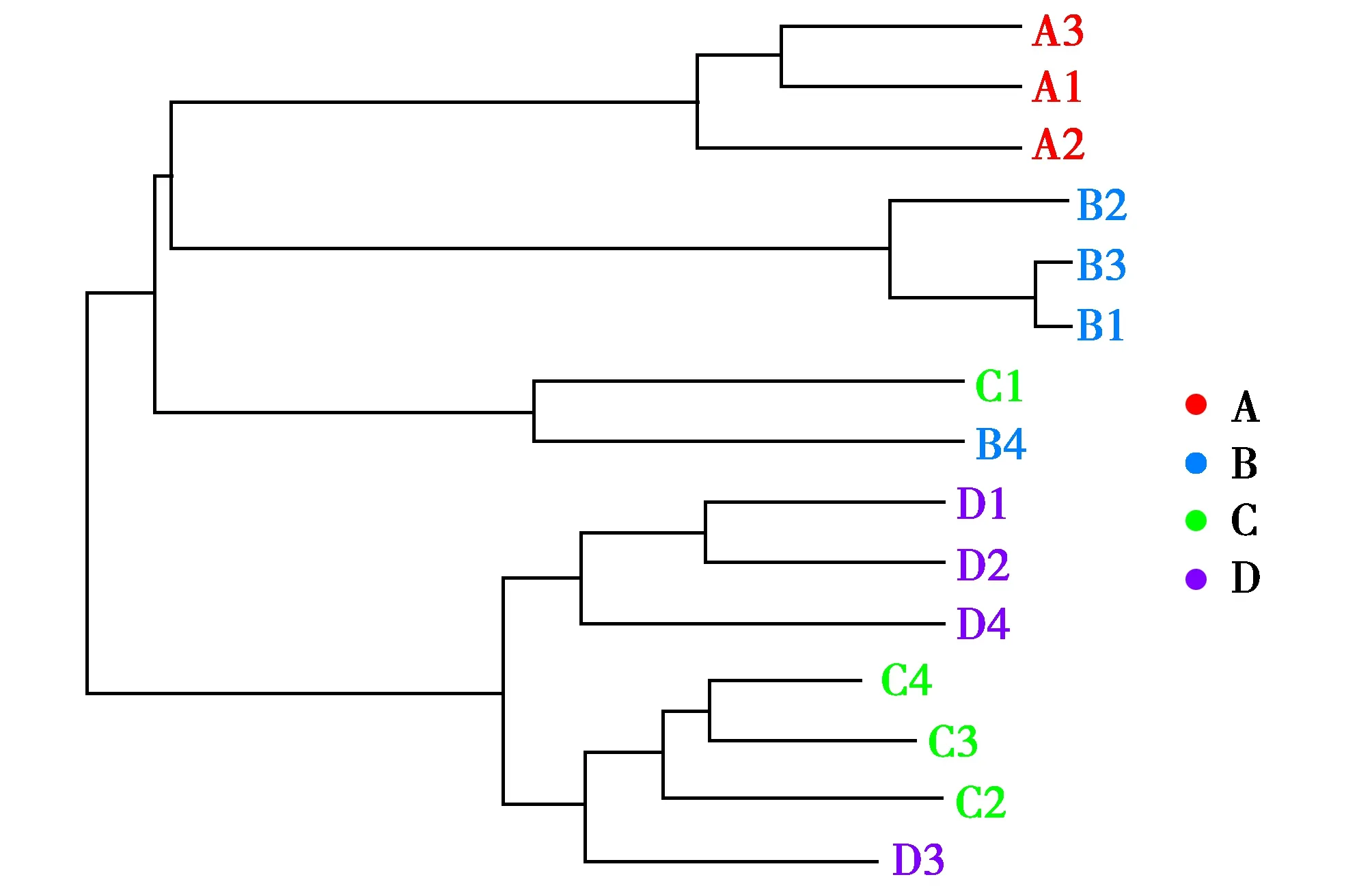
图6 样品间物种聚类分析图(weighted UniFrac)Fig.6 Analysis of species clustering among samples(weighted UniFrac)
2.6.1 门水平肠道菌群结构相对丰度差异分析
在门水平上(图7),各组样品所检测到的优势细菌类群(相对丰度>1%)结构间具有明显差异:A组的核心菌门由变形菌门(Proteobacteria,相对丰度为30.70%,下同)、蓝藻菌门(Cyanobacteria,22.41%)、厚壁菌门(Firmicutes,15.31%)、拟杆菌门(Bacteroidetes,14.38%)、放线菌门(Actinobacteria,11.78%)和梭杆菌门(Fusobacteria,2.35%)构成。B组的核心菌门由厚壁菌门(42.08%)、拟杆菌门(41.80%)、梭杆菌门(11.90%)和变形菌门(2.58%)构成。C组的核心菌门由厚壁菌门(77.48%)、梭杆菌门(13.53%)和变形菌门(7.328%)构成。D组的核心菌门由厚壁菌门(89.71%)、梭杆菌门(6.30%)、蓝藻菌门(1.67%)和变形菌门(1.45%)构成。
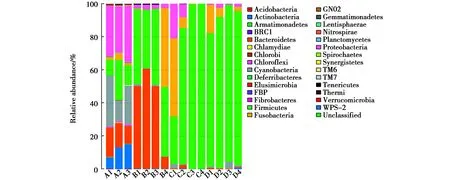
图7 门水平物种丰度柱状图Fig.7 Species abundance histogram of Phylum level
2.6.2 属水平肠道菌群结构相对丰度差异分析
在属水平上(图8),各组样品所检测到的优势细菌类群(相对丰度>4%)结构间具有明显差异:A组的核心菌属由氢噬胞菌属(Hydrogenophaga,相对丰度为4.62%,下同)、假鱼腥藻属(Pseudanabaena,4.07%)和普雷沃菌属(Prevotella,4.02%)构成。B组的核心菌属由拟杆菌属(Bacteroides,32.56%)、梭杆菌属(Fusobacterium,11.89%)、Turicibacter(7.09%)、梭菌属(Clostridium,4.73%)和巨单胞菌属(Megamonas,4.18%)构成。C组的核心菌属由链球菌属(Streptococcus,44.86%)、梭杆菌属(13.50%)、肠球菌属(Enterococcus,6.88%)、弯曲菌属(Campylobacter,5.87%)和Turicibacter(5.67%)构成。D组的核心菌属由Turicibacter(20.30%)、链球菌属(18.24%)、粪球菌属(Coprococcus,6.84%)、梭菌属(6.31%)、梭杆菌属(6.28%)和肠球菌属(4.19%)构成。
值得注意的是,在C组和D组的优势菌群中,链球菌属、梭杆菌属、肠球菌属、弯曲菌属、梭菌属的一些细菌能感染人类并导致疾病。
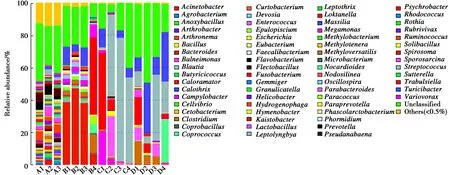
图8 属水平物种丰度柱状图Fig.8 Species abundance histogram of Genus level
2.7 天鹅肠道菌群的功能预测分析
因为很多细菌自身具有不同数目的16S rRNA基因拷贝数,利用PICRUS预测其宏基因组,与KEGG数据库进行比对,寻找同源基因,对可能存在的二级KEGG通路及丰度值进行预测,以柱状图表示,柱高表示相对丰度。4组中共鉴定出25条丰富的二级代谢途径,其中21条途径在各个组之间存在显著差异。较为常见的通路有氨基酸代谢、碳水化合物代谢、细胞过程和信号、能量代谢、膜运输、复制和修复等,这些途径大多与能量代谢、生长和发育有关(图9)。
各组差异代谢通路相对丰度中,C组和D组之间无显著差异。氨基酸代谢通路中A组与B组相似,显著高于C组和D组。跨膜转运通路A组与B组相似,显著低于C组和D组。能量代谢通路A组显著高于B组,B组显著高于C组和D组。脂质代谢通路A组显著高于其他3组(表2)。
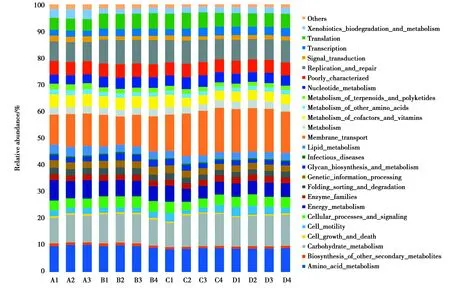
图9 KEGG代谢通路分类柱状图(二级)Fig.9 Classification histogram of KEGG metabolic pathway(level 2)
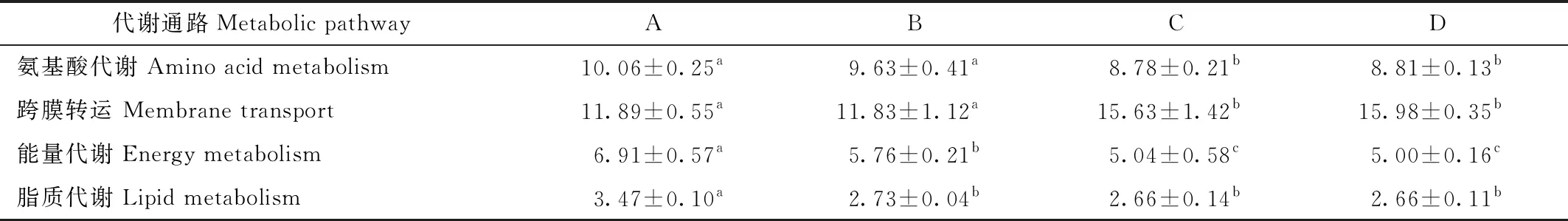
表2 各组部分差异代谢通路
3 讨论
天鹅是我国目前珍稀鸟类养殖业最受欢迎的经济野禽之一[8]。我国有大量天鹅的繁殖地和越冬地,以及人工养殖的天鹅。研究表明饮食营养和栖息地环境都是动物肠道微生物群落的潜在驱动因素[9-12]。但国内还没有关于天鹅的肠道菌群研究,而肠道菌群可以在营养和能量新陈代谢、免疫动态平衡和繁殖方面发挥着作用[13]。本研究利用Illumina HiSeq平台,基于16S rRNA基因高通量测序研究不同地点天鹅的肠道菌群差异。
本研究样品来源中,A组的黑天鹅饲养于学校人工湖中,每天定时投喂饲料,可以自由活动和觅食,能够接触外界环境;B组的黑天鹅饲养于动物园中,主要为人工喂养,活动面积较大,能够接触外界环境;C组和D组为迁徙至三门峡越冬的大天鹅,每年需要在繁殖地和越冬地之间迁徙,多为自主觅食。试验结果显示,4组之间的OTU个数均有差异,尤其是A组OTU数目远高于其他3组,这表明不同的生活环境会影响天鹅肠道菌群的丰富度。另外基于OTU水平的PCA分析结果显示,C组和D组的主成分在PC1和PC2方向上相似度都较高,这是由于这2组样品均来自迁徙越冬的大天鹅,具有相似的生活环境和食物来源,因此肠道菌群主成分较为相似;而A组和B组与其他组均有较大差异,特别是PC2方向上,这说明不同的生活环境能够影响天鹅肠道菌群的结构组成。
Chao1指数、ACE指数和Shannon指数是评价肠道菌群丰富度和多样性的指标,A组Chao1指数、ACE指数显著高于其他3组,说明其菌群丰度非常高,这可能是因为人工饲喂和能够自由的活动觅食的原因。Shannon指数中,A组显著高于其他3组,而且B组也显著高于C组和D组,说明A组的菌群多样性最高,B组其次。Beta多样性分析中,热图结果显示,C组和D组物种组成较为相似,这两组与A组和B组的物种组成差异较大,差异主要在PC1和PC2方向上;A组和B组之间的差异主要在PC2方向上。物种聚类分析图结果证明了beta多样性分析结果。以上结果表明,不同的生活环境及采食方式能够影响天鹅肠道菌群的物种多样性,主要原因可能是食物来源的差异,A组天鹅和B组天鹅饲养环境为人工饲养,可以自由活动觅食,而C组天鹅和D组天鹅饲养环境更接近自然条件。另外一些内生因素也可能对天鹅肠道菌群产生影响[14]。
先前研究发现,厚壁菌门和变形菌门是野生天鹅和养殖天鹅门水平上的优势菌群,相对丰度较高[14],它们通常存在于脊椎动物的胃肠道[15],在其他野生鸟类肠道菌群中同样是优势菌群,如鹦鹉(Amazonafarinosa,Araararauna,Arachloropterus)[16]、鸮鹦鹉(Strigopshabroptilus)[17]、鹈燕(Pelecanoidesurinatrix)[18]和斑头雁(Anserindicus)[19]。在本研究中这2种菌门同样是4组共有的优势菌群,两门细菌的相对丰度之和在每组均高于45%。除此之外,本研究中各组的其他优势菌门分别是A组的蓝藻菌门、拟杆菌门、放线菌门和梭杆菌门;B组的拟杆菌门、梭杆菌门;C组的梭杆菌门;D组梭杆菌门、蓝藻菌门。
在属水平上,各组的优势菌群组成差异明显。A组的核心菌属为氢噬胞菌属、假鱼腥藻属和普雷沃菌属,氢噬胞菌属和假鱼腥藻属常存在于水体的浮游细菌群落[20-21],这也说明了A组天鹅食物来源的广泛。B组的核心菌属拟杆菌属显著高于其他组,拟杆菌也存在于人类体内,是肠道微生物群落的主要成员,通过消化复杂的多糖产生有益物质,为宿主提供了多种益处[22]。链球菌属和肠球菌属细菌为C组和D组天鹅的优势菌群,两属细菌相对丰度之和分别为每组的51.74%和22.43%,显著高于其他组。其中,链球菌属细菌是化脓性球菌中常见的一大类革兰阳性球菌,有69个种和亚种,广泛分布于自然界和人体的鼻咽部、胃肠道等处,大多为正常菌群,部分为致病性链球菌,如牛链球菌(Streptococcusbovis)与人类中菌血症、感染性心内膜炎、结直肠癌和脑膜炎等疾病的发病有关[23],溶脂链球菌(St.gallolyticus)能导致人短暂性菌血症[24],肺炎链球菌(St.pneumoniae)会引起脑膜炎、中耳炎、菌血症和肺炎等疾病[25]等。而肠球菌属细菌普遍存在于人和动物的粪便中,并且在环境中持续存在,被认为是水中粪便污染的指示物[26]。肠球菌属细菌是机会性病原体,能引起败血症、心内膜炎和尿路感染等疾病,已成为医院和社区中获得性人类感染的主要原因之一[27]。肠球菌属目前有50多个种,以粪肠球菌(Enterococcusfaecalis)和屎肠球菌(En.faecium)为主,占分离株的80%以上[28],这两种细菌也是在人体肠道中检测到的最具代表性的肠球菌种类[29]。由于肠球菌属细菌在环境中有很强的适应性,因此需注意其公共卫生安全的意义。除此之外,C组和D组中的优势菌属中,梭杆菌属细菌被认为是一种与人类结直肠癌相关的主要肠道菌群[30];弯曲菌属(Campylobacter)是导致人类腹泻的最常见细菌之一,可引起一系列疾病,包括急性肠炎、肠道外感染和感染后并发症[31];梭菌属细菌与多种人类和动物疾病有关,例如艰难梭菌(Clostridiumdifficile,伪膜性肠炎)[32]、肉毒梭菌(Clostridiumbotulinum,婴儿型肉毒中毒)[33]、破伤风梭菌(Clostridiumtetani,破伤风)[34]和产气荚膜梭菌(Clostridiumperfringens,急性坏死性小肠结肠炎)[35],相关的病理归因于它们产生的毒素[36]。以上结果表明迁徙天鹅具有潜在的公共卫生威胁。先前研究发现,迁徙的天鹅肠道微生物群的核心类群经过长距离迁徙后高度保守[37],因此需要加强迁徙天鹅繁殖地和越冬地的环境卫生管理,避免其污染水源,造成人类感染。
本研究通过PICRUSt来预测相关的代谢通路,研究不同生活环境对天鹅肠道菌群的影响。4组较为常见的通路有氨基酸代谢、碳水化合物代谢、细胞过程和信号、能量代谢、膜运输、复制和修复等,这些途径大多与能量代谢、生长和发育有关。C组和D组之间的二级代谢通路相对丰度无显著差异,说明它们的肠道微生物组成相似,因此肠道微生物群的功能也相似。C组和D组中,氨基酸代谢、能量代谢通路丰度显著低于其他组,跨膜转运通路丰度显著高于其他组;A组脂质代谢通路丰度显著高于其他3组。差异较显著的代谢通路主要与能量相关代谢相关,氨基酸代谢是蛋白质水解生成的氨基酸的过程,氨基酸在体内有多种作用,如合成蛋白质和多肽、转化为糖和脂质、氧化释放能量和免疫调节作用[38];能量代谢是指在物质代谢过程中所伴随着的能量的贮存、释放、转移和利用过程;脂质代谢是脂类在体内消化、吸收和转运储存于脂肪组织,为机体供能和储能,脂类物质还能转变为各种衍生物参与代谢活动[39]。3种通路在C组和D组的丰度低于其他组,而A组和B组较高,因此推测栖息地环境和饮食的不同,迁徙天鹅摄入的营养物质较人工喂养天鹅的少,导致菌群结构的改变,影响天鹅能量相关代谢通路。跨膜转运是将物质通过细胞外表的质膜和细胞内的各种细胞器膜的过程[40],在各组比重较高(均大于11%),且在C组和D组大于A组和B组,说明其通过增强物质的跨膜转运来实现对物质的需求,这或许由于能量代谢不足而导致的。由于数据库及方法的限制,PICRUSt分析存在一定的缺陷,需要结合更多的方法进行肠道菌群功能的解读。
4 结论
本研究首次分析了在不同地点生活的天鹅肠道菌群的差异,证明了饮食营养和栖息地环境会影响天鹅肠道菌群组成,同时阐述了迁徙天鹅携带的人兽共患细菌,表明其对公共卫生安全具有威胁,同时发现迁徙天鹅的能量相关代谢通路丰度低于人工喂养天鹅。这为进一步研究天鹅肠道菌群多样性和天鹅在不同生活环境中的适应机制提供科学依据,并为加强公共卫生安全提供理论支持。

