白菜型油菜粒色主效基因BrTT1的调控机制分析
王艳花 荐红举 邱 晓 李加纳,*
白菜型油菜粒色主效基因的调控机制分析
王艳花1,2荐红举1邱 晓2李加纳1,*
1西南大学农学与生物科技学院/ 油菜工程研究中心, 重庆 400715;2萨斯喀彻温大学, 加拿大萨斯卡通 S7N5A8
白菜型油菜(L., 2= 20, AA)为十字花科芸薹属作物, 属于栽培油菜基本种。我国是白菜和白菜型油菜的起源中心, 与甘蓝型油菜相比, 其起源和栽培历史悠久, 遗传资源丰富, 具有天然而稳定的黄籽资源。大黄油菜是白菜型油菜中一种天然的黄籽资源, 其粒色鲜黄, 黄籽性状能够稳定遗传, 且具有大粒、含油量高、自交亲和性好等优点, 是研究油菜粒色性状的良好材料。本研究对白菜型油菜粒色主效基因进行了进一步的功能验证, 并对的粒色调控机制进行了初步解析。序列比较结果表明,在不同遗传背景白菜型油菜黄籽、红褐籽、黑籽品种中存在固定的差异位点, 同一粒色材料所得序列一致, 可用于预测粒色表型; 酵母双杂交分析表明, BrTT1可以与另外2个转录因子R2R3-MYB (BrTT2)和WD40 (BrTTG1)以及一个催化酶(BrTT3)相互作用。qRT-PCR结果表明, 超量和干扰的表达, 导致类黄酮合成路径结构基因及其他关键调节基因、的异常表达或不表达, 从而阻碍了原花色素的正常积累。研究结果进一步明确了在白菜型油菜粒色形成中的调节活性。
白菜型油菜;; 类黄酮代谢; 酵母双杂
油菜是世界上重要的油料作物, 菜籽油是重要的食用油和生物柴油来源, 菜籽饼粕是优良的动物蛋白饲料[1-4]。白菜型油菜作为在世界范围内广泛种植的芸薹属油料作物之一, 包含许多优异、稳定的天然黄籽资源。自20世纪70年代以来, 国内外许多研究者对油菜黄籽性状的大量研究表明, 在相同遗传背景下, 黄籽油菜的含油量、蛋白质含量均高于黒籽油菜, 且黄籽油菜具有皮壳率低、木质素和多酚含量低、抗营养物质(植酸、单宁、芥子碱、硫苷等)含量低等一系列优良品质性状[5-19]。通过对这些黄籽材料粒色性状的研究, 可以为白菜型黄籽油菜育种方案的制订提供指导, 促进白菜型油菜品种的改良。同时由于甘蓝型油菜中不存在天然的黄籽资源, 白菜作为甘蓝型油菜的供体亲本之一, 研究者常利用种间杂交或人工合成等方法将白菜型油菜中的黄籽性状导入甘蓝型油菜[20-26]。通过对白菜型油菜黄籽性状的研究, 以期为甘蓝型黄籽油菜遗传机理的分析奠定基础。
油菜种子的种皮色素、大小、成分影响油菜的产量和品质[24-25]。大黄油菜是白菜型油菜中一种天然的黄籽资源, 其粒色鲜黄, 黄籽性状能够稳定遗传, 且具有大粒、含油量高、自交亲和性好等优点, 是研究油菜粒色性状的良好材料[26]。目前对拟南芥透明种皮的形成机理较为清楚, 可为黄籽油菜种皮色素积累的研究提供借鉴, 但黄籽油菜种皮色素的形成及调控机理仍有待研究。Li等[27]对拟南芥和白菜型油菜种皮结构的解剖学观察证实了拟南芥与白菜型油菜种皮结构不同, 拟南芥与油菜种皮色素积累的方式应该存在潜在的差异。作为油菜中重要的粒色调控基因, 研究其调控机制具有重要意义[28-31]。然而, 目前关于及其他粒色主效基因在白菜型油菜粒色调控机制解析的报道较少。在前期研究中, 通过全基因组重测序结合遗传连锁图谱对白菜型黄籽油菜大黄的粒色基因进行了精细定位, 并对粒色候选基因进行克隆及功能分析, 初步鉴定了为控制大黄粒色的主效基因[30]。本研究将搜集不同来源的白菜型油菜黄、黑籽材料, 分析在其他白菜型油菜的序列差异, 并对在大黄油菜中的功能进行验证, 初步明确了该转录因子在类黄酮代谢路径中可能调控的靶基因,揭示下游粒色调控网络。解析对白菜型油菜粒色的分子调控机制, 以期为白菜型和甘蓝型油菜粒色研究奠定基础。
1 材料与方法
1.1 供试材料
不同遗传背景的白菜型油菜共22份, 主要包括粒色纯合的6份黄籽材料(Y1~Y6)、9份红褐籽材料(R1~R9)和7份黑籽材料(B1~B7), 均由西南大学油菜工程研究中心提供(附表1)。
1.2 不同遗传背景白菜型油菜中BrTT1的克隆及序列分析
选取Y1~Y6、R1~R9、B1~B7籽粒饱满的种子, 用纸盘发苗, 温度设置为22~25℃, 浇足水, 暗光培养2~3 d, 再移到光照培养室, 待长出4片叶子, 选取单株嫩叶, 采用CTAB (Cetyltrimethylammonium Ammonium Bromide)法[32]提取DNA, 利用紫外分光光度计检测每个单株的DNA浓度, 并稀释至50 ng μL-1, 冻于-20℃保存备用。采用TA克隆的方法[33]获得序列信息。取油菜授粉后21、28、35、42、49、56 d的种子, 取样后放-80℃冰箱备用, 采用北京Biomed柱式小量植物总RNA抽提试剂盒提取RNA。采用Geneious Basic 4.8.5软件进行基因序列的比对; 利用MEGA6.0软件及NCBI数据库BLASTP分析序列。
1.3 BrTT1互作蛋白预测
在BRAD (http://brassicadb.org/brad/)数据库获取白菜型油菜中与拟南芥类黄酮代谢相关基因的同源基因信息, 根据STRING (https://string-db.org/cgi/ input.pl)网站在线预测与BrTT1的互作蛋白, 选取预测分值大于0.5的互作蛋白做进一步验证。
1.4 酵母双杂分析BrTT1互作蛋白
采用Primer Premier 5.0软件设计引物Yb-F/-R (附表2), 克隆的CDS序列, 以pGBKT7为载体构建融合表达诱饵载体pGBKT7-BrTT1; 采用Primer Premier 5.0软件设计基因特异引物(附表2)分别克隆白菜型油菜类黄酮代谢相关基因、、、、、、、、、的CDS序列, 以pGADT7为载体构建融合表达捕获载体pGADT7-BrTT2、pGADT7-BrTT3、pGADT7-BrTT4、pGADT7-BrTT8、pGADT7-BrTT18、pGADT7-BrTTG1、pGADT7-BrTTG2、pGADT7-BrTT10、pGADT7-BrTT7、pGADT7-BrTT5与pGBKT7-BrTT1质粒分别共转入酵母细胞Y2HGold, 并涂布在SD/-Leu/-Trp/-Ade/-His及SD/-Leu/-Trp/-Ade/-His/+X-α-gal的平板上, 在30℃恒温培养3 d, 同时设阳性对照组(pGADT7-T + pGBKT7-53)和阴性对照组(pGADT7-T + pGBKT7-laminC), 筛选BrTT1的互作蛋白。
1.5 超量和抑制表达BrTT1载体构建及遗传转化
采用Primer Premier 5.0软件设计引物gCb-F/-R (附表2), 以白菜型油菜褐籽材料cDNA为模板, 采用TA克隆方法获得基因的CDS序列, Axygen胶回收试剂盒回收目的基因, 采用Gateway方法将胶回收片段连入骨干载体pEarlyGate101。连接产物转入大肠杆菌DH5α, 挑选阳性克隆送擎科生物测序。采用液氮冷激的方法将阳性克隆转化农杆菌菌株感受态细胞GV3101, 构建超量表达载体以pFGC5941为骨干载体, 构建干扰载体, 构建方法参照Ma等[34]。参考Cardoza和Stewart[35]的农杆菌介导的遗传转化体系, 将构建好的表达载体送武汉双螺旋生物公司完成遗传转化试验。
1.6 RNA提取和荧光定量分析方法
根据BRAD数据库信息, 利用白菜型油菜中已知类黄酮途径参考基因的CDS序列设计定量引物(附表3), 采集田间或温室不同发育时期(开花后21、28、35、42、48、56 d)的黄、黑籽种子, 用液氮冷冻后, 保存于-80℃超低温冰箱备用。待所有不同发育时期的种子取样完成后, 使用EASYspin RNA Rapid Plant Kit (Biomed)提取RNA, 使用PrimeScript RT reagent Kit with gDNA Eraser (TaKaRa, http://www.takara.com.cn/)合成cDNA。qRT-PCR反应体系为20 μL, 包含10 μL 2× Promega GoqPCR Master Mix、1 μL 20 μmol L-1正向引物、1 μL 20 μmol L-1反向引物、100 ng cDNA第一链, 添加ddH2O至20 μL。反应程序为94℃ 2 min; 94℃ 3 s, 60℃ 30 s, 40个循环; 60~95℃, 熔解曲线分析。使用2-DDCt法计算基因的表达量[36],基因作为内参基因。
2 结果与分析
2.1 芸薹属作物中TT1同源基因家族成员的序列差异分析
在BRAD (http://brassicadb.org/brad/)数据库下载获取芸薹属作物同源基因家族成员, 包括甘蓝型油菜和、白菜型油菜、甘蓝、芥菜型油菜的氨基酸序列, 采用ClustalW (http://www.ebi.ac.uk/clustalw)及GEN EDOC软件进行氨基酸序列比对分析。其中与的氨基酸序列存在9处差异, 一致性达95%, 而与的氨基酸序列存在4处差异,与的氨基酸序列存在7处差异, 相似度均大于97.5% (图1)。白菜型油菜与甘蓝型油菜的相似度大于甘蓝型油菜与之间的相似度, 且芸薹属作物不同种之间的氨基酸序列相似度均大于95%, 说明在进化过程中有一定的序列保守性。

图1 BrTT1同源基因氨基酸序列比对
2.2 不同遗传背景白菜型油菜中BrTT1的克隆及表达分析
从22份不同遗传背景的白菜型油菜(包括6个黄籽材料Y1~Y6、9个红褐籽材料R1~R9和7份黑籽材料B1~B7)中克隆了基因序列(图2-A, B)。根据BRAD数据库信息可知, 该基因包含有2个外显子和1个内含子。Geneious软件对测序得到的序列比对结果表明, 所有的黑籽材料(B1~B7)序列一致; 9份不同来源的红褐籽材料(R1~R9)扩增获得的序列表现一致, 且与黑籽材料序列相比,在第834 bp处(以起始密码子ATG为起点)多1个碱基A, 979 bp处存在1处碱基替换T→C; 黄籽材料Y2、Y3、Y4、Y5、Y6扩增得到的序列与前期对大黄油菜粒色主效基因的研究结果一致, 黄籽序列相同; 与序列相比,内含子区域存在17处SNP (single nucleotide polymorphisms)差异、2处片段缺失, 在外显子区域存在7处SNP差异, 分别是A→C、T→A、G→T、A→T、G→A、C→A、A→T, 发生有义突变的为A→C、C→A、A→T, 第2个外显子中的最后2个SNP (CNP, ANP)导致的DNA结合区域发生2个氨基酸变化(S氨基, H氨基), 这可能导致的功能丢失[31]。
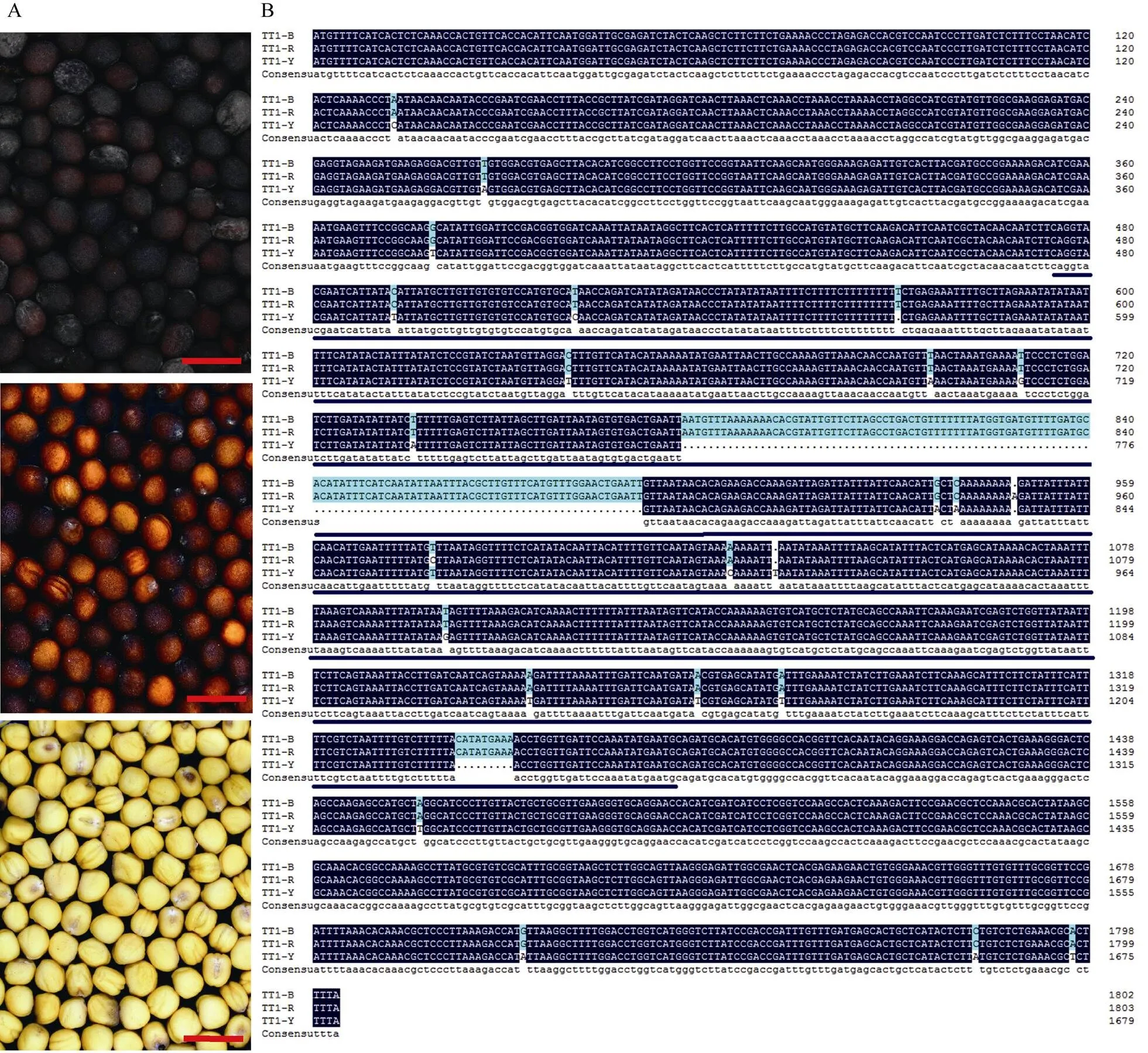
图2 不同粒色的BrTT1基因的核苷酸序列比对
A: 不同粒色的白菜型油菜种子表型(黑色、红褐色和黄色); B: 来自不同粒色的的核苷酸序列。TT1-B: 从黑籽中扩增得到的序列; TT1-R: 从红褐籽种子中扩增得到的序列; TT1-Y: 从黄籽中扩增得到的序列。核苷酸多态性通过颜色突出显示, 共有序列下方的线代表内含子区域。标尺= 5000 μm。
A: phenotypes oflines (black, reddish-brown, and yellow) with different seed colors; B: nucleotide sequences of.from the three lines with different seed color. TT1-B was-sequence amplified from the black seeds; TT1-R was-sequence amplified from the reddish-brown seeds; TT-Y was-Y sequence amplified from the yellow seeds. Nucleotide polymorphisms were highlighted by color, the line below the consensus sequence represents the intron region. Bar = 5000 µm.
2.3 BrTT1在白菜型油菜不同粒色形成期种子中的表达分析
在红褐、黑籽种子中的表达量均高于黄籽中, 且黑籽和红褐籽种子中的表达没有显著差异(图3)。表明-的内含子中2个片段的缺失以及外显子的SNP差异可能对在种子形成期的表达具有重要影响。前期研究已知全长序列为4586 bp, 包含启动子片段1796 bp、终止子下游序列986 bp、编码区序列1804 bp。等位基因序列分析发现,在黄、褐籽的启动子序列相同[37]。因此推测内含子中片段缺失以及外显子的SNP差异会影响到mRNA的剪接或稳定性。说明在白菜型油菜油菜中粒色调控过程中均发挥重要的作用。
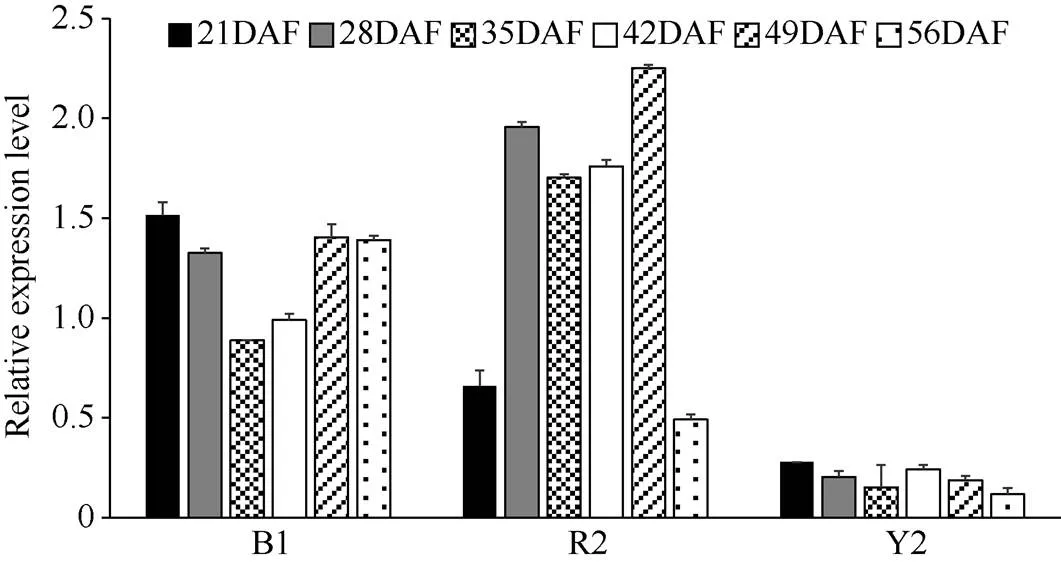
图3 BrTT1基因在3种不同粒色种子形成期的表达分析
B1: 黑色种子系; R2: 红褐籽种子系; Y2: 黄籽种子系。DAF: 开花后天数。
B1: a black seed line; R2: a reddish-brown seed line; Y2: a yellow seed line. DAF: days after flowering.
2.4 BrTT1互作蛋白预测
将BrTT1 (Bra028067)信息提交至STRING (https://string-db.org/cgi/input.pl)网站在线预测与BrTT1的互作蛋白(图4), 结果显示, 在白菜型油菜中与BrTT1互作的蛋白共有20个(表1), 其中类黄酮途径的互作蛋白有12个, 分别为BrTT2、BrTTG1、BrTT8、BrTTG2-A、BrTTG2-B、BrTT7、BrTT10-1A、BrTT10-2、BrTT12、BrTT5、BrTT3和BrTT18。
2.5 酵母双杂交筛选BrTT1互作蛋白
分别设计可能与BrTT1互作的类黄酮代谢基因(打分值在0.568~0.996)的特异引物(附表3), 克隆各基因CDS序列分别构建AD载体, 克隆基因CDS序列构建pGBKT7-BrTT1载体。将各捕获载体分别与诱饵载体pGBKT7-BrTT1共转化到酵母菌株Y2H Gold中, 同时设阳性对照组(pGADT7-T + pGBKT7-53)和阴性对照组(pGADT7-T + pGBKT7-laminC)。由图5可知, BrTT1与BrTT2、BrTT3、BrTTg1均有较强的互作, 而与类黄酮途径其他蛋白则没有发生互作, 推测BrTT1在类黄酮途径中可能的互作蛋白有BrTT2、BrTT3、BrTTg1。
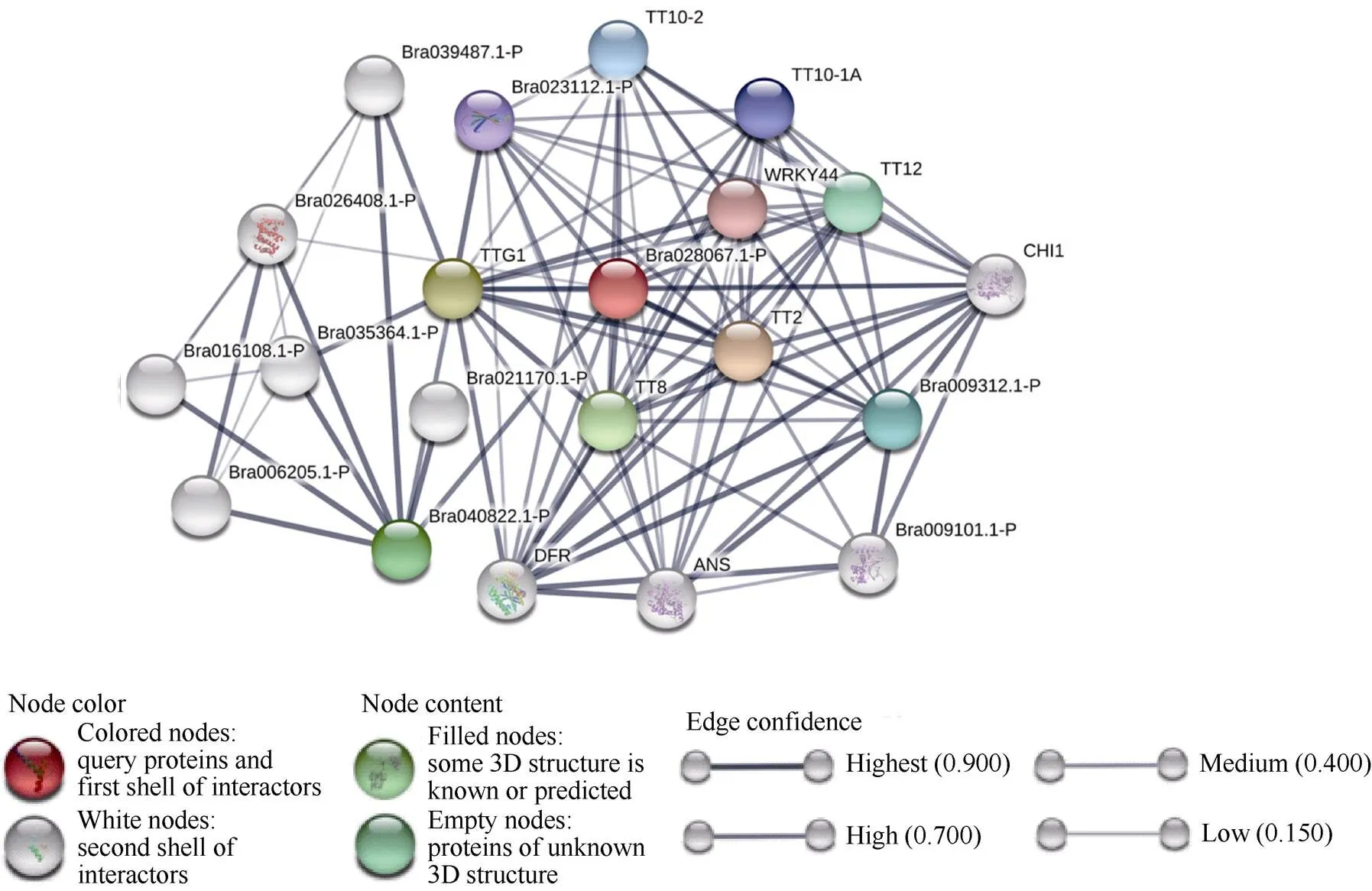
图4 白菜型油菜BrTT1的互作蛋白预测分析
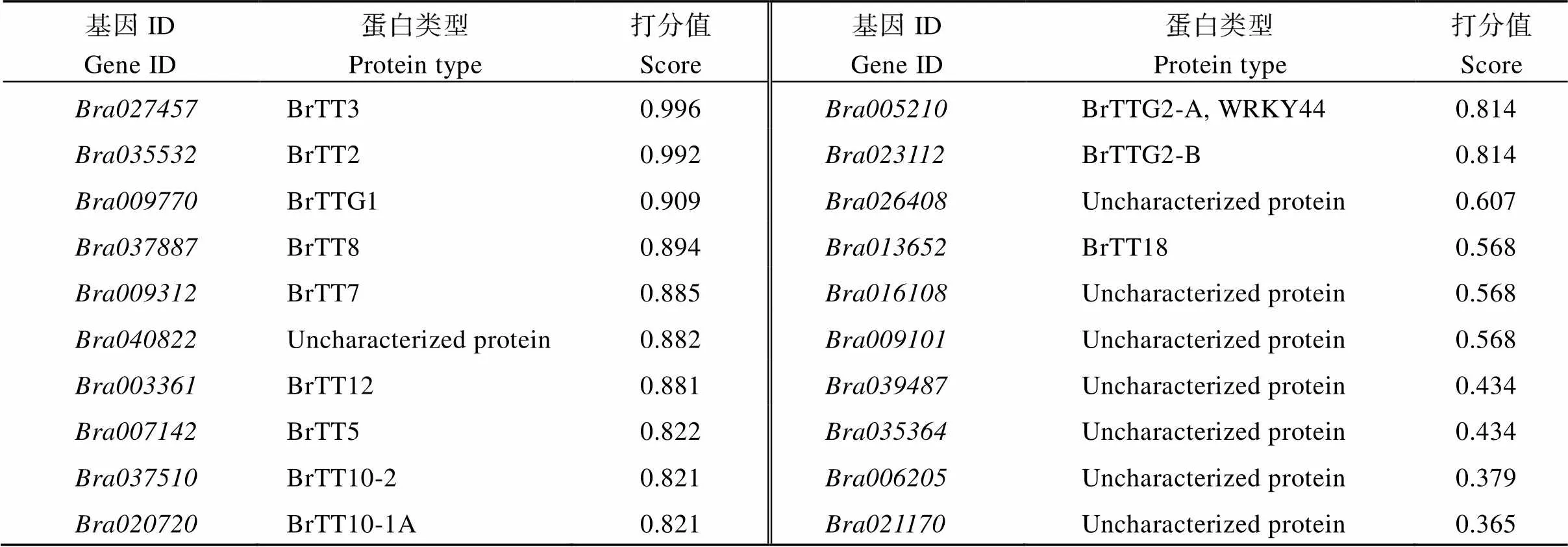
表1 预测白菜型油菜BrTT1的互作蛋白的信息

图5 酵母双杂筛选BrTT1可能的互作蛋白
酵母细胞涂布于-Leu/-Trp/-Ade/-His的培养基, pGADT7- T/pGBKT7-53作为阳性对照, pGADT7-T/pGBKT7-laminC作为阴性对照。X-α-gal的染色深度表示互作强弱。
Yeast cells were co-transformed and plated on -Leu/-Trp/-Ade/-His plates, with pGADT7-T/pGBKT7-53 as the positive control and pGADT7-T/pGBKT7-laminC as the negative control. X-α-gal activity was determined by colony-lift filter assay; the color intensity of the colonies depicts the qualitative binding strength of the interaction.
2.6 转基因株系中BrTT1的差异表达
为进一步探索的功能, 本研究构建了的超量表达载体和RNAi干扰载体转化白菜型油菜浩油11号。并通过PCR检测鉴定获得干扰表达的转基因株系(附图1)和超量表达的转基因株系(附图2)。在干扰株系中,基因的表达显著低于对照株系B (图6-A), 从而导致干扰株系转基因种子种皮色素积累减少(图6-B), 表明干扰可以减少转基因种子中黑色素的生物合成和沉积。与非转基因褐籽材料相比, 过量表达所得转基因种子中,的表达量及成熟种子的粒色并无显著差异(图6-A, B), 表明在白菜型油菜粒色中具有重要的调节功能。
2.7 转基因株系中类黄酮代谢相关基因的差异表达
为进一步了解可能的调控活性, 取过表达和抑制表达后, 转基因株系发育阶段的种子(开花后10、20、30、40 d)为材料, 通过qRT-PCR表达谱分析了类黄酮途径基因的表达水平。根据差异表达情况, 可将这些基因分为3类(图7), 第1类为正调控表达基因, 主要包括、、、、、和, 即的下调表达降低了这些基因的表达, 而的上调表达则提高了它们在发育中种子的表达, 表明这组基因可能受到的正向调控; 第2类包括、和, 其表达模式与第1类相反, 即下调表达可增加这些基因的表达, 而上调则可降低这组基因表达, 表明这些基因受负向调控; 第3组包括、、和, 其表达对表达的变化无反应, 在2种不同类型的转基因品系中均未观察到明显的变化, 表明这些基因未受调控。
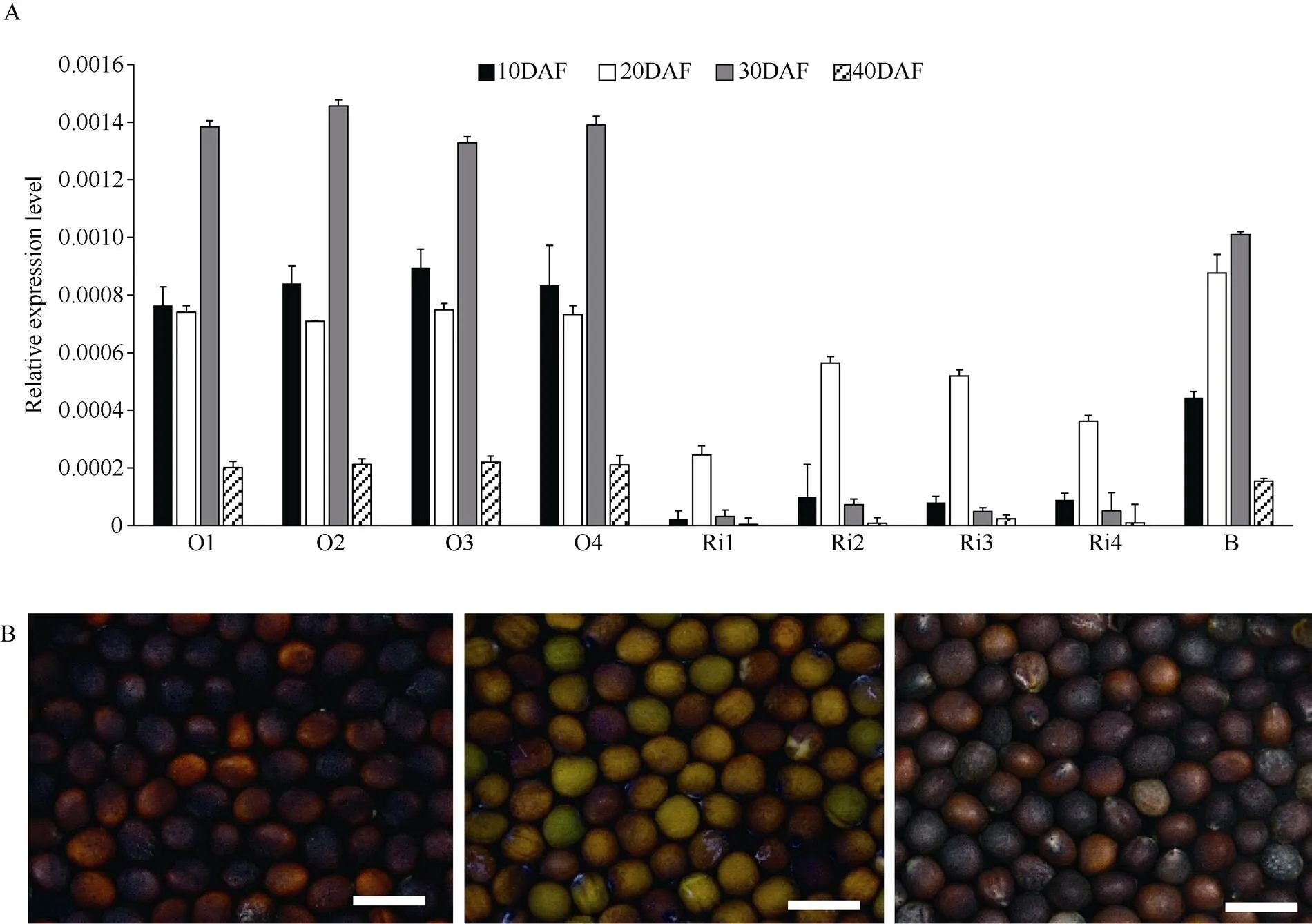
图6 过量和干扰表达BrTT1基因
A: 转基因种子中的差异表达; O1~O4: 过量表达转基因株系; Ri1~Ri4: 干扰表达转基因株系; B: 非转基因黑籽材料; DAF: 开花后天数。B: 左、中、右分别为过量表达、干扰表达基因、非转基因褐籽对照成熟种子表型。标尺= 5000 μm。
A: differential expression analysis ofin seeds of the transgenic lines; O1–O4: over-expression lines; Ri1–Ri4: RNAi expression lines; B: non-transgenic lines as the control; DAF: days after flowering. B: left, middle and right represent seed color phenotype in transgenic lines of over-expression, RNAi expression, and the non-transgenic black seed lines, respectively. Bar = 5000 μm.
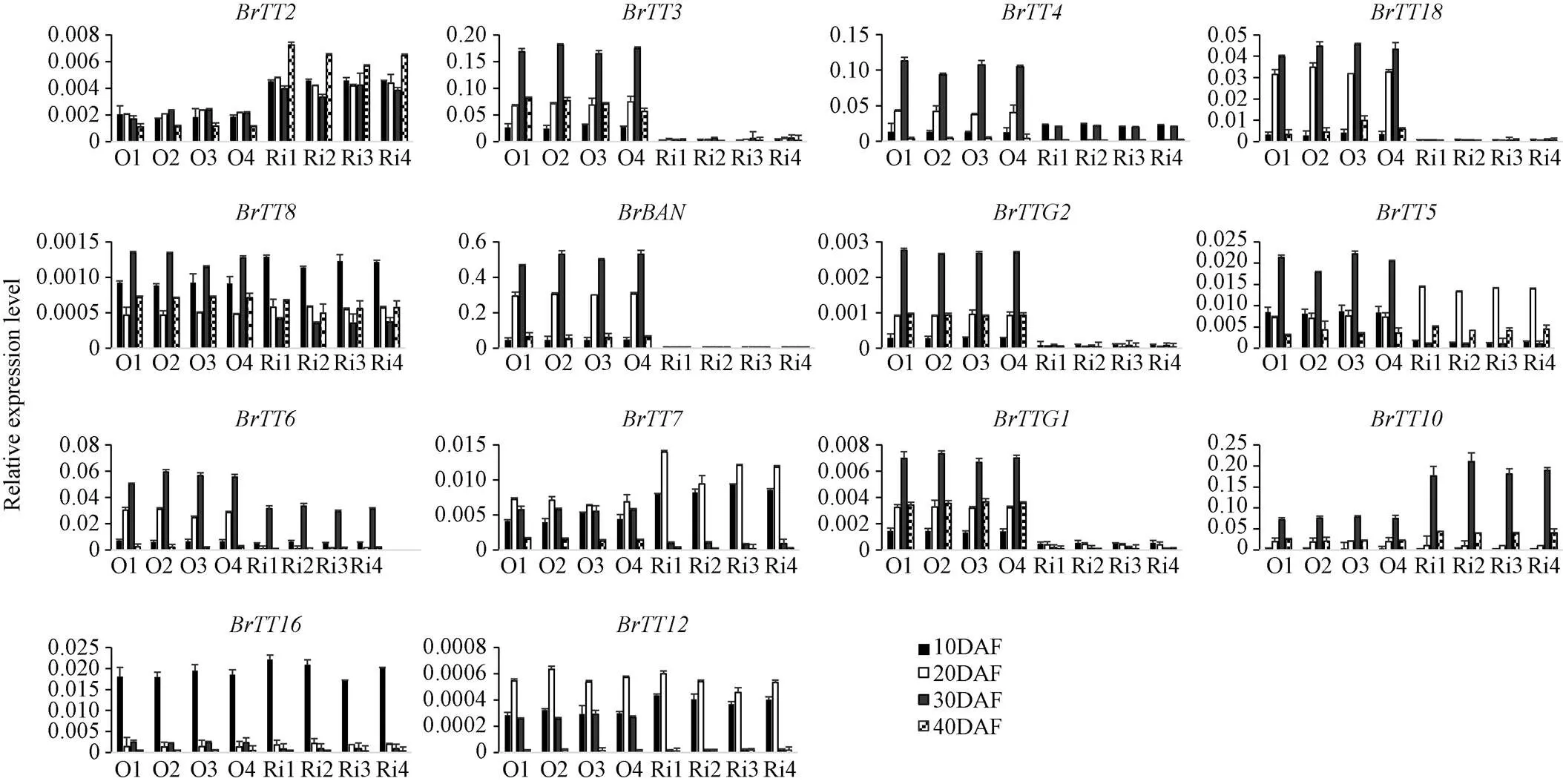
图7 RNAi和超量表达BrTT1基因的转基因株系中类黄酮相关基因的表达分析
O1~O4: 过量表达转基因株系; Ri1~Ri4: 干扰表达转基因株系; DAF: 开花后天数。
O1–O4: over-expression lines; Ri1–Ri4: RNAi expression lines; DAF: days after flowering.
3 讨论
原花色素是一种广泛存在于植物果实、树叶、种皮中的植物色素, 其生物合成及合成途径中主要基因功能的解析已在拟南芥、葡萄等植物中有较详细的研究, 主要由公共苯丙烷途径、类黄酮途径及原花色素途径组成的复合途径[38-46]。油菜的种皮颜色是由原花色素决定, 且黄籽油菜种皮中不含有原花色素[47-48], 在油菜中原花色素的合成路径及调控机制尚未完全清楚。模式植物拟南芥原花色素代谢途径主要涉及到12个结构基因、6个转录因子及3个转运蛋白的调控[49]。植物类黄酮代谢反应犹如一个链条式结构, 该结构中涉及的每一个酶的突变都可能导致整个代谢路径的中断, 原花色素积累受阻[50-51]。在白菜型油菜中, BrTT1被认为是调节种皮色素的生物合成和沉积的主要转录因子[30-31]。本研究对22份不同粒色(黑籽、红褐籽和黄籽)的白菜型油菜品系中进行了克隆测序。序列比较表明,序列在不同粒色材料中存在固定的差异位点。与黑籽和红褐籽中的相比, 来自黄籽的序列在2个外显子区域共有7个SNP位点, 而在内含子区域具有2个片段缺失, 这可能导致黄籽中调节活性丧失。qRT-PCR分析表明,在黄籽品系和RNAi转基因系中的表达非常低。这些结果进一步证明,在白菜型油菜粒色性状中具有重要的调控作用, 白菜型种子粒色表型可以通过序列多态性来确定, 与前研究结果在白菜和甘蓝型油菜中沉默或下调表达, 导致种皮中黄酮类化合物的减少, 从而形成黄籽的研究结果相一致[28-31]。在芥菜型油菜中, 从不同的粒色材料(黄籽和褐籽)克隆了, 并鉴定到8个单核苷酸多态性(SNP)位点, 但未检测到的差异表达[28]。
拟南芥AtTT1作为调节因子能够与转录因子R2R3-MYB ()和WD40转录因子()相互作用, 从而调节结构基因如()、()和()的表达[52-54]。酵母双杂分析表明, BrTT1可以与BrTT2、BrTT3和BrTTG1相互作用, 已有研究表明在拟南芥中TT1与TT2和TTG1的相互作用, 其中TT2的异位表达可以部分恢复突变体的表型[49]。此外, 本研究检测到BrTT1与BrTT3的相互作用, 在白菜型油菜中,()编码二氢黄酮醇4-还原酶,受2个转录因子BrMYB2-2和BrTT8调控[55-56]。尽管通过酵母双杂系统检测到白菜型油菜中BrTT1与BrTT3的相互作用, 且干扰和过量表达的转基因品系中的表达受到了明显的影响, 但仍需要更多试验来进一步证明BrTT1和BrTT3在白菜型油菜中是否存在相互作用。
在甘蓝型油菜中,、、、、和在黄籽种子中表达量较低, 黄籽与褐籽之间的表达差异显著。此外,的沉默表达降低类黄酮合成相关基因的表达, 如、、、和[31]。本研究表明, 白菜型RNAi转基因品系中下调的表达导致、、、、、和的表达降低。和编码2个转录因子(WD40和WRKY),编码查尔酮合酶(CHS), 这是类黄酮途径中催化丙二酰辅酶A和4-香豆酰辅酶A缩合的第一种酶[39-41]。、、和是类黄酮生物合成早期和中期重要的结构基因。这些基因的表达均受到的正向调控。此外, 与的表达呈负向调控的结构基因主要包括、和。编码黄烷酮3'-羟化酶(F3'H),编码漆酶15 (LAC15), 将原花青素(PA)聚合成更大的聚合物并进一步氧化原花色素, 从而使种皮呈现褐色[57], 这一类基因受负调控。根据我们之前对大黄油菜中相应的功能表达谱的研究,、、、、和在黄籽种子中表达较低, 但在近等基因系褐籽种子中表达较高。该结果与本研究RNAi转基因系中类黄酮代谢的下调表达的结果相一致。进一步表明是调节白菜型油菜粒色性状的关键基因之一。的调控活性的分子解析为芸薹属作物种皮中类黄酮的生物合成研究提供借鉴。
4 结论
前期研究对白菜型黄籽油菜大黄的粒色性状进行了基因精细定位、克隆及拟南芥中遗传转化分析, 初步明确了大黄油菜粒色性状受1对隐性基因控制。本研究在此基础上明确在其他白菜型油菜黄籽、红褐籽、黑籽品种中的序列及表达特性, 同一粒色材料所得序列完全一致, 且不同粒色材料存在固定的差异位点, 为更多不同遗传背景的白菜型油菜粒色研究奠定基础。通过酵母双杂及超量和干扰的表达, 解析控制粒色可能的顺式作用元件, BrTT1与R2R3-MYB转录因子(TT2)及TTG1转录因子互作影响类黄酮合成路径结构基因的表达, 初步解析调控油菜粒色的分子机制, 为白菜型油菜优质育种提供新思路, 同时也为甘蓝型油菜粒色调控网络的研究提供借鉴。
[1] He Y T, Tu J X, Fu T D, Li D R, Chen B Y. Genetic diversity of germplasm resources ofL. in China by RAPD markers., 2002, 28: 697–703.
[2] Chen X, Wu J, Liu K. Genetic diversity comparison between spring and weak-wintercultivars using single- locus SSR markers., 2010, 32: 6–13.
[3] Wang J L, Chang T J, Cheng H H, Fang H L. Study on character evolution and cladistic taxonomy of wild rapes (and) in Tibet., 2008,17: 10–17.
[4] Chrungu B, Verma N, Mohanty A, Pradhan A, Shivanna K R. Production and characterization of interspecific hybrids betweenand crop., 1999, 98: 608–613.
[5] Sensoz S, Angin D, Yorgun S. Influence of particle size on the pyrolysis of rapeseed (L.): fuel properties of bio-oil., 2000, 19: 271–279.
[6] Rahman M H, Joersbo M, Poulsen M H. Development of yellow-seededof double low quality., 2001, 120: 473–478.
[7] Rahman M H. Production of yellow-seededthrough interspecific crosses., 2001, 120: 463–472.
[8] Whetten R W, Mackay J J, Sederoff R R. Recent advances in understanding lignin biosynthesis., 1998, 49: 585–609.
[9] Meng J L, Shi S W, Gan L, Li Z Y, Qu X S. The production of yellow-seeded(AACC) through crossing interspecific hybrids of(AA) and(BBCC) with., 1998, 103: 329–333.
[10] Mukhlesur R M, Hirata Y. Homology of seed coat color specific marker ofwith brown seeded cultivar of., 2004, 4: 731–734.
[11] Shirzadegan M, Röbbelen G. Influence of seed color and hull proportion on quality properties of seeds inL., 1985, 87: 235–237.
[12] Akhov L, Ashe P, Tan Y F, Datla R, Selvaraj G. Proanthocyanidin biosynthesis in the seed coat of yellow-seeded, canola qualityYN01-429 is constrained at the committed step catalyzed by dihydroflavonol 4-reductase., 2009, 87: 616–625.
[13] Simbaya J, Slominski B A, Rakow G, Campbell L D, Downey R K, Bell J M. Quality characteristics of yellow-seededseed meals: protein, carbohydrate, and dietary fiber components., 1995, 43: 2062–2066.
[14] Ren Y J, He Q, Ma X M, Zhang L G. Characteristics of color development in seeds of brown- and yellow-seeded heading Chinese cabbage and molecular analysis of Brsc, the candidate gene controlling seed coat color., 2017, 8: 1410–1418.
[15] Ye X, Li J N, Tang Z L. Difference of seed coat color between black-and yellow-seeded inwith seed development changes of anthocyanin, phenylalanine and phenylalaine ammonia-lyase and their correlation analyses., 2002, 28: 638–643.
[16] Fu F Y, Liu L Z, Chai Y R, Chen L, Yang T, Meng Y J, Ma A F, Yan X Y, Zhang Z S, Li J N. Localization of QTLs for seed color using recombinant inbred lines ofin different environments., 2007, 50: 840–854.
[17] Schwetka A. Inheritance of seed colour in turnip rape (L.)., 1982, 62: 161–169.
[18] Vera C L, Woods D L, Downey R K. Inheritance of seed coat color in., 1979, 59: 635–637.
[19] Li X, Chen L, Hong M, Zhang Y, Zu F, Wen J, Yi B, Ma C Z, Sheng J X, Tu J X, Fu T D. A large insertion in bHLH transcription factorresulting in yellow seed coat in., 2012, 7: e44145.
[20] Dixon R A, Xie D Y, Sharma S B. Proanthocyanidins—a final frontier in flavonoid research., 2005, 165: 9–28.
[21] Alois H D, Klíma, Miroslav K, Viehmannová I, Milan O U, Eloy F C, Miroslava V. Efficient resynthesis of oilseed rape (L.) from crosses of winter types×via simple ovule culture and early hybrid verification., 2015, 120: 191–201.
[22] Deynze A E V, Landry B S, Pauls K P. The identification of restriction fragment length polymorphisms linked to seed colour genes in., 1995, 38: 534–542.
[23] Zhang Y, Li X, Ma C Z, Shen J X, Chen B Y, Tu J X, Fu T D. The inheritance of seed color in a resynthesizedline No. 2127-17 including a new epistatic locus., 2009, 31: 413–419.
[24] Rahman M, Mcvetty P B E, Li G. Development of SRAP, SNP and multiplexed SCAR molecular markers for the major seed coat color gene inL., 2007, 115: 1101–1107.
[25] Öztürk Ö. Effects of source and rate of nitrogen fertilizer on yield, yield components and quality of winter rapeseed (L.)., 2010, 70: 132–141.
[26] Ahmed S U, Zuberi M I. Effects of seed size on yield and some of its components in rapeseed,L. var Toria., 1973, 13: 119–120.
[27] Xiao L, Zhao Z, Du D, Yao Y M, Xu L, Tang G Y. Genetic characterization and fine mapping of a yellow-seeded gene in Dahuang (alandrace)., 2012, 124: 903–909.
[28] Yan M L. Cloning and SNP analysis ofgene in., 2010, 36: 1634–1641.
[29] Lian J P, Lu X C, Yin N W, Ma L J, Lu J, Liu X, Li J N, Lu J, Lei B, Wang R, Chai Y R. Silencing offamily genes affects seed flavonoid biosynthesis and alters seed fatty acid composition in., 2017, 254: 32–47.
[30] Wang Y H, Xiao L, Guo S M, An F Y, Du D Z. Fine mapping and whole-genome resequencing identify the seed coat color gene in., 2016, 11: e0166464.
[31] Wang Y H, Xiao L, Dun X L, Liu K D, Du D Z. Characterization of thegene responsible for seed coat color formation in Dahuang (L. landrace)., 2017, 37: 137–150.
[32] Yan M, Wei G, Pan X H, Ma H L. A method suitable for extracting genomic DNA from animal and plant-modified CTAB method., 2008, 36: 500–504.
[33] Li J G, Han G Y, Li X M, Sun J J, Song K J, Zhang T. Improvement of TA cloning method to facilitate direct directional cloning of PCR products., 2014, 565: 3–8.
[34] Ma L J, Feng Y, Jiang L P, Shen M, Chai Y R. Modification of pFGC5941 and construction of RNAi vector ofgene () family., 2010, 18: 1189–1190.
[35] Cardoza V, Stewart C N. Increased-mediated transformation and rooting efficiencies in canola (L.) from hypocotyl segment explants., 2003, 21: 599–604.
[36] Schmittgen T D, Livak K J. Analyzing real-time PCR data by the comparative CT method., 2008, 3: 1101–1108.
[37] 王艳花. 大黄油菜粒色性状候选基因的定位克隆及功能分析. 青海大学博士学位论文, 青海西宁, 2017. Wang Y H. Positional Cloning and Functional Study of Seed Coat Color Gene in Dahuang (L. landrace). PhD Dissertation of Qinghai University, Xining, Qinghai, China, 2017 (in Chinese with English abstract).
[38] Feinbaum R L, Ausubel F M. Transcriptional regulation of thechalcone synthase gene., 1988, 8: 1985–1992.
[39] Marek M, Sebastian K, Takayuki T, Federico M G, Olivia W, Malcolm M C, Alisdair R F, Björn U, Zoran N, Staffan P. PlaNet: combined sequence and expression comparisons across plant networks derived from seven species., 2011, 23: 895–910.
[40] Hartmann U, Valentine W J, Christie J M, Hays J, Jenkins G I, Weisshaar B. Identification of UV/blue light-response elements in thechalcone synthase promoter using a homologous protoplast transient expression system., 1998, 36: 741–754.
[41] Li X, Bonawitz N D, Weng J K, Clint C. The growth reduction associated with repressed lignin biosynthesis inis independent of flavonoids., 2010, 22: 1620–1632.
[42] Jiang W B, Yin Q G, Wu R R, Zheng G S, Liu J Y, Dixon R A, Pang Y Z. Role of a chalcone isomerase-like protein in flavonoid biosynthesis in., 2015, 66: 7165–7179.
[43] Pelletier M K. Molecular and Biochemical Genetics of 2-oxoglutarate-dependent Dioxygenases Required for Flavonoid Biosynthesis in. PhD Dissertation of Virginia Tech, Blacksburg, Virginia, America, 1997.
[44] Han Y P, Sornkanok V, Ruth E S G, Sergio R M, Zheng D M, Anatoli V L, Schuyler S K. Ectopic expression of apple F3'H genes contributes to anthocyanin accumulation in themutant grown under nitrogen stress., 2010, 153: 806–820.
[45] Abrahams S, Tanner G J, Ashton L A R. Identification and biochemical characterization of mutants in the proanthocyanidin pathway in., 2002, 130: 561–576.
[46] Sato S, Tabata S. The complete genome sequence of., 2001, 46: 61–65.
[47] Matsui K, Tanaka H, Ohme-Takagi M. Suppression of the biosynthesis of proanthocyanidin inby a chimeric PAP1 repressor., 2004, 2: 487–493.
[48] Gonzalez A, Zhao M, Leavitt J M, Lloyd A M. Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex inseedlings., 2008, 53: 814–827.
[49] Appelhagen I, Lu G H, Huep G, Schmelzer E, Weisshaar B, Sagasser M.interacts with R2R3 MYB factors and affects early and late steps of flavonoid biosynthesis in the endothelium ofseeds., 2011, 67: 406–419.
[50] Quattrocchio F, Baudry A, Lepiniec L, Grotewold E. The regulation of flavonoid biosynthesis., 2006, 179: 79–86.
[51] Shijun S. The study of seed coat color in yellow-seeded., 2003, 22: 608–612.
[52] Baudry A, Heim M A, Dubreucq B, Caboche M, Weisshaar B, Lepiniec L.,, andsynergistically specify the expression of BANYULS and proanthocyanidin biosynthesis in., 2010, 39: 366–380.
[53] Su F, Hu J, Zhang Q L, Luo Z R. Isolation and characterization of a basic Helix–Loop–Helix transcription factor gene potentially involved in proanthocyanidin biosynthesis regulation in persimmon (Thunb.)., 2012, 136: 115–121.
[54] Xu W J, Dubos C, Lepiniec L. Transcriptional control of flavonoid biosynthesis by MYB-bHLH-WDR complexes., 2015, 176–185.
[55] Wendell D L, Anoumid V, Gurbaksh S. The gene encoding dihydroflavonol 4-reductase is a candidate for the anthocyaninless locus of rapid cycling(Fast Plants Type)., 2016, 11: e0161394.
[56] Ahmed N U, Park J I, Jung H J, Yang T J, Hur Y K, Nou I S. Characterization of dihydroflavonol 4-reductase (DFR) genes and their association with cold and freezing stress in., 2014, 550: 46–55.
[57] Zhang K, Lu K, Qu C M, Liang Y, Wang R, Chai Y R, Li J N. Gene silencing offamily genes causes retarded pigmentation and lignin reduction in the seed coat of., 2013, 8: e61247.

附图1 PCR扩增检测RNAi得到的阳性转基因植株
Supplementary Fig. 1 PCR amplification of a transgene fragment in transgenic plants withdown-regulated by RNAi using genomic DNA isolated from the seedlings as templates
检测引物为F35S3ND/FBTT1I (附表2), 检测片段1.0 kb。+表示阳性对照。
The primer of F35S3ND/FBTT1I (Supplementary Table 2) was used for the amplification of a fragment size at about 1.0 kb. + is a positive control.
附图2 PCR扩增检测过表达得到的阳性转基因植株
Supplementary Fig. 2 PCR amplification of a transgene fragment in transgenic plants over-expressingusing genomic DNA isolated from the seedlings as templates
检测引物为F35S3ND/FBTT1I (附表2), 检测片段1.0 kb。+表示阳性对照。
The primer of F35S3ND/CB-R (Supplementary Table 2) was used for the amplification of a fragment size at about 1.0 kb. + is a positive control.
附表1 不同来源的白菜型油菜黄、红、黑籽材料
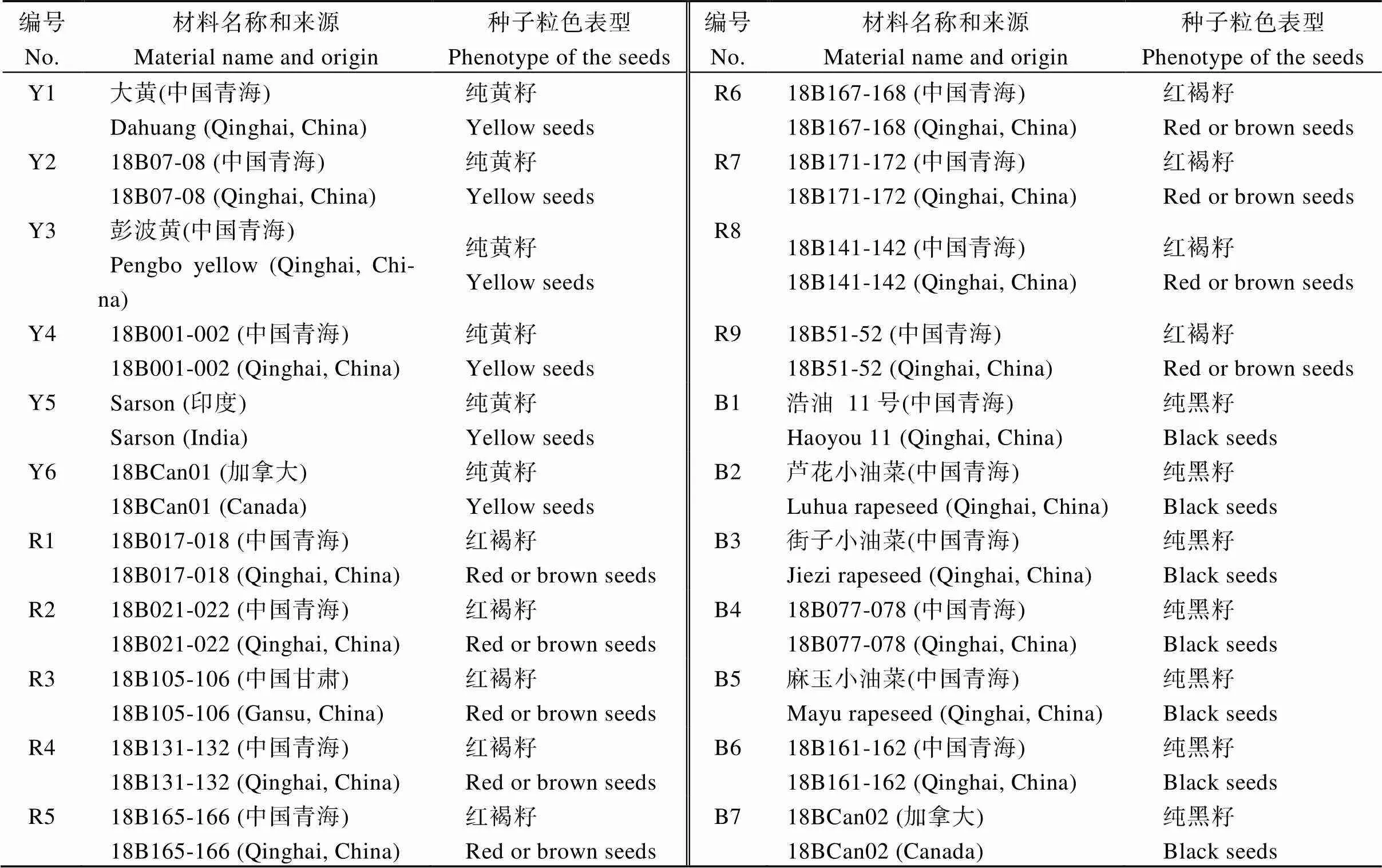
Supplementary Table 1 Yellow- and red- and black-seeded materials in B. rapa
附表2 用于载体构建的引物序列

Supplementary Table 2 Primers sequences for vector construction
附表3 用于差异表达分析的引物序列

Supplementary Table 3 Primer sequences for differential expression analysis
Regulatory mechanism of the seed coat color geneinL.
WANG Yan-Hua1,2, JIAN Hong-Jiu1, QIU Xiao2, and LI Jia-Na1,*
1College of Agronomy and Biotechnology, Southwest University/ Chongqing Engineering Research Center for Rapeseed, Chongqing 400715, China;2University of Saskatchewan, Saskatoon S7N5A8, Canada
(L., 2= 20, AA) is a specie ofgenus, belonging to the basic species of cultivated rapeseed. China is the original center of Chinese cabbage and. Compared with, it has a long history of origin and cultivation and rich genetic resources, which has natural and stable yellow seed resources. Dahuang has the natural yellow seed resource in. Its seed coat color is bright yellow, the yellow seed trait can be stably inherited, and Dahuang has the advantages of large grain, high oil content and good self-adhesiveness. Sequence comparison showed that nucleotide polymorphisms were solely found insequences from different seed color lines (yellow, red or brown, and black), which could be used to predict seed color phenotype. Yeast two-hybrid analysis indicated BrTT1 could interact with two other transcriptional factorsR2R3-MYB (BrTT2) and WD40 (BrTTG1), and one catalytic enzyme (BrTT3). Quantitative RT-PCR analysis of transgeniclines with the gene down-regulated by RNA interference and up-regulated by overexpression revealed that two contrasting groups of genes were regulated byin the biosynthesis and deposition of flavonoids pigments in the seed of. These results further define the regulatory activity ofin seed coat color formation inspecies.
;; flavonoid; yeast two-hybrid
10.3724/SP.J.1006.2020.04036
本研究由中国博士后科学基金面上项目(2019M653319), 重庆市自然科学基金博士后科学基金项目(cstc2019jcyj-bsh0102)和高等学校学科创新引智基地项目(“111”项目)(B12006)资助。
This study was supported by the Project of China Postdoctoral Science Foundation (2019M653319), the Project of Chongqing Natural Science Foundation Postdoctoral Science Foundation (cstc2019jcyj-bsh0102), and Project of Intellectual Base for Discipline Innovation in Colleges and Universities (“111” Project) (B12006).
李加纳, E-mail: ljn1950@swu.edu.cn, Tel: 023-68250642
E-mail: hawer313@163.com
2020-02-17;
2020-07-02;
2020-07-15.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20200714.1154.004.html

