基于多重PCR和第二代高通量测序技术快速检测下呼吸道感染病原微生物方法的建立和应用*
郑凯文,黄晓园,陈渡波,张俊杰,王菊芳△,徐鸿绪▲
(1.华南理工大学生物科学与工程学院,广东广州 510006;2.中山大学附属第一医院医学检验科,广东广州 510080)
下呼吸道感染是临床最常见的感染性疾病之一,具有很高的病死率[1]。根据世界卫生组织(WHO)2018年公布的2016年统计数据,全球前十位死亡原因中下呼吸道感染高居第四位,在传染性疾病中位列第一,共导致约300万人死亡[2]。传统的培养法是检测下呼吸道感染病原体的“金标准”,但整个过程通常包括病原体培养、鉴定和抗菌药物敏感性试验,需要2~4 d甚至更长时间[3]。血清学方法也是下呼吸道病原体检测的重要方法之一,但存在通量低,易出现假阳性结果等缺陷[4]。为了及时控制病情,临床上常常采取经验性用药,待得到培养结果及药敏测试结果后再进行调整。但这也容易导致抗菌药物使用不当,使病原微生物耐药性出现及蔓延的概率增加[5],导致下呼吸道感染的诊断和治疗更为棘手,成为当今全球共同面临的重大卫生挑战[6-7]。因此,开发新的、更准确及高通量的下呼吸道感染病原微生物检测方法具有重要意义。
本研究采用生物信息学方法,针对肺炎克雷伯菌、化脓性链球菌、鲍曼不动杆菌等20种下呼吸道感染常见病原微生物设计40对特异性引物,通过PCR方法验证引物的敏感性和特异性,建立多重PCR联合第二代高通量测序技术,即AC-Seq检测体系。将该方法应用于临床肺泡灌洗液标本的检测并与临床培养结果进行对比,旨在确立一种检测下呼吸道感染常见病原微生物的快速、高通量方法,为下呼吸道感染病原体检测技术的开发提供参考。
1 资料与方法
1.1一般资料 根据《下呼吸道感染实验诊断规范》中下呼吸道感染病原谱[8]、中国细菌耐药监测网(CHINET)以及一些大型三甲医院的统计数据[9-12],本研究选择了20种下呼吸道感染常见病原微生物作为研究对象,分别是肺炎克雷伯菌、化脓性链球菌、鲍曼不动杆菌、阴沟肠杆菌、铜绿假单胞菌、卡他莫拉菌、金黄色葡萄球菌、黏质沙雷菌、流感嗜血杆菌、洋葱伯克霍尔德菌、嗜麦芽窄食单胞菌、产气肠杆菌、大肠埃希菌、奇异变形杆菌、肺炎链球菌、嗜肺军团菌、屎肠球菌、粪肠球菌、白色念珠菌、近平滑假丝酵母菌。所有标准菌株均购自广东省微生物菌种保藏中心。34份临床肺泡灌洗液来自2018年11-12月中山大学附属第一医院医学检验科的送检标本。依据国家卫生健康委员会2001年颁布的《医院感染诊断标准(试行)》[13]对下呼吸道感染病例进行诊断入组,排除临床资料不完整的病例。
1.2仪器与试剂 PCR仪(Bio-Rad,美国)、DNA电泳仪(Bio-Rad,美国)、Qubit 4荧光计(ThermoFisher Scientific,美国)、凝胶成像仪(广州博鹭腾,中国)、全自动研磨仪(上海净信,中国)、自动化核酸提取仪(西安天隆,中国)、VITEK 2全自动微生物鉴定系统(BioMérieux,法国)、基因组核酸提取试剂盒(广州美基,中国)、Agencourt AMPure XP磁珠(Beckman Coulter,美国),其他试剂均为国产分析纯等级。
1.3方法
1.3.1病原微生物培养与核酸提取 在无菌环境分别接种20种标准菌株于相应培养平板上,在适宜温度下培养12~24 h。挑取单个菌落,接种于5 mL液体培养基,过夜培养。取1 mL菌液使用全自动研磨仪破碎细胞,用基因组核酸提取试剂盒及自动化核酸提取仪提取核酸,-20 ℃保存备用。
1.3.2引物设计 本研究采用生物信息学方法筛选基因序列的保守区段,每个病原微生物设计两对引物,共40对特异性引物,片段长度为90~110 bp,以便于衔接后续的第二代高通量测序。
1.3.3引物验证 敏感性验证:利用每对引物对目标菌株基因组模板进行单重PCR,将PCR产物经琼脂糖凝胶电泳检测,观察条带是否单一、明亮,并将扩增产物送至第三方检测机构进行DNA测序,验证引物敏感性。特异性验证:除去待验证目标菌株基因组,将其余19种目标菌株基因组和人基因组等量混合,使用待验证目标菌株的特异性引物进行扩增,以PCR产物有无非特异性条带验证引物特异性。根据模板混合物中包含的目标,选择两对相应引物在同样条件下进行PCR作为阳性对照,以证实PCR能否正常进行。PCR扩增:98 ℃预变性 3 min;98 ℃ 10 s,60 ℃ 15 s,72 ℃ 10 s,循环30次;最后72 ℃延伸10 min。反应结束后取5 μL PCR产物,进行电泳检测,通过紫外凝胶成像系统观察结果。
1.3.4AC-Seq法检测方程 (1)多重PCR体系建立。目标序列扩增:2×多重PCR Taq酶(预混和)20 μL,引物混合物(200 nmol/L)4 μL,DNA模板xμL(0.1≤x≤0.5),ddH2O(16-x)μL。PCR反应参数:95 ℃预变性 3 min;95 ℃ 20 s,60 ℃ 4 min,循环19 次;最后72 ℃延伸4 min;10 ℃结束反应。PCR产物纯化:使用Agencourt AMPure XP磁珠,按说明书操作。(2)测序文库制备。引入测序接头:2×多重PCR Taq酶(预混和)15 μL,接头F (10 μmol/L) 1 μL,接头R (10 μmol/L) 1 μL,DNA模板为上轮PCR纯化产物,ddH2O 13 μL。PCR反应参数:95 ℃预变性 3 min;95 ℃ 15 s,58 ℃ 15 s,72 ℃ 1 min,循环8次;最后72 ℃延伸10 min;10 ℃结束反应。后重复PCR产物纯化步骤。(3)文库质检及测序:PCR产物文库质检及测序由海普洛斯(深圳)生物科技有限公司完成。
1.3.5灵敏度检测 以金黄色葡萄球菌ATCC 43300、肺炎链球菌ATCC 49619、鲍曼不动杆菌ATCC 19606、大肠杆菌ATCC 35150、白色念珠菌ATCC 10231为目标,通过PCR方法从目标菌基因组中获取目标扩增片段。然后将扩增片段依次梯度稀释为106、105、104、103、102、101、100copy/μL,取5 μL模板进行多重PCR扩增,测序文库制备流程见1.3.4)。每个梯度设置重复试验3组。空白对照组模板均为无菌水。
1.3.6临床标本鉴定 将临床标本分成3份,一份均匀涂布在血平板上,培养12~24 h后挑取主要单菌落进行纯培养。病原微生物鉴定采用VITEK 2全自动微生物鉴定系统。一份用于AC-Seq法检测,流程见1.3.4。另一份保存至-80 ℃冰箱备用。对两种检测方法结果不一致的标本进行单重PCR验证,选择的通用引物序列如下:细菌27F 5′-AGAGTTTGATCCTGGCTCAG-3′,1492R 5′-GGTTACCTTGTTAC GACTT-3′;真菌ITS1 5′-TCCGTAGGTGAACCT GCGG-3′,ITS4 5′-TCCTCCGCTTATTGATA TGC-3′。
1.4统计学处理 数据处理软件采用Microsoft Excel 2016。统计分析软件使采用SPSS22.0。计数资料以例数或百分比表示。AC-Seq和临床培养两种检测方法的结果比较采用配对χ2检验,以P<0.05为差异有统计学意义。
2 结 果
2.1引物敏感性和特异性验证 对引物进行扩增敏感性评估,2%琼脂糖凝胶电泳分析显示40对引物的扩增产物均获得明亮、单一、清晰的DNA条带,大小约为150 bp(含约50 bp测序接头序列)。将扩增后的PCR产物采用Sanger测序,BLAST比对确定为相应菌株的核酸片段,表明40对引物均具有良好的扩增敏感性,见图1。对引物进行扩增特异性评估,2%琼脂糖凝胶电泳分析显示40对引物均未获得非特异性扩增产物,且阳性对照条带明亮清晰,表明40对引物均具有良好的扩增特异性,见图2。
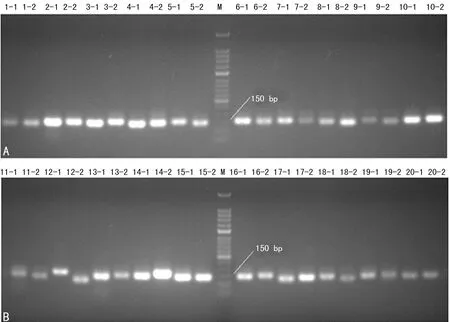
注:M表示50 bp DNA Ladder;图A中,1-1、1-2为肺炎克雷伯菌;2-1、2-2为化脓性链球菌;3-1、3-2为鲍曼不动杆菌;4-1、4-2为阴沟肠杆菌;5-1、5-2为铜绿假单胞菌;6-1、6-2为卡他莫拉菌;7-1、7-2为金黄色葡萄球菌;8-1、8-2为黏质沙雷菌;9-1、9-2为流感嗜血杆菌;10-1、10-2为洋葱伯克霍尔德菌;图B中,11-1、11-2为嗜麦芽窄食单胞菌;12-1、12-2为产气肠杆菌;13-1、13-2为大肠埃希菌;14-1、14-2为奇异变形杆菌;15-1、15-2为肺炎链球菌;16-1、16-2为嗜肺军团菌;17-1、17-2为屎肠球菌;18-1、18-2为粪肠球菌;19-1、19-2为白色念珠菌;20-1、20-2为近平滑假丝酵母菌。
2.2灵敏度检测 灵敏度检测结果显示,模板分别为5×106、5×105、5×104、5×103、5×102、5×101、5×100、0拷贝,对应检出的平均reads依次为501 536.00、399 369.67、301 303.33、102 015.67、8 009.67、527.67、1.00、0.00。因此在40 μL扩增体系中,本研究所建立方法的最低检出模板量为5×100拷贝。见图3。
2.3AC-Seq法检测临床标本结果 AC-Seq法给出了34例临床标本的病原微生物分布及丰度。阳性标本以深色方框表示,颜色越深表示标本中该病原微生物核酸的检出reads越多。白色表示未检出。见图4。
2.4临床标本两种方法检测结果比较 34例临床标本中AC-Seq共检出13种病原微生物,其中检出嗜麦芽窄食单胞菌16例,占比47.06%;鲍曼不动杆菌11例,占比32.35%;黏质沙雷菌8例,占比23.53%;铜绿假单胞菌6例,占比17.65%;屎肠球菌5例,占比14.71%;大肠埃希菌2例,占比5.88%;肺炎克雷伯菌、阴沟肠杆菌、流感嗜血杆菌、洋葱伯克霍尔德菌、肺炎链球菌、产气肠杆菌、粪肠球菌各1例,均占比2.94%。
培养、鉴定方法共检出8种病原微生物,其中检出鲍曼不动杆菌7例,占比20.59%;嗜麦芽窄食单胞菌、铜绿假单胞菌各2例,均占比5.88%;肺炎克雷伯菌、阴沟肠杆菌、流感嗜血杆菌、产气肠杆菌、白色念珠菌各1例,均占比2.94%。
AC-Seq法检出阳性共计27例,总阳性率为79.41%;培养法检出阳性共计11例,总阳性率为32.35%。两种方法检测结果一致的标本共17例,总一致率为50%。经配对χ2检验得出,二者间差异有统计学意义(χ2=0.195,P<0.05)。不一致结果经第三方测序验证后,统计得出AC-Seq检测方法的灵敏度为100.00%,特异度为29.17%。
在检测时间方面,传统培养法需将样品在平板培养24~48 h后,挑取主要单菌落进行纯培养再通过VITEK 2全自动微生物鉴定系统确定病原微生物种类。AC-Seq法可在6 h完成32例标本从核酸提取到多重PCR产物文库构建过程,18 h内完成测序及结果分析,周转时间不超过24 h。

注:M表示50 bp DNA Ladder;泳道0-1、0-2为阳性对照;1-1、1-2为肺炎克雷伯菌;2-1、2-2为化脓性链球菌;3-1、3-2为鲍曼不动杆菌;4-1、4-2为阴沟肠杆菌;5-1、5-2为铜绿假单胞菌;6-1、6-2为卡他莫拉菌;7-1、7-2为金黄色葡萄球菌;8-1、8-2为黏质沙雷菌;9-1、9-2为流感嗜血杆菌;10-1、10-2为洋葱伯克霍尔德菌;11-1、11-2为嗜麦芽窄食单胞菌;12-1、12-2为产气肠杆菌;13-1、13-2为大肠埃希菌;14-1、14-2为奇异变形杆菌;15-1、15-2为肺炎链球菌;16-1、16-2为嗜肺军团菌;17-1、17-2为屎肠球菌;18-1、18-2为粪肠球菌;19-1、19-2为白色念珠菌;20-1、20-2为近平滑假丝酵母菌。
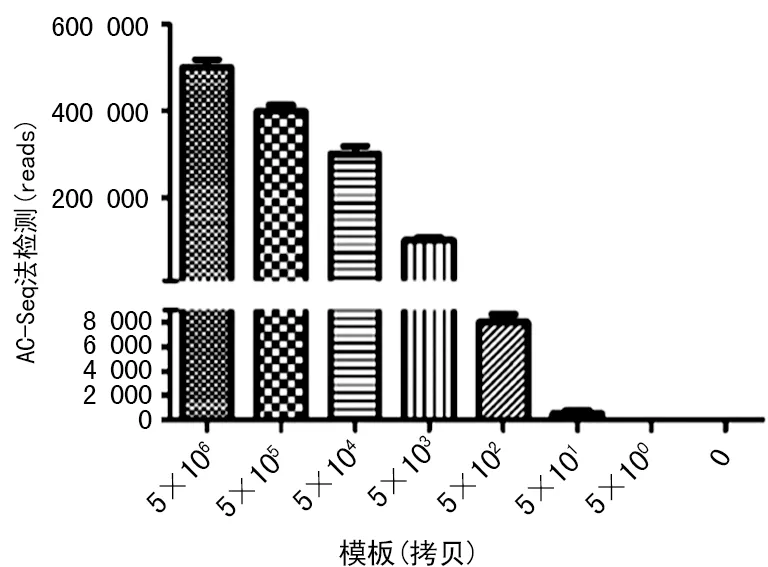
图3 灵敏度检测结果

图4 AC-Seq法检测临床标本病原微生物丰度热图
3 讨 论
目前下呼吸道感染病原微生物的筛查主要依靠分离培养法及自动化的微生物鉴定系统,虽然具有操作简便,成本低等优点,但也存在通量低、耗时长、分离过程易受主观因素影响等缺点[14-15]。其他检测方法,如基于免疫荧光法的呼吸道九联检试剂盒,具有检测速度快、灵敏度高等优点,但主要针对的是呼吸道病毒、肺炎支原体和肺炎衣原体等病原体的检测[16];环介导等温扩增是一种基于有链置换活性的DNA聚合酶的等温快速核酸扩增方法[17]。应用于下呼吸道病原体检测,该方法有灵敏度高、特异性强、反应速度快,不需要特殊仪器等优点[18],但是该方法对引物的设计有特殊要求,因此在多重PCR及高通量检测方面存在一定的缺陷。微流控技术具有小型化、集成化与自动化的优势,且试剂用量少、能耗低,污染少,但该方法技术成本高,不利于临床大规模的病原微生物筛查及在中小医院推广[19]。
本研究通过自主设计特异性引物,联合多重PCR与第二代高通量测序技术实现了20种下呼吸道感染常见病原微生物的高通量、快速检测,可以至少覆盖90%的临床下呼吸道病原微生物,尤其适合多种病原微生物感染的检测。本研究一方面通过多重PCR技术有效扩增并富集病原微生物核酸,同时在纯化环节去除人基因组及其他大片段非目标核酸,弥补了宏基因组测序的缺陷[20];另一方面,通过第二代高通量测序技术可以直接、客观、高通量地鉴别感染病原微生物。
本研究建立的方法使用全自动研磨仪和全自动核酸提取仪直接从临床标本中提取核酸,在 1 h内可处理32个标本,提高了检测效率,整个检测过程耗时不超过24 h,可基本满足临床上大批量标本病原微生物初筛的需求。此外通过高通量测序还可以获得标本中不同病原微生物的相对丰度信息,为临床精准治疗提供参考。和培养鉴定法相比,检测阳性率差异显著及检测特异性低的原因可能是多重PCR联合第二代高通量测序技术大大提高了检测的灵敏度;对于一些不一致的阳性结果,可能需要与临床进一步讨论。在后续的研究中,会进行更大规模的测试,以进一步评估和优化本方法。此外,还会将本方法应用于耐药基因、毒力基因的检测,为临床精准用药提供更全面的参考。
4 结 论
本研究采用生物信息学方法设计了40对特异性引物,通过PCR验证了引物具有良好的敏感性和特异性。建立了可进行第二代高通量测序技术的多重PCR扩增体系。把建立的方法应用于临床肺泡灌洗液标本的检测并与临床培养结果进行对比。结果表明,与传统培养法相比,本方法具有高灵敏度、高通量、短周期等优势。成功建立了一种对20种下呼吸道感染常见病原微生物检测的快速、高通量方法。

