甘蓝型油菜响应低氮胁迫的表达谱分析
肖 燕 姚珺玥 刘 冬 宋海星 张振华
甘蓝型油菜响应低氮胁迫的表达谱分析
肖 燕**姚珺玥**刘 冬 宋海星 张振华*
湖南农业大学资源环境学院 / 南方粮油作物协同创新中心, 湖南长沙 410128
随着人们对作物产量的需求不断提高, 氮肥被过量施用, 而作物的氮素利用率(NUE)却在不断降低。本研究从低氮胁迫下油菜的生理变化入手, 结合高通量的数字基因表达谱测序技术, 分析了油菜在低氮0、3、72 h下的转录组差异响应, 鉴定了氮的吸收﹑转运﹑分配和转录因子等方面的差异表达基因。结果表明, 甘蓝型油菜在低氮处理后, 氮优先分配到地上部, 硝酸还原酶(NR)活性显著降低, 而谷氨酞胺合成酶(GS)活性升高, 油菜植株总氮浓度降低, NUE升高。基因基因本位论(GO)功能与京都基因和基因组百科全书(KEGG)代谢通路分析表明, 地上部差异表达基因主要是参与代谢过程、蛋白结合、离子结合、阴离子结合等, 根中差异表达基因主要是参与分子功能、初级代谢过程、离子结合、阴离子结合等。基因表达谱分析表明, 低氮胁迫72 h后, 根中家族基因表达大部分升高; 根中和的基因表达量降低;、和亚家族中的大部分基因的表达量降低;家族中的大部分基因表达上调; 在和亚家族中, 根中(BnaA06g04560D)、(BnaA06g04570D)、(BnaA02g11760D)、(BnaC02g16150D)的表达上调。同时, 地上部和根发生外显子跳跃(SE), 外显子选择性跳跃(MXE)类型的可变剪接积极加强对低氮的适应。总而言之, 在低氮处理下, 甘蓝型油菜可以通过调控家族基因提高NUE以及调控、家族和可变剪接积极适应低氮胁迫。
甘蓝型油菜; 低氮胁迫; NO3-; 可变剪接
氮(N)是植物生物量和作物产量必需的营养元素[1]。植物必须从土壤中获取丰富的氮资源, 以满足植物正常生理发育。在我国农业生产中, 氮素利用效率(NUE)比较低, 每年需要施用大量氮肥以维持作物生产力。而氮肥的过量施用造成一定的资源浪费, 并对环境构成污染, 因此在减少施氮量的同时提高NUE是可持续农业和生态系统良性发展的关键。
低氮胁迫下, 植物可以通过诱导根生长来增加对氮的吸收[2], 氮高效的水稻品种通过诱导NO3-转运子使地上部累积更多的氮, 来促进光合作用, 提高碳、氮同化效率[3]。与谷类作物不同, 甘蓝型油菜(L.)对最优种子产量具有相对较高的N营养需求[4-5], 合适的氮水平可以促进光合效率, 并增加干物质累积量[7], 而适当的低氮处理可以增加油菜的NUE[6]。事实上, 尽管油菜具有强大的NO3-吸收能力, 但油菜仍然是目前已知作物中NUE最低的[7]。
甘蓝型油菜(, AnAnCnCn, 约1345 Mb, 2= 4= 38)属于十字花科芸薹属油料作物[8], 全基因组包含约十万多个蛋白编码基因, 由白菜(, ArAr, 约485 Mb, 2= 2= 20)和甘蓝(, CoCo, 约630 Mb, 2= 2= 18)这2个二倍体基本种通过天然远缘杂交形成的异源四倍体作物[9]。本研究中采用的油菜品种为湘油15号, 是由湘油10号为父本, 湘油11号为母本杂交, 经多年选育的双低油菜, 是一种高NUE品种[10]。
近年来, 伴随测序技术的迅猛发展和测序成本的大幅下降, 高通量测序已经成为植物基因组和转录组(或数字基因表达谱)等众多组学研究的首选分析测试手段, 极大地推进了基因组学和分子生物学等多种学科领域的研究进展。已有学者对多份谷子核心种质在低氮处理下的表型进行了分析, 并选择耐低氮的品种进行了转录组测序分析, 进而筛选到可能参与低氮胁迫响应的基因并进行后续试验分析[11]。并且不少研究表明, 转录因子在植物生命活动中起着非常重要的作用, 转录因子可以通过调控相应基因的表达来适应非生物胁迫[12-14]。
虽然越来越多的研究从组学入手, 深入解析植物在低氮胁迫下的响应[15-16], 但是关于NUE高效油菜在低氮胁迫下的转录响应的研究比较少。本研究鉴定了在低氮处理下甘蓝型油菜产生的异常形态与生理生化紊乱。随后, 利用高通量的数字基因表达谱测序技术分析了油菜在低氮0、3、72 h下的转录组差异响应, 鉴定了氮的吸收﹑转运﹑分配和转录因子等方面的差异表达基因。该研究旨在提高对油菜响应低氮胁迫的分子机制认识, 为鉴定和培育适应低氮的NUE高效的作物种质资源提供部分理论基础。
1 材料与方法
1.1 试验材料
选用油菜品种湘油15号 (XY15), 由国家油料改良中心湖南分中心提供。
1.2 营养液培养试验
试验在本课题组油菜光照培养室中进行。温度设置为22℃, 光照周期为14 h光照/10 h黑暗, 光照强度为300~320 μmol m-2s-1, 湿度为60%~75%。选取大小一致、籽粒饱满的油菜种子, 使用1%的NaClO灭菌处理10 min; 将种子表面冲洗干净后, 4℃避光条件下用超纯水浸泡种子24 h。将浸泡过后的种子均匀播于纱布上用超纯水育苗。育苗1周后, 将长势一致的幼苗移栽至盛有10 L营养液的黑色塑料盆中。
营养液试验中的大量元素营养液采用Hoagland配方, 微量元素营养液采用Arnon配方[17]。每隔5 d更换1次营养液, 初始营养液为1/4浓度营养液, 随后为1/2浓度营养液, 最后为全量营养液。试验设置正常氮水平(NO3-9.0 mmol L-1)和低氮水平(NO3-0.3 mmol L-1) 2个氮水平。
1.3 取样及测定方法
采用Hoagland溶液进行水培油菜幼苗, 在整个试验过程中不断曝气, 每隔5 d更新一次[18]。以正常氮水平(NO3-9.0 mmol L-1)水培油菜品种湘油15号(XY15)的幼苗10 d, 然后转移到低氮水平(NO3-0.3 mmol L-1)。在0、3和72 h后分别对XY15幼苗的地上部和根进行取样。将新鲜根和叶研磨成细粉(约100 mg), 然后提取并用Ehlting等[19]描述的分光光度法测定NR活性。用Wang等[20]报道的方法测定GS活性。根据Patterson等[21]的研究, 在410 nm处用分光光度法测定了油菜根和叶中NO3-的浓度。用Wang等[22]报道的方法测定了油菜的总氮含量。根据Li等[23]的计算方法, NUE = 总生物量/总氮积累。
1.4 数字基因表达谱分析
采用高通量的数字基因表达谱测序进行全基因组的基因表达分析, 测序平台为Illumina HeSeq 4000平台上的高通量转录组测序(NooGeNe, 中国北京), 测序方式为单端测(single-end, SE), 单条reads读长为50 bp, 单个样品的测序reads数目为20 M条, 单个样品获得的数据量约为1.2 Gb。
由于原始测序数据可能包含低质量序列和接头序列等, 为保证信息分析结果的可靠性, 需要经过一系列数据处理来过滤这些包含杂质的raw reads, 具体步骤如下: (1)去除含adapter的reads; (2)去除含N (表示无法确定碱基信息)比例大于10%的reads; (3)去除低质量reads (质量值≤5的碱基数占整条read的50%以上)。过滤后的数据称为clean reads。
利用Euclidean距离算法计算各个样品基因的表达量距离, 利用离差平方和(Ward)算法计算样品间的距离, 根据距离大小建立聚类图。聚类图能直观的反映出样品之间的距离关系和差异关系, 聚类图的纵坐标表示聚类树中的高度, 高度接近的生物学样品容易聚在一起。在基因表达的聚类分析中, 本研究利用Cluster软件[24], 以欧氏距离为矩阵计算公式, 对样品表达基因和样品方案同时进行分层聚类分析, 聚类结果用JavaTreeview[25]显示。在基因表达的聚类图中, 每列代表一个生物学样本, 每行代表一个基因, 不同表达量用不同的颜色表示, 红色表示高表达, 蓝色表示低表达。基因的GO功能注释使用WEGO软件[26]进行分析, 从宏观上认识基因的功能分布; 本研究使用KEGG[27]公共数据库对基因Pathway富集分析。使用NOISeq方法鉴定不同时间低氮处理之间的差异表达基因[28], 其筛选标准为: |log2Fold-change|≥2且FDR < 0.05。
基因表达定量的结果以FPKM为单位, 具体计算公式如下: FPKM=106/(NL103)。设FPKM(A)为基因A的表达量, 则为比对到基因A的Fragments数,为比对到参考基因的总Fragments数,为基因A编码区的碱基数。FPKM法能够消除基因长度和测序量差异对基因表达的影响, 计算得到的基因表达量可直接进行不同样品间的基因差异表达分析。基因表达谱的热图谱由Multiexperiment Viewer (MEV, http://www.tm4.org/ mev.html)描绘[29]。
1.5 数据处理
采用SPSS 19.0统计分析软件进行方差分析和相关分析, 不同处理间采用最小显著差数法(LSD法)进行差异显著性检验(< 0.05), 采用Microsoft Excel进行数据整理和作图。
2 结果与分析
2.1 甘蓝型油菜响应低氮胁迫的生理差异
当NO3-供应不足时, 植物对有限的氮生长条件产生一系列适应性反应[30]。以油菜为试材, 生长10 d后, 在低氮(0.30 mmol L-1NO3-)条件下进行水培试验, 测定了油菜对氮限制的生理反应。详细研究了植物对短期(3 h)和长期(72 h)低氮(0.30 mmol L-1)胁迫的反应。由图1-A可知, 在低氮处理下, 氮资源优先分配到地上部, 以满足光合作用的需要。氮代谢相关酶分析结果表明, NR活性在根和地上部中均显著降低(图1-E), 而GS活性在低氮处理下升高(图1-F)。低氮处理3 h生物量较低氮0 h无显著差异, 低氮处理72 h后, 其生物量较低氮0 h和3 h显著增加(图1-D), 在低NO3-条件下处理3 h后, 随着氮素限制时间的延长, 油菜植株总氮浓度显著降低(图1-B), 而氮素耗竭则提高了油菜植株的NUE (图1-C)。与充足的NO3-供应相比, 低氮诱导了油菜植株显著的生理变化。
2.2 甘蓝型油菜全基因组数字基因表达谱
为了鉴定甘蓝型油菜在全基因组水平上对低氮胁迫的分子响应, 我们利用Illumina HeSeq 4000 (NooGeNe, 中国北京)进行高通量的数字基因表达谱测序。其中转录组原始序列测序数据已经上传至NCBI (https://www.ncbi.nlm.nih.gov/)的SRA (https:// www.ncbi.nlm.nih.gov/sra/?term=)中, 其中BioProject ID为PRJNA340053。在去除接头序列和低质量的reads后, 每个测序样品获得4.2×107至5.7×107条reads, 碱基质量值Q20均在96%以上, Q30均在91%以上, GC含量也均在45%以上, 错误率均在0.02%以下(表1)。
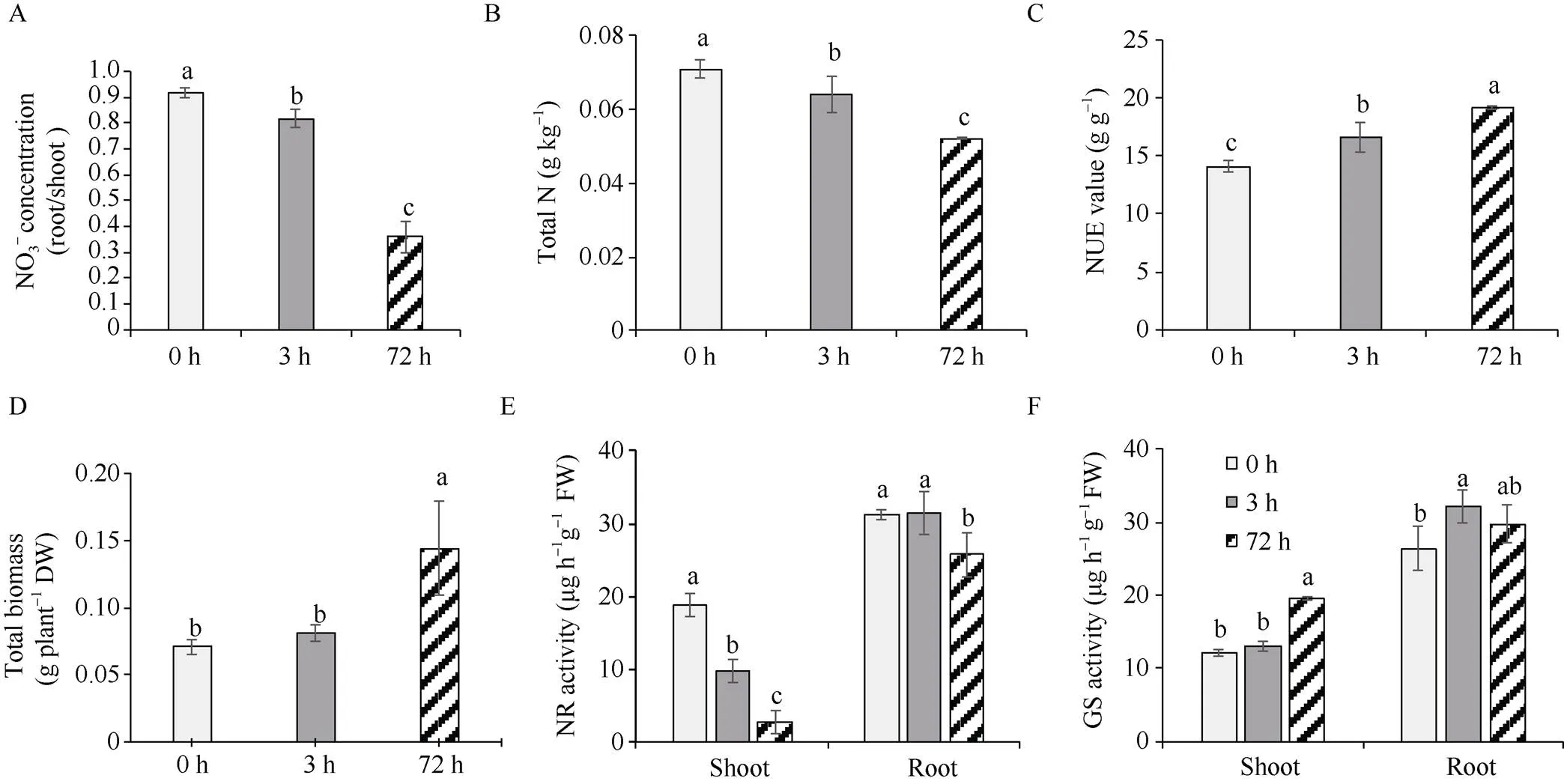
图1 油菜对氮(N)胁迫的生理响应
A: 根部NO3-浓度与地上部NO3-浓度的比值; B: 植株总N浓度; C: 氮利用效率(NUE), NUE=总干重/总N含量; D: 总生物量(单株植物干重 DW); E: 地上部和根部硝酸还原酶(NR)活性; F: 地上部和根部谷氨酰胺合成酶(GS)活性。数值表示平均值(= 5), 误差条表示标准误差(SE)值。
A: the ratio of root NO3-concentration to shoot NO3-concentration; B: the total N concentration; C: the nitrogen use efficiency (NUE) value, NUE = total dry weight / total N content; D: the total biomass (per plant dry weight DW); E: the nitrate reductase (NR) activity in shoots and roots; F: glutamine synthase (GS) activity in shoots and roots. Data present means (= 5), the error bars denote the standard error (SE) value.
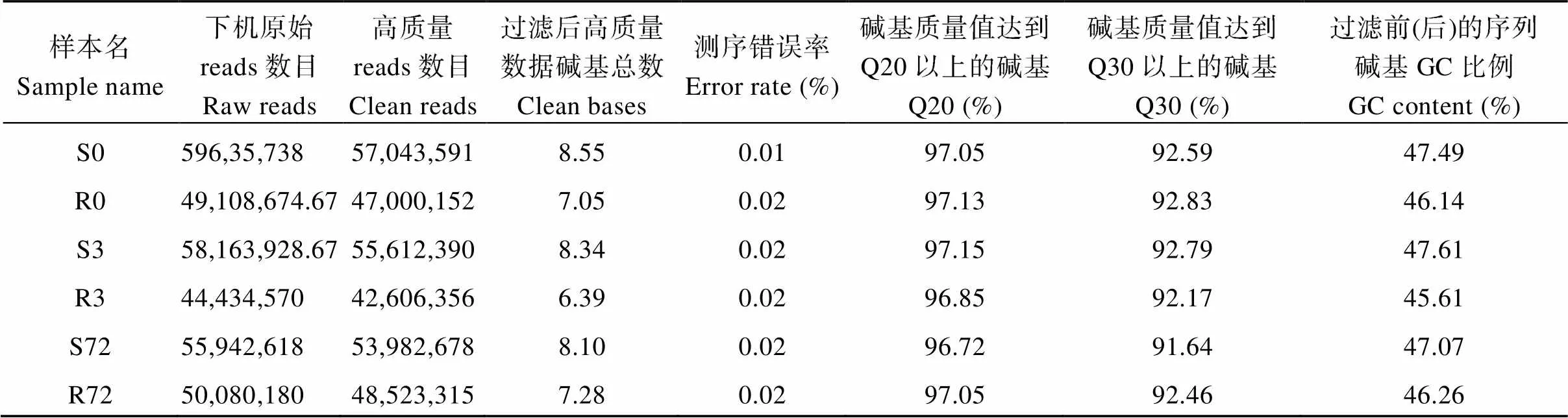
表1 数字基因表达谱测序数据概况
S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。
S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h under low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h under low nitrogen treatment, respectively.
为了评估低氮处理同一时间下3个生物学材料之间的相关性, 通过每2个生物学重复之间标准化的表达谱, 计算得出它们的Pearson相关性系数。结果表明, 在同一处理下的地上部或者根系中, 每2个生物学重复之间的相关性系数基本都在0.95以上(图2)。
基因表达差异的分析以低氮处理0 h作为对照, 低氮处理3 h和72 h后, 地上部分别检测到12,719个和10,066个差异表达基因; 根中分别检测到9204个和13,400个差异表达基因。低氮3 h和72 h下, 分别在地上部特异性地检测到5677个和3550个差异表达基因; 根中特异性地检测到3359个和5832个差异表达基因(图3)。
为了进一步鉴定差异表达基因的生物学功能, 本研究进行了基因GO功能与KEGG代谢通路分析。差异表达基因富集分析结果表明, 低氮处理72 h的条件下, 地上部差异表达基因主要是参与代谢过程、蛋白结合、离子结合、阴离子结合等(图4-A), 根中差异表达基因主要是参与分子功能、初级代谢过程、离子结合、阴离子结合等(图4-B)。KEGG代谢通路分析结果表明, 低氮处理72 h, 地上部的差异表达基因涉及最多的通路是代谢通路(图4-C), 该结果与差异表达基因的GO富集分析具有较高的一致性。根中的差异表达基因涉及最多的通路是与剪接体、核蛋白体以及碳代谢相关(图4-D)。
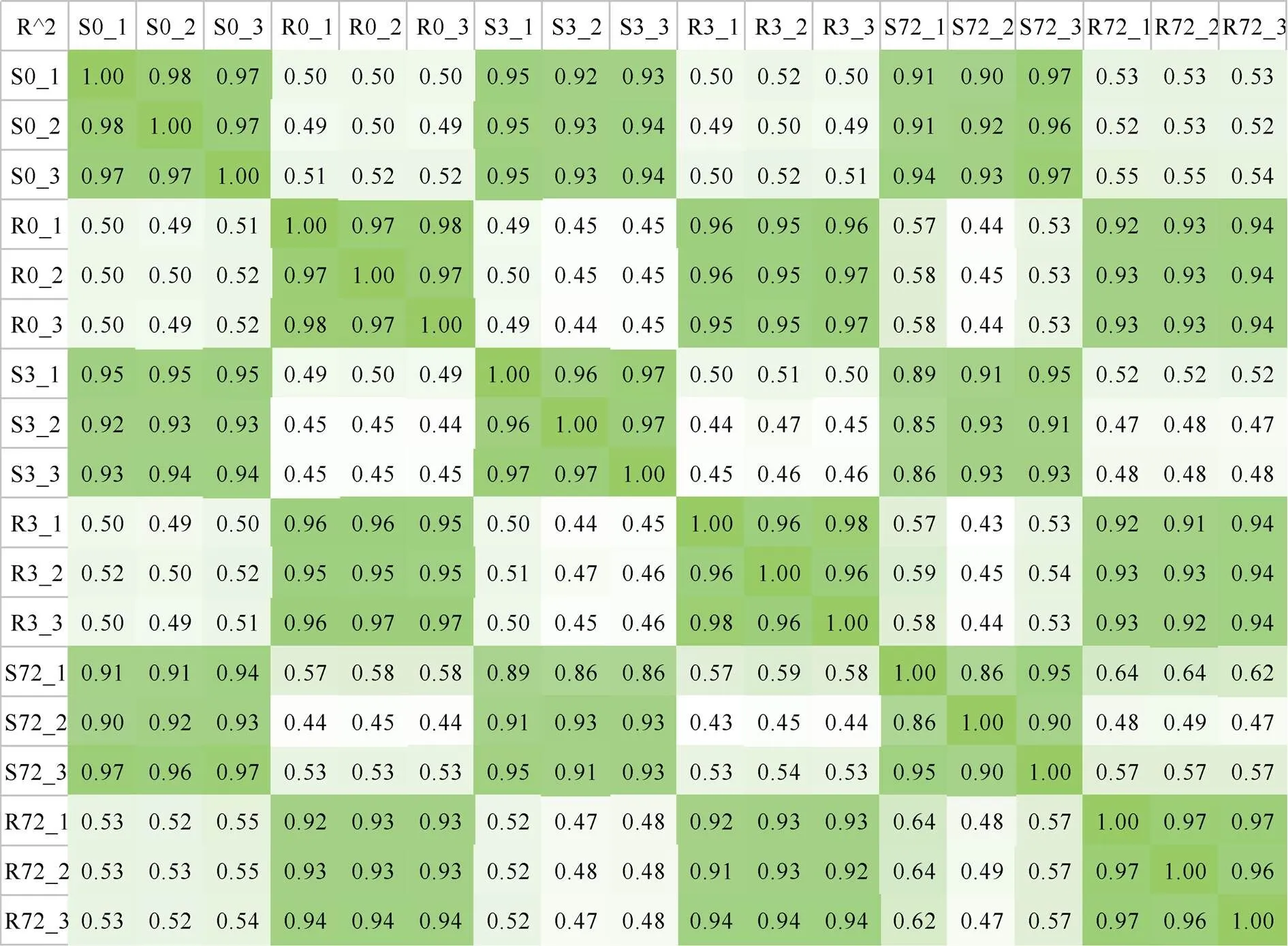
图2 油菜幼苗地上部和根系的Pearson相关性系数热图
S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。
S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h under low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h under low nitrogen treatment, respectively.
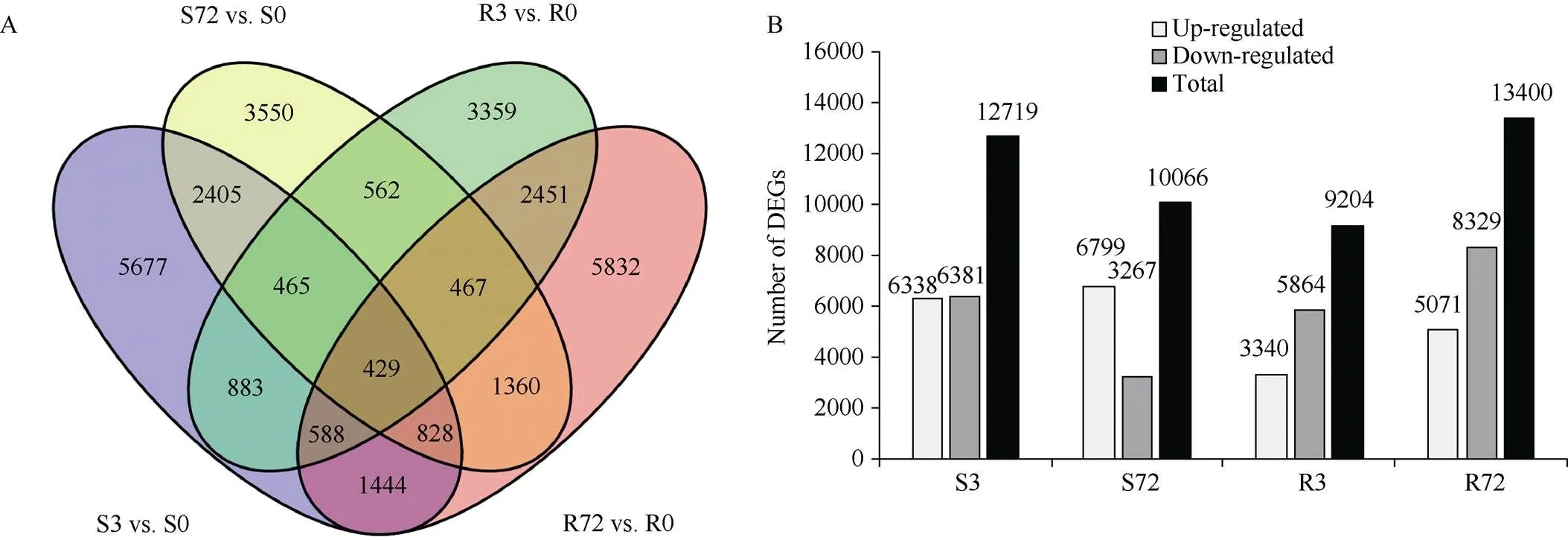
图3 低氮0, 3, 72 h下油菜地上部和根的差异表达基因
A: 差异表达基因的维恩图分析; B: 差异表达基因数目。S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。
A: the venn-graph analysis of the DEGs; B: the number of the DEGs. S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h under low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h under low nitrogen treatment, respectively.
2.3 油菜GLNs家族基因对低氮胁迫的表达反应
在拟南芥全基因组中, GLNs家族共存在6个成员(~,), 目前已证明其功能是调控谷氨酰胺合成酶[31]。本研究用拟南芥的GLNs基因序列在甘蓝型油菜Database (BRAD)数据库中进行BLASTn比对, 用以检索油菜基因组中的家族基因。在油菜全基因组范围内, 共同源比对到20个家族基因, 包括2个﹑2个﹑6个﹑4个﹑2个﹑4个。
本研究共检测到19个家族基因响应低氮处理。总体上,家族基因主要在根部和地上部均有表达(图5-A)。低氮处理后, 在s亚家族中,(BnaA04g08160D)在根中表达量显著上升,(BnaC04g30250D)地上部和根中表达均有显著上升(图5-B);在根中的表达量在低氮72 h后有显著上升(图5-C); 在亚家族中,(BnaA03g 34780D)和(BnaC03g40250D)在根中的表达量低氮72 h后显著上升, 且地上部的(BnaC03g40250D)在低氮72 h后表达量也显著上升(图5-D);亚家族在受到低氮胁迫后, 部分基因的根中表达量上升(图5-E);(BnaC06g02010D)在地上部和根中的表达量受到低氮胁迫的显著诱导(图5-F);亚家族基因主要在地上部表达, 根中(BnaC04g29670D)在低氮胁迫后表达上调(图5-G)。家族基因在低氮处理后总体呈上升趋势, 与GS活性基本一致(图1-E)。
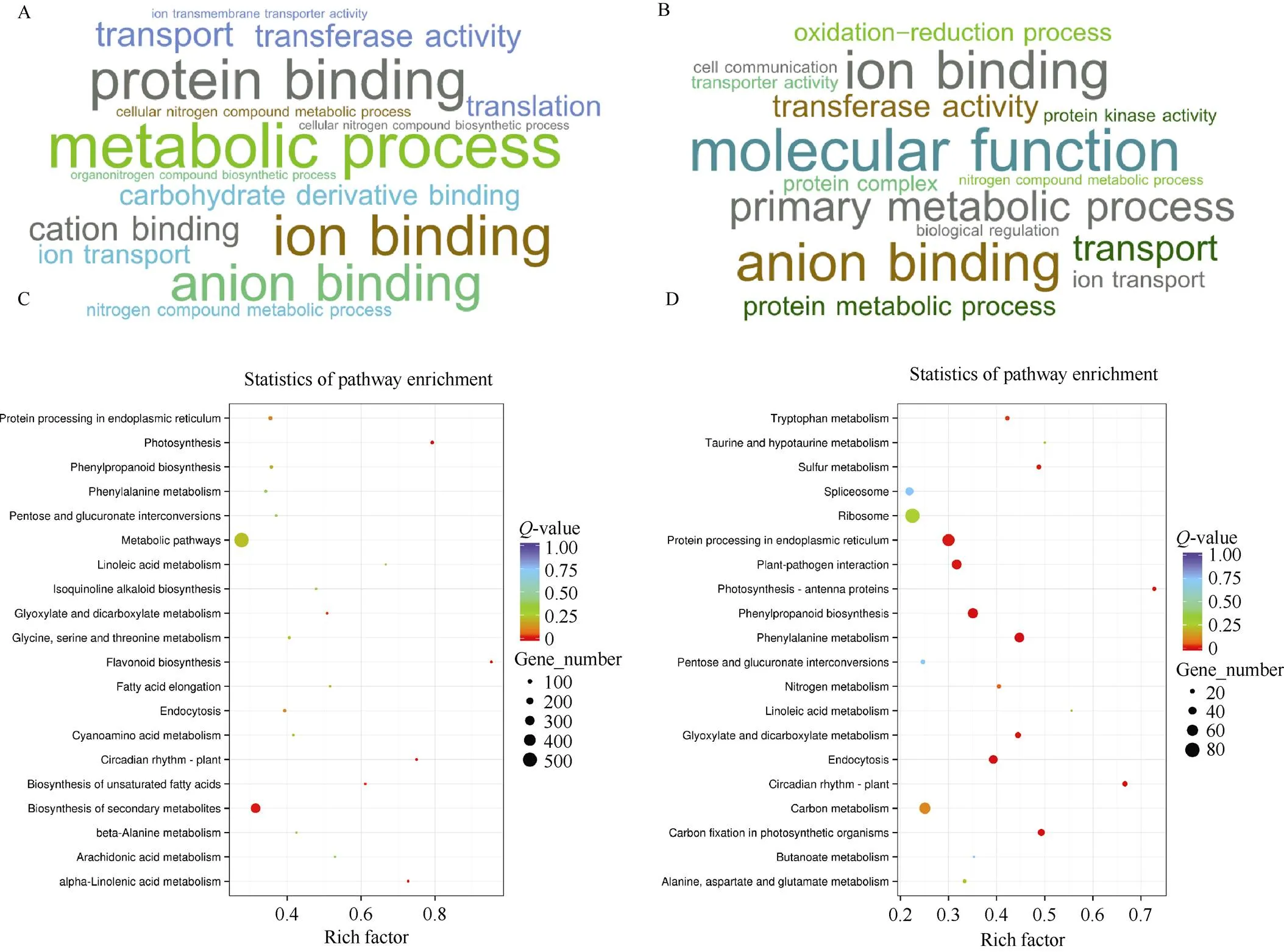
图4 油菜地上部和根系中差异表达基因的GO、KEGG富集分析
A: 在低氮72 h条件下, 地上部中的差异表达基因的GO富集分析; B: 在低氮72 h条件下, 根系中差异表达基因的GO富集分析; C: 在低氮72 h条件下, 地上部中的差异表达基因的KEGG富集分析; D: 在低氮72 h条件下, 根系中的差异表达基因的KEGG富集分析。
A: GO enrichment analysis of the differentially expressed genes in the shoots after low nitrogen treatment for 72 h; B: GO enrichment analysis of the differentially expressed genes in the roots after low nitrogen treatment for 72 h; C: KEGG enrichment analysis of the differentially expressed genes in the shoots after low nitrogen treatment for 72 h; D: KEGG enrichment analysis of the differentially expressed genes in the roots after low nitrogen treatment for 72 h.
2.4 油菜WRKYs家族基因对低氮胁迫的表达反应
WRKY转录因子在植物的生物和非生物胁迫、生长发育以及代谢的响应过程中发挥重要作用[32]。甘蓝型油菜全基因组共有287个WRKYs家族成员[33]。
家族基因主要在根部表达(图6-A), 在的差异表达基因中, 我们主要关注了(图6-B)和(图6-C), 发现其在受到低氮胁迫时, 根中表达量均显著降低。
2.5 油菜MYBs家族基因对低氮胁迫的表达反应
油菜的MYBs家族基因主要在根部表达(图7-A)。在的差异表达基因主要在根中响应缺氮处理。在亚家族受到低氮胁迫时, 根中表达量显著降低(图7-B); 在亚家族受到低氮胁迫时, 根中表达量基本是降低趋势, 部分在低氮72 h时与0 h相比达到显著差异(图7-C)。在亚家族中,(BnaA06g 12700D)在根中的表达量显著受低氮胁迫的诱导, 其他基因表达量在低氮72 h后显著降低(图7-D), 说明(BnaA06g12700D)可能与其他家族基因功能相反。
2.6 油菜NIAs家族基因对低氮胁迫的表达反应
NIA转录因子在调控硝酸还原酶的过程具有重要作用[34]。甘蓝型油菜的NIAs在叶片的根部均有表达(图8-A)。在亚家族中,(BnaA02g18610D)和(BnaA07g33680D)在根中的表达量受低氮72 h显著诱导,(BnaA07g33690D)在根中和叶片中的表达量均是在低氮3 h下降, 在低氮72 h上升,(BnaC06g38300D)在根中的表达量在低氮3 h后下降(图8-B)。在亚家族中,(BnaA07g20840D)在地上部和根中的表达量在低氮处理后均受到显著诱导,(BnaC06g 20690D)在根中的表达量在低氮3 h后下降, 低氮72 h上升(图8-C)。

图5 BnaGLNs家族基因的数字基因表达谱热图
A:s家族基因的总体表达谱; B:亚家族基因表达谱; C:亚家族基因表达谱; D:亚家族基因表达谱; E:亚家族基因表达谱; F:亚家族基因表达谱; G:亚家族基因表达谱。S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。差异表达基因(S3 vs. S0, S72 vs. S0; R3 vs. R0, R72 vs. R0)用星号标注。
A: the total DGE profiling offamily genes; B: the DGE profiling ofsubfamily genes; C: the DGE profiling ofsubfamily genes; D: the DGE profiling ofsubfamily genes; E: the DGE profiling ofsubfamily genes; F: the DGE profiling ofsubfamily genes; G: the DGE profiling ofsubfamily genes. S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h of low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h of low nitrogen treatment, respectively. The DEGs (S3 vs. S0, S72 vs. S0, R3 vs. R0, and R72 vs. R0) are labelled by asterisks.
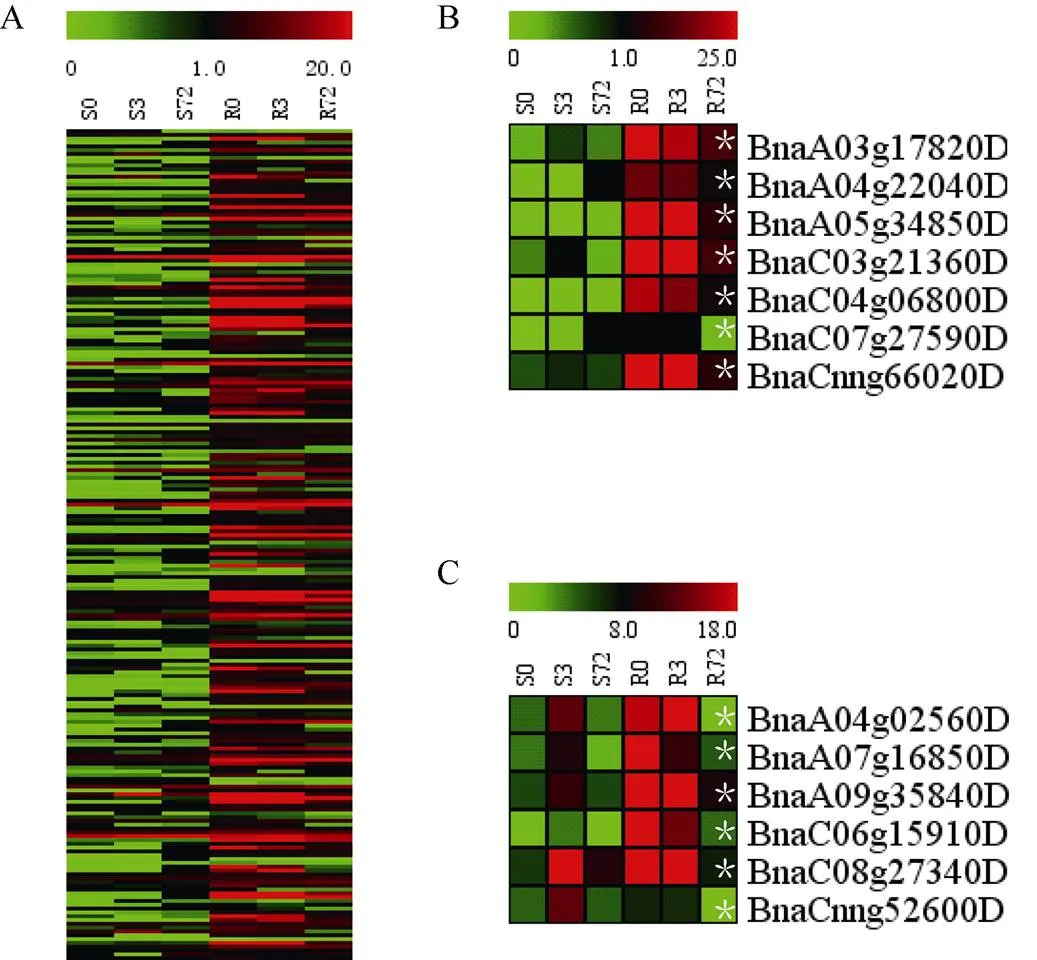
图6 WRKYs家族基因的数字基因表达谱热图
A: 油菜家族基因的总体表达谱; B:亚家族基因表达谱; C:亚家族基因表达谱。S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。差异表达基因(S3 vs. S0, S72 vs. S0; R3 vs. R0, R72 vs. R0)用星号标注。
A: the total DGE profiling offamily genes; B the digital gene expression profiling ofsubfamily genes; C the digital gene expression profiling ofsubfamily genes. S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h of low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h of low nitrogen treatment, respectively. The DEGs (S3 vs. S0, S72 vs. S0; R3 vs. R0, and R72 vs. R0) are labelled by asterisks.
2.7 油菜NRTs家族基因对低氮胁迫的表达反应
NRT (nitrate transporter)家族成员在植物体内硝酸盐的吸收和运输中发挥多功能作用[35-36], 对植物的生长发育具有重要意义。其中本文关注了和的差异表达基因, 发现其主要在根中响应缺氮处理(图9-A)。在亚家族中,(BnaA06g04560D)和(BnaA06g04570D)在根中的表达量在低氮72 h显著上调(图9-B)。在亚家族中,(BnaA02g11760D)和(BnaC02g16150D)受到低氮胁迫3 h和72 h后的表达量均显著上升(图9-C), 这可能与低氮处理后硝酸盐向地上部转运有关。
2.8 甘蓝型油菜可变剪接鉴定及分析
可变剪接是调节基因表达和产生蛋白质组多样性的重要机制, 是导致真核生物基因和蛋白质数量较大差异的重要原因, 是真核生物中必不可少的过程。可变剪接可以通过单个前mRNA不同的剪接方式来增加基因表达多样性和蛋白质组多样性[37]。
在低氮处理3 h和72 h后, 地上部和根中均有可变剪接发生, 具体为外显子跳跃(SE) 3个, 外显子选择性跳跃(MXE) 2个, 其他类型无可变剪接发生(图10)。因此低氮处理后, 一些基因可以翻译出不同的蛋白质, 进而增加其低氮胁迫下系统的复杂性和适应性。
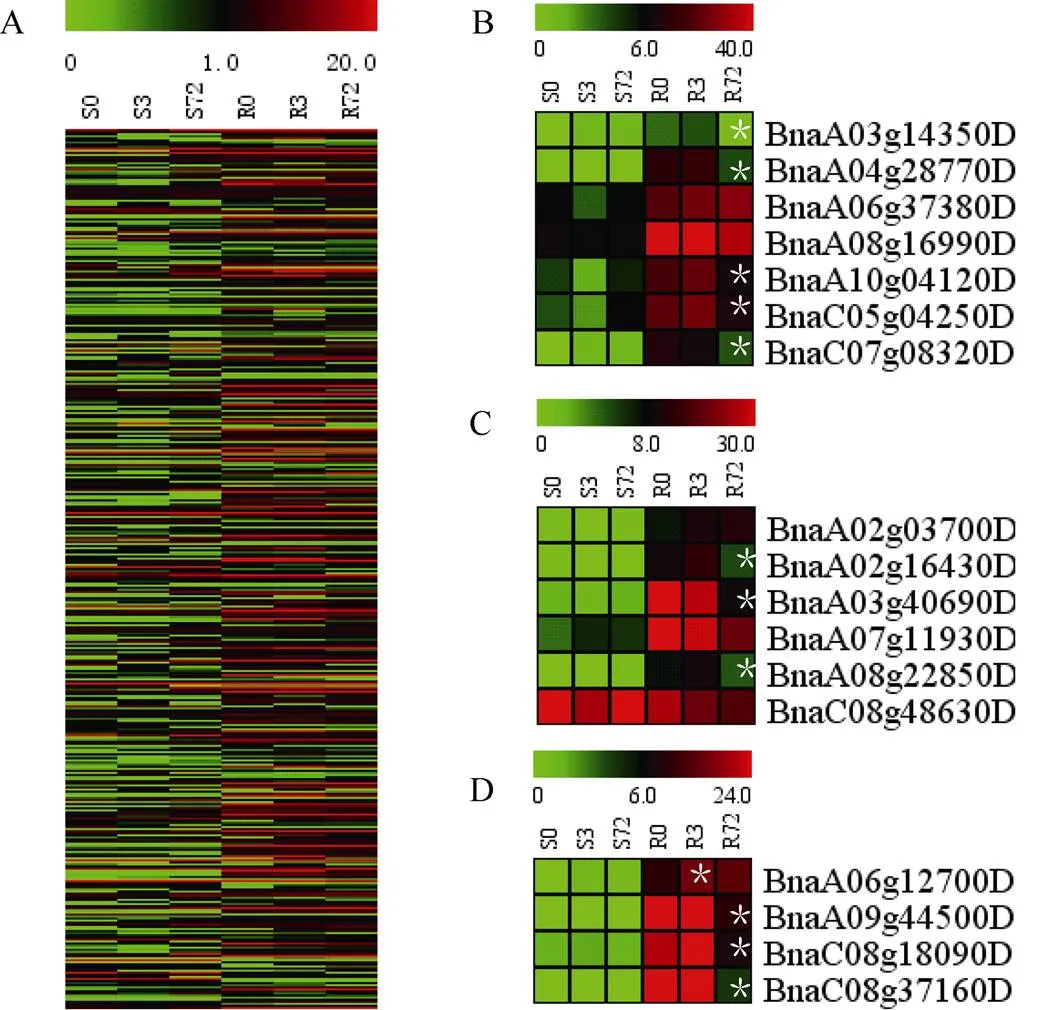
图7 MYBs家族基因的数字基因表达谱热图
A: 油菜家族基因的总体表达谱; B:亚家族基因表达谱; C:亚家族基因表达谱; D:亚家族基因表达谱。S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。差异表达基因(S3 vs. S0, S72 vs. S0; R3 vs. R0, R72 vs. R0)用星号标注。
A: the total DGE profiling offamily genes; B: the DGE profiling of; C: the DGE profiling of; D: the DGE profiling ofsubfamily genes. S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h of low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h of low nitrogen treatment, respectively. The DEGs (S3 vs. S0, S72 vs. S0; R3 vs. R0, and R72 vs. R0) are labelled by asterisks.
3 讨论
与谷物不同, 甘蓝型油菜生长发育过程中对氮素营养需求相对较高[4], 尽管它对氮限制条件极其敏感。国内外经常施用过量的氮肥以提高作物产量[5]。但由于50%~70%的施用氮不能被作物吸收, 过量施用氮肥必然会增加作物生产成本, 并导致环境污染[38], 因此如何在低氮情况下提高油菜NUE仍是当前农业生产的重要内容。
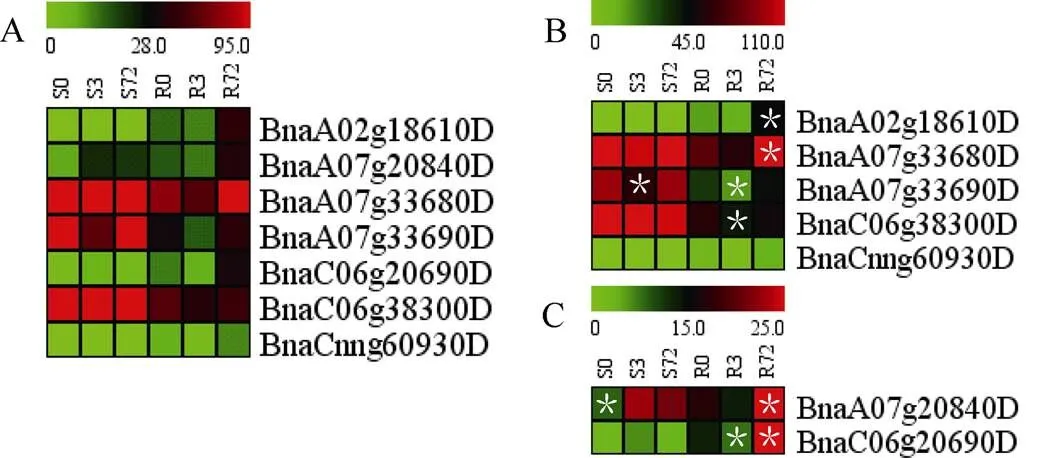
图8 NIAs家族基因的数字基因表达谱热图
A: 油菜家族基因的总体表达谱; B:亚家族基因表达谱; C:亚家族基因表达谱。S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。差异表达基因(S3 vs. S0, S72 vs. S0; R3 vs. R0, R72 vs. R0)用星号标注。
A: the total DGE profiling offamily genes; B: the DGE profiling of; C: the DGE profiling ofsubfamily genes. S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h of low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h of low nitrogen treatment, respectively. The DEGs (S3 vs. S0, S72 vs. S0; R3 vs. R0, and R72 vs. R0) are labelled by asterisks.
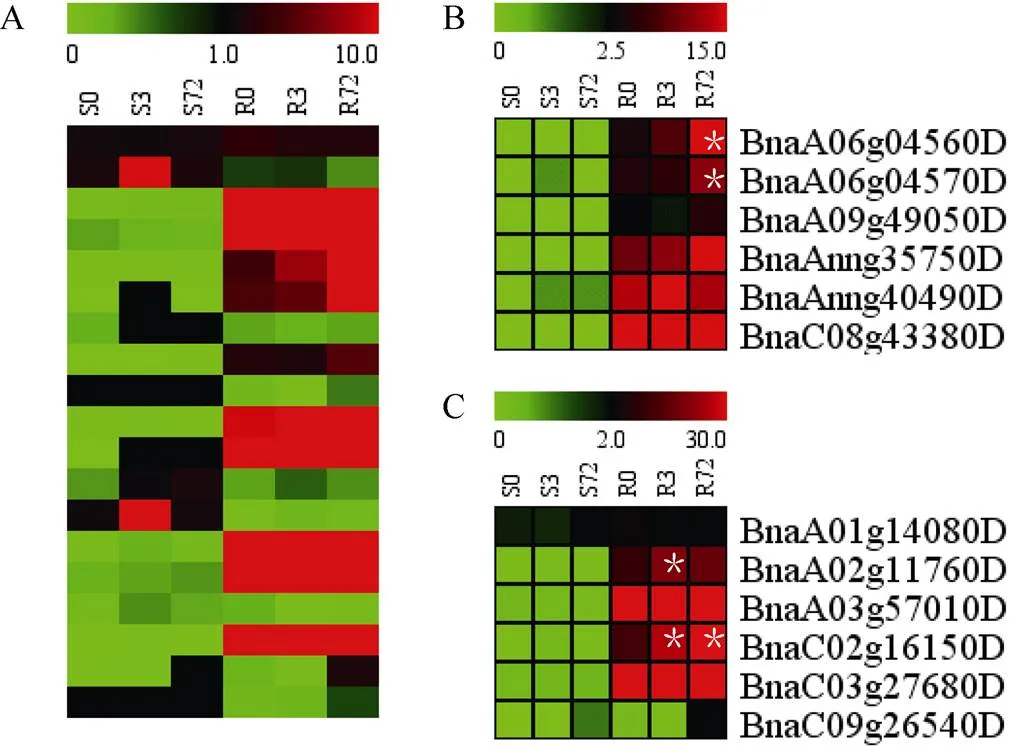
图9 NRTs 家族基因的数字基因表达谱热图
A: 油菜和家族基因的总体表达谱; B:亚家族基因; C:亚家族基因在油菜幼苗地上部和根系中的表达谱。S0和R0分别表示未做低氮处理的地上部和根; S3和R3分别表示低氮处理3 h后的地上部和根; S72和R72分别表示低氮处理72 h后的地上部和根。差异表达基因(S3 vs. S0, S72 vs. S0; R3 vs. R0, R72 vs. R0)用星号标注。
A: the total DGE profiling ofandfamily genes; B: the DGE profiling of; C: the DGE profiling ofsubfamily genes. S0 and R0 represent the shoots and roots without low nitrogen treatment, respectively; S3 and R3 indicate the shoots and roots after 3 h of low nitrogen treatment, respectively; S72 and R72 denote the shoots and roots after 72 h of low nitrogen treatment, respectively. The DEGs (S3 vs. S0, S72 vs. S0; R3 vs. R0, and R72 vs. R0) are labelled by asterisks.
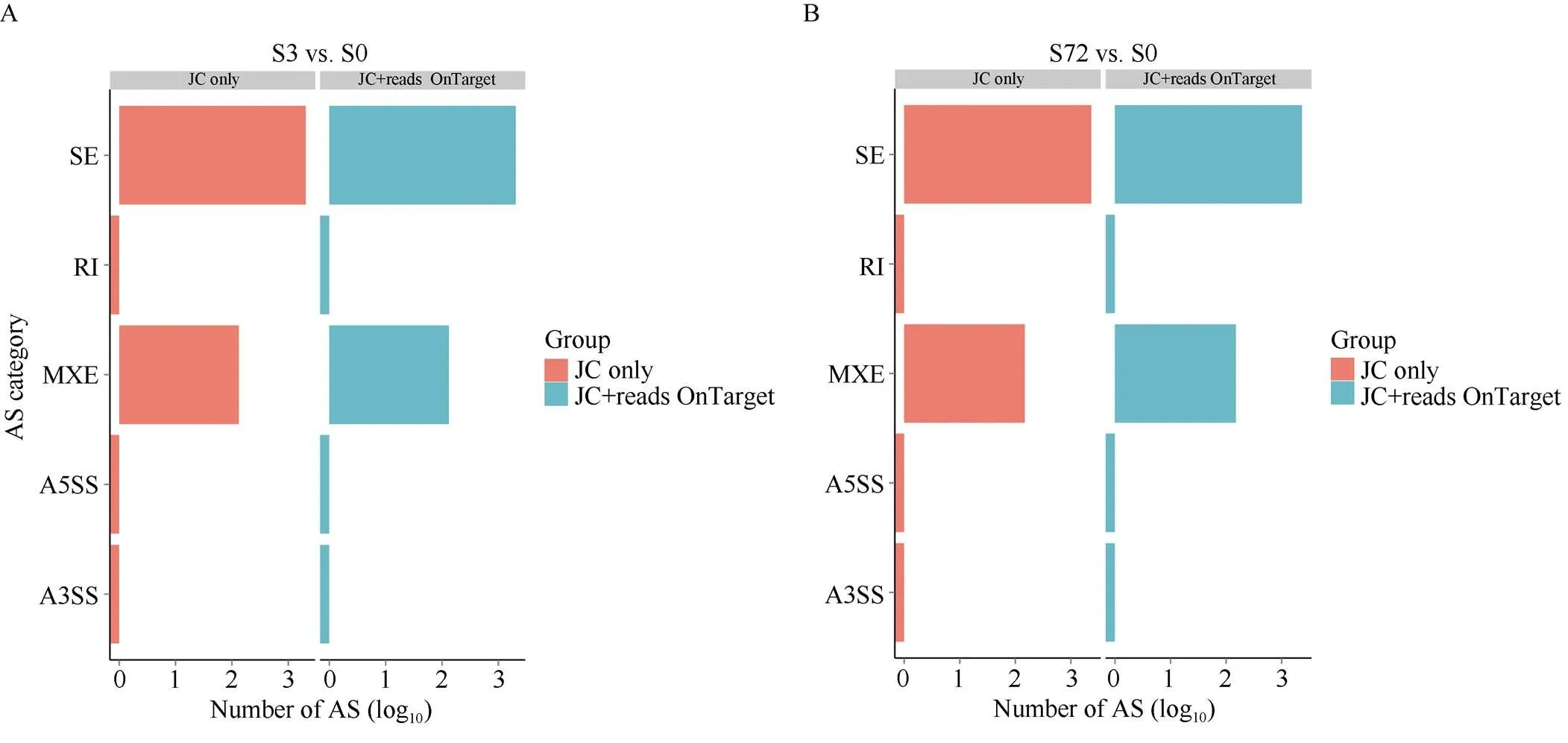
(图10)
A: 在低氮3 h后, 地上部的可变剪接类型及数量; B: 在低氮72 h后, 地上部的可变剪接类型及数量; C: 在低氮3 h后, 根的可变剪接类型及数量; D: 低氮72 h后, 根的可变剪接类型及数量。JC only表示只使用Junction Counts进行AS事件检测, JC + reads OnTarget表示同时使用Junction Counts 和 reads on target进行AS事件检测。
A: the type and number of alternative splicing in shoots after 3 h under low nitrogen treatment; B: the type and number of alternative splicing in shoots after 72 h under low nitrogen treatment; C: the type and number of alternative splicing in roots after 3 h under low nitrogen treatment; D: the type and number of alternative splicing in roots after 72 h under low nitrogen treatment. JC only represent that only Junction Counts are used for AS event detection, and JC + reads OnTarget represent that both Junction Counts and reads on target are used for AS event detection.
油菜适应氮胁迫的生理生化变化包括生长和光合作用的降低、氮素从根向地上部的转移增加(图1-A)和氮同化相关酶活性的改变(图1-D, E)。而油菜在低氮胁迫下的转录水平上, 代谢过程、蛋白结合、离子结合、阴离子结合、分子功能等相关基因均发生差异表达(图4), 植株可能在面临低氮胁迫时, 通过改变相关基因表达, 影响地上地下部的代谢过程, 从而更好地适应低氮环境。此外, 本研究发现, 在氮胁迫下, 油菜的NUE显著升高(图1-C), 这表明提高作物品种对有限氮的适应性可能是提高农业生产中NUE的有效策略。
与生理数据一致, 高通量转录组学也揭示, 氮胁迫显著改变参与硝酸还原酶和谷氨酰胺合成酶相关的基因的表达(图5和图8)。目前已证明NIA家族的功能是调控硝酸还原酶[34], GLN家族的功能是调控谷氨酰胺合成酶[31]。NRT2.1是高亲和的硝酸盐转运蛋白[39], 当植株处在低氮条件下时, NRT2.1活性增强(图9-B), 以适应低浓度硝酸盐条件下对氮素的吸收, 随着植株体内硝酸盐含量下降, 硝酸还原酶活性降低, NIA的基因表达量部分升高, 部分降低(图8), 可能是由于短时间内的低氮使硝酸还原酶活性降低, 但是由于氮的同化加强, 需要更多NH4+, 反向调节了NIA的表达。为满足植物生长需要, 植株通过调节GLNs的表达, 调节谷氨酰胺合成酶的活性, 使谷氨酰胺合成酶活性增强, 加强氮的同化。
前人研究发现,是番茄灰霉病水杨酸(SA)途径的负调节因子, 此外, 还参与氧化还原稳态、水杨酸(SA)信号、乙烯(ET)-茉莉酸(JA)介导的交叉互作[40]。0在决定SA依赖和JA依赖防御途径之间的平衡中起着关键作用[41]。前人的研究暗示,和可能在非生物胁迫下存在协同作用, 本研究利用高通量的数字基因表达谱测序, 发现油菜中和在低氮72 h下的表达量均显著下降, 基于这个结果, 我们猜测和在氮素利用过程中的调控机制发挥一定作用, 但是其调控机制有待进一步探究。
MYB转录因子可以调节次生代谢产物的生物合成[42]。拟南芥MYB转录因子属于R2R3-MYB家族, 与非生物胁迫反应和发育有关[42-43]。在拟南芥中,是抑制肉桂酸4-羟化酶(C4H)基因表达的转录抑制剂, 该基因编码羟基肉桂酸酯生物合成的关键酶来调控重要次生代谢物质芥子酸酯的生物合成[44]。在拟南芥中迅速能被茉莉酸甲酯(MeJA)快速诱导[45], 并通过直接控制激活SA介导的防御和抑制JA介导的防御来调节拮抗相互作用[46]。研究表明,/(HIGH INDOLIC GLUCOSINOLATE1)、/(HIGH INDO LICGLUCOSINOLATE2)和/(ALTERED TRYPTOPHAN REGULATION1)参与了吲哚硫代葡萄糖苷(IG)生物合成的调控[47-49]。这3个MYB转录因子在标准生长条件下是IG产生所必需的[50]。在本研究中,和在低氮处理72 h之后, 表达量均显著下调(图6-C和图7-C), 研究表明,可以直接调控[33],基因与其他植物基因在核苷酸和氨基酸水平上具有较高的同源性(80%以上), 同时在基因结构上与相似[51]。而与同样具有抗病性[52]。因此, 我们猜测在油菜中和在氮素利用过程中的调控机制, 可能与拟南芥的调控机制类似, 但是具体的调控机制有待进一步探究。
选择性剪接是导致转录组多样性增加的关键转录后事件[53], 不少研究表明可变剪接在植物对非生物胁迫响应起重要作用[54-55]。在低氮处理3 h和72 h后, 地上部和根中发生外显子跳跃(SE) 3个, 外显子选择性跳跃(MXE) 2个, 暗示在低氮条件下, 甘蓝型油菜可以通过可变剪接的发生来适应低氮胁迫。
4 结论
在低氮胁迫下, 甘蓝型油菜一方面通过调控将氮资源优先分配到地上部, 并通过调控NR和GS的活性, 来提高油菜的NUE; 另一方面可以通过对、相关转录因子及可变剪接的调控来积极适应低氮胁迫。
[1] Konishi M, Yanagisawa S. Emergence of a new step towards understanding the molecular mechanisms underlying nitrate- regulated gene expression., 2014, 65: 5589–5600.
[2] Hermans C, Hammond J P, White P J, Verbruggen N. How do plants respond to nutrient shortage by biomass allocation?, 2006, 11: 610–617.
[3] 冯洋, 陈海飞, 胡孝明, 周卫, 徐芳森, 蔡红梅. 我国南方主推水稻品种氮效率筛选及评价. 植物营养与肥料学报, 2014, 20: 1051–1062.Feng Y, Chen H F, Hu X M, Zhou W, Xu F S, Cai H M. Nitrogen efficiency screening of rice cultivars popularized in South China., 2014, 20: 1051–1062 (in Chinese with English abstract).
[4] Grant C A, Bailey L D. Fertility management in canola production., 1993, 73: 651–670.
[5] Rathke G W, Christen O, Diepenbrock W. Effects of nitrogen source and rate on productivity and quality of winter oilseed rape (L.) grown in different crop rotations., 2005, 94: 103–113.
[6] 荣楠, 韩永亮, 荣湘民, 宋海星, 彭建伟, 谢桂先, 张玉平, 张振华. 油菜NO3-的吸收, 分配及氮利用效率对低氮胁迫的响应. 植物营养与肥料学报, 2017, 23: 1104–1111. Rong N, Han Y L, Rong X M, Song H X, Peng J W, Xie G X, Zhang Y P, Zhang Z H. Response of NO3-uptake and distribution and nitrogen use efficiency in oilseed rape to limited nitrogen stress., 2017, 23: 1104–1111 (in Chinese with English abstract).
[7] Clément G, Moison M, Soulay F, Reisdorf-Cren M, Masclaux-Daubresse C. Metabolomics of laminae and midvein during leaf senescence and source-sink metabolite management inL. leaves., 2017, 69: 891–903.
[8] Wang X, Wang H, Wang J, Sun R, Wu J, Liu S, Bai Y, Mun J H, Bancroft I, Cheng F. The genome of the mesopolyploid crop species., 2011, 43: 1035.
[9] Bayer P E, Hurgobin B, Golicz A, Chan C K, Yuan Y, Lee H T, Renton M, Meng J, Li R, Long Y, Zou J, Bancroft I, Chalhoub B, King G J, Batley J, Edwards D. Assembly and comparison of two closely related Brassica napus genomes., 2017, 10: 1–9.
[10] Han Y L, Liu Q, Gu J D, Gong J M, Guan C Y, Lepo J E, Rong X M, Song H X, Zhang Z H. V-ATPase and V-PPase at the tonoplast affect NO3-content inby controlling distribution of NO3-between the cytoplasm and vacuole., 2015, 34: 22–34.
[11] 薛飞洋. 谷子苗期低氮胁迫转录组测序及蛋白磷酸酶2C(PP2C)基因家族的特性分析. 西北农林科技大学硕士学位论文, 陕西杨陵, 2013.Xue F Y. Transcriptome of Seeding Foxtall Millet Response to Low Nitrogen Stress and Characteristics Analysis of Protein Phosphatease 2C () Gene Family in Foxtall Millet. MS Thesis of Northwest A&F University, Yangling, Shaanxi, China, 2013 (in Chinese with English abstract).
[12] Chen W, Provart N J, Glazebrook J, Katagiri F, Chang H S, Eulgem T, Budworth P R. Expression profile matrix oftranscription factor genes suggests their putative functions in response to environmental stresses., 2002, 14: 559–574.
[13] Tran L S P, Nakashima K, Sakuma Y, Simpson S D, Fujita Y, Maruyama K, Yamaguchi-Shinozaki K. Isolation and functional analysis ofstress-inducible NAC transcription factors that bind to a drought-responsive cis-element in the early responsive to dehydration stress 1 promoter., 2004, 16: 2481–2498.
[14] Singh K B, Foley R C, Oñate-Sánchez L. Transcription factors in plant defense and stress responses., 2002, 5: 430–436.
[15] Zhao M H, Zhang W Z, Xu Z J, Wang J Y, Zhang L, Chen W F. Altered expression of transcription factor genes in rice flag leaf under low nitrogen stress., 2012, 19: 100–107.
[16] Shamloo-Dashtpagerdi R, Razi H, Ebrahimie E, Niazi A. Molecular characterization ofstress related transcription factors,and, selected based on comparative analysis ofandtranscriptomes., 2018, 45: 1111–1124.
[17] Hoagland D R, Arnon D I. The water-culture method for growing plants without soil., 1950, 347: 1–39.
[18] Hua Y P, Zhang D D, Zhou T, He M L, Ding G D, Shi L, Xu F S. Transcriptomics assisted quantitative trait locus fine mapping for the rapid identification of a nodulin 26-like intrinsic protein gene regulating boron efficiency in allotetraploid rapeseed., 2016, 39: 1601–1618.
[19] Ehlting B, Dluzniewska P, Dietrich H, Selle A, Teuber M, Hänsch R, Gessler A. Interaction of nitrogen nutrition and salinity in Grey poplar ()., 2007, 30: 796–811.
[20] Wang L, Zhou Q, Ding L, Sun Y. Effect of cadmium toxicity on nitrogen metabolism in leaves ofL. as a newly found cadmium hyperaccumulator., 2008, 154: 818–825.
[21] Patterson K, Cakmak T, Cooper A, Lager I, Rasmusson A G, Escobar M A. Distinct signalling pathways and transcriptome response signatures differentiate ammonium- and nitrate-supplied plants., 2010, 33: 1486–1501.
[22] Wang R C, Xing X J, Wang Y, Tran A, Crawford N M. A genetic screen for nitrate regulatory mutants captures the nitrate transporter gene., 2009, 151: 472–478.
[23] Li H, Li M, Luo J, Cao X, Qu L, Gai Y, Polle A, Peng C, Luo Z B. N-fertilization has different effects on the growth, carbon and nitrogen physiology, and wood properties of slow- and fast-growingspecies., 2012, 63: 6173–6185.
[24] Jain A K, Murty M N, Flynn P J. Data clustering: a review., 1999, 31: 264–323.
[25] Saldanha A J. Java Treeview-extensible visualization of microarray data., 2004, 20: 3246–3248.
[26] Ye J, Fang L, Zheng H K, Zhang Y, Chen J, Zhang Z J, Wang J, Li S T, Li R Q, Bolund L, Wang J. WEGO: a web tool for plotting GO annotations., 2006, 34: D293–D297.
[27] Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M, Itoh M, Katayama T, Kawashima S, Okuda S, Tokimatsu T, Yamanishi Y. KEGG for linking genomes to life and the environment., 2008, 36: D480–D484.
[28] Tarazona S, García-Alcalde F, Dopazo J, Ferrer A, Conesa A. Differential expression in RNA-seq: a matter of depth., 2011, 21: 2213–2223.
[29] Eisen M B, Spellman P T, Brown P O, Botstein D. Cluster analysis and display of genome-wide expression patterns., 1998, 95: 14863–14868.
[30] Ding L, Wang K J, Jiang G M, Biswas D K, Xu H, Li L F, Li Y H. Effects of nitrogen deficiency on photosynthetic traits of maize hybrids released in different years., 2005, 96: 925–930.
[31] Tobin A K, Yamaya T. Cellular compartmentation of ammonium assimilation in rice and barley., 2001, 52: 591–604.
[32] Jiang Y, Duan Y, Yin J, Ye S, Zhu J, Zhan, F, Luo K. Genome-wide identification and characterization of the populus WRKY transcription factor family and analysis of their expression in response to biotic and abiotic stresses., 2014, 65: 6629–6644.
[33] He Y J, Mao S S, Gao Y L, Zhu L Y, Wu D M, Cui Y X, Li J N, Qian W. Genome-wide identification and expression analysis of WRKY transcription factors under multiple stresses in., 2016, 11: e0157558.
[34] Cheng C L, Dewdney J, Nam H G, Den Boer B G, Goodman H M. A new locus () inencoding nitrate reductase., 1988, 7: 3309–3314.
[35] Wang Y Y, Hsu P K, Tsay Y F. Uptake, allocation and signaling of nitrate., 2012, 17: 458–467.
[36] Feng H, Yan M, Fan X, Li B, Shen Q, Miller A J, Xu G. Spatial expression and regulation of rice high-affinity nitrate transporters by nitrogen and carbon status., 2011, 62: 2319–2332.
[37] Lopez A J. Alternative splicing of pre-mRNA: developmental consequences and mechanisms of regulation., 1998, 32: 279–305.
[38] Good A G, Shrawat A K, Muench D G. Can less yield more is reducing nutrient input into the environment compatible with maintaining crop production?, 2004, 9: 597–605.
[39] Filleur S, Daniel-Vedele F. Expression analysis of a high-affinity nitrate transporter isolated fromby differential display., 1999, 207: 461–469.
[40] Birkenbihl R P, Diezel C, Somssich I E.is a key transcriptional regulator of hormonal and metabolic responses towards Botrytis cinerea infection., 2012, 159: 266–285.
[41] Li J, Brader G, Kariola T, Tapio P E.modulates the selection of signaling pathways in plant defense., 2006, 46: 477–491.
[42] Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in., 2001, 4: 447–456.
[43] Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L. MYB transcription factors in., 2010, 15: 573–581.
[44] 王玉明, 曹廷, 冯瑜, 柴友荣. 甘蓝型油菜基因RNA干扰载体构建. 作物杂志, 2010, (2): 23–27. Wang Y M, Cao T, Feng Y, Chai Y R. Construction of RNAi vector ofgene., 2010, (2): 23–27 (in Chinese with English abstract).
[45] Jung C, Seo J S, Han S W, Koo Y J, Kim C H, Song S I, Nahm B H, Choi Y D, Cheong J J. Overexpression ofenhances stomatal closure to confer abiotic stress tolerance in transgenic., 2008, 146: 623–635.
[46] Shim J S, Jung C, Lee S, Min K, Lee Y W, Choi Y, Choi Y D.regulatesexpression and modulates antagonistic interaction between salicylic acid and jasmonic acid signaling., 2013, 73: 483–495.
[47] Celenza J L, Quiel J A, Smolen G A, Merrikh H, Silvestro A R, Normanly J, Bender J. TheATR1 MYB transcription factor controls indolic glucosinolate homeostasis., 2005, 137: 253–262.
[48] Gigolashvili T, Yatusevich R, Berger B, Müller C, Flügge U I. The R2R3-MYB transcription factor/is a regulator of methionine-derived glucosinolate biosynthesis in., 2007, 51: 247–261.
[49] Malitsky S, Blum E, Less H, Venger I, Elbaz M, Morin S, Eshed Y, Aharoni A. The transcript and metabolite networks affected by the two clades ofglucosinolate biosynthesis regulators., 2008, 148: 2021–2049.
[50] Frerigmann H, Gigolashvili T.,, anddistinctly regulate indolic glucosinolate biosynthesis in., 2014, 7: 814–828.
[51] Kirik V, Kölle K, Miséra S, Bäumlein H. Two novel MYB homologues with changed expression in late embryogenesis-defectivemutants., 1998, 37: 819–827.
[52] Sun Q, Lin L, Liu D, Wu D, Fang Y, Wu J, Wang Y. CRISPR/Cas9-mediated multiplex genome editing of theandgenes inL., 2018, 19: 2716.
[53] Boue S, Letunic I, Bork P. Alternative splicing and evolution., 2003, 25: 1031–1034.
[54] Staiger D, Brown J W S. Alternative splicing at the intersection of biological timing, development, and stress responses., 2013, 25: 3640–3656.
[55] Filichkin S, Priest H D, Megraw M, Mockler T C. Alternative splicing in plants: directing traffic at the crossroads of adaptation and environmental stress., 2015, 24: 125–135.
Expression profile analysis of low nitrogen stress in
XIAO Yan**, YAO Jun-Yue**, LIU Dong, SONG Hai-Xing, and ZHANG Zhen-Hua*
College of Resource and Environment, Hunan Agricultural University / Southern Regional Collaborative Innovation Center for Grain and Oil Crops in China, Changsha 410128, Hunan, China
The nitrogen fertilizer was overapplied with people’s increased demand for crop yield, but the nitrogen utilization efficiency (NUE) of crops was decreasing. In this study, the differentially expressed genes (DEGs) including the nitrogen absorption, transport, distribution and transcription factors were screened under low nitrogen treatment of 0, 3, and 72 h by the physiological changes and RNA-Seq in rapeseed. The results showed that nitrogen were preferentially allocated to the shoots with the increased glutamine synthetase (GS) and NUE activities and the decreased nitrate reductase (NR) activity and the total nitrogen concentration under low nitrogen treatment. The analysis of Gene Ontology (GO) enrichment and the Kyoto encyclopedia of genes and genomes (KEGG) metabolic pathway showed that DEGs of the shoots were mainly involved in metabolic process, protein binding, ion binding, and anion binding, while DEGs of the roots were mainly involved in molecular function, primary metabolic process, ion binding, and anion binding. The gene expression profile analysis indicated that after low nitrogen treatment for 72 h, the expression of most genes inincreased; the expression ofandshowed significantly decreased in roots; the expression of most genes in,, anddecreased in the roots; the expression of most genes infamily was up-regulated in roots; and in the subfamily ofand, the expression of(BnaA06g04560D),(BnaA06g04570D),(BnaA02g11760D), and(BnaC02g16150D) increased significantly in roots. At the same time, skipped exon (SE) and mutually exclusive exons (MXE) type occurred in shoots and roots in order to have a better adaptation under low nitrogen stress. In conclusion, the NUE activity was increased by regulating,andgenes, and the,genes and alternative splicing favoredto adapt the low nitrogen stress.
; low nitrogen stress; NO3-; alternative splicing
10.3724/SP.J.1006.2020.94197
本研究由国家重点研发计划项目(2017YFD0200100, 2017YFD0200103)资助。
This study was supported by the National Key Research and Development Program of China (2017YFD0200100, 2017YFD0200103).
张振华, E-mail: zhzh1468@163.com
**同等贡献(Contributed equally to this work)
肖燕, E-mail: xiaoxiaoY8432@163.com; 姚珺玥, E-mail: yjy950606@126.com
2019-12-16;
2020-04-15;
2020-05-07.
URL: http://kns.cnki.net/kcms/detail/11.1809.s.20200506.1831.006.html

