川麦42和川农16抗穗发芽QTL定位及聚合效应分析
王琴,刘泽厚,万洪深,魏会廷,龙海,李涛,邓光兵,李俊,杨武云
川麦42和川农16抗穗发芽QTL定位及聚合效应分析
王琴1,2,刘泽厚1,2,万洪深1,2,魏会廷2,3,龙海4,李涛4,邓光兵4,李俊1,2,杨武云1,2
(1四川省农业科学院作物研究所,成都 610066;2农业部西南地区小麦生物学与遗传育种重点实验室,成都 610066;3四川省农业科学院植物保护研究所,成都 610066;4中国科学院成都生物研究所,成都 610041)
【】小麦穗发芽严重影响小麦产量和品质,是全球小麦生产面临的重大问题之一。通过鉴定挖掘抗穗发芽QTL,聚合穗发芽抗性位点,选育抗穗发芽小麦品种,为四川小麦穗发芽抗性改良提供技术和材料支撑。以川麦42/川农16重组自交系(RIL,F8)为材料,于2016—2018年分别在2个环境下对RIL群体进行籽粒发芽指数(GI,2016和2018)、籽粒发芽率(GR,2016和2018)和整穗发芽率(SGR,2017和2018)3个穗发芽指标测定。利用90K SNP芯片构建的遗传图谱检测全基因组穗发芽相关QTL,并分析抗性QTL聚合效应。双亲间GI、GR和SGR指标值差异显著,亲本川农16穗发芽抗性明显优于亲本川麦42。共检测到11个与穗发芽抗性有关的QTL,主要分布在2B、2D、3A、3D、4A、5A、5B和6B染色体上。5B染色体上检测到的单个环境表达的整穗发芽QTL解释的表型变异率最大,达到29%;在2D和3A染色体上检测到的整穗发芽主效QTL,以及5A染色体上检测到的与种子休眠相关的籽粒发芽主效QTL,在2个环境下均能表达,其抗穗发芽等位变异均来源于川农16。基因型分析发现,RIL群体中不同株系聚合抗性QTL的数量变幅为1—9个,表现为抗穗发芽的株系均携带4—9个与穗发芽相关的抗性QTL。重组自交系群体中6个株系GI、GR和SGR值均在15%以下,表现出高抗穗发芽特性;这6个优异株系聚合了多个与穗发芽相关的抗性QTL,且均聚合了川麦42在4A染色体上的微效QTL(和),以及川农16在2D和5B染色体上的主效QTL(和);编号为104和125的优异株系已通过审定,定名为川麦104和川麦64。其中,川麦104于2012年同时通过国家和四川省审定,其抗穗发芽能力强,产量、品质、抗病等优良性状突出,聚合了7个正向穗发芽QTL,包括2B、2D和5B染色体上来源于川农16的4个抗性QTL(、、和),以及4A和6B染色体上来源于川麦42的3个QTL(、和);近年来,川麦104已成为西南麦区小麦育种的核心亲本,育成小麦品种(系)18个。共检测到11个抗穗发芽QTL,其中3个来源于川麦42,8个来源于川农16;RIL群体中的抗穗发芽株系均携带4—9个抗性QTL,优异株系川麦104和川麦64高抗穗发芽,均聚合了7个穗发芽抗性QTL。
小麦;穗发芽;QTL定位;QTL聚合;聚合效应
0 引言
【研究意义】小麦收获前期如遇连续阴雨或湿热环境,其籽粒在麦穗上萌动、发芽的现象即为穗发芽(pre-harvest sprouting,PHS)[1]。小麦穗发芽在全世界小麦种植区均有发生,全球每年由小麦穗发芽引起的直接经济损失约为10亿美元[2-3];中国约有83%的小麦种植区均发生过严重的穗发芽危害,尤其是长江中下游麦区、西南麦区和东北春麦区频繁发生,黄淮冬麦区和北方冬麦区也时有发生,严重影响中国小麦的安全生产[1,4-6]。四川盆地在小麦成熟期降雨较多,特别是近2年,小麦收获前2—3 d连续阴雨,生产上,穗发芽敏感的品种穗发芽发生严重,引起其内部籽粒蛋白质和储藏物质分解,导致产量和品质严重降低,给种植大户和企业造成巨大的经济损失。因此,挖掘抗穗发芽基因,培育抗穗发芽小麦品种己成为小麦育种的重要目标。【前人研究进展】穗发芽是一个复杂的、由多基因控制的数量性状,遗传基础复杂且易受环境影响[7]。研究表明,种子休眠特性、穗部形态、籽粒性状、颖壳内发芽抑制物质、α-淀粉酶活性以及光照、温度等均不同程度影响穗发芽抗性[8]。目前,与穗发芽抗性相关的QTL已被定位到小麦21条染色体上[9-16],其中,第2、3和4同源群是穗发芽主效抗性基因的富集区域[14-15,17-21]。随着小麦基因组测序和生物信息学的快速发展,7个与穗发芽或休眠相关的基因已被克隆,包括第2同源群上的关键基因,第3同源群的关键基因、、和,第4同源群的关键基因和[22-30]。【本研究切入点】目前,已定位的小麦穗发芽QTL数量众多,随着候选基因的挖掘,一些与穗发芽抗性相关的功能标记也被开发应用;但不同群体、不同环境下均能有效应用的标记仍较少,且大多标记为与种子休眠特性相关的标记,从而限制了标记检测结果与穗发芽表型的吻合性。因此,穗发芽研究任重道远,挖掘更多穗发芽抗性基因及其优异等位变异,聚合不同效应的抗穗发芽优异等位基因将有助于提高栽培小麦穗发芽抗性。川麦42是四川省农业科学院作物研究所利用CIMMYT人工合成六倍体小麦育成的高产小麦品种,具有大穗大粒、抗条锈病等优异特性,但其对穗发芽和花期低温敏感。为了改良其穗发芽和花期冻害特性,四川省农业科学院小麦种质资源创新团队对收集的资源通过系统评价,发现四川农业大学培育的小麦品种川农16分蘖力强、抗白粉病、对花期低温和穗发芽具有较强抗性,其重要性状与川麦42互补性强。因此,四川省农业科学院小麦种质资源创新团队利用川麦42与川农16杂交,以单粒传的方式构建了重组自交系群体(RILs)。【拟解决的关键问题】本研究通过籽粒发芽和整穗发芽试验,利用中国科学院成都生物研究所构建的川麦42/川农16 RIL群体SNP遗传连锁图谱,挖掘与籽粒发芽和整穗发芽相关的QTL,寻找聚合双亲抗穗发芽QTL且表现抗穗发芽的优异材料。
1 材料与方法
1.1 试验材料
小麦品种川麦42、川农16及其构建的127个重组自交系(RIL,F8),以及穗发芽敏感对照川麦45。川麦42和对照川麦45由四川省农业科学院作物研究所提供,川农16由四川农业大学提供;川麦42和川农16为红粒小麦,川麦45为白粒小麦;重组自交系由四川省农业科学院作物研究所利用川麦42与川农16杂交,采用单粒传法构建而成,通过昆明、成都两地穿梭种植稳定成系。
川麦42、川农16及其构建的127个重组自交系群体,穗发芽敏感对照川麦45分别种植于广汉和成都。2016年种植于广汉(2016GH),每个小区种植3行,随机区组设计,3次重复,行长1.5 m,行距20 cm,每行15窝,采用免耕、撬窝、稻草覆盖栽培方式;2017和2018年均种植于成都网室(2017CD和2018CD),试验设计和种植方式与广汉相同,播种后采用细土覆盖。田间管理与当地小麦生产一致。
1.2 穗发芽鉴定
籽粒发芽:对2016年和2018年环境下的RIL群体及其亲本进行籽粒发芽测定,于生理成熟期随机取中间一行的5个穗子,室内阴干,7 d后-20℃保存。将保存于-20℃的5个穗子手工脱粒,用0.5%的NaClO消毒冲洗后,取100粒完整种子置于培养皿(培养皿底部置有湿润的滤纸)中;每个材料2次重复,试验第2天开始统计发芽种子数,并将已发芽种子移除,连续计数7 d。计算发芽率(seed germination rate,GR)和发芽指数(seed germination index,GI)。GR=(发芽粒数∕总粒数)×100%;GI=(7×n1+6×n2+5×n3+4×n4+3×n5+2×n6+1×n7)/(7×N)×100%,N表示籽粒总数;n1、n2、…、n7分别表示第1天至第7天的发芽籽粒数。
整穗发芽:对2017年和2018年成都点的RIL群体及其亲本进行人工模拟降雨田间整穗发芽测定。采用田间自动喷灌系统人工模拟降雨,于生理成熟期连续喷洒7 d,确保所有试验材料穗子一直保持湿润,待对照穗子籽粒90%以上萌动后,随机收获中间一行的10个穗子,快速烘干后手工脱粒,统计总粒数和发芽粒数,计算整穗发芽率(seed germination rate of in each spike,SGR)。SGR=发芽粒数/总粒数×100%。
1.3 分子标记及遗传图谱的构建
供试材料基因组DNA由中国科学院成都生物研究所采用CTAB方法提取,DNA质检、小麦90K SNP芯片检测工作由北京博奥公司完成。RIL群体SNP数据分型及遗传图谱均由中国科学院成都生物研究所分析完成,遗传连锁图谱采用IciMapping 4.1软件完成。
1.4 数据统计及QTL分析
利用SPSS 13.0 软件对表型进行统计分析,通过QTL分析软件IciMapping v3.0(http://www.isbreeding. net/software.html)分别计算各环境下不同性状的广义遗传力。参考前人研究以抗性品种发芽率鉴定筛选抗穗发芽材料[1,5-6,8],结合实际情况,将GI、GR和SGR值低于15%定为高抗穗发芽品种,15%—25%定为抗穗发芽品种。
利用QTL分析软件ICIMapping v3.0对RIL群体的GI、GR和SGR进行QTL分析。采用逐步回归法的完备区间作图法(inclusive composite interval mapping,ICIM),基于1 cM的步移速度进行QTL的鉴定分析,最小QTL效应的LOD阈值设为2.5。
2 结果
2.1 穗发芽分析
2016、2017和2018年对亲本川麦42、川农16及其RIL群体进行了穗发芽鉴定,3个指标籽粒发芽指数(GI)、籽粒发芽率(GR)和整穗发芽率(SGR)用于分析其穗发芽抗性(表1)。结果显示,2个环境下,亲本川麦42的3个指标值(GI、GR和SGR)均显著高于亲本川农16,表明川农16穗发芽抗性显著优于川麦42;RIL群体中穗发芽抗性呈连续分布,存在超亲分离现象。不同环境下穗发芽抗性参数的遗传力不同,2016年籽粒发芽率的遗传力最高(0.82)。
相关性分析表明(表2),所有穗发芽参数间均表现正相关;单环境下,GI、GR和SGR之间表现为显著或极显著正相关,GI与GR相关系数较大,而GI、GR分别与SGR间的相关系数较小;不同环境下籽粒发芽参数相关性不显著。SGR在2个环境下极显著正相关,但相关系数小于0.5。结果表明,小麦穗发芽抗性易受环境影响,但在不同年份间,SGR表现出相对稳定的趋势。
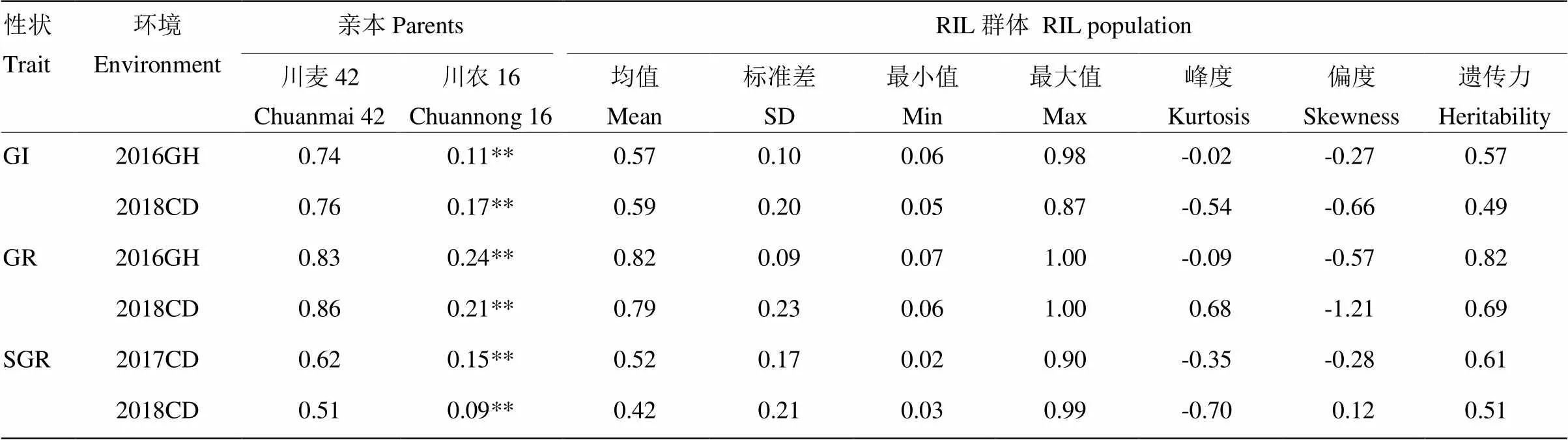
表1 RIL群体及其亲本穗发芽性状参数统计
GI:籽粒发芽指数;GR:籽粒发芽率;SGR:整穗发芽率;GH:广汉;CD:成都。**表示川麦42和川农16在0.01水平差异极显著。下同
GI: Seed germination index; GR: Seed germination rate; SGR: Seed germination rate of in each spike; GH: Guanghan; CD: Chengdu. ** Indicate significant difference at<0.01 between Chuanmai 42 and Chuannong 16. The same as below
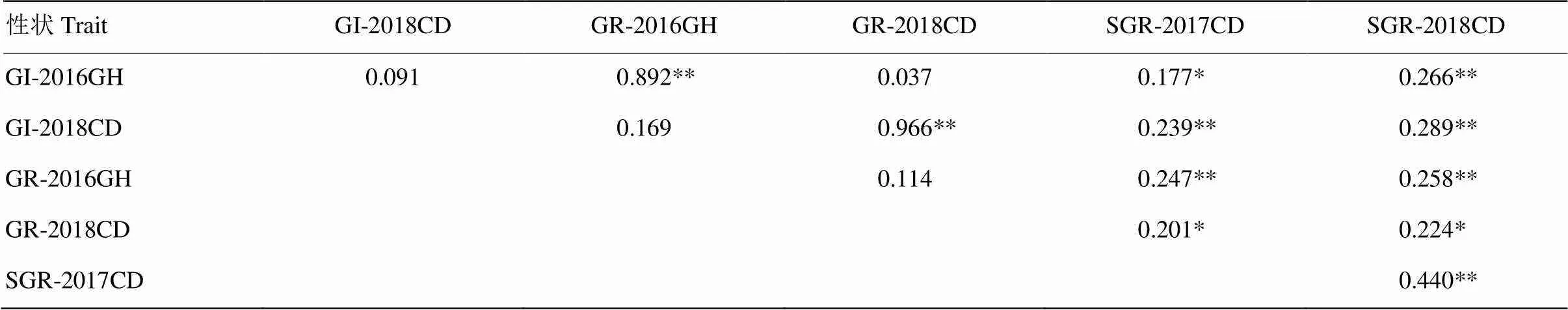
表2 RIL群体中穗发芽性状间的相关性
*表示在0.05水平上差异显著* represent significance at 0.05
2.2 QTL分析
利用RIL群体在不同环境下穗发芽相关性状的表型数据,结合SNP芯片数据对穗发芽相关QTL进行分析。共检测到11个与穗发芽抗性有关的QTL(表3和图1),贡献率为4.28%—29.03%,主要分布在2B、2D、3A、3D、4A、5A、5B和6B染色体上;除4A和6B染色体上的3个QTL增加穗发芽抗性的等位变异来自川麦42外,其余8个QTL增加穗发芽抗性的等位变异均来自亲本川农16。5B染色体上—区段检测到的与整穗发芽相关的QTL()贡献率最大(29.03%),但仅在单个环境中表达。2017和2018年2个环境下,在2D和3A染色体上均检测到SGR的QTL,不同环境下的QTL贡献率差异明显,最高为10.86%。5A染色体上—区段检测到与休眠相关的GI和GR的QTL,均能在2个环境下表达,贡献率最高为18.80%。2B和4A染色体上仅检测到在单个环境表达的与休眠相关的微效QTL,其贡献率均较小。
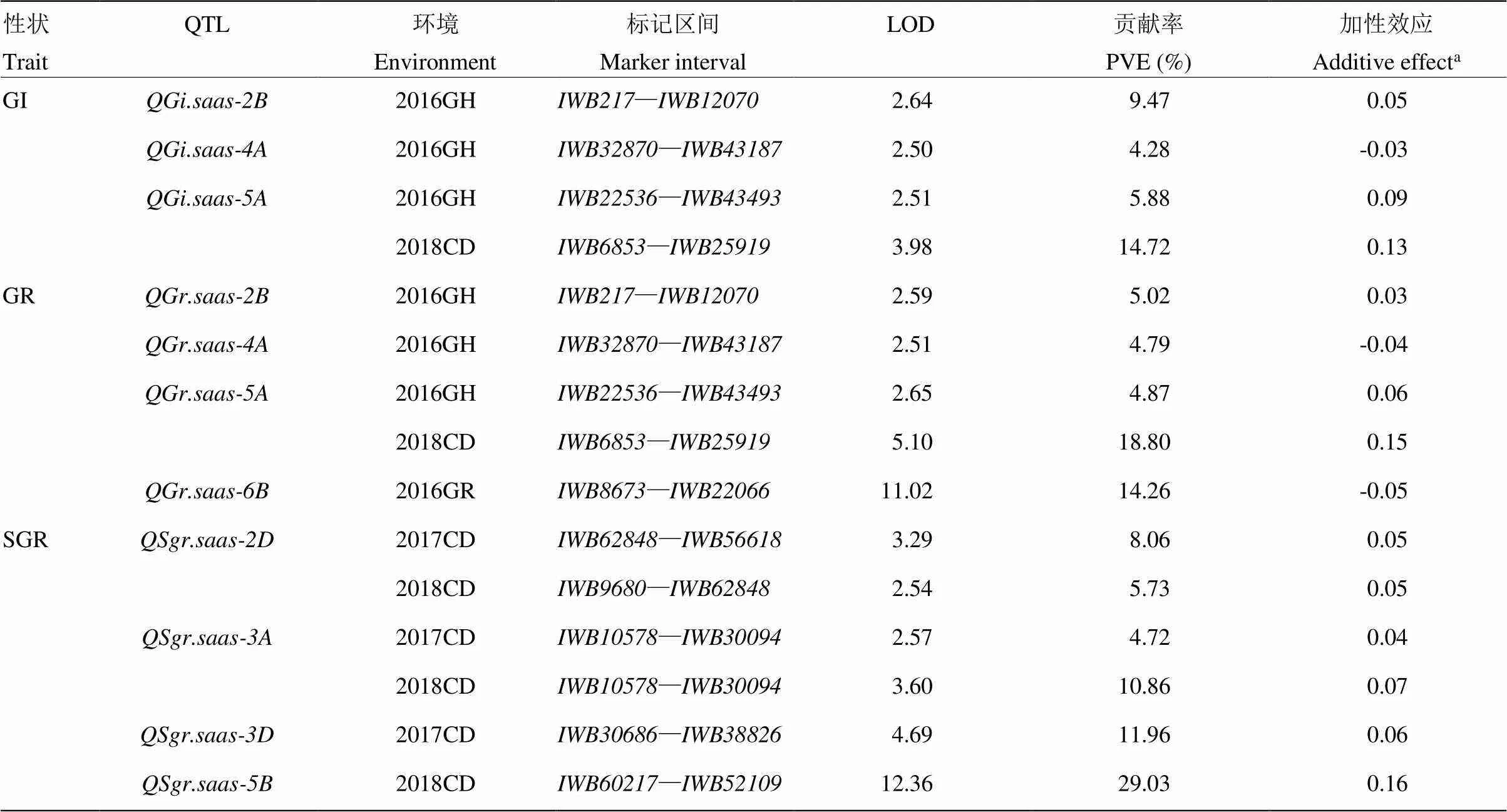
表3 川麦42/川农16重组自交系群体中检测到的穗发芽相关性状QTL
a:加性效应正值表示增强穗发芽抗性的等位变异来自亲本川农16,负值表示增强穗发芽抗性的等位变异来自亲本川麦42
a: Positive values of additive effect indicate that alleles from Chuannong 16 increase the resistance to PHS, and negative values indicate that alleles from Chuanmai 42 increase the resistance
2.3 抗性QTL聚合效应分析
基因型分析发现,在RIL群体中,普遍存在穗发芽抗性QTL聚合现象,聚合的QTL数量变幅在1—9个,其中,聚合4个抗性QTL的株系数最多(图2)。根据3个环境下穗发芽表型数据,发现RIL群体中GI、GR和SGR值低于15%的株系分别为13、12和14个,GI、GR和SGR值在15%—25%的株系分别为31、32和36个;其中6个株系的GI、GR和SGR值均低于15%,表现为高抗穗发芽(表4)。分析其基因型,发现抗穗发芽的株系携带4—9个穗发芽相关性状的抗性QTL;6个优异株系携带7或8个抗性QTL,编号为12和74的株系携带QTL数最多(8个)。6个高抗株系同时聚合了川麦42在4A染色体上的微效QTL,以及川农16在2D和5B染色体上的主效QTL。株系12、74和125在2个环境下SGR值均小于10%,均携带2D、3D和5B染色体上的SGR抗性QTL。高抗穗发芽的株系104已于2012年同时通过国家和四川省审定,定名为川麦104;株系125于2013年通过四川省审定,定名为川麦64。
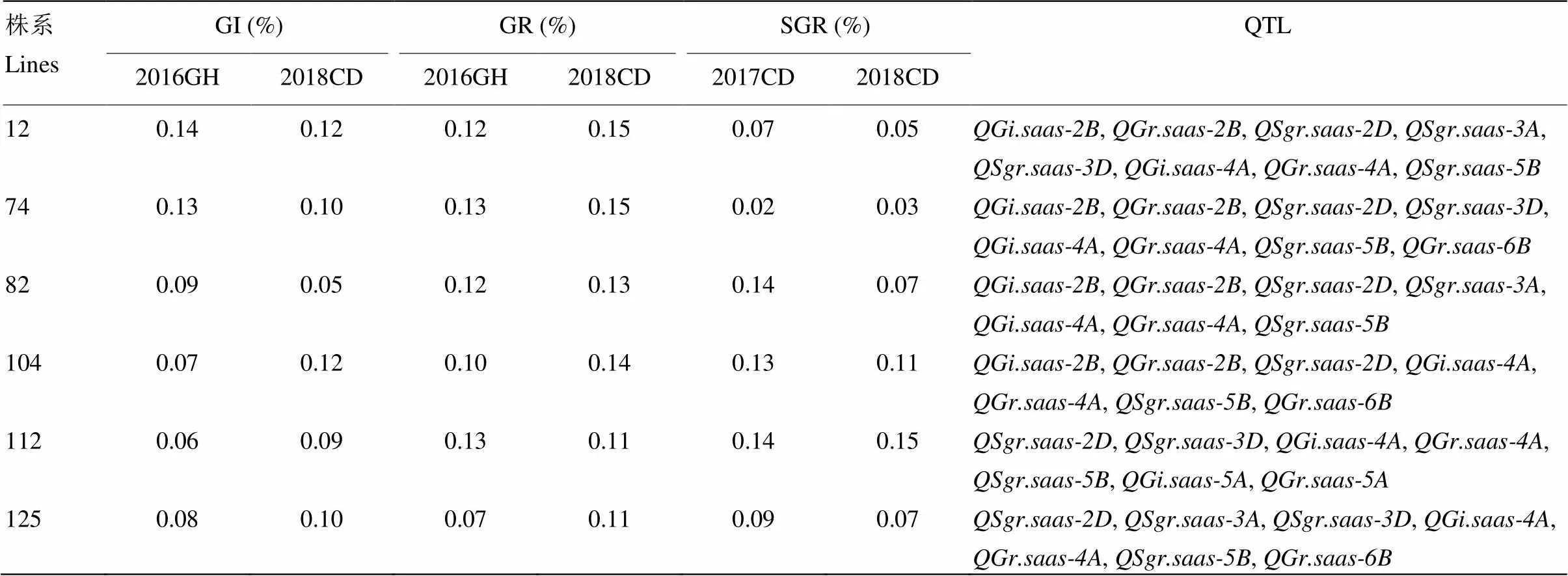
表4 6个高抗穗发芽株系携带的抗性QTL

三角形表示峰值所在位置。无填充的三角形表示2016年的QTL,斜线填充的三角形表示2017年的QTL,黑色填充的三角形表示2018年的QTL
The triangle indicates the position of QTL peak. The unfilled triangles represent QTLs for PHS in 2016, the diagonal filled triangles represent QTLs for PHS in 2017, and the black triangles represent QTLs for PHS in 2018
图1 穗发芽相关性状QTL在8条染色体上的分布
Fig. 1 QTLs for PHS related traits on 8 chromosomes
3 讨论
3.1 人工模拟降雨整穗发芽法效果
小麦穗发芽的鉴定方法有很多,如整穗发芽、籽粒发芽、α-淀粉酶活性鉴定、田间穗发芽鉴定、沉降值测量等,常用的主要是籽粒发芽、整穗发芽法和田间发芽鉴定[1,8]。籽粒发芽法为简化的穗发芽鉴定方法,是生产上大规模鉴定评价的最普遍的方法,但其只能检测种子的休眠特性;整穗发芽法和田间发芽鉴定法既能检测种子的休眠性,又能从穗部形状、颖壳特性、外源抑制物质等方面全面直观地反映品种的抗穗发芽特性,是最符合生产实际的遗传研究方法。本研究同时利用籽粒发芽法和整穗发芽法鉴定评价川麦42/川农16重组自交系群体及其亲本穗发芽发生程度,更能全面地挖掘抗穗发芽QTL,鉴定出更适合大田生产需要的育种材料。本研究同一材料整穗发芽率普遍比籽粒发芽率和发芽指数低,这与前人研究结果一致[31];相关性分析表明,穗发芽抗性易受环境影响,但在不同年份间,SGR表现出相对稳定的趋势。因此,人工模拟降雨整穗发芽法也能较准确地评价穗发芽抗性,通过2种鉴定方法评价筛选抗穗发芽材料,更能优选出适合大田生产实际的资源。
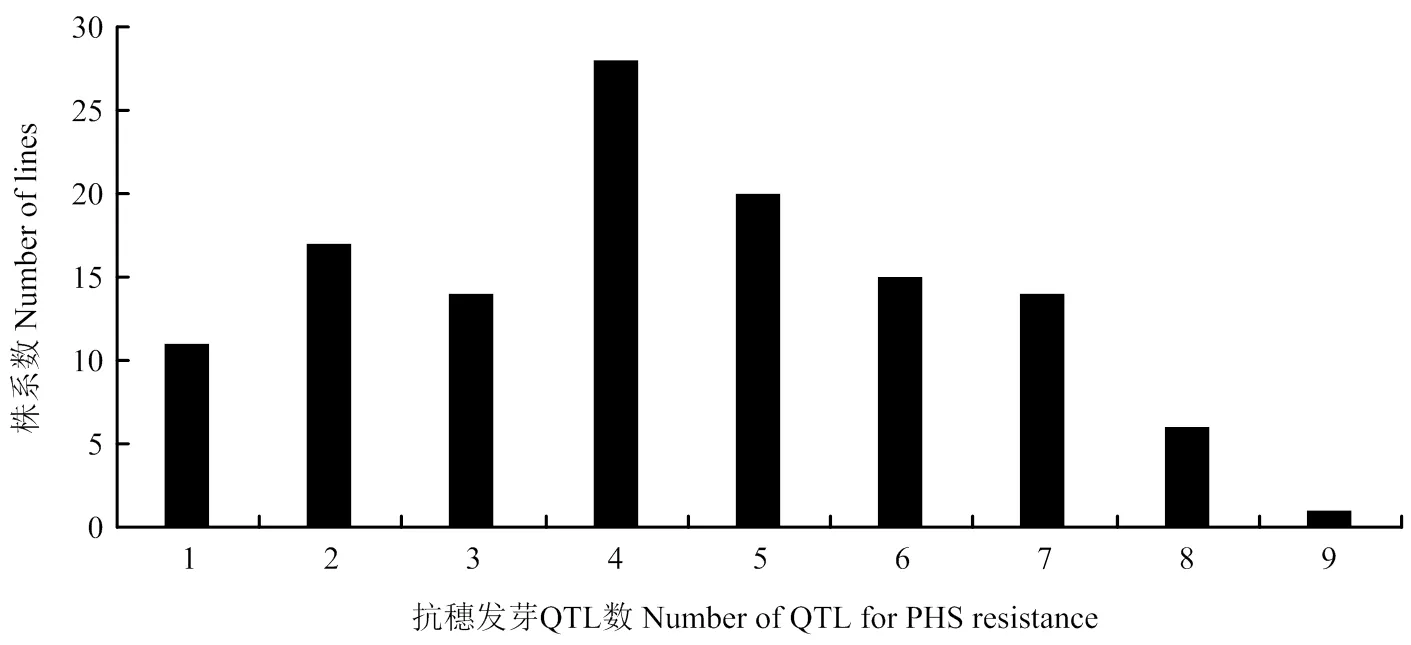
图2 川麦42/川农16 RILs中穗发芽抗性QTL聚合频率分布
3.2 川麦42和川农16抗穗发芽QTL比较
前人利用不同研究群体已将小麦穗发芽和种子休眠相关QTL定位在所有染色体上,其中,第2、3和4同源群上的穗发芽相关基因在不同研究中均被重复定位到[9-16,32-35]。本研究利用中国科学院成都生物研究所构建的SNP遗传连锁图谱,根据穗发芽相关的表型数据,在2B、2D、3A、3D、4A、5A、5B和6B染色体上检测到11个与穗发芽抗性相关的QTL,其中,2D、3A和5A上检测到的主效QTL在2个环境下均能稳定表达。前人利用染色体代换系、DH群体等研究也发现位于2D、3A、4A和5A染色体上与穗发芽相关的主效QTL[7,21,36-41],但由于这些研究所用标记方法与本研究不同,不能直接比较,因此,无法确定本研究在2D、3A、4A和5A染色体上检测到的QTL是否与前人定位的一致。Lin等[42]利用SSR和GBS-SNP技术在5A染色体上发现一个与种子休眠相关的QTL,与SSR标记距离最近;Kumar等[37]通过籽粒发芽的方法将与籽粒休眠相关的QTL定位在5A染色体上SSR标记—;Groos等[36]通过整穗发芽的方法也将穗发芽QTL定位到与Kumar等[37]发现的5A染色体上同一区域。本研究在5A染色体上检测到的与种子休眠相关的QTL在2个环境下均表达,贡献率为4.87%—18.8%,但是否与前人研究结果位于同一区域还需进一步增加SSR标记扫描验证。Zhang等[25-26]利用比较基因组学法,在小麦中对水稻休眠基因进行同源克隆,获得休眠基因,并开发了和的分子标记;周勇[43]在3A染色体上发现地方品种中休眠相关QTL与SNP标记-连锁,毗邻籽粒颜色相关的转录因子基因。目前,2D、3A和4A染色体上与穗发芽相关基因/QTL多次被报道,其关键基因已经被克隆[3,7,17-19,21,29,38-39,41,44-46],位于第3和第4同源群的、、和是目前最有效的小麦穗发芽相关基因[47];本研究在2D、3A和4A染色体上检测到的穗发芽相关主效QTL是否是克隆的基因或邻近已知基因,还需利用有效标记检测及SNP序列比较分析进一步验证。
3.3 抗穗发芽QTL聚合及衍生品种川麦104抗性表现
本研究利用双亲染色体重组筛选出一批抗穗发芽材料,聚合了多个穗发芽抗性QTL。其中,株系104于2012年同时通过国家和四川省审定,定名为川麦104;川麦104国家区试平均产量6 130.5 kg·hm-2,比对照川麦42增产8.42%;四川省区试平均产量6 116.1 kg·hm-2,比对照绵麦37增产14.12%。基因型分析发现,川麦104聚合了双亲7个正向穗发芽QTL,具有较强的抗穗发芽能力;李式昭等[48]利用穗发芽抗性等位基因分子标记,对2000年以来四川育成的105份小麦品种进行鉴定,发现川麦104同时含抗穗发芽基因。Tang等[49]控制性试验表明,生理成熟期川麦104的籽粒发芽指数在所有参试品种中最低,成熟后延迟1—2周收获,其降落值降低幅度小,仍接近优质小麦标准(300s),显著高于其他多数参试品种。2018年小麦成熟后持续淋雨3 d,30个参试品种平均籽粒发芽率20.8%,最高68.9%,而川麦104籽粒发芽率仅2.3%;2019年成熟期遭遇持续降雨,各地调查川麦104基本未出现发芽,而大多数品种穗发芽严重。由于川麦104产量、品质、抗病及抗穗发芽等优良性状突出,其已成为西南麦区小麦育种的核心亲本,近年来,已育成小麦品种(系)18个。因此,双亲抗穗发芽QTL聚合可以提高穗发芽抗性,本研究鉴定出的抗穗发芽优异株系是小麦穗发芽育种改良的重要基因资源。
4 结论
共检测到11个与穗发芽抗性相关的QTL,其中3个来源于川麦42,8个来源于川农16。2D、3A和5A染色体上检测到的穗发芽主效QTL,在2个环境下均能表达,其抗穗发芽等位变异均来源于川农16。在RIL群体中,抗穗发芽的株系均携带4—9个与穗发芽相关的QTL,6个优异株系聚合了7或8个抗性QTL;编号为104和125的优异株系(国家、四川省审定,定名为川麦104和川麦64)聚合了7个正向穗发芽QTL,包括4个来源于川农16的抗性QTL和3个来源于川麦42的QTL。
[1] 肖世和, 闫长生, 张海萍, 孙果忠. 小麦穗发芽研究. 北京: 中国农业科学技术出版社, 2002.
XIAO S H, YAN C S, ZHANG H P, SUN G Z..Beijing: China Agricultural Science and Technology Press, 2002. (in Chinese)
[2] DERERA N F, BHATT G M, MCMASTER G J. On the problem of preharvest sprouting of wheat., 1977, 26(2): 299.
[3] OGBONNAYA F C, IMTIAZ M, YE G, HEARNDEN P R, HERNANDEZ E, EASTWOOD R F, GINKEL M V, SHORTER S C, WINCHESTER J M. Genetic and QTL analyses of seed dormancy and preharvest sprouting resistance in the wheat germplasm CN10955., 2008, 116(7): 891-902.
[4] XIAO S H, ZHANG X Y, YAN C S, LIN H. Germplasm improvement for preharvest sprouting resistance in Chinese white-grained wheat: An overview of the current strategy., 2002, 126(1): 35-38.
[5] 原亚萍, 陈孝, 肖世和. 小麦穗发芽的研究进展. 麦类作物学报, 2003, 23(3): 136-139.
YUAN Y P, CHEN X, XIAO S H. Advances in the study on wheat pre-harvest sprouting., 2003, 23(3): 136-139. (in Chinese)
[6] 闫长生, 张海萍, 海林, 张秀英, 胡琳, 胡汉桥, 蒲宗君, 肖世和. 中国小麦品种穗发芽抗性差异的研究. 作物学报, 2006, 32(4): 580-587.
YAN C S, ZHANG H P, HAI L, ZHANG X Y, HU L, HU H Q, PU Z J, XIAO S H. Differences of preharvest sprouting resistance among chinese wheat cultivars., 2006, 32(4): 580-587. (in Chinese)
[7] IMTIAZ M, OGBONNAYA F C, OMAN J, GINKEL M V. Characterization of quantitative trait loci controlling genetic variation for preharvest sprouting in synthetic backcross-derived wheat lines., 2008, 178(3): 1725-1736.
[8] 刘莉, 王庆海, 陈国志. 小麦穗发芽研究进展. 作物杂志, 2013(4): 6-11.
LIU L, WANG Q H, CHEN G Z. Advances on resistance to pre-harvest sprouting in wheat., 2013(4): 6-11. (in Chinese)
[9] ANDERSON J A, SORRELLS M E, TANKSLEY S D. RFLP analysis of genomic regions associated with resistance to preharvest sprouting in wheat., 1993, 33(3): 453-459.
[10] ROY J K, PRASAD M, VARSHNEY R K, BALYAN H S, BLAKE T K, DHALIWAL H S, EDWARDS K J, GUPTA P K. Identification of a microsatellite on chromosomes 6B and a STS on 7D of bread wheat showing an association with preharvest sprouting tolerance., 1999, 99(1): 336-340.
[11] ARIF M R, NEUMANN K, NAGEL M, KOBILJSKI B, LOHWASSER U, BÖRNER A. An association mapping analysis of dormancy and pre-harvest sprouting in wheat., 2012, 188: 409-417.
[12] FLINTHAM J, ADLAM R, BASSOI M, HOLDSWORTH M, GALE M. Mapping genes for resistance to sprouting damage in wheat., 2002, 126: 39-45.
[13] JAISWAL V, MIR R R, MOHAN A, BALYAN H S, GUPTA P K. Association mapping for pre-harvest sprouting tolerance in common wheat (L.)., 2012, 188: 89-102.
[14] KULWAL P L, SINGH R, BALYAN H S, GUPTA P K. Genetic basis of pre-harvest sprouting tolerance using single-locus and two-locus QTL analyses in bread wheat., 2004, 4(2): 94-101.
[15] MARES D, RATHJEN J, MRVA K, CHEONG J. Genetic and environmental control of dormancy in white-grained wheat (L.)., 2009, 168(3): 311-318.
[16] SINGH A K, KNOX R E, CLARKE J M, CLARKE F R, SINGH A, DEPAUW R M, CUTHBERT R D. Genetics of pre-harvest sprouting resistance in a cross of Canadian adapted durum wheat genotypes., 2014, 33(4): 919-929.
[17] REN X B, LAN X J, LIU D C, WANG J L. LIANG Z Y. Mapping QTLs for pre-harvest sprouting tolerance on chromosome 2D in a synthetic hexaploid wheat × common wheat cross., 2008, 49(4): 333-341.
[18] ZHANG X Q, LI C D, TAY A, LANCE R, MARES D, CHEONG J, CAKIR M, MA J H, APPELS R. A new PCR-based marker on chromosome 4AL for resistance to pre-harvest sprouting in wheat (L.)., 2008, 22(2): 227-236.
[19] CHEN C X, CAI S B, BAI G H. A major QTL controlling seed dormancy and pre-harvest sprouting resistance on chromosome 4A in a Chinese wheat landrace., 2008, 21(3): 351-358.
[20] SOMYONG S, ISHIKAWA G, MUNKVOLD J D, TANAKA J, BENSCHER D, CHO Y G, SORRELS M E. Fine mapping of a preharvest sprouting QTL interval on chromosome 2B in white wheat., 2014, 127(8): 1843-1855.
[21] WANG X Y, LIU H, MIA M S, SIDDIQUE K H M, YAN G J. Development of near-isogenic lines targeting a major QTL on 3AL for pre-harvest sprouting resistance in bread wheat., 2018, 69(9): 864-872.
[22] WANG J R, LIU Y X, WANG Y, CHEN Z H, DAI S, CAO W G, FEDAK G, LAN X J, WEI Y M, LIU D C, ZHENG Y L. Genetic variation of, 2011, 33(2): 139-146.
[23] NAKAMURA S, ABE F, KAWAHIGASHI H, NAKAZONO K, TAGIRI A, MATSUMOTO T, UTSUGI S, OGAWA T, HANDA H, ISHIDA H, MORI M, KAWAURA K, OGIHARA Y, MIURA H. A wheat homolog of MOTHER OF FT and TFL1 acts in the regulation of germination., 2011, 23(9): 3215-3229.
[24] LIU S B, SEHGAL S K, LI J R, LIN M, TRICK H N, YU J M, GILL B S, BAI G H. Cloning and characterization of a critical regulator for preharvest sprouting in wheat., 2013, 195(1): 263-273.
[25] ZHANG Y J, MIAO X L, XIA X C, HE Z H. Cloning of seed dormancy genes () associated with tolerance to pre-harvest sprouting in common wheat and development of a functional marker., 2014, 127(4): 855-866.
[26] ZHANG Y J, XIA X C, HE Z H. The seed dormancy alleleassociated with pre-harvest sprouting tolerance is mainly present in Chinese wheat landraces., 2016, 130(1): 81-89.
[27] ZHOU S H, FU L, WU Q H, CHEN J J, CHEN Y X, XIE J Z, WANG Z Z, WANG G X, ZHANG D Y, LIANG Y, ZHANG Y, YOU M S, LIANG R Q, HAN J, LIU Z Y. QTL mapping revealedconferred pre-harvest sprouting resistance in wheat population Yanda 1817×Beinong 6., 2017, 16(2): 435-444.
[28] DONG Z D, CHEN J, LI T CHEN F, CUI D Q. Molecular survey ofgenes and their association with grain color and germinability in Chinese wheat and., 2015, 94(3): 453-459.
[29] BARRERO J M, CAVANAGH C, VERBYLA K L, TIBBITS J F G, VERBYLA A P, HUANG B. E, ROSEWARNE G M, STEPHEN S, WANG P, WHAN A, RIGAULT P, HAYDEN M J, GUBLER F. Transcriptomic analysis of wheat near-isogenic lines identifiesandas candidates for a major dormancy QTL., 2015, 16: 93.
[30] TORADA A, KOIKE M, OGAWA T, TAKENOUCHI Y, TADAMURA K, WU J Z, MATSUMOTO T, KAWAURA K, OGIHARA Y. A causal gene for seed dormancy on wheat chromosome 4A encodes a MAP kinase kinase., 2016, 26(6): 782-287.
[31] 李玉营, 马东方, 王晓玲, 方正武. 小麦穗发芽鉴定方法的比较与分析. 广西植物, 2016, 36(3): 261-266.
LI Y Y, MA D F, WANG X L, FANG Z W, Comparison and analysis of wheat pre-harvest sprouting screening methods., 2016, 36(3): 261-266. (in Chinese)
[32] KULWAL P L, KUMAR N, GAUR A, KHURANA P, KHURANA J P, TYAGI A K, BALYAN H S, GUPTA P K. Mapping of a major QTL for pre-harvest sprouting tolerance on chromosome 3A in bread wheat., 2005, 111(6): 1052-1059.
[33] JIANG Y F, WANG J R, LUO W, WEI Y M, QI P F, LIU Y X, JIANG Q T, PENG Y Y, CHEN G Y, DAI S F, ZHENG Y L, LAN X J. Quantitative trait locus mapping for seed dormancy in different post-ripening stages in a Tibetan semi-wild wheat (ssp.Shao)., 2015, 203(3): 557-567.
[34] LIN M, ZHANG D D, LIU S B, ZHANG G R, YU J M, FRITZ A K, BAI G H. Genome-wide association analysis on pre-harvest sprouting resistance and grain color in U.S. winter wheat., 2016, 17(1): 794-810.
[35] MOHAN A, KULWAL P, SINGH R, KUMAR V, MIR R R, KUMA J, PRASAD M, BALYAN H S, GUPTA P K. Genome-wide QTL analysis for pre-harvest sprouting tolerance in bread wheat., 2009, 168(3): 319-329.
[36] GROOS C, GAY G, PERRETANT M R, GERVAIS L, BERNARD M, DEDRYVER F, CHARMET G. Study of the relationship between pre-harvest sprouting and grain color by quantitative trait loci analysis in a white×red grain bread-wheat cross., 2002, 104(1): 39-47.
[37] KUMAR S, KNOX R E, CLARKE F R, POZNIAK C J DEPAUW RM, CUTHBERT R D, FOX S. Maximizing the identification of QTL for pre-harvest sprouting resistance using seed dormancy measures in a white-grained hexaploid wheat population., 2015, 205(1): 287-309.
[38] MARES D J, MRVA K, CHEONG J, WILLIAMS K, WATSON B, STORLIE E, SUTHERLAND M, ZOU Y. A QTL located on chromosome 4A associated with dormancy in white- and red-grained wheats of diverse origin., 2005, 111(7): 1357-1364.
[39] TAN M K, SHARP P J, LU M Q, HOWES N. Genetics of grain dormancy in a white wheat., 2006, 57: 1157-1165.
[40] MUNKVOLD J D, TANAKA J, BENSCHER D, SORRELLS M E. Mapping quantitative trait loci for preharvest sprouting resistance in white wheat., 2009, 119(7): 1223-1235.
[41] LIU S B, BAI G H, CAI S B, CHEN C X. Dissection of genetic components of preharvest sprouting resistance in white wheat., 2011, 27(4): 511-523.
[42] LIN M, CAI S, WANG S, LIU S B, ZHANG G R, BAI G H. Genotyping-by-sequencing (GBS) identified SNP tightly linked to QTL for pre-harvest sprouting resistance., 2015, 128(7): 1385-1395.
[43] 周勇. 中国小麦地方品种穗发芽抗性评价及全基因组关联分析[D]. 成都: 四川农业大学, 2017.
ZHOU Y. Genome-wide association study for pre-harvest sprouting resistance in a large germplasm collection of Chinese wheat landraces [D]. Chengdu: Sichuan Agricultural University, 2017. (in Chinese)
[44] SINGH R, MATUS-CÁDIZ M, BÅGA M, HUCL P, CHIBBAR R N. Identification of genomic regions associated with seed dormancy in white-grained wheat., 2010, 174(3): 391-408.
[45] CABRAL A L, JORDAN M C, MCCARTNEY C A, YOU F M, HUMPHREYS D G, MACLACHLAN R, POZNIAK C J. Identification of candidate genes, regions and markers for pre-harvest sprouting resistance in wheat (L.)., 2014, 14(1): 340.
[46] KATO K, NAKAMURA W, TABIKI T, MIURA H, SAWADA S. Detection of loci controlling seed dormancy on group 4 chromosomes of wheat and comparative mapping with rice and barley genomes., 2001, 102(6/7): 980-985.
[47] Justin M V, Robert N S,John M M,Michael J G. Revealing the genetic mechanisms of pre-harvest sprouting in hexaploid wheat (L.)., 2019, 281: 180-185.
[48] 李式昭, 郑建敏, 伍玲, 李俊, 万洪深, 杨漫宇, 罗江陶, 刘廷辉, 杨开俊, 蒲宗君. 四川小麦品种籽粒硬度和穗发芽抗性相关基因的分子标记鉴定. 西南农业学报, 2018, 31(4): 641-645.
LI S Z, ZHENG J M, WU L, LI J, WAN H S, YANG M Y, LUO J T, LIU T H, YANG K J, PU Z J. Identification of grain hardness and resistance to pre-harvest sprouting in Sichuan wheat cultivars with molecular markers., 2018, 31(4): 641-645. (in Chinese)
[49] TANG Y L, LI C S, YANG W Y, WU Y Q, WU X L, WU C, MA X L, LI S Z, ROSEWARNE G M. Quality potential of synthetic-derived commercial wheat cultivars in south-western China., 2016, 67(6): 583-593.
Identification and pyramiding of QTLs for traits associated with Pre-harvest sprouting resistance in two wheat cultivars Chuanmai 42 and Chuannong 16
WANG Qin1,2, LIU ZeHou1,2, WAN HongShen1,2, WEI HuiTing2,3, LONG Hai4, LI Tao4, DENG GuangBing4, LI Jun1,2, YANG WuYun1,2
(1Crop Research Institute, Sichuan Academy of Agricultural Sciences, Chengdu 610066;2Key Laboratory of Wheat Biology and Genetic Improvement on Southwestern China, Ministry of Agriculture, Chengdu 610066;3Plant Protection Institute, Sichuan Academy of Agricultural Sciences, Chengdu 610066;4Chengdu Institute of Biology, Chinese Academy of Sciences, Chengdu 610041)
【】 Pre-harvest sprouting (PHS) is one of the serious problems for wheat production, which significantly reduces grain yield and end-use quality, especially in rainy or high humidity regions. The objective of this study is to identify and aggregate quantitative trait loci (QTLs) for traits associated with PHS resistance, which will provide a theoretical basis for improving PHS resistance in Sichuan wheat cultivars.【】A recombinant inbred line (RIL) population derived from Chuanmai 42 and Chuannong 16 was used to detect QTL and assess the new germplasm resources for PHS resistance. 127 lines in RIL population were analyzed by phenotypic and genetic identification for PHS-related traits. Seed germination index (GI), seed germination rate (GR) and seed germination rate of in each spike (SGR) in two different environments were used to evaluate PHS resistance. All QTLs for PHS resistance were mapped by an available high-density single nucleotide polymorphism (SNP, 90K). The pyramiding of the resistant QTL was also analyzed according to the genotype of every line in RILs. 【】There were significant difference in GI, GR and SGR between two parents. PHS resistance of Chuannong 16 was superior than that of Chuanmai 42. A total of 11 QTLs for PHS were detected on chromosomes 2B, 2D, 3A, 3D, 4A, 5A, 5B and 6B.was significantly associated with PHS resistance in single environment and explained 29.03% phenotypic variation.,,andcould express stably in two environments, and the alleles of enhancing PHS resistance were from Chuannong 16. The results of genotype analysis showed that the number of resistant QTL in different lines ranged from one to nine. Six excellent lines in RILs with high resistance carried seven or eight additive QTLs for PHS resistance. These additive QTLs included the minor QTLs on chromosome 4A from Chuanmai 42 and the major QTLs on chromosomes 2D and 5B from Chuannong 16. No. 104 and No. 125 in RIL population were released in China or Sichuan province because of its high yield and PHS resistance, and were named Chuanmai 104 and Chuanmai 64, respectively. Chuanmai 104 showed high yield and good resistance for stripe rust, powdery mildew and PHS in the Sichuan provincial trials and the national trails for Upper and Middle Yangtze River region in 2010 and 2012. The QTL analysis for PHS resistance revealed that Chuanmai 104 carried seven QTLs, including four QTLs on chromosomes 2B, 2D and 5B from Chuannong 16 and three QTLs on chromosomes 4A and 6B from another parent Chuanmai 42. The pyramiding of these additive QTL alleles from each parent led directly to the character of high PHS resistance in Chuanmai 104. In recent years, Chuanmai 104 was widely used to wheat improvement in Southwest China, and 18 wheat varieties (lines) have been bred. 【】Eleven QTLs for PHS resistance, including three QTLs from Chuanmai 42 and eight QTLs from chuannong16, were detected in this study. Four to nine resistant QTLs were generally carried by the resistant lines in RIL population. Two pyramiding lines (Chuanmai 104 and Chuanmai 64) with high PHS resistance carried seven resistant QTLs.
wheat; pre-harvest sprouting (PHS); QTL mapping; QTL pyramiding; pyramiding effect

10.3864/j.issn.0578-1752.2020.17.001
2019-10-21;
2020-02-08
国家小麦产业技术体系(CARS-03)、四川省科技计划(2016NYZ0049)、四川省财政创新能力提升工程(2016ZYPZ-016)
王琴,E-mail:wq860728@163.com。通信作者李俊,E-mail:lijunchd@126.com。通信作者杨武云,E-mail:yangwuyun@126.com
(责任编辑 李莉)

