植物CDF转录因子功能研究进展
许达为 鲍恩财 邹佳宁 曹凯

摘要:CDF(cycling dof factors)蛋白是植物DOF家族中一类特有的转录因子,在植物生长发育中扮演重要角色。CDF蛋白在N-末端有一个由52个氨基酸残基组成的高度保守的C2-C2单锌指结构域,能够特异性地识别植物启动子序列中的AAAG/CTTT作用元件;在C-末端包含有21、22和33个氨基酸组成的3个保守序列,这是与其他蛋白发生相互作用的关键位点。研究发现,CDF基因具有生物钟节律且表达量受光周期调控,其编码的蛋白在调控植物生长发育和响应逆境胁迫方面起着重要作用,就CDF转录因子的序列特点和开花、产量以及响应非生物胁迫方面的功能进行了综述。
关键词:CDF转录因子;开花;光合作用;产量;非生物胁迫
中图分类号:S184 文献标志码: A
文章编号:1002-1302(2020)14-0012-05
植物中基因转录水平的调控在许多生物学进程中发挥着极其重要的作用,比如细胞的形态建成、信号跨膜转导和环境胁迫响应等[1]。DOF(DNA binding with one finger)家族是植物特有的一类转录因子,分别通过对拟南芥、水稻、大麦、短柄草属的36、30、26、27个DOF蛋白进行系统分类,根据C-末端的转录调控结构域以及其内含子-外显子结构,将它们分为A~D 4个亚族[2-3]。其中,CDF(cycling dof factors)蛋白位于进化树的D族,编码该蛋白基因的转录水平受到光周期的调控[4],以下对CDF转录因子的结构与功能进行综述。
1 CDF转录因子的结构
CDF转录因子N-末端包含由52个保守的氨基酸残基组成的C2-C2型单锌指结构域,可以与目标基因启动子的AAAG/CTTT序列特异性结合[5]。AAAG/CTTT序列任意位点的突变都会导致CDF蛋白丧失对目的基因的结合能力[6-8]。单锌指结构域中有4个绝对保守的Cys残基与1个Zn2+共价结合,Zn2+和Cys残基是CDF蛋白保持活性所必需的,二价离子螯合剂的存在和Cys残基的替换都会导致CDF蛋白失活[6-9]。CDF转录因子C-末端区域包含21、22和33个氨基酸组成的3个保守序列(图1),是与F-BOX蛋白FKF1和LKP2的 C- 末端KELCH重复域相互作用的关键结构域[10]。另外,这3个保守序列也被发现在其他物种的同源蛋白质中,例如麻疯树JcDOF3,二穗短柄草BdDOF4、BdDOF11、BdDOF16、BdDOF20和BdDOF22以及马铃薯StCDF1[3,11-12]。这3个鉴定的C-末端保守序列是CDF蛋白的共同特征,其生物学功能需要进一步研究确定。
2 CDF转录因子的生物学功能
CDF蛋白由于其富含Cys残基的单锌指保守结构域以及C-末端3个保守序列元件,导致其功能的多样性。CDF蛋白不仅可以发生蛋白质之间的相互作用,还可以与植物大多数特异性启动子结合,调控其表达。大量研究表明,CDF转录因子在响应光周期控制开花、提升产量及抵御非生物胁迫方面发挥着重要作用[13-15]。
2.1 CDF转录因子对植物开花的影响
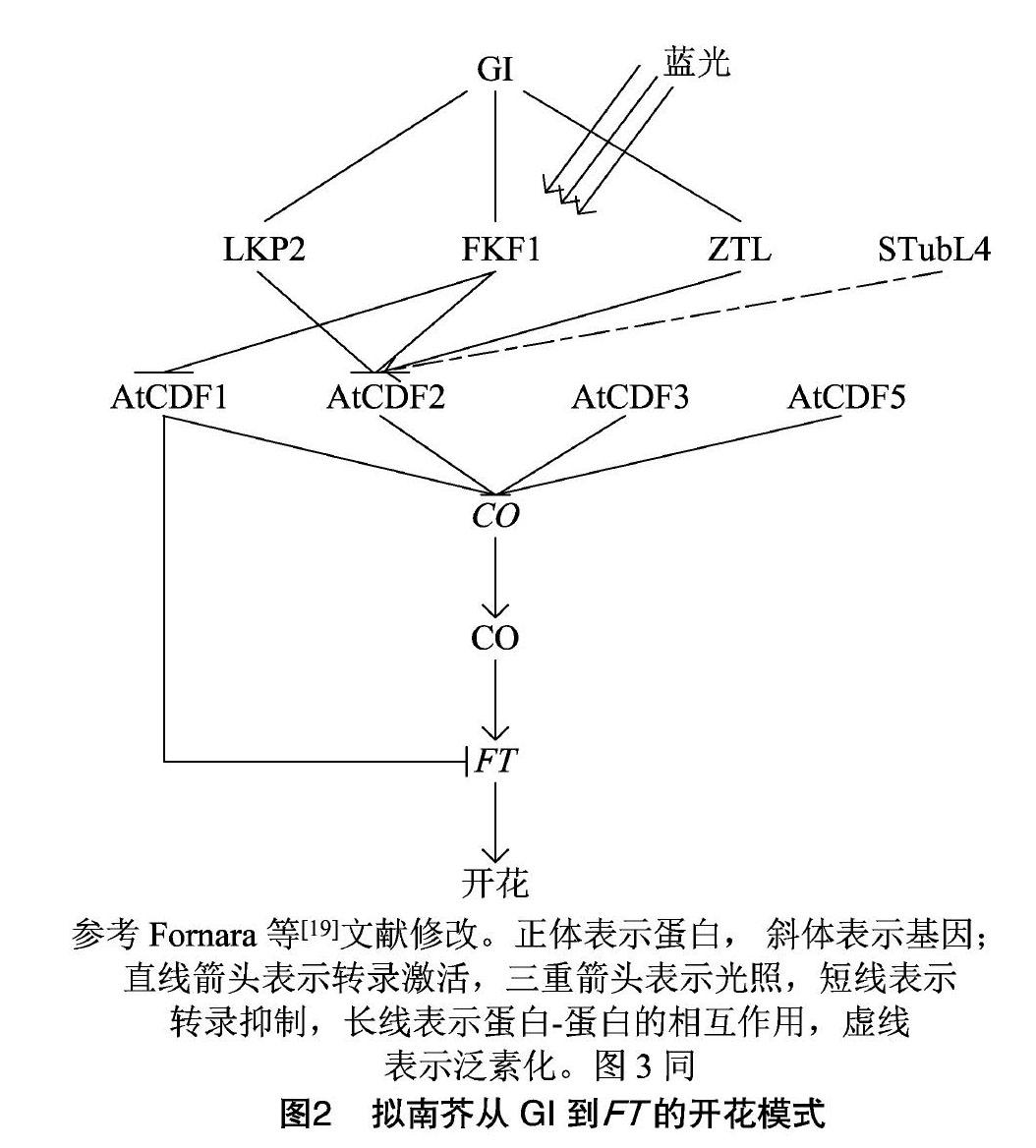
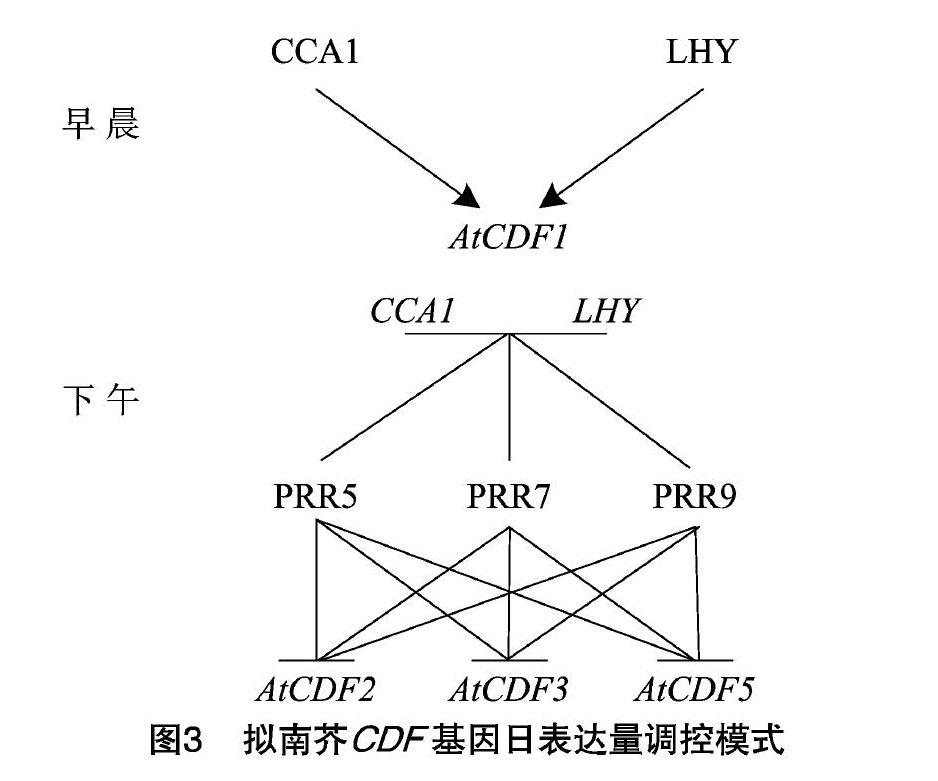
对开花植物而言,从营养生长到生殖生长是植物生命周期的重要阶段。大量研究揭示了模式植物拟南芥開花分子调控机制,其开花时间在很大程度上取决于FT(flowering基因编码的成花素含量。长日照条件能诱导拟南芥FT基因高水平表达,缩短开花时间,短日照条件下则相反[16]。同时,FT基因的表达水平受转录激活因子CO(CONSTANS)和转录抑制因子AtCDF的调控[4,10,17-18]。在蓝光诱导的条件下,FKF1(FLAVIN-BINDING、KELCH REPEAT F-BOX 1)和GI(GIGANTEA)形成稳定的复合物,降解AtCDF1和AtCDF2蛋白,从而解除了对CO基因的转录抑制,导致提前开花。有趣的是,AtCDF1还可以直接和FT基因启动子结合,延迟开花(图2)[4,10,13,18]。CDF基因日表达量变化模式是控制开花时间的关键,在拟南芥中存在多个生物钟因子来调控AtCDF基因的表达[19-23]。2个与光照相关的Myb转录因子分别为CCA1(CIRCADIAN CLOCK ASSOCIATED 1)和LHY(LATEELONGATED HYPOCOTYL)在早晨诱导AtCDF1的表达[4,10,24]。同时,AtCDF1在早晨表达变化模式是拟南芥区分日长的关键,例如拟南芥atcdf1235突变体对光周期不敏感[13]。AtCDF基因的转录水平在下午受转录阻遏蛋白PRR(PSEUDO-RESPONSEREGULATOR)家族的调控,其中PRR5、PRR7和PRR9蛋白结合至少3种AtCDF(AtCDF2、AtCDF3和AtCDF5)基因的启动子,CCA1和LHY基因的表达也受到PRR蛋白抑制(图3)[22-23,25]。
其他开花植物中也发现了类似的CDF转录因子调控开花的分子机制。如在日中性植物番茄中,短日照条件下稍早开花[26]。光照影响番茄开花时间这个过程被认为是通过内部同化竞争来控制[27-28]。最近的研究发现,在番茄中有5个SlCDFs基因,分别为SlCDF1、SlCDF2、SlCDF3、SlCDF4、SlCDF5,它们的表达量受光周期影响,在长日照条件和全日照条件下产生了2种表达模式。拟南芥中过表达SlCDF3导致长日照条件下延迟开花,且TCOL(CO同源蛋白)转录因子和FT-like蛋白(FT同源蛋白)的mRNA水平降低,与拟南芥AtCDF1功能相似[29]。短日照植物水稻OsDof12基因是AtCDF的同源基因,受昼夜节律调控。水稻中过表达OsDof12基因在长日照条件下提前开花,且Hd3a(heading date3a,FT[CM(11*5]同源基因)的表达量明显升高[30-31]。OsDof4与OsDof12同处于一个亚族,水稻中过表达OsDof4导致其在长日照条件下提前开花,在短日条件下延迟开花,同时长日照条件下Hd3a、RFT1和Ehd1的表达量明显升高,与短日照条件下相反[32]。另外,在其他物种的同源蛋白质中也发现相似的变化规律,如甘蓝型油菜中的BnCDF1;小立碗藓中的PpDof3、PpDof4;麻风树中的JcDof1、JcDof3[33-35]。CDF蛋白作为转录因子可以直接或间接地调控FT基因的表达,但大多数植物调控开花的分子机制尚不清楚。
2.2 CDF转录因子对植物光合和产量的影响
21世纪以来,通过改善农业生产方式来提升产量效果不显著,而提升光合作用效率和经济系数逐渐成为增产的主要方式[36]。研究发现,CDF基因过表达植株在提升光合效率,协调碳氮平衡,促进植物生长,提升单株果数和单果质量等多方面发挥着重要作用,为应用转基因手段提升作物产量提供了思路[6,37]。
过表达番茄SlCDF3基因的拟南芥叶片中,花瓣、心皮和长角果明显增大,进一步代谢分析显示,蔗糖水平升高,氨基酸变化程度显著。这表明SlCDF3在增加氮同化方面与之前报道的其他DOF转录因子作用结果一致[6]。过表达SlCDF3的番茄显示出更高的气孔导度,增加了叶肉细胞中CO2的扩散速率,同时表现出更高的PSⅡ的有效量子产率和赤霉素(GA)活性[14],前者通过PSII的电子传递,提升了Rubisco的羧化率,最终优化了过表达植株的光合效率,后者会诱导纤维素合成酶和膨胀素的合成,参与细胞伸长生长,高活性的GA还可以增加番茄坐果量,促进早期果实发育,代谢物分析显示SlCDF3能诱导番茄糖分组成和有机酸含量的变化,改善果实风味与品质[38-40],在拟南芥中过表达AtCDF也观察到类似现象[13]。此外,水稻osdof12突变体谷粒的大小和千粒质量明显减小,马铃薯stcdf1.2和stcdf1.3突变体C-末端受损,块茎发育和植株成熟度显著下降[12,31]。综上所述,CDF蛋白可以通过增加气孔导度,提升光合效率,控制碳氮和激素代谢平衡,最终影响植株的产量品质。同时通过调控下游一系列生长发育相关基因的表达,为转基因育种提供了理论支持。
2.3 CDF转录因子的非生物胁迫响应
非生物胁迫如干旱、盐碱和极端温度是限制植物生长、发育和产量的重要环境因素之一。越来越多的研究发现,CDF转录因子参与诱导应激响应基因的表达,积累渗透调节物质,维持碳/氮平衡和内源激素平衡,在响应非生物胁迫、提高作物应激耐受能力方面发挥着重要作用。
2.3.1 诱导应激响应基因表达及内源激素平衡
拟南芥突变体atcdf3-1对干旱和低温胁迫更敏感,而AtCDF3过表达植株在遭受干旱、寒冷和渗透胁迫时的耐受性明显增强。进一步研究发现,AtCDF3在植物响应极端温度、干旱和渗透胁迫时调控一系列非生物胁迫响应基因的表达如CBFs、DREB2A和ZAT12[29]。同时AtCDF3的功能在空间上可能是分开的,例如在韧皮部伴胞中控制开花时间,在其他部位与不同的转录因子结合响应非生物胁迫[14]。拟南芥atcdf1235突变体表现出更高的氧化应激敏感性[13]。番茄SlCDF3过表达植株上调了参与生长和胁迫的基因PIF1、MYB44的表达水平,使其在盐胁迫下保持较高的光合速率,过表达番茄SlCDF1和SlCDF3的拟南芥中也发现类似现象[14-15]。过表达甘蓝型油菜BnCDF1的拟南芥中发现非生物胁迫响应基因CBF1、CBF2、COR15A和RD29A的表达量上升,耐寒性也显著增加。上述研究指出,CDF蛋白在接受外界胁迫环境信号之后,作为转录因子直接或间接地调控非生物胁迫响应基因的表达。
植物在分子水平上响应逆境胁迫的明显变化之一,就是积累大量作为信息传递媒介的胁迫激素。其中调节植物抵御非生物胁迫的主要激素有ABA(脱落酸)、ET(乙烯)、JA(茉莉酸)、SA(水杨酸)、GA(赤霉素)[41-45]。在过表达AtCDF3的番茄中,活性GA的含量明显增加[14]。并且在盐胁迫条件下IAA和GA含量降低,ABA增加趋势显著,为盐胁迫条件下番茄的产量与品质提供保证。CDF蛋白提升植物胁迫耐受性的主要方式是通过与非生物胁迫相关激素合成途径上关键基因的上游启动子顺式作用元件结合,调控内源激素平衡,提升植物对非生物胁迫的适应能力。
2.3.2 渗透调节物质的积累及维持碳/氮平衡
许多非生物胁迫都能导致植物体的光合速率下降,导致碳同化紊乱,水分亏缺。脯氨酸作为渗透调节物质的含量一定程度反映了植物抵御胁迫的能力[46-47]。过表达AtCDF3显著诱导了与胁迫耐受性和氮同化增加相关的糖和氨基酸的积累,如蔗糖、葡萄糖、L- 亮氨酸、L-天冬酰胺、L-谷氨酰胺、GABA和L-脯氨酸,与之前报道的玉米ZmDOF1[6]功能一致。同时参与糖酵解和糖异生途径的PK(丙酮酸激酶)和PEPCK(PEP羧激酶)活性增强,提升逆境下植物代谢的稳定性。AtCDF3参与植物响应不同的非生物胁迫,根据环境信号的变化调控具体的目的蛋白[15]。在拟南芥中过表达SlCDF3发现同样的规律,例如脯氨酸、谷氨酰胺、GABA和蔗糖水平增加,通过调节pH值和渗透势提升植株的干旱和耐盐性[14]。BnCDF1在冷胁迫条件下脯氨酸含量是野生型拟南芥的1.5倍[33]。CDF蛋白通过诱导糖酵解和三羧酸循环中相关酶的合成代谢,调控植物碳/氮平衡,合成脯氨酸和可溶性糖等一系列渗透调节物质,提升植物抗逆性。
3 结论与展望
在蔬菜栽培中,植株開花的早晚会影响后期产量和果实品质,在大豆、蚕豆和西红花中都得到了验证。CDF蛋白作为编码成花素基因FT-like的上游转录因子,在调控植物开花时间,光合产物源库分配中扮演重要角色。自然环境中旱涝、盐渍、高温、低温等非生物胁迫同样也影响植物的生长发育,并导致减产[48-51]。研究发现,CDF蛋白在冷害、高温、盐碱胁迫下含量迅速上升,通过诱导非生物胁迫响应基因的表达,合成响应胁迫的内源激素,建立新的糖氮代谢平衡,积累脯氨酸等渗透调节物质来提升植物的抗逆性。CDF蛋白在植物转录调控中发挥重要作用,对了解植物生长发育,解决生产实践中的问题有着重要意义。
根据CDF蛋白响应光周期信号的特点,可以利用设施栽培中光周期的可控性,精准调控设施蔬菜的开花成熟时间,可以有效避免极端天气,保证其产量和品质。还可以通过生物工程手段培育CDF转基因植物,改良农作物的抗性,为提高农作物的产量和品质提供新的思路和方法。随着基因组学和生物信息学的发展,越来越多的CDF蛋白将会被发现、克隆及鉴定。目前,对CDF家族的研究仅停留在相关基因和蛋白的层面上,对其如何参与植物生长发育以及在非生物胁迫中发挥的作用机制尚不清楚,还需要进一步的研究阐明。
參考文献:
[1]Riechmann J L,Heard J,Martin G,et al. Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes[J]. Science,2000,290(5499):2105-2110.
[2]Lijavetzky D,Carbonero P,Vicente-Carbajosa J. Genome-wide comparative phylogenetic analysis of the rice and Arabidopsis Dof gene families[J]. BMC Evolutionary Biology,2003,3(1):17.
[3]Hernando-Amado S,Gonzalez-Calle V,Carbonero P,et al. The family of DOF transcription factors in Brachypodium distachyon:phylogenetic comparison with rice and barley DOFs and expression profiling[J]. BMC Plant Biology,2012,12(1):202.
[4]Imaizumi T,Schultz T F,Harmon F G,et al. FKF1F-BOX protein mediates cyclic degradation of a repressor of CONSTANS in Arabidopsis[J]. Science,2005,309(5732):293-297.
[5]Yanagisawa S,Schmidt R J. Diversity and similarity among recognition sequences of Dof transcription factors[J]. Plant Journal,1999,17(2):209-214.
[6]Yanagisawa S. Dof domain proteins:plant-specific transcription factors associated with diverse phenomena unique to plants[J]. Plant and Cell Physiology,2004,45(4):386-391.
[7]Kim H S,Kim S J,Abbasi N,et al. The DOF transcription factor Dof5.1 influences leaf axial patterning by promoting revoluta transcription in Arabidopsis[J]. Plant Journal,2010,64(3):524-535.
[8]Wei P C,Tan F,Gao X Q,et al. Overexpression of AtDOF4.7,an Arabidopsis DOF family transcription factor,induces floral organ abscission deficiency in Arabidopsis[J]. Plant Physiology,2010,153(3):1031-1045.
[9]Yanagisawa S. A novel dna-binding domain that may form a single zinc-finger motif[J]. Nucleic Acids Research,1995,23(17):3403-3410.
[10]Sawa M,Nusinow D A,Kay S A,et al. FKF1 and GIGANTEA complex formation is required for day-length measurement in Arabidopsis[J]. Science,2007,318(5848):261-265.
[11]Yang J,Yang M F,Zhang W P,et al. A putative flowering-time-related Dof transcription factor gene,JcDof3,is controlled by the circadian clock in Jatropha curcas[J]. Plant Science,2011,181(6):667-674.
[12]Kloosterman B,Abelenda J A,Gomez M D C,et al. Naturally occurring allele diversity allows potato cultivation in northern latitudes[J]. Nature,2013,495(7440):246-250.
[13]Fornara F,Panigrahi K C S,Gissot L,et al. Arabidopsis DOF transcription factors act redundantly to reduce CONSTANS expression and are essential for a photoperiodic flowering response[J]. Dev Cell,2009,17(1):75-86.
[14]Renau-Morata B,Molina R V,Carrillo L,et al. Ectopic expression of CDF3 genes in tomato enhances biomass production and yield under salinity stress conditions[J]. Frontiers in Plant Science,2017,8:660.
[15]Corrales A R,Carrillo L,Lasierra P,et al. Multifaceted role of cycling DOF factor 3 (CDF3) in the regulation of flowering time and abiotic stress responses in Arabidopsis[J]. Plant Cell Environ,2017,40(5):748-764.
[16]Kobayashi Y,Kaya H,Goto K,et al. A pair of related genes with antagonistic roles in mediating flowering signals[J]. Science,1999,286(5446):1960-1962.
[17]Valverde F,Mouradov A,Soppe W,et al. Photoreceptor regulation of CONSTANS protein in photoperiodic flowering[J]. Science,2004,303(5660):1003-1006.
[18]Song Y H,Smith R W,To B J,et al. FKF1 conveys timing information for CONSTANS stabilization in photoperiodic flowering[J]. Science,2012,336(6084):1045-1049.
[19]Nakamichi N,Kita M,Niinuma K,et al. Arabidopsis clock-associated pseudo-response regulators PRR9,PRR7 and PRR5 coordinately and positively regulate flowering time through the canonical CONSTANS-dependent photoperiodic pathway[J]. Plant and Cell Physiology,2007,48(6):822-832.
[20]Ito S,Niwa Y,Nakamichi N,et al. Insight into missing genetic links between two evening-expressed pseudo-response regulator genes and PRR5 in the circadian clock-controlled circuitry in Arabidopsis thaliana[J]. Plant and Cell Physiology,2008,49(2):201-213.
[21]Nakamichi N,Kiba T,Henriques R,et al. PSEUDO-RESPONSE REGULATORS 9,7,and 5 are transcriptional repressors in the Arabidopsis circadian clock[J]. Plant Cell,2010,22(3):594-605.
[22]Gendron J M,Pruneda-Paz J L,Doherty C J,et al. Arabidopsis circadian clock protein,TOC1,is a DNA-binding transcription factor[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(8):3167-3172.
[23]Huang W,Perez-Garcia P,Pokhilko A,et al. Mapping the core of the Arabidopsis circadian clock defines the network structure of the oscillator[J]. Science,2012,336(6077):75-79.
[24]Rosas U,Mei Y,Xie Q G,et al. Variation in Arabidopsis flowering time associated with cis-regulatory variation in CONSTANS[J]. Nature Communications,2014,5(1):3651.
[25]Nakamichi N,Kiba T,Kamioka M,et al. Transcriptional repressor PRR5 directly regulates clock-output pathways[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(42):17123-17128.
[26]Kinet J M. Effect of light conditions on the development of the inflorescence in tomato[J]. Scientia Horticulturae,1977,6(1):15-26.
[27]Uzun S. The quantitative effects of temperature and light on the number of leaves preceding the first fruiting inflorescence on the stem of tomato (Lycopersicon esculentum,Mill.) and aubergine (Solanum melongena L.)[J]. Scientia Horticulturae,2006,109(2):142-146.
[28]Dieleman J A,Heuvelink E. Factors affecting the number of leaves preceding the 1st inflorescence in the tomato[J]. Journal of Horticultural Science,1992,67(1):1-10.
[29]Corrales A R,Nebauer S G,Carrillo L,et al. Characterization of tomato cycling dof factors reveals conserved and new functions in the control of flowering time and abiotic stress responses[J]. J Exp Bot,2014,65(4):995-1012.
[30]Iwamoto M,Higo K,Takano M. Circadian clock- and phytochrome-regulated Dof-like gene, is associated with grain size in rice[J]. Plant Cell Environ,2009,32(5):592-603.
[31]Li D J,Yang C H,Li X B,et al. Functional characterization of rice OsDof12[J]. Planta,2009,229(6):1159-1169.
[32]Wu Q,Liu X,Yin D D,et al. Constitutive expression of OsDof4,encoding a C2-C2 zinc finger transcription factor,confesses its distinct flowering effects under long- and short-day photoperiods in rice (Oryza sativa L.)[J]. BMC Plant Biology,2017,17(1):166.
[33]Xu J Y,Dai H B. Brassica napus cycling dof factor1 (BnCDF1) is involved in flowering time and freezing tolerance[J]. Plant Growth Regulation,2016,80(3):315-322.
[34]Ishida T,Sugiyama T,Tabei N,et al. Diurnal expression of CONSTANS-like genes is independent of the function of cycling DOF factor (CDF)-like transcriptional repressors in Physcomitrella patens[J]. Plant Biotechnology,2014,31(4):293-U125.
[35]Yang J,Yang M F,Wang D,et al. JcDof1,a Dof transcription factor gene,is associated with the light-mediated circadian clock in Jatropha curcas[J]. Physiologia Plantarum,2010,139(3):324-334.
[36]Paul M J,Foyer C H. Sink regulation of photosynthesis[J]. J Exp Bot,2001,52(360):1383-1400.
[37]Saibo N J M,Lourenco T,Oliveira M M. Transcription factors and regulation of photosynthetic and related metabolism under environmental stresses[J]. Annals of Botany,2009,103(4):609-623.
[38]Kordel B,Kutschera U. Effects of gibberellin on cellulose biosynthesis and membrane-associated sucrose synthase activity in pea internodes[J]. Journal of Plant Physiology,2000,156(4):570-573.
[39]Mariotti L,Picciarelli P,Lombardi L,et al. Fruit-set and early fruit growth in tomato are associated with increases in indoleacetic acid,cytokinin,and bioactive gibberellin contents[J]. Journal of Plant Growth Regulation,2011,30(4):405-415.
[40]Ariizumi T,Shinozaki Y,Ezura H. Genes that influence yield in tomato[J]. Breeding Sci,2013,63(1):3-13.
[41]劉娟娟,汪惠丽. 植物中脱落酸对非生物胁迫的耐受性研究进展[J]. 安徽农业科学,2017,45(16):11-12,15.
[42]赵 赫,陈受宜,张劲松. 乙烯信号转导与植物非生物胁迫反应调控研究进展[J]. 生物技术通报,2016,32(10):1-10.
[43]忽雪琦,李东阳,严加坤,等. 干旱胁迫下外源茉莉酸甲酯对玉米幼苗根系吸水的影响[J]. 植物生理学报,2018,54(6):991-998.
[44]刘燕敏,周海燕,王 康,等. 植物对非生物胁迫的响应机制研究[J]. 安徽农业科学,2018,46(16):35-37,62.
[45]牛亞利,赵 芊,张肖晗,等. 赤霉素信号在非生物胁迫中的作用及其调控机制研究进展[J]. 生物技术通报,2015,31(10):31-37.
[46]田晓艳,刘延吉,郭迎春. 盐胁迫对NHC牧草Na+、K+、Pro、可溶性糖及可溶性蛋白的影响[J]. 草业科学,2008,25(10):34-38.
[47]薛腾笑,任子蓓,任士福. NaCl胁迫对美国金钟连翘生理特性的影响[J]. 江苏农业科学,2018,46(11):104-108.
[48]尤 超,沈 虹,张营营,等. 油桃生理特性对水分胁迫的响应[J]. 江苏农业科学,2018,46(16):98-101.
[49]李学孚,倪智敏,吴月燕,等. 盐胁迫对‘鄞红葡萄光合特性及叶片细胞结构的影响[J]. 生态学报,2015,35(13):4436-4444.
[50]韩光明,蓝家样,陈全求,等. 高温对棉花生殖生长及其生理生化过程的影响[J]. 棉花科学,2018,40(3):12-17.
[51]黄伟超,范宇博,王泳超. 低温胁迫对玉米幼苗抗氧化系统及渗透调节物质的影响[J]. 中国农学通报,2018,34(24):6-12.
收稿日期:2019-07-23
基金项目:国家自然科学基金青年科学基金(编号:31801903);江苏省农业科学院基本业务费(编号:ZX195004)。
作者简介:许达为(1994—),男,山西太原人,硕士,主要从事设施植物生理生态研究。E-mail:13772050385@163.com。
通信作者:曹 凯,博士,助理研究员,主要从事设施植物生理生态研究。E-mail:243706299@qq.com。

