雅浦海沟海参(Synallactidae)肠道微生物多样性及系统进化分析
高志远杨继超刘文凤何培青陈 颢*
(1.自然资源部 第一海洋研究所,山东 青岛266061;2.青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室,山东 青岛266061;3.国家深海基地管理中心,山东 青岛266061)
雅浦海沟位于雅浦海脊和雅浦群岛东侧,南北长650 km,最深可达8 527 m,是典型的超深渊环境。雅浦海沟具有低温、高压、低光/暗、高盐、低溶解氧和寡营养等特征[1-4],这些特征制约着碳、氮、硫等生源要素分布和生物群落组成,形成超深渊环境独特的物质循环和能量流动体系[5]。微生物在沉积环境物质循环和能量传递中具有重要作用,目前已有相关研究揭示了雅浦海沟沉积物中的微生物组成。Zhang等[6]通过宏基因组测序技术发现,雅浦海沟表层沉积物中奇古菌门(48.4%)和γ-变形菌纲(16.9%)占绝对优势;Fu等[7]利用Miseq高通量测序技术发现,在雅浦海沟超深渊深层沉积物中,微生物群落以变形菌门和奇古菌门为主,其中细菌丰度大于古菌丰度;何高阳等[8]通过传统培养方法研究了雅浦海沟沉积物中真菌多样性及其反硝化能力,发现子囊菌门(Ascomycota)和担子菌门(Basidiomycota)中的硝酸盐还原类群占优势。这些微生物类群,通过对不同生物要素转化,对超深渊环境乃至全球生物地球化学循环产生重要影响。近几年,“蛟龙号”载人潜水器在雅浦海沟科学考察中,还观察到海参、海绵、海星和海蛇尾等底栖生物[9],但在极端高压环境中的采样技术限制了这些底栖生物的采集以及进一步研究。
海参属于棘皮动物门(Echinodermata)、海参纲(Holothuroidea),营底栖生活,具有简单的管状消化道,可容纳大量沉积物。海参通过摄取沉积物中的有机物,如细菌、硅藻、大型藻类碎屑和原生动物等[10-11]获得能量。在寡营养海域,表层沉积物缺乏容易被利用的硅藻和大型藻类碎屑,微生物生物量相对较高,可能是海参的直接营养来源,或间接为宿主提供其他途径无法获得的必需营养元素[12-15]。利用脂肪酸生物标志物追踪证明,细菌是日本刺参(Apostichopusjaponicus)最主要的食物来源,占其能量需求量的70%以上[16]。丁斯予等[17]利用Miseq高通量测序技术发现黄海刺参(Apostichopusjaponicus)肠道微生物群落结构与渤海的不同;陆振[18]利用MiSeq高通量测序技术,也发现大连、烟台、霞浦和莆田地区,养殖仿刺参(Apostichopusjaponicus)肠道微生物丰度和优势类群存在较大差异;王轶南等[19]的研究表明,养殖海参肠道中的微生物群落结构与其栖息沉积环境中的微生物群落结构具有相似之处[20]。上述研究说明海参肠道微生物组成反映了海参摄食途径、沉积物营养水平和栖息环境。
雅浦海沟具有“V”形构造(越靠近底部越陡峭),促进了海沟上部颗粒物沿海沟侧壁向下沉降,使海沟底部营养物质汇集,而海沟侧坡营养相对匮乏[21]。这种地形特征,限制海底平原生物向海沟下潜,导致该区域生物量相对较低[22]。郭承秧等[9]研究发现雅浦海沟真光层中的浮游植物、浮游动物与大型底栖生物相比,碳、氮的稳定同位素比率(‰)(δ13C和δ15N)差异超过了1个营养级,这说明作为底栖生物初始食物来源的浮游植物和浮游动物在向下输送的过程中,经历了食物链传递和细菌的降解过程。由于雅浦海沟底栖生物的营养来源相对匮乏,微生物可能是主要、直接的能量来源。而目前雅浦海沟海参及其肠道微生物研究未见报道。
2017-06-13,我国载人深潜器“蛟龙”号在执行38航次152潜次雅浦海沟科学考察中,于6 622 m海底捕获一只海参。本研究利用MiSeq高通量测序技术对其肠道微生物多样性及系统进化进行了分析,研究结果可为超深渊环境物质能量循环及生命进化提供理论参考。
1 材料与方法
1.1 样品采集
2017-06-13“蛟龙”号深潜器在雅浦海沟(137°84′E,8°30′N)水深6 622 m 处海域进行采集(图1a),水温1.7℃,采集后获得海参,回收上船时由于压力的变化,体壁已爆裂,仅有完整的肠道和部分体壁组织(图1b)。将回收样本直接置于液氮中,后转移至-80℃冰箱保存。养殖海参于2018-12在青岛黄海海域的黄岛滨海海参养殖场采集(120°18′E,35°97′N),采集海域水深2 m,水温10℃;采集样品大小均匀、色泽正常、体质健康且体表无损伤(图1c);样品采集后直接置于液氮中,后转移至-80℃冰箱保存。

图1 雅浦海沟海参和黄海养殖海参Fig.1 Sea cucumber from the Yap Trench and from aquaculture in the Yellow Sea
1.2 样品处理
在实验室采用无菌生理盐水(0.9%)分别冲洗雅浦海沟海参(YP-SPE)体壁和黄海养殖海参(BY)表皮后,采用无菌解剖刀剖开海参体壁,将体壁组织置于无菌离心管中,用于提取海参基因组。取雅浦海沟海参和2只黄海养殖海参的肠道内容物,分别编号为YP-SPE-1,B5-A1和B6-B1,用于提取肠道微生物基因组。
1.3 海参基因组提取及基因扩增
分别取海参体壁组织30 mg,使用海洋动物DNA提取试剂盒(天根,北京)提取总DNA,采用紫外可见分光光度计(BioSpectrometer®basic,Eppendorf)检测DNA浓度和纯度,使用1%琼脂糖凝胶电泳检测DNA片段大小和提取的完整性。分别利用16SA-F/16SB-R[23]引物和COIe-F/COIe-R[24]引物对16S核糖体小亚基基因(16S rDNA)和细胞色素氧化酶亚基I基因(COI)进行PCR扩增。PCR反应采用的DNA聚合酶为TransStartTMTaq DNA Polymerase(全式金,北京),PCR扩增程序为:95℃预变性60 s;95℃变性30 s,50℃退火30 s,72℃延伸80 s,40个循环;72℃延伸10 min[25]。PCR产物由上海美吉生物医药科技有限公司完成测序。
使用DNA Star软件中的Clustal W将基因扩增片段与GenBank中近缘序列进行多重比对,使用MEGA 6.0软件的相邻连接法(Neighbor-Joining)构建系统发育树,自举值设为1 000。将测序原始序列提交到NCBI,获得序列号为:MH572268,MK002697,MN401255和MN427946。
1.4 肠道细菌、古菌16S rDNA基因扩增
分别取肠道内容物200 mg,使用粪便DNA提取试剂盒(天根,北京)提取总DNA,采用紫外可见分光光度计(BioSpectrometer®basic,Eppendorf)检测DNA浓度和纯度,采用1%琼脂糖凝胶电泳检测DNA的片段大小和提取的完整性。利用338F/806R[26]引物对细菌16S r DNA的V3-V4可变区域进行PCR扩增,PCR反应体系参考文献[27],扩增程序为:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸30 s,27个循环;72℃延伸10 min。利用524F/958R[28]引物对古菌16S r DNA的V4-V5可变区域进行PCR扩增,PCR反应体系与扩增程序参考文献[28]。通过Illumina公司MiseqPE300平台进行测序(上海美吉生物医药科技有限公司)。原始数据上传至NCBI数据库中(序列号:SAMN13705046~SAMN13705049)。本研究所有扩增引物序列见表1。

表1 扩增引物序列Table 1 Sequences of primers
1.5 数据处理
原始测序序列使用Trimmomatic软件质控,FLASH软件进行拼接:1)设置50 bp的窗口,如果窗口内的平均质量值低于20,需窗口前端位置截去该碱基后端所有序列,再去除质控后长度低于50 bp的序列;2)根据重叠碱基将两端序列进行拼接,拼接时重叠碱基之间的最大错配率为0.2,长度需大于10 bp;3)根据序列首尾两端的标签和引物将序列拆分至每个样本,标签需精确匹配,引物允许2个碱基的错配,去除存在模糊碱基的序列。使用UPARSE软件(version7.1,http:∥drive5.com/uparse/),根据97%的相似度对序列进行分类操作单元(Operational Taxonomic Unit,OTU)聚类,在聚类的过程中去除单序列和嵌合体。利用RDP classifier(Ribosomal Database Project classifier,http:∥rdp.cme.msu.edu/)对OTUs里的每一条代表序列(默认丰度最高)进行物种分类注释,比对Silva数据库(SSU123),设置比对阈值为70%。基于分类结果统计各样品中微生物类群相对丰度。
1.6 Alpha多样性分析
使用Mothur软件分析序列的覆盖率和Alpha多样性。Alpha多样性包括:丰富度实际观测值(the Observed Richness,Sobs)、ACE指数(ACE Index)、Chao1指数(Chao1 Index)、辛普森指数(Simpson Index)和香农指数(Shannon Index),其中香农和辛普森指数可估计样本群落多样性,ACE指数和Chao1指数可估计样本群落丰富度。
2 结果与分析
2.1 海参的分子鉴定和系统进化树分析
通过引物扩增获得了458 bp的16S r DNA基因片段和558 bp的COI基因片段。系统发育分析的结果表明,基于16S rDNA基因序列片段(图2),雅浦海沟海参(YP-SPE)与来自深海环境的海参聚为一个分支,并与辛那参科(Synallactidae)的粗拟刺参(Pseudostichopusvillosus)的进化关系最近(相似度为99.1%),其次为Molpadiodemasinvolutus(相似度98.2%);黄海养殖海参(BY)与近海养殖场的海参聚为1个分支,亲缘关系较近,其中与同仿刺参(Apostichopusjaponicus)进化关系最近,相似度高于99.4%。基于COI基因序列片段(图3),雅浦海沟海参(YP-SPE)与低温高压深海环境的辛那参科(Synallactidae)的海参聚为一个分支,与Molpadiodemasmorbillus的进化关系最近,相似度为96.4%,与雅浦海沟海参亲缘关系较近的其它2个分支中也均来自于深海环境;养殖海参(BY)与4只近海养殖海参聚为1个大分支(图3)。综合考虑,本研究将采集的雅浦海沟海参鉴定为辛那参科(Synallactidae)。雅浦海沟海参的16S r DNA和COI基因序列与黄海养殖海参的相似度不高,分别为79.8%和92.3%,说明它们的进化关系差异与基因序列差异有关。

图2 基于海参16S r DNA基因片段的系统进化树Fig.2 Molecular phylogenetic tree of sea cucumbers based on 16S r DNA gene sequences

图3 基于海参COI基因片段的系统进化树Fig.3 Molecular phylogenetic tree of sea cucumbers based on COI gene sequences
2.2 海参肠道微生物多样性
2.2.1 海参肠道微生物16S r DNA测序结果
雅浦海沟海参和2只黄海养殖海参肠道的细菌16S r DNA的原始序列为41 575~50 733条,古菌为90 102条。经质量控制、去除单序列和嵌合体之后,获得有效序列分别为35 935~42 405条和78 883条。有效序列百分比均达80%以上,达到后续微生物多样性分析的要求。雅浦海沟海参和黄海养殖海参肠道细菌16S r DNA序列的平均长度分别为459.6,420.0和420.7 bp;古菌16S r DNA序列的平均长度为448.2 bp。
2.2.2 海参肠道微生物的Alpha多样性
雅浦海沟海参和黄海养殖海参肠道微生物测序覆盖率为99.7%~99.9%,表明测序结果代表样本真实情况。在OTU水平上(97%),2个采样区域的海参肠道微生物测序稀释曲线均趋于平坦,说明测序深度足以反映微生物多样性。雅浦海沟海参肠道细菌的香农指数、ACE指数、Chao1指数和辛普森指数均比养殖海参肠道细菌的低,表明雅浦海沟海参较养殖海参的肠道细菌多样性和菌群丰度低(表2)。

表2 雅浦海沟海参和养殖海参肠道细菌及古菌(OTU,97%)Alpha多样性分析Table 2 Alpha diversity analysis of bacteria and archaea(OTU,97%)of sea cucumber intestines from the Yap Trench and the Yellow Sea
2.3 细菌组成
雅浦海沟海参肠道细菌共获得18个门、36个纲、80个目、107个科、140个属和206个种;养殖海参肠道的细菌共获得23个门、48个纲、122个目、157个科、238个属和211个种。
雅浦海沟海参肠道细菌主要为变形菌门(Proteobacteria,55.2%),包括:γ-变形菌纲(Gammaproteobacteria,34.1%),α-变形菌纲(Alphaproteobacteria,16.0%),δ-变形菌纲(Deltaproteobacteria,4.9%)。其次为拟杆菌门(Bacteroidetes,24.3%),浮霉菌状门(Planctomycetes,9.0%),放线菌门(Actinobacteria,3.7%)和硝化螺菌门(Nitrospirae,1.8%)等(图4)。
养殖海参肠道微生物优势类群也为变形菌门(Proteobacteria,平均76.2%),包括:δ-变形菌纲(Deltaproteobacteria,平均28.1%),γ-变形菌纲(Gammaproteobacteria,平均23.7%),α-变形菌纲(Alphaproteobacteria,平均12.1%),ε-变形菌纲(Epsilonbacteraeota,平均13.7%),其次为拟杆菌门(Bacteroidetes,平均9.0%),放线菌门(Actinobacteria,平均4.0%),绿弯菌门(Chloroflexi,平均3.3%)和酸杆菌门(Acidobacteria,平均2.0%)(图4)等。变形菌门和拟杆菌门是2个区域海参肠道主要的微生物类群。
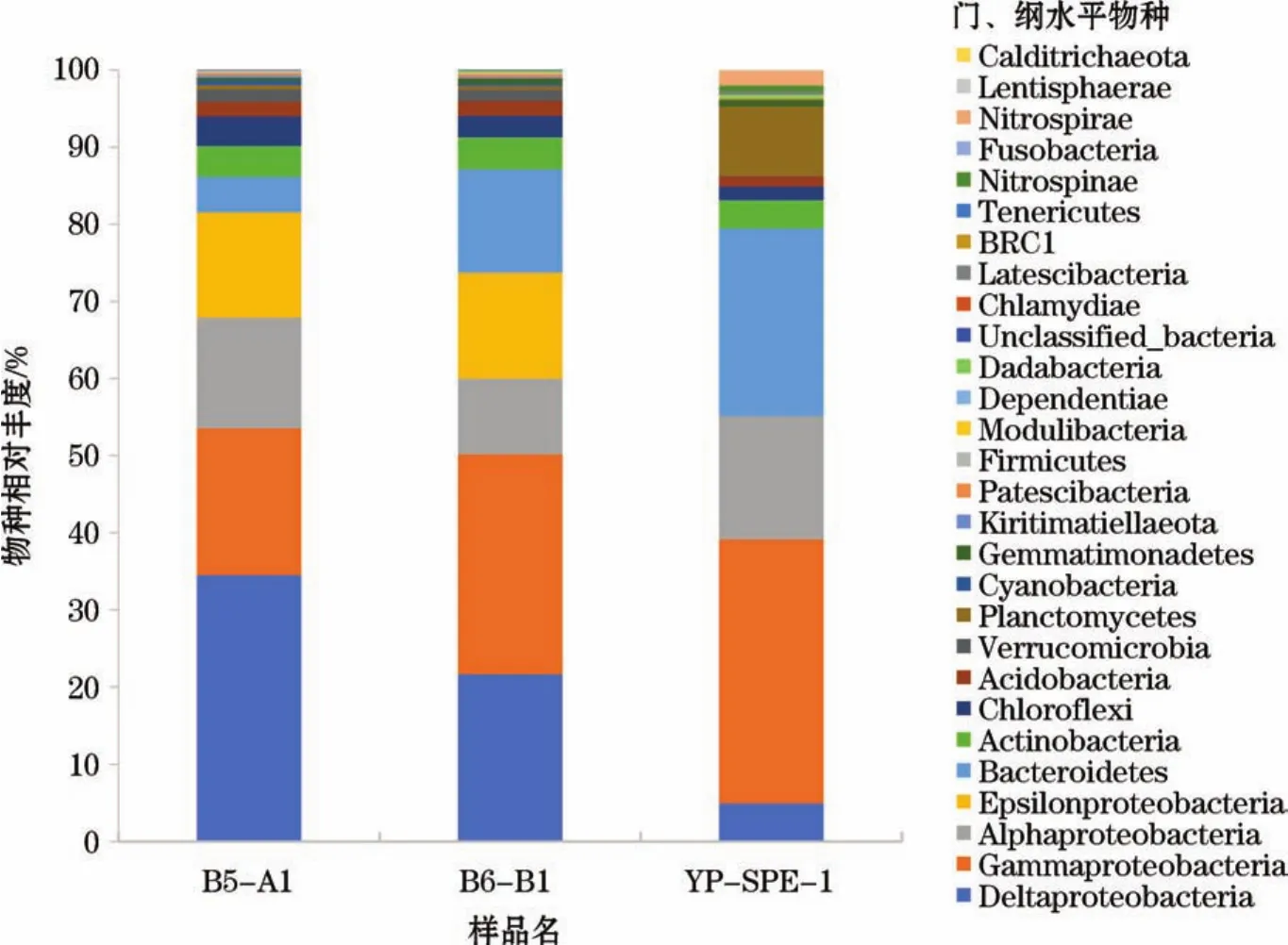
图4 雅浦海沟和黄海海参肠道细菌组成Fig.4 Composition of bacteria of sea cucumber intestines from the Yap Trench and the Yellow Sea
2.3.1 变形菌门(Proteobacteria)
1)γ-变形菌(Gammaproteobacteria)
在雅浦海沟海参肠道微生物中,γ-变形菌纲占绝对优势。γ-变形菌纲也是近海海参肠道中最主要且普遍存在的类群[29-32],在促进养殖环境中的碳、氮循环中具有重要作用[33]。雅浦海沟海参肠道中γ-变形菌的丰度高于黄海养殖海参的,这主要由于β-变形杆菌目(Betaproteobacteriales)的嗜甲基细菌甲基娇养杆菌属(Methylotenera)占总丰度的10.6%。Kalyuhznaya等[34]和Bec等[35]研究发现甲基娇养杆菌属的甲醇代谢与反硝化过程密切相关。通过16S r DNA序列相似度比对和系统发育进化树的构建(图5),发现来源于深海蟹类肠道[36]、海洋河口沉积物[37]以及深海烃渗出沉积物[38]的4株细菌16S r DNA序列与雅浦海沟海参肠道的甲基娇养杆菌属代表菌株(MethyloteneramobilisYP-SPE-1)16S r DNA序列聚为1个进化分支,而与污染环境中7株细菌的16S r DNA序列亲缘关系较远。在β-变形杆菌目中,雅浦海沟海参肠道的亚硝化单胞菌科(Nitrosomonadaceae,1.4%)丰度也高于黄海养殖海参(平均0.03%)的丰度。据报道亚硝化单胞菌目中的微生物可将氨氧化为亚硝酸盐,在氮循环中发挥着重要作用[39]。
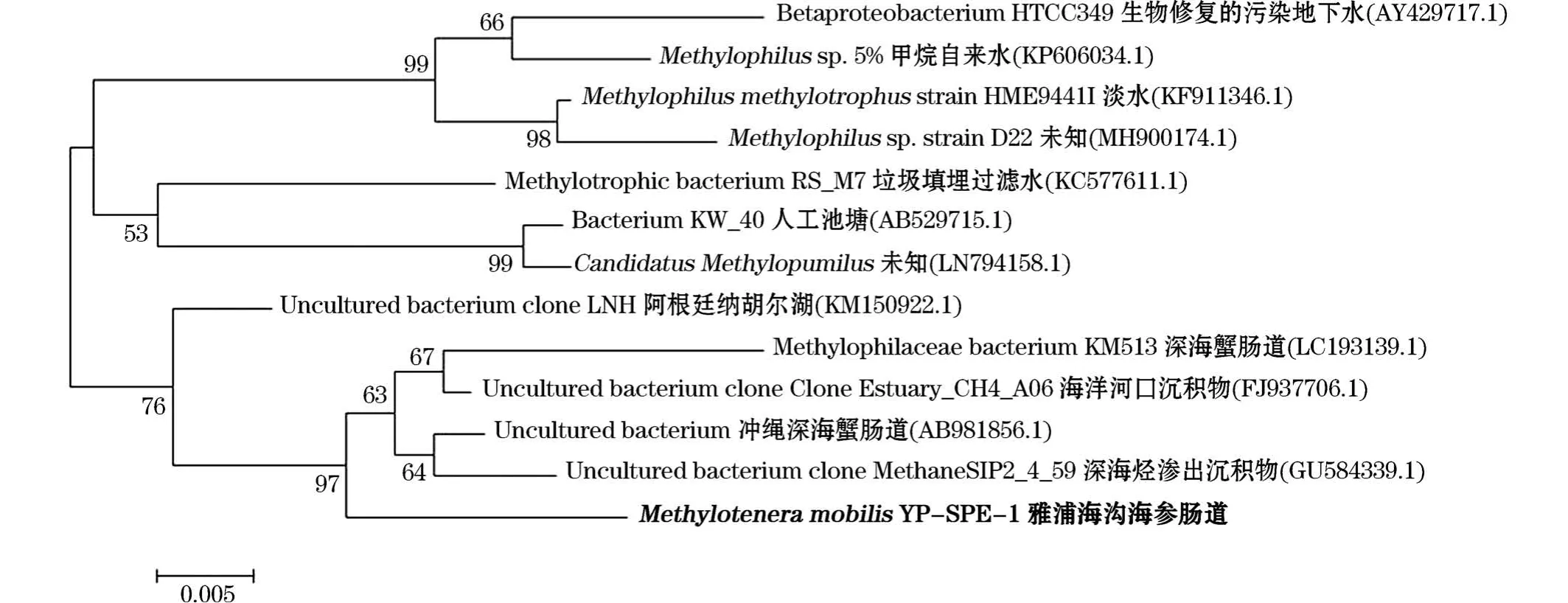
图5 基于Methylotenera 16S rDNA基因片段的系统发育树Fig.5 Phylogenetic tree of Methylotenera based on 16S rDNA gene sequences
γ-变形菌纲中Halieaceae科在黄海养殖海参中丰度最高,平均值为7.3%,主要包括海球菌(Halioglobus,6.2%);在雅浦海沟海参肠道中,仅占0.2%。海球菌也普遍存在于其他养殖海参(如仿刺参)以及海洋沉积物中[12-13]。
2)α-变形菌(Alphaproteobacteria)
雅浦海沟海参肠道的α-变形菌主要包括小棒菌目(Parvibaculales,3.9%),红细杆菌目(Rhodobacterales,3.3%),红弧菌目(Rhodovibrionales,1.9%)和根瘤菌目(Rhizobiales,2.2%);黄海养殖海参肠道的α-变形菌主要为红细杆菌目(Rhodobacterales,7.0%)。
3)δ-变形菌(Deltaproteobacteria)
黄海养殖海参肠道微生物δ-变形菌主要包括脱硫盒菌目(Desulfarculales),脱硫杆菌目(Desulfobacterales),脱硫单胞菌目(Desulfuromonadales)及脱硫弧菌目(Desulfovibrionales)的硫酸盐还原菌,总丰度平均为28.1%,脱硫盒菌目脱硫叶菌属(Desulfobulbus,10.2%)丰度较高;而δ-变形菌在雅浦海沟海参肠道中总丰度平均仅为4.9%。研究表明硫卵形菌属以乳酸、乙醇、丙酮酸及其他脂肪酸为电子受体和能源物质将硫酸盐还原为硫化氢并产生乙酸,属于不完全氧化型的硫酸盐还原菌[40]。
4)ε-变形菌(Epsilonproteobacteria)
黄海养殖海参肠道微生物ε-变形菌平均丰度为13.7%,由硫卵形菌属(Sulfurovum)构成。硫卵形菌属可以单质硫、硫化物和硫代硫酸盐作为电子供体,以硝酸盐作为电子受体,进行化能自养过程[41-42]。在雅浦海沟海参肠道中未发现硫卵形菌属。
2.3.2 拟杆菌门(Bacteroidetes)
Cottrell,Kirchman[43]和Osullivan[44]研究发现拟杆菌门与脂类、蛋白质等有机物质的转化密切相关,在维持宿主肠道稳态方面发挥重要的作用[45]。雅浦海沟海参肠道中拟杆菌门丰度高于黄海养殖海参的拟杆菌门丰度,其中几丁质噬菌体目(Chitinophagales,12.2%)和黄杆菌目(Flavobacteriales,10.7%)为主要类群,可以将氧化二氮还原为氮气,参与反硝化过程[46]。
2.3.3 浮霉菌门(Planctomycetes)
雅浦海沟海参肠道中,浮霉菌门丰度占9.0%,其中浮霉菌纲(Phycisphaerae)为5.4%;在黄海养殖海参肠道中浮霉菌门仅占0.03%。研究表明浮霉菌刚细菌在活性污泥中参与脱氮作用[47]。
2.3.4 放线菌门(Actinobacteria)
放线菌在有机质分解和矿化作用的过程中起重要作用[48],黄海养殖海参肠道微生物的放线菌丰度高于雅浦海沟海参肠道微生物的放线菌丰度,其中沉积岩杆菌属(Ilumatobacter)占绝对优势,且该属也在河口和海滨沙滩分布[49-50],可以利用柠檬酸,葡萄糖,麦芽糖等多种碳源进行生长代谢。
2.3.5 硝化螺菌门(Nitrospirae)和硝化刺菌门(Nitrospinae)
雅浦海沟海参肠道中分布硝化螺菌门(Nitrospirae,1.8%)和硝化刺菌门(Nitrospinae,0.8%),而在黄海养殖海参肠道中未检测到。这2个类群是黑暗海洋中化能自养的亚硝酸盐氧化菌最主要的类群[51-53]。
2.4 微生物古菌组成
雅浦海沟海参肠道古菌共获得奇古菌门、泉古菌门和广古菌门三个门,包括6个纲,9个目,14个科,16个属,18个种。其中,奇古菌门丰度高达98.8%,主要由氨氧化古菌亚硝化侏儒菌属(Nitrosopumilus,95.6%)组成。氨氧化古菌广泛分布于海洋和陆地生境,可以将氨氧化成亚硝酸盐,是黑暗海洋中最丰富的化能自养类群之一[54-58],在全球氮循环中具有重要作用。
本研究从GeneBank中选取雅浦海沟海参肠道亚硝化侏儒菌属代表菌株(NitrosopumilusYP-SPE-1)的16S r DNA近缘序列,并构建系统发育进化树(图6),亚硝化侏儒菌属(NitrosopumilusYP-SPE-1)与来自超深渊或深海沉积物的3株古菌聚为一个小分支;而与非深海海洋环境中4株菌株的亲缘关系较远;来自陆地的3株菌株聚为一个独立分支,位于进化树的基部。

图6 基于Nitrosopumilus 16S rDNA基因片段的系统发育树Fig.6 Phylogenetic tree of Nitrosopumilus based on 16S r DNA gene sequences
3 讨 论
雅浦海沟等超深渊环境具有独特的“V”形地质构造、水动力环境及物质循环和能量流动体系,具有相比其他大洋环境更高的有机碳等生源要素的沉降通量和储碳效率,是海洋初级生产的巨大“捕获器”和有机碳的沉积汇。雅浦海沟还是连接深部生物圈与海洋的窗口和通道,在海洋和全球碳循环中扮演重要角色[5]。研究雅浦海沟超深渊生态系统中物质循环过程、生物形态结构和生态功能将有助于加深人类对超深渊系统的认识。
通过生物的进化关系分析,JAMIESON等[59]发现即使在相邻近的海沟,同种类生物(如,海参和片脚类等)也具有独特的特征,表明不同海域海沟生物群落在地理空间上的特异性[59]。本研究通过构建16S r DNA和COI基因片段的系统发育进化树,分析了雅浦海沟超深渊海参与和养殖海参的系统进化关系。在对雅浦海沟超深渊海参与养殖海参基于16S r DNA和COI基因序列的相似度和系统发育关系对比后发现,超深渊与近海地理隔离造成了物种差异,这种基因序列的差异可能是导致生物形成独特特征的原因。
雅浦海沟微生物的系统进化特征,也反映了地理空间特异性。甲基娇养杆菌系统进化分析表明,甲基娇养杆菌(MethyloteneraYP-SPE-1)与深海环境中的3株甲基娇养杆菌(2株来自深海蟹类肠道)亲缘关系最近,而与人为污染环境中甲基娇养杆菌亲缘关系较远。亚硝化侏儒菌系统进化结果表明,亚硝化侏儒菌(NitrospumilusYP-SPE-1)与深海沉积物的亚硝化侏儒菌属亲缘关系最近,其次为近海来源的亚硝化侏儒菌属,而与陆源的亲缘关系最远。Ren等[60]通过构建了奇古菌系统发育进化树,推断出氨氧化古菌(Ammon-Oxidizing Archaea,AOA)进化的3个阶段:1)起源于陆地上的非AOA的奇古菌;2)在浅海海水定殖;3)向深海海水扩张。本研究系统进化树的研究结果发现,陆地沉积物中的奇古菌位于进化树的基部,进化程度最古老,其次为近海沉积物和海水,进化程度最晚的分支来源于盆地和深海沉积环境。本研究的进化分支与Ren等[60]构建的水环境奇古菌3大进化分支相一致,因此,我们推断沉积环境中的氨氧化古菌进化也遵循从陆地到深海进化的理论,这也证实并补充了Ren等[60]的理论。
海参肠道微生物为宿主提供营养来源[20],也反映了宿主栖息环境的微生物组成[14-15]。雅浦海沟海参肠道古菌中,奇古菌门相对丰度达98.8%,其中,氨氧化古菌亚硝化侏儒菌高达95.6%。Zhang等[6]利用宏基因组测序手段,研究了雅浦海沟微生物群落结构和代谢特性,也发现奇古菌门丰度达41%~49%。由此可见,雅浦海沟海参肠道中的奇古菌门反映了栖息环境氨氧化类群的分布特征以及氨氧化生成亚硝酸盐的化能自养过程。雅浦海沟海参肠道中还发现了硝化螺菌门(Nitrospirae)和硝化刺菌门(Nitrospinae),可通过亚硝酸盐氧化进行碳固定作用。雅浦海沟海参肠道微生物中,化能自养的硝酸盐还原菌甲基娇养杆菌属为γ-变形菌纲的优势类群。在α-变形菌纲、拟杆菌门、浮霉菌门等类群中也发现多种自养和异养的硝酸盐还原菌。何高阳等[8]发现子囊菌门和担子菌门中的硝酸盐还原类群在雅浦海沟沉积物可培养真菌中占优势。本研究未对黄海养殖海参肠道的古菌进行特异性扩增和分析,但雅浦海沟海参肠道微生物宏基因组数据中,亚硝化侏儒菌丰度较高,而在养殖海参肠道微生物宏基因组数据中,并未发现亚硝化侏儒菌序列(未发表数据),说明亚硝化侏儒菌不是养殖海参肠道的主要微生物类群。我们推测,雅浦海沟环境中的化能自养微生物,通过氨氧化、亚硝酸盐氧化和硝酸盐还原作用,完成氮的循环过程,这些化能自养的类群还通过暗固碳作用合成有机物,这可能是寡营养的生存环境中能量的高效获取机制,也在全球氮循环中起着重要作用。这些微生物类群同时为海参提供了食物来源,将物质和能量向更高一级食物链传递。
黄海养殖海参肠道微生物硫酸盐还原菌(δ-变形菌纲)和硫卵形菌属(ε-变形菌纲)的丰度均高于雅浦海沟海参的,这可能反映了近岸富营养的沉积物中硫的循环过程。氮循环和硫循环在超深渊和近海海参肠道微生物中的差异,指示了栖息环境物质循环和能量流动的特征。
4 结 语
本研究首次分析了雅浦海沟海参及其肠道微生物多样性及系统进化特征,为揭示超深渊环境的物质能量循环及生命进化提供理论参考。
雅浦海沟海参及其肠道微生物均与深海环境种类的亲缘关系最近,而与陆栖环境或近海环境的物种亲缘关系远,这反映了超深渊与近海环境地理隔离导致的基因序列及进化选择的差异性。根据亚硝化侏儒菌的16S r DNA进化树,我们推断沉积环境中的氨氧化奇古菌也同海水中的一样,遵循从陆地到深海环境进化的理论。根据肠道微生物种类,推断可知,雅浦海沟中的化能自养微生物,通过氨氧化,亚硝酸盐氧化和反硝化作用,完成氮的循环过程,在全球氮循环中起着重要作用;而近海富营养的沉积环境促进了硫酸盐的还原及硫的氧化过程。未来需要获得更多数量及不同种类的超深渊底栖生物进行深入研究,从而更全面地认识超深渊底栖生物肠道微生物的多样性,及其与极端环境的相互作用机制。

