小立碗藓扩展蛋白基因家族的鉴定与生物信息学分析
蓝雨纯 黄彬 韦娇 姜山


摘 要: 扩展蛋白(Expansins, EXP)是一类基因家族,几乎参与了植物发育的全过程,从种子萌发到果实成熟都有扩展蛋白的参与。该研究利用生物信息学的方法对小立碗藓(Physcomitrella patens)Expansin基因家族成员进行鉴定,分析了其基因结构、染色体定位以及系统发生关系。结果表明:小立碗藓基因组中含有Expansin A(EXPA)32个、Expansin-like A(EXLA)6个,并未发现Expansin-like B(EXLB)及Expansin B(EXPB)。扩展蛋白氨基酸序列长度在228~290 aa之间,编码蛋白质具有两个保守的结构域Pollen_allerg_1和DPBB_1。蛋白质亚细胞定位预测结果表明:运用CELLO在线工具预测发现小立碗藓中约4/5的EXP家族基因定位于细胞外;而Euk-mPLoc预测结果则显示小立碗藓EXP基因家族成员全定位于细胞外。基因结构分析表明,小立碗藓中约68% Expansin 基因有含有1~3个内含子。以上结果可为深入研究小立碗藓扩展蛋白基因的分子进化与生物学功能奠定基础。
关键词: 小立碗藓, 扩展蛋白, 基因家族, 生物信息学
中图分类号: Q943 文献标识码: A
文章编号: 1000-3142(2020)06-0854-10
开放科学(资源服务)标识码(OSID) :
Abstract: Expansins (EXP) is a family of genes that are involved in the whole process of plant development. From seed germination to fruit ripening, there are Expansins involved. Bioinformatics methods were used to identify the members of the Expansin gene family of Physcomitrella patens, and their gene structures, chromosomal locations and phylogenetic relationships were analyzed. The results were as follows: Genome of the P. patens contains 32 Expansin A (EXPA) and 6 Expansin-like A (EXLA), but Expansin-like B (EXLB) and Expansin B (EXPB) were not found. The Expansin amino acid sequence is between 228-290 aa in size, and the encoded protein has two conserved domains, Pollen_allerg_1 and DPBB_1. The results of protein subcellular localization prediction showed that about 4/5 of the EXP family genes in the P. patens were located outside the cell using the CELLO online tool; while the Euk-mPLoc prediction showed that the EXP gene family member was fully localized in extracellular. Gene structure analysis showed that about 68% of the Expansin gene in P. patens contains 1-3 introns. This study analyzed the basic information of the P. patens extended protein gene family, which lays a foundation for further study of the molecular evolution and biological functions of its extended protein genes.
Key words: Physcomitrella patens, Expansin, gene family, bioinformatics
植物细胞壁是植物细胞中是一种极其重要的结构。细胞壁能够决定细胞的形状,为细胞提供基本的机械支撑和硬度,是细胞抗击病原体的最后一道屏障(Cosgrove, 2005)。扩展蛋白,即植物细胞壁松弛蛋白(Expansins, EXP),是一类细胞壁蛋白,主要存在于各植物細胞组织中,通过引起细胞壁组分间的松驰和细胞壁柔韧性增加,参与植物的许多生长发育过程。
在对黄瓜(Cucumic sativus)下胚轴细胞壁酸诱导伸展的研究实验中,Cosgrove et al. (2002)首次发现EXP。实验结果显示:EXP可以使经热钝化后的细胞壁恢复伸展活性,但不具有溶菌酶活性,并且达到几乎和离体细胞壁本身伸展活性相当的程度,以此推测其具有调节植物细胞壁的伸展性的功能(Javier et al., 1992)。实验证实EXP与许多生长发育过程相关,例如:Cosgrove et al. (2002)实验发现果实成熟软化和花粉管延伸有关;Belfield et al. (2005)研究表明EXP与叶片脱落有关;除此之外, Han et al. (2004)研究显示EXP基因在植物抗逆中也有重要作用。早期研究表明,EXP以酶催化的作用方式,使细胞壁组分间疏松,细胞伸展,增强细胞柔韧性,以此缓解细胞在不良环境下的压力(Cosgrove et al., 1997)。近年来,随着基因组测序技术的普及,越来越多的扩展蛋白在其他物种中被鉴定。依据进化关系,可将扩展蛋白基因家族划分为四个亚家族:α、β、γ和δ。如今,科学研究在多种植物的不同生长阶段都发现扩展蛋白存在。α- 和β-expansin家族分别被重命名为EXPA与EXPB,γ-expansin称为Expansin-like A(EXLA)和δ-expansin称为Expansin-like B(EXLB)(金慧清等,2006)。EXPA和EXPB亚家族蛋白具有细胞壁伸展活性,参与细胞壁伸长和其他发育过程(Hans et al., 2004);但EXLA和EXLB两类亚家族仅仅知道其基因序列,暂无相关实验证明EXLA和EXLB两类亚家族蛋白具有增强细胞壁韧性的功能(Sampedro et al., 2006)。
小立碗藓(Physcomitrella patens)是非维管束植物类群的模式植物,进化地位特殊,全基因组也已测序完成,是研究功能基因组学、发育生物学、植物生理、系统进化理想材料。小立碗藓基因组大小为511 Mb,共含27条染色体(Rensing et al., 2002),有着易与外源基因发生同源重组、易培养且生长周期较短、表型易观察等优势。本研究基于2007年小立碗藓的全基因组序列测序工作完成(http: //www.cosmoss.org/)(Rensing et al., 2008),利用生物信息学的方法筛选鉴定小立碗藓扩展蛋白基因家族成员,并对其进行进化树构建,染色体定位、基因结构分析等。为后续研究该基因家族提供了参照。
1 材料与方法
1.1 材料
以模式植物小立碗藓为研究对象,小立碗藓全基因组数据下载于Ensembl Plants数据库(Bolser et al., 2017)(http://plants.ensembl.org/index.html)中最新数据。
1.2 小立碗藓Expansin基因家族的鉴定
由Ensembl数据库(http://asia.ensembl.org/index.html)中获得小立碗藓基因组相关数据;从Pfam数据库(http://pfam.xfam.org/)下载蛋白保守结构域的隐马科夫模型文件(*.hmm)。利用HMMER软件(Finn et al., 2011)中hmmsearch工具搜索小立碗藓全基因组预测蛋白序列中含有Pollen_allerg_1(PF01357)和DPBB_1(PF03330)保守结构域的序列(李昊阳等,2014),筛选E-value < 1 × 10-20的序列作为初筛序列。利用初筛序列及HMMER软件重新构建小立碗藓的特异性hmm文件,并利用该hmm文件再次进行筛选(E-value<0.01),筛选所得序列为备用序列。为保证搜索结果的准确性,通过SMART网站(http://smart.embl-heidelberg.de/)对备用蛋白进行手动筛选(Letunic et al., 2012),以手动剔除不含Pollen_allerg_1和DPBB_1两个保守结构域的序列。
1.3 小立碗藓扩展蛋白基因家族的蛋白特征分析及亚细胞定位
将鉴定得到的小立碗藓扩展蛋白家族蛋白序列,通过NCBI(https://www.ncbi.nlm.nih.gov/)数据库查找其分子量(Mw)、等电点(PI)等数据。通过Cello在线工具(http://cello.life.nctu.edu.tw/)及Euk-mPLoc在线网站(http://www.csbio.sjtu.edu.cn/bioinf/euk-multi-2/)预测小立碗藓EXP基因家族的亚细胞定位。
1.4 小立碗藓Expansin家族系统进化树的构建
利用小立碗藓Expansin基因的蛋白序列,与TAIR据库(https://www.arabidopsis.org/)及NCBI数据库(https://www.ncbi.nlm.nih.gov/)搜索到的拟南芥(Arabidopsis thaliana)Expansin基因蛋白序列,应用多序列比对工具ClustalX构建系统进化树,以氨基酸全序列联配的结果为基础,用MEGA 7.0程序生成。采用程序Maximum Likelihood法,校验参数为Bootstrap=1 000。
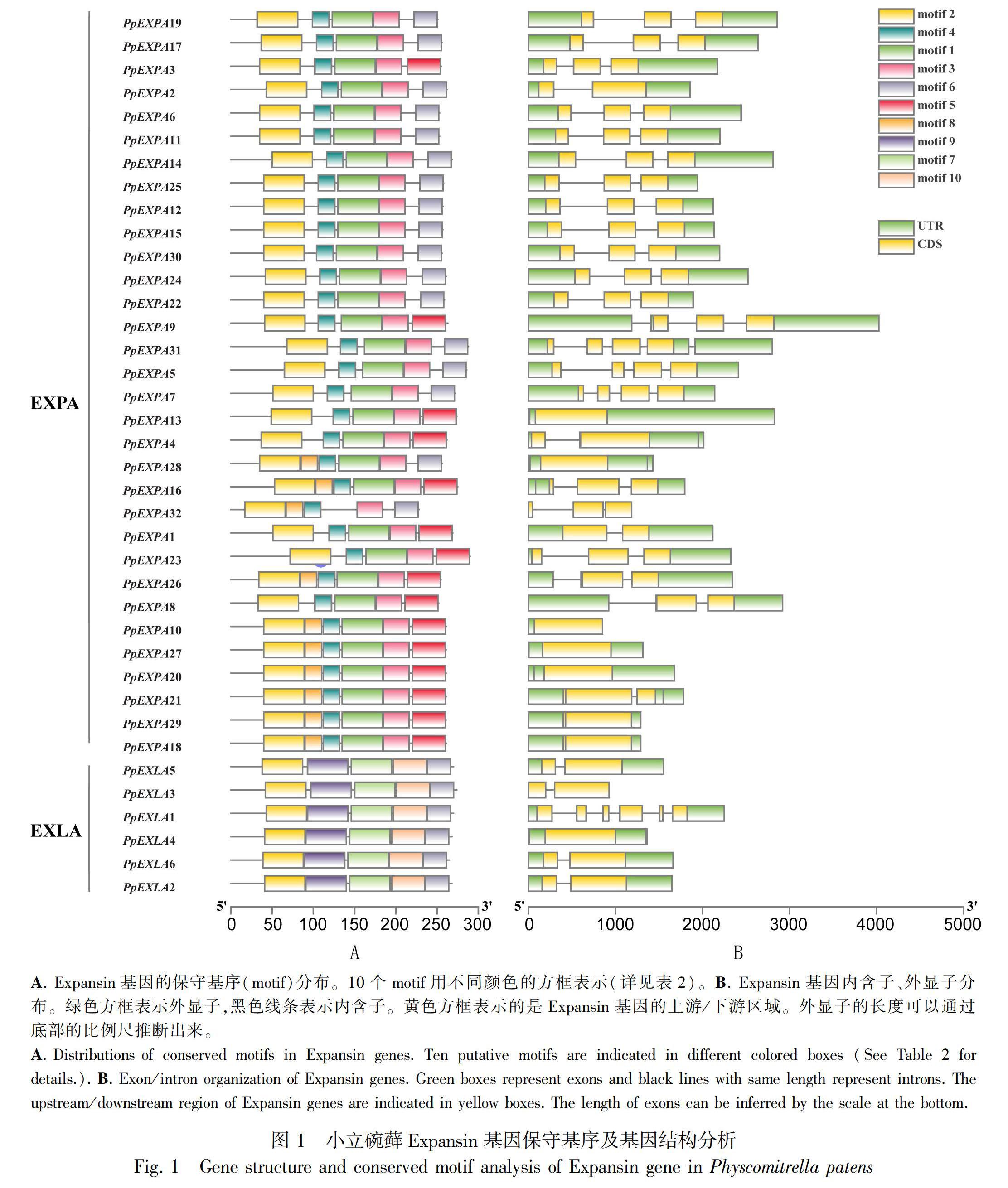
1.5 小立碗藓Expansin基因保守基序及基因结构分析
通过MEME网站检测,小立碗藓Expansin家族基因中所存在相似度较高的基序(motif);并利用基因组注释文件,获得小立碗藓Expansin基因家族的内含子及外显子分布情况;最后利用TBtools软件对其进行可视化分析。
1.6 小立碗藓Expansin基因的染色体位置图
以Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html)提供的相关基因信息, 确定Expansin基因的染色体定位。然后通过MapInspect软件作图(易吉明等,2015)。
2 结果与分析
2.1 小立碗藓 Expansin 基因家族成员信息
通過 HMMER软件对小立碗藓全基因组预测蛋白序列进行搜索,共获得91个备用蛋白。运用SMART 网站手动鉴定备用蛋白后,最终获得38个小立碗藓扩展蛋白(表1)。与双子叶模式植物拟南芥中鉴定的扩展蛋白数量持平(Seader et al., 2016)。
通过蛋白特征分析,小立碗藓38个Expansin基因的分子量为25.14 kDa到73.95 kDa,其中约87%的扩展蛋白分子量处于20~30 kDa,等电点最小为4.14。这些小立碗藓Expansin基因编码的蛋白包含228 ~290个氨基酸。亚细胞定位预测结果显示:运用CELLO在线工具预测发现小立碗藓中约1/5的EXP家族基因定位于细胞周质;而Euk-mPLoc预测结果则显示小立碗藓EXP基因家族全定位于细胞外。Pfam数据库(http://pfam.xfam.org)验证结果显示38个扩展蛋白同时具有Pollen_allerg_1和DPBB_1特征结构域。
2.2 小立碗藓 Expansin 基因保守结构域与基因结构分析
通过MEME分析小立碗藓Expansin基因序列,获得10个保守性较高的motif(图1:A)。同时,motif之间的位置具有重要规律,其中发现小立碗藓EXPA亚家族除PpEXPA32出现motif 1缺失外,该亚家族其余基因具有motif 2-(motif 8)-motif 4-motif 1-motif 3的相对稳定结构;而EXLA亚家族则具有motif 2-motif 9-motif 7-motif 10-motif 6的稳定结构,并未出现motif缺失、增加或替换。
分析38个小立碗藓EXP基因的内含子、外显子结构(图1:B),发现EXPA中约49%的基因含有2个内含子、约1/5的基因仅含有1个内含子、9%的基因含有3个内含子、有约22%的EXPA亚家族基因不含内含子。在分析小立碗藓EXLA亚家族时发现,EXLA几乎仅含1个内含子,除PpEXLA1和PpEXLA4外。其中PpEXLA1含有5个内含子,为Expansin基因家族中含有内含子数目最多的基因。
2.3 小立碗藓扩展蛋白基因家族的系統进化分析
对小立碗藓Expansin基因家族进行了氨基酸多序列比对和系统树构建(图2),分析表明,小立碗藓的Expansin基因家族系统进化树有3个非常明显的分枝,且小立碗藓Expansin基因家族的两个亚基因家族都出现了独立的进化分支。其中EXPA亚家族包含两个进化分支,说明EXPA亚家族的基因在长期的进化过程中,出现了不同的进化方式。另外,小立碗藓中有些Expansin基因分支较长,说明这些基因在很早的时候就发生了分化,相应基因序列也已经发生较大的分化,但仍然可以肯定它们之间具有一定的演化关系。
为研究模式植物小立碗藓与拟南芥基因组中EXP之间的进化关系,利用38个小立碗藓EXP基因和38个拟南芥扩展蛋白的氨基酸序列构建了系统发育树(图3)。从系统发育树可知,小立碗藓38个EXP基因被明显分为2个亚家族。共鉴定种内的直系同源蛋白12对,其中有4对直系同源蛋白间的Bootstrap值为99,而物种内的旁系同源蛋白数目为1。
2.4 小立碗藓EXP基因家族的染色体位置
通过Phytozome数据库获得小立碗藓EXP基因家族染色体定位信息,将小立碗藓中38个扩展蛋白定位在15条染色体上,图4显示小立碗藓第8条、第14条染色体上均定位了6个EXP基因。根据基因簇的定义可发现小立碗藓EXP基因家族在第14条染色体上出现了小规模的基因簇(Bai et al., 2002)。另7条染色体上均只含1个基因,3条染色体含有2个基因,可见小立碗藓38个Expansin基因在染色体上分布是不均匀的。
3 讨论
扩展蛋白是植物细胞壁的重要组分,主要参与细胞扩张以及一系列发生细胞壁修饰的发育过程以及植物抗病的重要生理过程。研究鉴定小立碗藓中含有38个扩展蛋白,可进一步分类为EXPA亚家族及 EXLA亚家族。小立碗藓EXP基因的两个亚家族之间在基因内含子外显子分布、motif结构等方面具有一定的保守性,代表了亚家族的重要特征, 研究发现EXPA亚家族中约70%的基因含有1~ 2个内含子,另有约22%的EXPA亚家族基因不含内含子;而EXLA亚家族几乎仅含1个内含子。基序分析表明,EXPA亚家族及EXLA亚家族均含有 motif 2;EXLA亚家族基因均含有motif 2-motif 9-motif 7-motif 10-motif 6的稳定结构,比EXPA亚家族更具有保守性。同一亚家族内的EXP基因含有保守的内含子与外显子结构和motif特征,而不同亚家族之间motif的多样化也表明了小立碗藓EXP基因在一定程度上参与细胞内的多种代谢途径。通过染色体定位发现,在小立碗藓Expansin基因随机分布15条染色体上,除14号染色体外,基本不形成基因簇。
EXP基因在植物体的生长发育及逆境胁迫中具有重要的生理意义,已成为植物体基因功能研究热点之一,但其在小立碗藓中的基因功能尚不明确。本研究通过对已有数据库数据的分析,使用生物学信息的方法手段,筛选得到38个小立碗藓扩展蛋白基因家族成员,分析其蛋白特征、进化关系、基因结构及染色体定位等。从基因水平上展示小立碗藓种属的特征,为后续研究扩展蛋白基因功能提供了理论依据。
参考文献:
BAI J, PENNILL LA, NING JC, et al., 2002. Diversity in nucleotide binding site-leucine-rich repeat genes in cereals [J]. Cytogenet Genom Res, 12(12): 1871-1884.
BOLSER DM, RUQURTI B, ROBERTS J, et al., 2017. Ensembl plants: Integrating tools for visualizing, mining, and analyzing plant genomic data [J]. Methods Mol Biol, 1533: 1-31.
BEDINGER P, DURACHKO DM, 1997. Group I allergens of grass pollen as cell wall-loosening agents [J]. Proc Natl Acad Sci USA, 94(12): 6559-6564.
COSGROVE DJ, 2000. Loosening of plant cell walls by expansins [J]. Nature, 407(6802): 321-326.
COSGROVE DJ, 2005. Growth of the plant cell wall [J]. Nat Rev Mol Cell Biol, 6(11): 850-861.
COSGROVE DJ, LI LC, CHO HT, et al., 2002. The growing world of expansins [J]. Plant Cell Physiol, 43(12): 1436-1444.
ERIC JB, BENEDETTO R, JEREMY AR, et al., 2005. Changes in expansin activity and gene expression during ethylene-promoted leaflet abscission in Sambucus nigra [J]. J Exp Bot, 56(413): 817-823.
HAN YY, LI AX, LI F, et al., 2012. Characterization of a wheat (Triticum aestivum L.) expansin gene, TaEXPB23, involved in the abiotic stress response and phytohormone regulation [J]. Plant Physiol Biochem, 54: 49-58.
HANS K, KENT JB, DAVID AB, et al., 2004. Nomenclature for members of the expansin superfamily of genes and proteins [J]. Plant Mol Biol, 55(3): 311-314.
IVICA L, TOBIAS D, PEER B, 2012. SMART 7: Recent updates to the protein domain annotation resource [J]. Nucl Acids Res, 40(Database issue): 302-305.
JAVIER S, ROBERT EC, DANIEL JC, 1992. Two endogenous proteins that induce cell wall extension in plants [J]. Plant cell, 4(undefined): 1425-1433.
JIN HQ, CHEN YH, JIN YF, 2006. The development of expansin (cell wall expansion protein) [J]. Chin Bull Life Sci, 18(2): 168-174. [金慧清, 陳英豪, 金勇丰, 2006. Expansin(细胞壁松弛蛋白)的发展 [J]. 生命科学, 18(2): 168-174.]
LI HY, SHI Y, DING YN, et al., 2014. Bioinformatics analysis of expansin gene family in poplar genome [J]. J Beijing For Univ, 36(2):59-67. [李昊阳, 施杨, 丁亚娜, 等, 2014. 杨树扩展蛋白基因家族的生物信息学分析 [J]. 北京林业大学学报, 36(2):59-67.]
ROBERT DF, JODY C, SEAN RE, 2011. HMMER web server: Interactive sequence similarity searching [J]. Nucl Acids Res, 39(Web Server issue): 29-37.
RENSING SA, ROMBAUTS S, VAN DEP, et al., 2002. Moss transcriptome and beyond [J]. Trends Plant Sci, 7(12): 535-538.
STEFAN AR, DANIEL L, ANDREAS Z, et al., 2008. The Physcomitrella genome reveals evolutionary insights into the conquest of land by plants [J]. Science, 319(5859): 64-69.
SAMPEDRO J, CAREY RE, COSGROVE DJ, 2006. Genome histories clarify evolution of the expansin superfamily: New insights from the poplar genome and pine ESTs [J]. J Plant Res, 119(1): 11-21.
VICTORIA HS, JENNIFER MT, ROBERT EC, 2016. Utility of the Amborella trichopoda expansin superfamily in elucidating the history of angiosperm expansins [J]. J Plant Res, 129(2): 199-207.
YI JM, HUANG T, HUANG Y, et al., 2015. Phylogenetic analysis of MADS-box gene family in Physcomitrella patens [J]. Plant Physiol J, 51(2): 197-206. [易吉明, 黄婷, 黄勇, 等, 2015. 小立碗藓MADS-box基因家族的系统进化分析 [J]. 植物生理学报, 51(2): 197-206.]
(责任编辑 周翠鸣)

