甘薯基因组NBS-LRR类抗病家族基因挖掘与分析
黄小芳 毕楚韵 石媛媛 胡韵卓 周丽香 梁才晓 黄碧芳 许 明1, 林世强1,,* 陈选阳1,,5,*
甘薯基因组NBS-LRR类抗病家族基因挖掘与分析
黄小芳1,2,**毕楚韵1,2,**石媛媛2胡韵卓3周丽香4梁才晓4黄碧芳4许 明1,2林世强1,4,*陈选阳1,2,5,*
1福建农林大学作物生物技术福建省高校重点实验室, 福建福州 350002;2福建农林大学农学院, 福建福州 350002;3福建农林大学植物保护学院, 福建福州 350002;4福建农林大学生命科学学院, 福建福州 350002;5福建农林大学教育部作物遗传育种与综合利用重点实验室, 福建福州 350002
NBS-LRR类基因家族是植物抗病基因(Resistance gene)数量最多的一类, 具有NBS (Nucleotide-binding site)和LRR (Leucine-leucine-repeat)结构域。甘薯()栽培种基因组已完成测序, 但尚未注释, 本研究对甘薯基因组序列进行外显子预测, 得到甘薯染色体组全基因组蛋白序列, 在此基础上进一步对NBS-LRR家族基因鉴定和分析表明, 甘薯基因组中含有379个NBS-LRR家族基因, 占全基因组基因总数的0.212%, 其中N型亚家族120个, NL型103个, CNL型133个, TNL型22个, PN型1个。所有染色体上均有NBS-LRR家族基因分布, 但数量明显不同, 其中有60.9%的NBS-LRR基因序列呈簇状分布。NBS-LRR基因序列有15个保守结构域, 在N端较为保守。研究结果为甘薯进一步开展NBS-LRR家族基因的功能研究和抗性育种提供了参考。
甘薯; NBS-LRR;基因; 基因家族; 生物信息学
在植物育种过程中, 通常利用抗病基因(resistance gene)来控制植物病害[1-3]。基因编码的抗性蛋白通过直接或间接的方式识别对应的由病原微生物无毒基因Avr (Avirulence gene)编码的无毒蛋白后, 启动效应因子触发的免疫系统(effector- triggered immunity, ETI), 激活植物体内抗病信号途径, 在侵染部位产生局部的细胞和组织过敏性坏死(hypersensitive response, HR)反应, 最终抵御病原菌的侵染及进一步扩散[4-5]。
目前克隆的抗病R基因大多数都属于NBS-LRR家族[6]。这类基因家族根据其N-末端是否含有TIR结构可分为2个亚类, 一个亚类为TNL型, 含有TIR-NBS-LRR结构, 这个亚类R基因编码的蛋白N-末端有一个白细胞介素受体(toll/interleukin-1 receptor, TIR)的同源结构域; 另一个亚类为非TIR- NBS-LRR (non-TNL)结构, 这类基因编码的蛋白N-末端不含有TIR结构, 但通常被编码的CC (Coiled-Coil)替代, 称为CC-NBS-LRR(CNL)型[7]。
我国是世界上甘薯种植面积最大的国家, 甘薯总产量稳居世界首位。据联合国粮食及农业组织FAO (http://www.fao.org/faostat/zh/#data)统计, 2018年中国甘薯栽培面积和总产分别占世界的30.0%和58.0%。尽管我国甘薯种植总面积和占薯类比重持续下降, 但种植效益仍十分可观[8]。甘薯在种植过程中受到各种病害、虫害和病毒病的威胁[9-10], 而选育和应用抗病品种是减少病虫害损失的最有效手段。近年来, 关于甘薯抗病基因的挖掘逐步得到重视, NBS类抗病基因的克隆也有了一些有限研究和报道[11], 但从甘薯全基因组中发掘NBS-LRR抗病基因的研究还未见报道。
2017年中国科学院上海辰山植物科学研究中心和植物生理生态研究所, 联合德国马克斯普朗克分子遗传研究所和分子植物生理研究所, 利用Illumina测序技术完成对甘薯全基因组序列测序[12], 为甘薯的基础和应用研究建立了扎实的基础。本研究通过生物信息学方法对甘薯全基因组序列进行了CDS (Coding Domain Sequence)区预测, 检索了NBS- LRR家族基因, 并对其进行染色体定位、分类、结构分析和系统进化树研究, 以期为甘薯抗病机制研究及甘薯抗病遗传育种提供科学参考。
1 材料与方法
1.1 甘薯全基因组序列
从NCBI (https://www.ncbi.nlm.nih.gov/genome/? term= Ipomoea+batatas)下载甘薯全基因组序列, 得到15条染色体的DNA序列。
1.2 snap基因注释
通过snap程序应用拟南芥(At.hmm)、线虫(Ce.hmm)、水稻(Os.hmm)的HMM模型对甘薯染色体序列CDS进行检索[13], 分别得到3个.zff文件, 分别从中随机抽取500组合并, 作为训练甘薯特异HMM模型的序列文件。将甘薯特异的HMM模型文件(Ib.hmm)作为检索工具, 通过snap程序检索预测甘薯染色体CDS, 得到甘薯全基因组蛋白质序列。
1.3 含NB-ARC结构域基因预测
从Pfam网站[14-15](http://pfam.xfam.org/)下载NB-ARC(ID:PF00931)家族序列信息, 保存为NB- ARC.hmm文件。通过hmmsearch程序[16-17]用下载的NB-ARC.hmm文件对甘薯全基因组蛋白质序列进行NB-ARC结构域预测。从中选取E-value值≤1E-60的序列, 利用Samtools程序[18-19]新建甘薯的NB-ARC蛋白质序列文件(Ib-NBS.fasta)[20]。利用Clustal Omega[21]对Ib-NBS.fasta文件进行多重序列比对, 得到Ib-NBS-clustalo.fasta文件。利用hmmbuild程序建立Ib-NBS-clustalo.fasta文件的HMM模型, 获得甘薯特异的NB-ARC HMM模型(NB-ARC-specific.hmm)。利用甘薯特异的HMM模型再次检索甘薯全基因组蛋白质序列预测NB-ARC结构域, 选取E-value<0.01的蛋白质序列保存为甘薯高特异的NB-ARC序列信息(Ib-NBS-specific- aa.fasta)。去除甘薯高特异的NB-ARC序列信息中长度小于200的蛋白质序列, 随后将其导入NCBI Conserved Domains Tool[22-25]和Interproscan[26](http:// www.ebi.ac.uk/interpro/search/sequence/)进行筛选, 去除结构域缺失严重的序列, 最终得到甘薯NB-ARC基因家族蛋白质序列数据库。
1.4 NBS关联的保守结构域分析分类
NBS-LRR家族基因在其N-末端含有CC、TIR[7]、RPW8[27]等结构域, 在C-末端通常含有LRR结构域[28]。从Pfam数据库[14-15](http://pfam.xfam.org/)中下载TIR HMMs (PF01582)、RPW8 HMMs (PF05659)和LRR HMMs (PF00560、PF07723、PF07725和PF12799)模型, 合并后用hmmpress程序建立TIR/RPW8/LRR HMM模型(TRL.hmm)。通过hmmscan程序用TRL.hmm文件对甘薯NBS-LRR家族基因信息库进行结构域预测, 同时结合Interproscan得到结果鉴别出不同类型的TIR/ RPW8/LRR结构域, 统计其类型和重复数。通过Pepcoil程序[29]预测甘薯NBS-LRR家族基因氨基酸序列信息中的CC结构域(coiled-coil domain), 利用 Paircoil2 (值临界值为0.03, 氨基酸长度临界值为21)[30]检测CC结构域。
1.5 甘薯NBS-LRR基因染色体定位
统计甘薯15条染色体上NBS-LRR家族基因的位置信息, 参考Jupe等[31]的方法绘制NBS-LRR家族基因在染色体上的位置。
1.6 甘薯CNL、TNL、RPW8亚家族motif分析
将甘薯NBS-LRR家族基因分类整理后, 从中筛选出CC-NBS-LRR、TIR-NBS-LRR、RPW8-NBS三种NBS亚家族类型的90个氨基酸序列[32], 通过MEME程序[33]进行motif分析和识别(设置搜索功能域数为15)。将MEME程序运行后得到的.xml文件利用TBtools软件[34]处理。
1.7 甘薯CNL、TNL、RPW8亚家族进化树构建
将筛选得到的CC-NBS-LRR、TIR-NBS-LRR、RPW8-NBS三种NBS-LRR亚家族类型的90个氨基酸序列利用Clustal Omega程序[21]进行多序列比对后, 使用Gblocks[35-36]提取保守序列(允许序列保留50%的缺口数), 进一步利用Jalview[37-38]手动矫正提取到的保守序列, 保存为.fa序列文件。根据比对结果为基础, 应用MEGA X[39]软件, 选择Maximum Likehood法, 设置运行参数模式WAG with Freqs.(+F)model, 校验参数Bootstrap=500, 生成甘薯NBS-LRR基因家族系统进化树。
2 结果与分析
2.1 甘薯基因组中含有NB-ARC结构域的基因鉴别及其分类
采用snap程序对甘薯全基因组序列进行CDS区识别预测, 得到甘薯15条染色体CDS序列178,458个。利用hmmsearch程序筛选甘薯基因组中含有NB-ARC结构域的序列, 以E-value≤0.01为标准, 获得相关蛋白序列432个。从中选取高特异蛋白质序列(E-value≤1E-60)构建甘薯特异的NB-ARC HMM模型, 利用甘薯特异的HMM模型再次预测甘薯全基因组序列的NB-ARC保守结构域, 获得甘薯含有NB-ARC结构域的特异蛋白序列735个(E-value≤0.01)。去除氨基酸长度小于200以及结构域缺失严重的序列, 最终得到379个甘薯NBS-LRR家族基因, 占甘薯全基因组基因数目(178,458)的0.212%。根据TIR/RPW8/LRR结构域和CC结构域分为N (NBS)、NL (NBS-LRR)、CNL (CC-NBS- LRR)、TNL (TIL-NBS-LRR)和PN (RPW8-NBS) 4个亚家族类型(表1), 其中N型120个, NL型103个, CNL型133个, TNL型22个, PN型1个。分析发现, NBS/LRR结构域重复数和位置存在差异, 在序列结构域读取文件中含有RX-CC_like结构域, 此结构域为马铃薯抗X病毒和类似蛋白的Coiled-coil结构域(Coiled coil domain of the potato virus X resistance protein and similar proteins)[40], 记为Cx结构域, 以区分Pepcoil程序得到的CC结构域。因此, 根据NBS/LRR结构域重复数和位置差异、CC结构域类型进一步分为34个小类(表1)。
在NBS-LRR家族基因的N末端区域含有TIR (Toll/inter-leukin-1 receptor)保守结构域的蛋白称为TNL 蛋白, 其他的称为非TNL蛋白。大部分非TNL蛋白的N末端有卷曲螺旋结构(Coiled-coil), 称为CNL蛋白[41]。目前已进行NBS-LRR家族基因信息分析的植物有木薯[42]、水稻[43]、拟南芥[44]、乌拉尔图小麦[45]等, 其中CNL亚家族数目占其NBS家族基因数比例分别为木薯(35.78%)[42]、水稻(32.71%)[43]、拟南芥(24.64%)[44]、乌拉尔图小麦(19.18%)[45]。在甘薯NBS-LRR家族基因序列结构域分类中, 同时含有CC、NBS、LRR结构域的序列有77个, 占NBS-LRR家族基因总数的20.32%; 同时含有TIR、NBS、LRR结构域的序列有10个, 占总基因家族信息数2.64%。

表1 甘薯编码NBS-LRR蛋白的基因数量及其分类
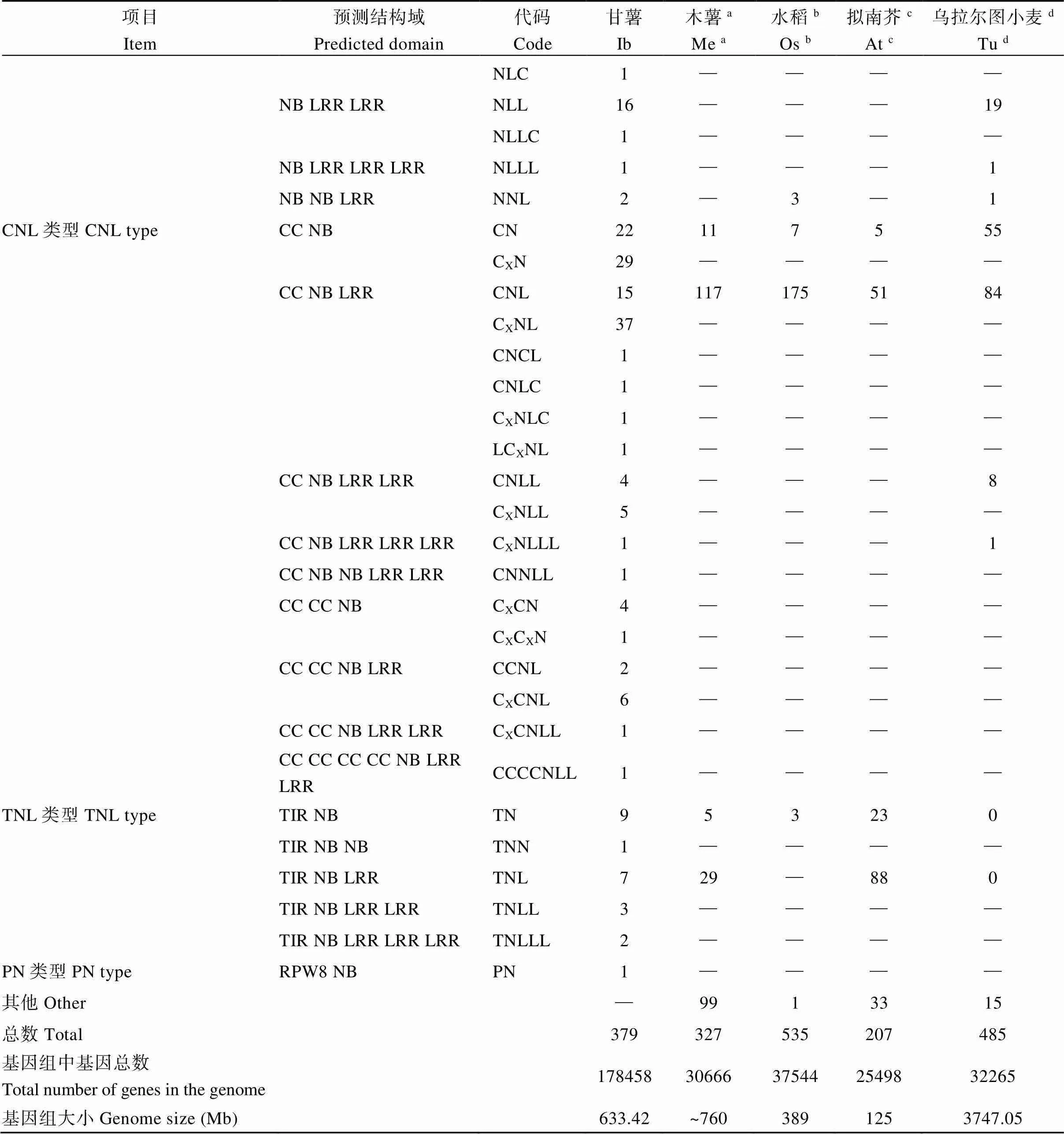
(续表1)
a: 数据来源于[42]; b: 数据来源于[43]; c: 数据来源于[44]; d: 数据来源于[45]。
a: data were from [42]; b: data were from [43]; c: data were from [44]; d: data were from [45]. Ib:; Me:; Os:; At:; Tu:.
2.2 甘薯NBS-LRR家族基因在染色体上的分布
NBS-LRR家族基因有不同的亚家族, 包括N型(黑色)、NL型(红色)、CNL型(绿色)、TNL型(蓝色)、PN型(青色)(图1)。基因有正链编码(图1中染色体右侧)和负链编码(图1中染色体左侧)。NBS-LRR家族基因数量在甘薯15条染色体上分布并不均匀, 在11号染色体上仅有3个, 而在13号染色体上则有54个。在不同染色体上的基因以单基因或基因簇的形式存在。基因簇是指在200 kb的核苷酸单位中含有的基因群[46-47], 甘薯NBS-LRR家族基因中共有231个基因是以基因簇的形式存在。这231个基因分布在81个基因簇中, 占NBS-LRR家族基因总数的60.9%, 平均每个基因簇含2.9个基因。在这些基因簇中含有2个基因的基因簇数目最多, 有46个, 占总基因簇数的56.8%。其次是3~4个基因的基因簇, 有18个, 12个基因簇含有4个基因。含5个、6个和8个基因的基因簇均只有1个。含有最大基因数的基因簇有2个, 分别分布在9号和13号染色体上, 簇中含有9个基因。

(图1)
甘薯基因组中, 单独分布在染色体上的NBS- LRR家族基因有148个, 占NBS-LRR基因总数39.1% (图1和表2)。在14号染色体上的9个NBS- LRR家族基因均呈单基因分布, 其中含RPW8结构域的NBS-LRR家族基因也分布于14号染色体。7号染色体基因簇最多达16个, 据此推测7号染色体可能发生NBS-LRR家族抗病基因的大规模复制, 导致该染色体上NBS-LRR家族基因的高密度分布[46]。
2.3 甘薯NBS-LRR家族基因CNL、TNL亚家族motif分析
分别将甘薯NBS-LRR家族基因中CNL和TNL亚家族序列信息利用MEME程序检测, 筛选得到15个相似度较高的结构域。77个CNL亚家族基因的结构域分布(图2)存在以下规律: motif 10-motif 11- motif 2(P-loop)-motif 4(RNBS-A)-motif 1(Kinase 2)- motif 13(RNBS-B)-motif 5(GLPL)-motif 15-motif 9(RNBS-D)-motif 6(RNBS-D)-motif 12-motif 3(MHDV)-motif 8-motif 14-motif 7。其中motif 15(motif 5和motif 9之间)仅存在于21个序列中, 即位于GLPL结构域和RNBS-D结构域之间。其中RNBS-D保守基序对应于2个结构域, 分别为motif 9和motif 6, 共有70个序列含有motif 6, 其中有17个序列缺失motif 9。TNL亚家族结构域分布(图3)存在以下规律: motif 10(TIR-1)-motif 3-motif 1(TIR-2)-motif 5-(TIR-3)-motif 8(TIR-4)-motif 2(P- loop)-motif 9(Kinase 2)-motif 4(RNBS-B)-motif 13- (RNBS-C)-motif 7(GLPL)-motif 11-motif 15-motif6-(MHDV)-motif 12-motif 14或motif 10(TIR-1)- motif 3-motif 1(TIR-2)-motif 5-(TIR-3)-motif 8(TIR- 4)-motif 2(P-loop)-motif 9(Kinase 2)-motif 4(RNBS- B)-motif 13-(RNBS-C)-motif 7(GLPL)-motif 11-motif 6-(MHDV)-motif 12(LRR)-motif 14(LRR)-。大部分motif 15存在于motif 11和motif 6之间, 即MHDV结构域之前, 仅有3个序列motif 15位于motif 14之后。
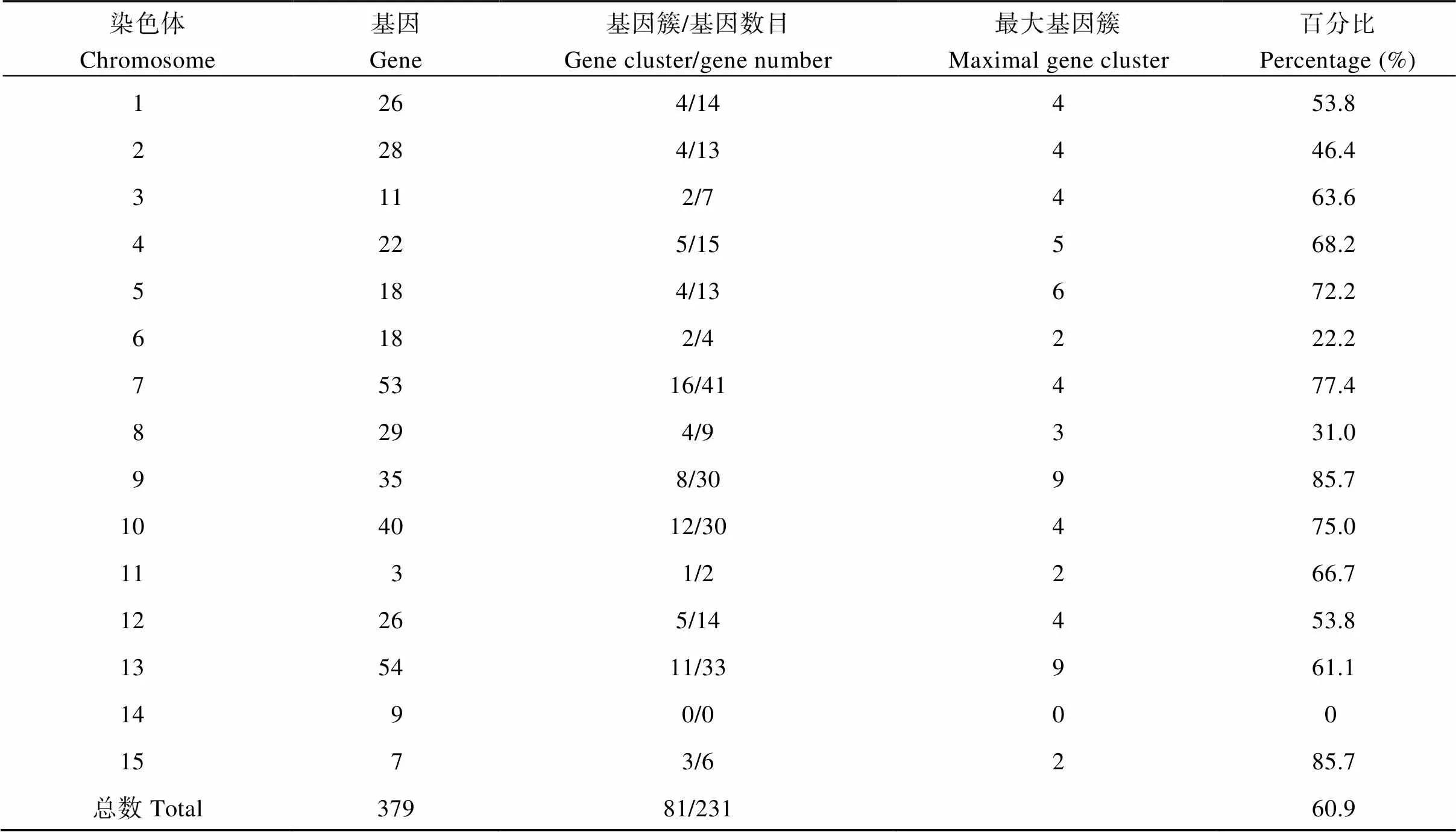
表2 甘薯NBS-LRR基因家族基因簇统计表
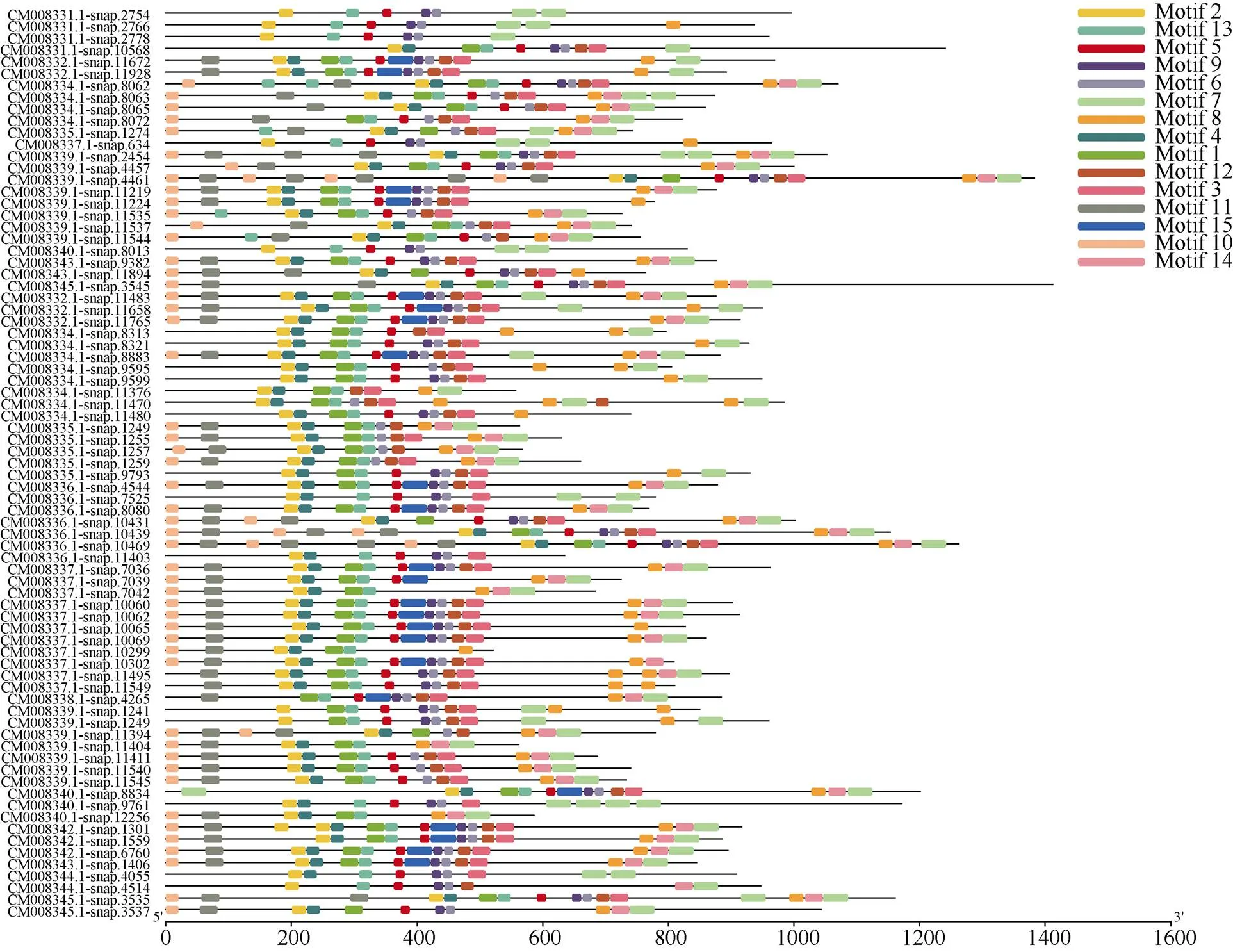
图2 甘薯NBS-LRR家族中CNL亚家族保守结构域分布
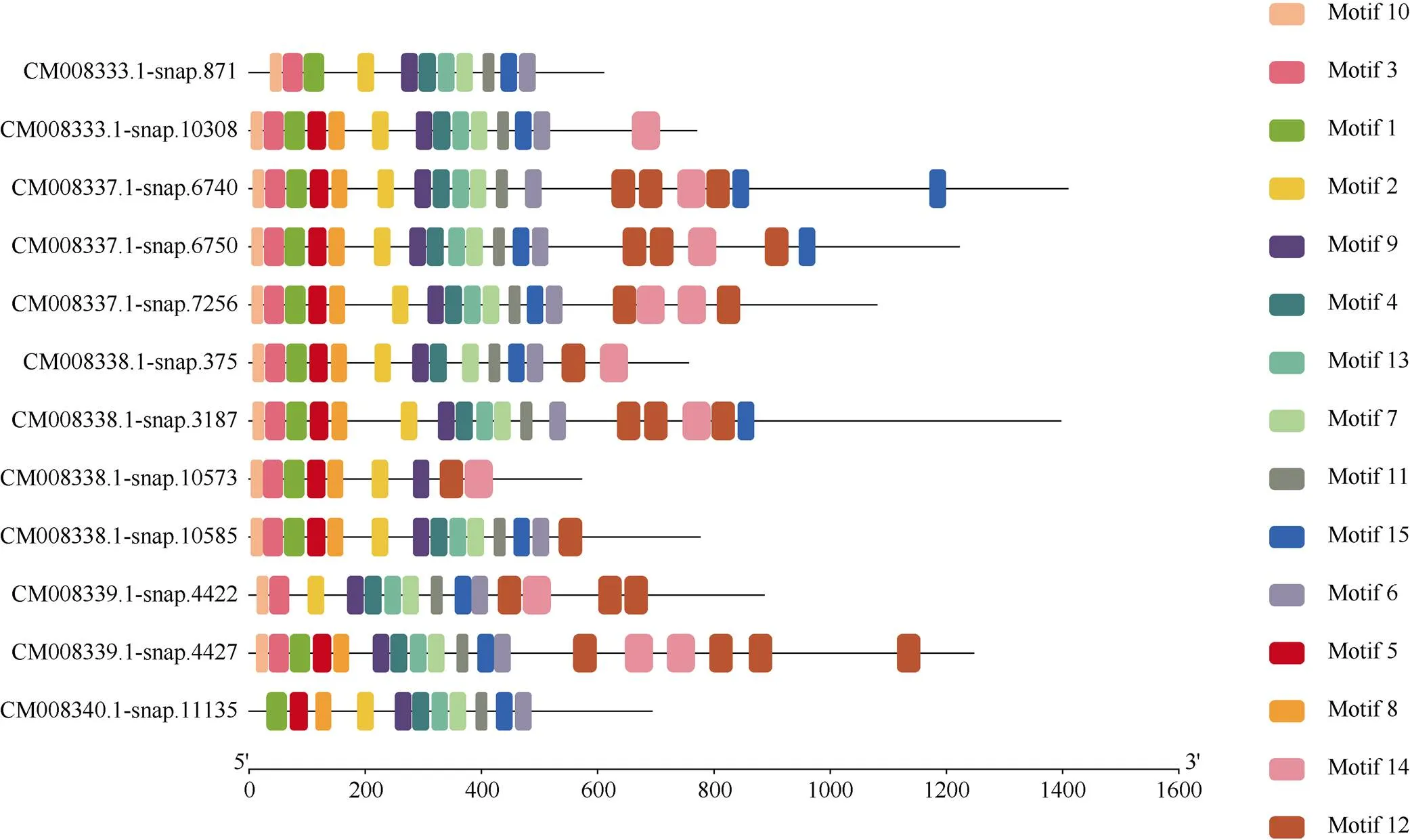
图3 甘薯NBS-LRR家族中TNL亚家族保守结构域分布
2.4 CNL、TNL亚家族NB-ARC结构域保守性分析
植物NBS-LRR家族基因中的NB-ARC区域相对保守, 通常有P-loop、RNBS-A、Kinase2、RNBS-B、RNBS-C、GLPL、RNBS-D和MHDV 8个保守基序[48]。对获得的甘薯NBS-LRR家族中CNL和TNL亚家族序列利用Clustal Omega进行多重比对发现, 具有5个保守性较高的序列结构域[44,49-50](图4), 分别为P-loop (Kinase 1)、Kinase 2、RNBS-B、GLPL、MHDV。其中P-loop最可能氨基酸序列为SIVLA (画横线部分为氨基酸保守性较强序列), Kinase 2最可能氨基酸序列为YLIW, RNBS-B最可能的氨基酸序列为I+LLR, GLPL最可能的氨基酸序列为IVSY+VVVAKRL, MHDV最可能的氨基酸序列为K+TIRMLLRDMGR。
根据MEME结构域搜索结果, 对CNL和TNL亚家族基因结构域进行基因定位和识别[51](图5), 发现TNL亚家族和CNL亚家族中分别有10个和7个保守结构域, 其中TNL亚家族NB-ARC结构域中保守基序分别为P-loop、Kinase 2、RNBS-B、RNBS-C、GLPL和MHDV, 缺失RNBS-A以及RNBS-D保守基序; CNL亚家族NB-ARC结构域保守基序分别为P-loop、RNBS-A、Kinase 2、RNBS-B、GLPL、RNBS-D和MHDV, 缺失RNBS-C保守基序。TNL亚家族和CNL亚家族中共有的保守基序分别为P-loop (Kinase 1)、Kinase 2、RNBS-B、GLPL以及MHDV, 与多重比对结果相同。此外, 在TNL亚家族中TIR结构域检测到4个保守基序(TIR1-4)[52]。其中9个NBS-LRR家族基因含有完整的TNL结构域, 占总TNL亚家族基因数的75%, 有2个序列同时缺失TIR 3和TIR 4, 有一个序列缺失TIR 1。
2.5 甘薯CNL、TNL、RPW8亚家族系统进化树分析
将甘薯NBS-LRR家族基因中CC/TIR/RPW8/ NBS/LRR结构域的序列提取后, 使用MEGA X构建系统进化树。进化树结果中(图6)有3个明显的分支, 分别为CNL型(绿色)、TNL型(蓝色)、PN型(青色)。3种NBS-LRR家族的亚家族存在于不同的进化分支中, 但不存在单独的分支, 说明这3类基因存在相同的进化方式。进化树分支中有些基因分支路径很长, 如CM008340.1-snap12256、CM008331.1- snap10568等, 推测这类基因的“祖先”基因在很早的时候就发生了分化, 相应的基因序列也发生了较大的分化。有些基因序列的分支距离较近, 如CM008334.1-snap8062、CM008342.1-snap6760、CM008334.1-snap11470等, 推测这些基因在短期内发生了复制[46]。在CNL亚家族分支CC结构域中, 马铃薯X病毒类似蛋白[40]的CC结构域(Coiled-coil domain of the potato virus X resistance protein and similar proteins)大致分布于同一小分支, 说明马铃薯X病毒类似蛋白的CC结构域(表1中Cx)与普通CC结构域(表1中C)存在进化关系上的差异。
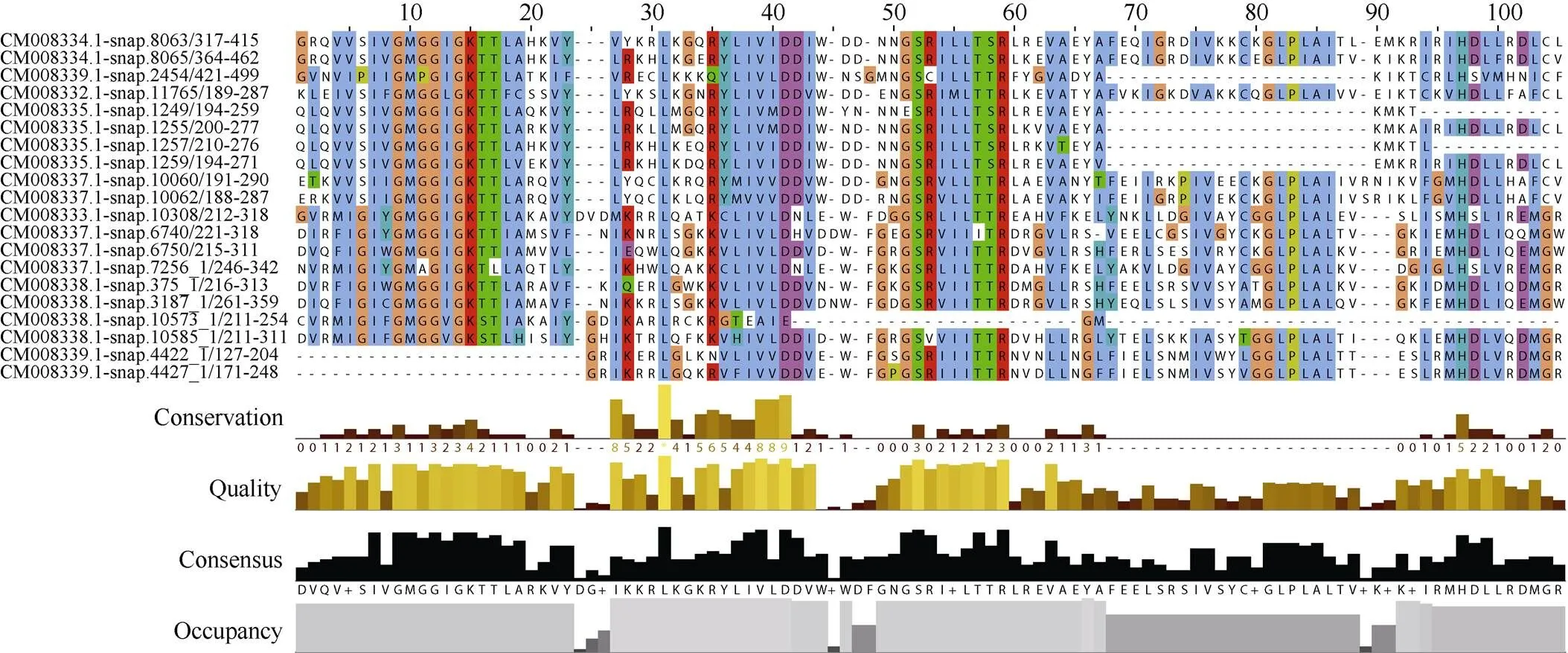
图4 甘薯NB-ARC保守性分析
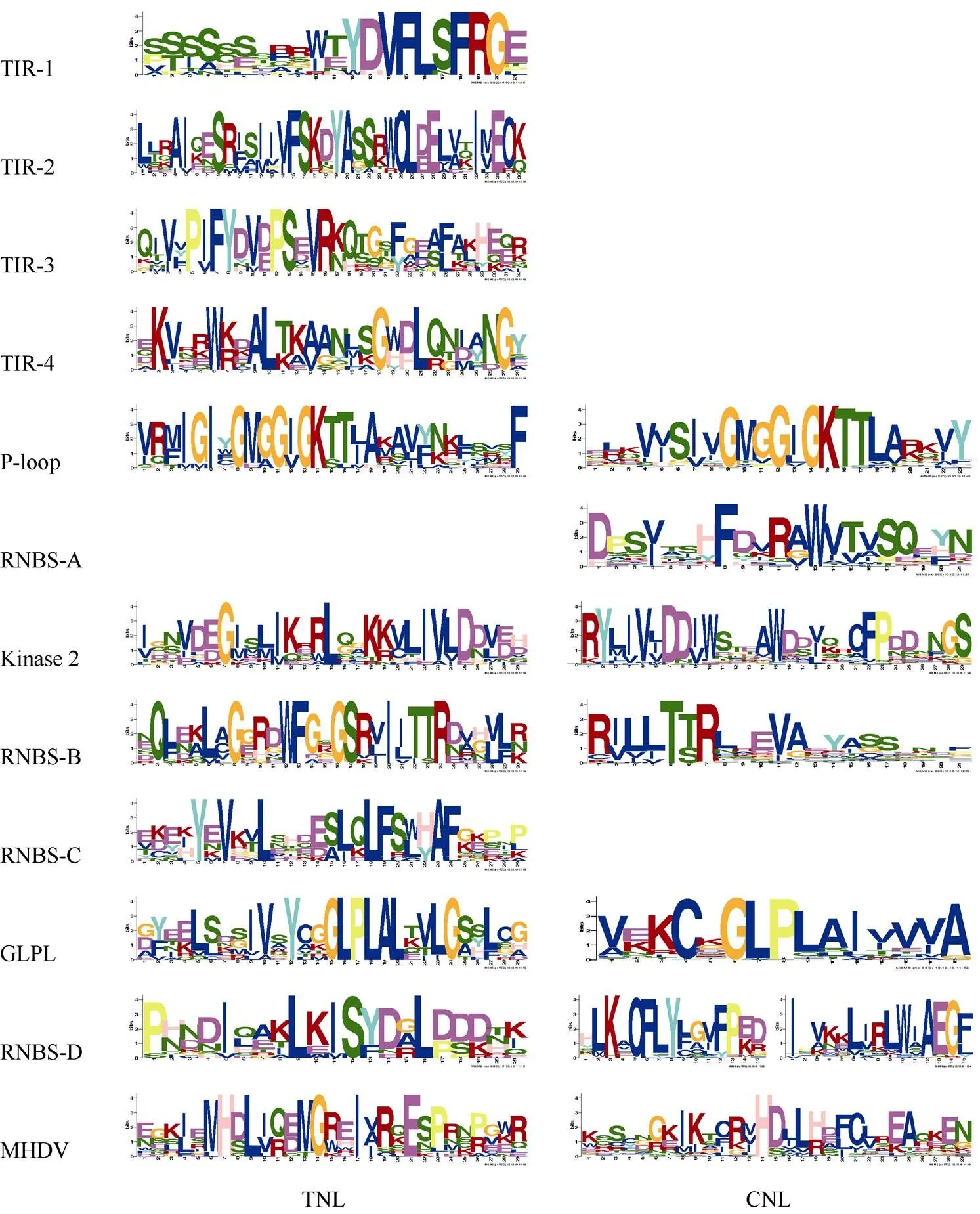
图5 甘薯NBS-LRR家族基因保守结构域及其氨基酸保守性分析
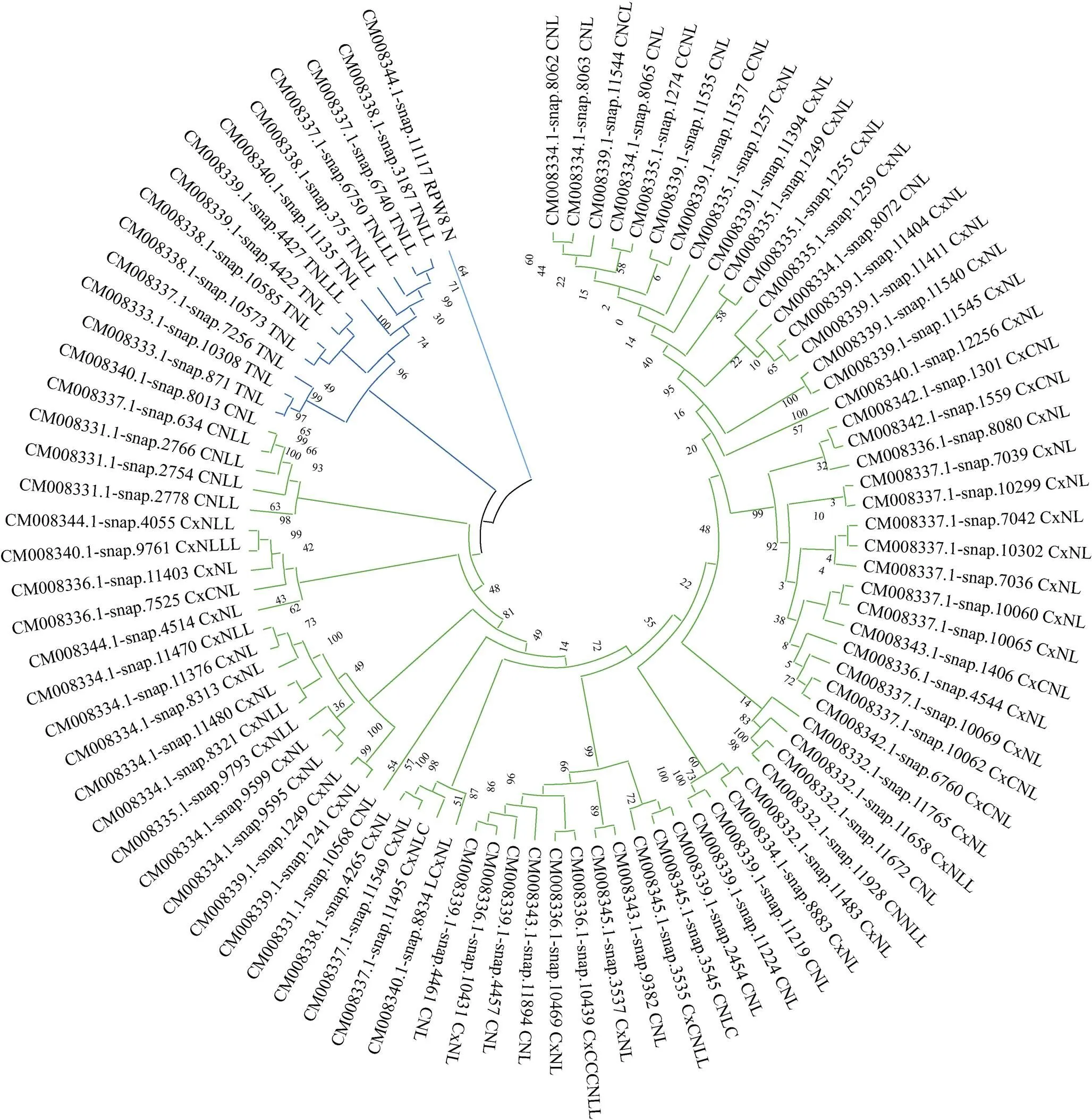
图6 甘薯NBS-LRR家族基因系统进化树
3 讨论
生物信息学方法分析植物抗病基因已被广泛运用于许多作物, 甘薯是重要的粮食作物, 然而, 甘薯基因组尚未注释, 无法直接查找与分析其基因组的基因。本研究应用snap软件对甘薯全基因组序列进行了外显子预测, 得到甘薯CDS区蛋白质序列, 为甘薯基因的搜索及其他相关生物信息学分析提供了序列基础。
本研究利用HMMER程序对甘薯全基因组序列进行NBS-LRR家族基因检索, 得到735个甘薯NBS-LRR家族基因。经过筛选、验证、剔除等步骤, 最终得到379个序列完整的NBS-LRR家族基因, 占甘薯全基因组数目0.2%, 但与木薯(1.1%)[42]、水稻(1.4%)[43]、拟南芥(0.8%)[44]、乌拉尔图小麦(1.5%)[45]相比数量明显偏少。此外, 甘薯NBS-LRR家族中TNL亚家族基因有22个, 与其他作物有较大差别, 如水稻和乌拉尔图小麦中NBS-LRR家族基因无TNL类型[43,45], 在其他禾本科和草本的单子叶植物中TNL亚家族基因很少[53-54]。本研究分析了甘薯NBS-LRR家族基因的NB-ARC结构域保守性, 分别对CNL和TNL亚家族进行比较, 发现两者在P-loop、Kinase 2、RNBS-B、GLPL、MHDV具有较高的相似性。甘薯P-loop和GLPL基序保守性最高, 其基序分别为SIVLA 和IVSY+VVVAKRL, 且GMGGIGKTT区域保守性最高。其次为Kinase 2和RNBS-B, Kinase 2结构域序列为YLI, 77个CNL类型中, 有70个Kinase 2序列末端氨基酸为W, TNL类型中末端氨基酸主要为E或D, 未发现W。研究表明, Kinase 2序列末端氨基酸W是区分CNL亚家族和TNL亚家族的关键因素[42,55]。同时, TNL类型motif位置的分布比较保守, 为TIR(1-4)-P loop-Kinase 2-RNBS B- RNBS C-GLPL-motif 11。CNL类型motif之间位置的排布规律遵循motif 10-motif 11-P loop-RNBS C-Kinase 2-RNBS B-GLPL的顺序。
值得注意的是, 本研究所分析的甘薯基因组是基于二代测序组装的单倍体型基因组[12], 存在数据不完整和一些错误组装的局限性[56], 本研究结果可能难以完整覆盖甘薯栽培种基因组中NBS-LRR家族的所有基因, 在对相关基因进一步深入分析和研究应用时, 还可以结合三浅裂野牵牛()基因组。但是本研究的分析方法和结果, 可为甘薯NBS-LRR基因发掘、抗性相关机制研究和抗病育种提供参考。
4 结论
预测了甘薯基因组序列的外显子, 得到甘薯染色体组全基因组蛋白序列, 从中筛选了379个NBS-LRR家族基因, 并进行了染色体定位, 发现NBS-LRR家族基因在不同染色体上的分布数量差异很大, 其中有60.9%的NBS-LRR基因序列呈簇状分布。NBS-LRR基因序列有15个保守结构域, 在N端较为保守, 根据对基因的结构分析构建了系统进化树。研究结果可为甘薯进一步开展NBS-LRR家族基因的功能研究和抗性育种提供参考。
[1] Staskawicz B J, Ausubel F M, Baker B J, Ellis J G, Jones J D. Molecular genetics of plant disease resistance., 1995, 268: 661–667.
[2] 李楠洋. 棉花抗黄萎病基因筛选及NBS-LRR类抗病基因功能研究. 中国农业科学院博士学位论文, 北京, 2017. Li N Y. Screening of Cotton Anti Verticillium Wilt Genes and Functional Study on NBS-LRR Resistance Gene. PhD Dissertation of Chinese Academy of Agricultural Sciences, Beijing, China, 2017 (in Chinese with English abstract).
[3] Changkwian A, Venkatesh J, Lee J H, Han J W, Kwon J K, Siddique M I, Solomon A M, Choi G J, Kim E, Seo Y, Kim Y H, Kang B C. Physical localization of the root-knot nematode () resistance locusin pepper ()., 2019, 10: 886.
[4] 房卫平, 谢德意, 李志芳, 李武, 赵付安, 孙瑶, 段峥峥, 杨晓杰. NBS-LRR类抗病蛋白介导的植物抗病应答分子机制. 分子植物育种, 2015, 13: 469–474. Fang W P, Xie D Y, Li Z F, Li W, Zhao F A, Sun Y, Duan Z Z. Yang X J. Molecular mechanism on NBS-LRR proteins-mediated plant disease response.2015, 13: 469–474 (in Chinese with English abstract).
[5] Jones J D G, Dangl J L. The plant immune system., 2006, 444: 323–329.
[6] Hammond-Kosack K E, Parker J E. Deciphering plant-pathogen communication: fresh perspectives for molecular resistance breeding., 2003, 14: 177–193.
[7] 尹玲, 方辉, 黄羽, 卢江, 曲俊杰. 植物TIR-NB-LRR类型抗病基因各结构域的研究进展. 广西植物, 2017, 37: 186–190. Yin L, Fang H, Huang Y, Lu J, Qu J J. Research progress on domains of plant TIR-NB-LRR resistance genes., 2017, 37: 186–190 (in Chinese with English abstract).
[8] 陆建珍, 汪翔, 秦建军, 戴起伟, 易中懿. 我国甘薯种植业发展状况调查报告(2017年)——基于国家甘薯产业技术体系产业经济固定观察点数据的分析. 江苏农业科学, 2018, 46(23): 393–398. Lu J Z, Wang X, Qin J J, Dai Q W, Yi Z Y. Investigation report on the development of sweet potato cultivation in China (2017): Analysis based on the data of industrial economy fixed observation point of the national sweet potato industry technology system., 2018, 46(23): 393–398 (in Chinese with English abstract).
[9] 赵永强, 张成玲, 孙厚俊, 徐振, 陈晓宇, 谢逸萍. 甘薯病毒病复合体(SPVD)对甘薯产量的影响. 西南农业学报, 2012, 25: 909–911. Zhao Y Q, Zhang C L, Sun H J, Xu Z, Chen X Y, Xie Y P. Effects of viruses (SPVD) on yield of sweet potato., 2012, 25: 909–911 (in Chinese with English abstract).
[10] 赵冬兰, 唐君, 张安, 周志林, 曹清河, 戴习彬. 甘薯病毒病对不同基因型甘薯产量和品质的影响. 江西农业学报, 2018, 30(11): 62–65. Zhao D L, Tang J, Zhang A, Zhou Z L, Cao Q H, Dai X B. Effects of virus diseases on yield and quality of different sweet potato genotypes., 2018, 30(11): 62–65 (in Chinese with English abstract).
[11] 屈满义, 查向东, 王钰, 杨金环, 蒋琳, 阮龙. 甘薯NBS-LRR类抗病基因同源序列的克隆、分析及数目研究. 热带作物学报, 2008, 29: 610–617. Qu M Y, Zha X D, Wang Y, Yang J H, Jiang L, Ruan L. Study on cloning, analysis and number of NBS-LRR resistance genes of sweet potato., 2008, 29: 610–617 (in Chinese with English abstract).
[12] Yang J, Moeinzadeh M H, Kuhl H, Helmuth J, Xiao P, Haas S, Liu G, Zheng J, Sun Z, Fan W, Deng G, Wang H, Hu F, Zhao S, Fernie A R, Boerno S, Timmermann B, Zhang P, Vingron M. Haplotype-resolved sweet potato genome traces back its hexaploidization history., 2017, 3: 696–703.
[13] Ian K. Gene finding in novel genomes., 2004, 5: 59.
[14] Finn R D, Bateman A, Clements J, Coggill P, Eberhardt R Y, Eddy S R, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer E L, Tate J, Punta M. Pfam: the protein families database., 2014, 42: 222–230.
[15] Madeira F, Park Y M, Lee J, Buso N, Gur T, Madhusoodanan N, Basutkar P, Tivey A R N, Potter S C, Finn R D, Lopez R. The EMBL-EBI search and sequence analysis tools APIs in 2019., 2019, 47: 636–641.
[16] Eddy S R. Profile Hidden Markov Models., 1998, 14: 755–763.
[17] Pottter S C, Luciani A, Eddy S R, Park Y, Lopez R, Finn R D. Web server issue., 2018, 46: 200–204.
[18] Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R, Genome Project Data Processing, Subgroup. The sequence alignment/map format and SAMtools., 2009, 25: 2078–2079.
[19] Li H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data.. 2011, 27: 2987–2993.
[20] Brendolise C, Montefiori M, Dinis R, Peeters N, Storey R D, Rikkerink E H. A novel hairpin library-based approach to identify NBS–LRR genes required for effector-triggered hypersensitive response in Nicotiana benthamiana., 2017, 13: 32.
[21] Sievers F, Wilm A, Dineen D, Gibson T J, Karplus K, Li W Z, Lopez R, McWilliam H, Remmert M, Söding J, Thompson J D, Higgins D G. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega., 2011, 7: 539.
[22] Marchler-Bauer A, Bryant S H. CD-search: protein domain annotations on the fly., 2004, 32: 327–331.
[23] Marchler-Bauer A, Lu S N, Anderson J B, Chitsaz F, Derbyshire M K, De Weese-Scott C, Fong J H, Geer L Y, Geer R C, Gonzales N R, Gwadz M, Hurwitz D I, Jackson J D, Ke Z, Lanczycki C J, Lu F, Marchler G H, Mullokandov M, Omelchenko M V, Robertson C L, Song J S, Thanki N, Yamashita R A, Zhang D C, Zhang N G, Zheng C J, Bryant S H. CDD: a conserved domain database for the functional annotation of proteins., 2011, 39: 225–229.
[24] Marchler-Bauer A, Derbyshire M K, Gonzales N R, Lu S N, Chitsaz F, Geer L Y, Geer R C, He J, Gwadz M, Hurwitz D I, Lanczycki C J, Lu F, Marchler G H, Song J S, Thanki N, Wang Z X, Yamashita R A, Zhang D C, Zheng C J, Bryant S H. CDD: NCBI’s conserved domain database., 2015, 43: 222–226.
[25] Marchler-Bauer A, Bo Y, Han L Y, He J N, Lanczycki C J, Lu S N, Chitsaz F, Derbyshire M K, Geer R C, Gonzales N R, Gwadz M, Hurwitz D I, Lu F, Marchler G H, Song J S, Thanki N, Wang Z X, Yamashita R A, Zhang D C, Zheng C J, Geer L Y, Bryant S H. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures., 2017, 45: 200–203.
[26] Quevillon E, Silventoinen V, Pillai S, Harte N, Mulder N, Apweiler R, Lopez R. InterProScan: protein domains identifier., 2005, 33: 116–120.
[27] Barragan C A, Wu R, Kim S T, Xi W Y, Habring A, Hagmann J, Van de Weyer A L, Zaidem M, Ho W W H, Wang G, Bezrukov I, Weigel D, Chae E. RPW8/HR repeats control NLR activation in., 2019, 15: e1008313.
[28] 刘云飞, 万红建, 李志邈, 叶青静, 王荣青, 阮美颖, 姚祝平, 周国治, 韦艳萍, 杨悦俭. 植物NBS-LRR抗病基因的结构、功能、进化起源及其应用. 分子植物育种, 2014, 12: 377–389. Liu Y F, Wan H J, Li Z M, Ye Q J, Wang R Q, Ruan M Y, Yao Z P, Zhou G Z, Wei Y P, Yang Y J. The structure, function, evolutional origin and application of plant NBS-LRR resistance genes., 2014, 12: 377–389 (in Chinese with English abstract).
[29] Lupas A, Dyke M V, Stock J, Predicting coiled coils from protein sequences., 1991, 252: 1162–1164.
[30] McDonnell A V, Jiang T, Keating A E, Berger B. Paircoil2: improved prediction of coiled coils from sequence., 2006, 22: 356–358.
[31] Jupe F, Pritchard L, Etherington G J, MacKenzie K, Cock P J, Wright F, Sharma S K, Bolser D, Bryan G J, Jones J D, Hein I. Identification and localisation of the NB-LRR gene family within the potato genome., 2012, 13: 75.
[32] 蒋卉, 张晶, 符真珠, 董晓宇, 王慧娟, 李艳敏, 高杰, 王利民, 张和臣. 蝴蝶兰NBS-LRR家族基因挖掘和生物信息学分析. 分子植物育种, 2018, 16: 2786–2794. Jiang H, Zhang J, Fu Z Z, Dong X Y, Wang H J, Li Y M, Gao J, Wang L M, Zhang H C. Mining and bioinformatics analysis of NBS-LRR gene family in phalaenopsis., 2018, 16: 2786–2794 (in Chinese with English abstract).
[33] Bailey T L, Williams N, Misleh C, Li W W. MEME: discovering and analyzing DNA and protein sequence motifs., 2006, 34: 369–373.
[34] Chen C J, Chen H, He Y H, Xia R. TBtools, a Toolkit for Biologists integrating various biological data handling tools with a user-friendly interface., 2018. doi: https://doi.org/10. 1101/289660.
[35] Castresana J. Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis., 2000, 17: 540–552.
[36] Talavera G, Castresana J. Improvement of phylogenies after removing divergent and ambiguously aligned blocks from protein sequence alignments., 2007, 56: 564–577.
[37] Waterhouse A M, Procter J B, Martin D M A, Clamp M, Barton G J. Jalview Version 2: a multiple sequence alignment editor and analysis workbench., 2009, 25: 1189–1191.
[38] Troshin P V, Procter J B, Barton G J. Java bioinformatics analysis web services for multiple sequence alignment—JABAWS: MSA., 2011, 27: 2001–2002.
[39] Kumar S, Stecher G, Li M, Knyaz C, Tamura K. MEGA X: molecular evolutionary genetics analysis across computing platforms., 2018, 35: 1547–1549.
[40] Hao W, Collier S M, Moffett P, Chai J. Structural basis for the interaction between the potato virus X resistance protein (Rx) and its cofactor Ran GTPase-activating protein 2 (RanGAP2)., 2013, 288: 35868–35876.
[41] Tarr D E, Alexander H M. TIR-NBS-LRR genes are rare in monocots: evidence from diverse monocot orders., 2009, 2: 197.
[42] Lozano R, Hamblin M T, Prochnik S, Jannink J L. Identification and distribution of the NBS-LRR gene family in the Cassava genome., 2015, 16: 360.
[43] Zhou T, Wang Y, Chen J Q, Araki H, Jing Z, Jiang K, Shen J, Tian D. Genome-wide identification of NBS genes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes., 2004, 271: 402–415.
[44] Meyers B C, Kozik A, Griego A, Kuang H, Michelmore R W. Genome-wide analysis of NBS-LRR-encoding genes in., 2003, 15: 809–834.
[45] 刘小芳, 袁欣, 聂迎彬, 张晶. 乌拉尔图小麦NBS-LRR家族生物信息学分析. 分子植物育种, 2018, 16: 7587–7597. Liu X F, Yuan X, Nie Y B, Zhang J. Bioinformatics Analysis of NBS-LRR gene family in., 2018, 16: 7587–7597 (in Chinese with English abstract).
[46] 王岩, 李兆阳, 唐心龙, 卢姗, 许鹏, 张静, 方奎, 席景会. 拟南芥基因组NBS-LRR类基因家族的生物信息学分析. 中国农学通报, 2009, 25(15): 40–45. Wang Y, Li Z Y, Tang X L, Lu S, Xu P, Zhang J, Fang K, Xi J H. Bioinformatic analysis of the NBS-LRR gene family in., 2009, 25(15): 40–45 (in Chinese with English abstract).
[47] Holub E B. The arms race is ancient history in Arabidopsis, the wildflower., 2001, 2: 516–527.
[48] Liu Z C, Xie J M, Wang H P, Zhong X H, Li H L, Yu J H, Kang J G. Identification and expression profiling analysis of NBS-LRR genes involved inf. spconglutinans resistance in cabbage., 2019, 9: 202.
[49] Kohler A, Rinaldi C, Duplessis S, Baucher M, Geelen D, Duchaussoy F, Meyers B C, Boerjan W, Martin F. Genome-wide identification of NBS resistance genes in., 2008, 66: 619–636.
[50] Ahlenstiel G, Lozano R, Ponce O, Ramirez M, Mostajo N, Orjeda G. Genome-wide identification and mapping of NBS-encoding resistance genes ingroup phureja., 2012, 7: e34775.
[51] Li T G, Wang B L, Yin C M, Zhang D D, Wang D, Song J, Zhou L, Kong Z Q, Klosterman S J, Li, J J, Adamu S, Liu T L, Subbarao K V, Chen J Y, Dai X F. TheTIR-NBS-LRR genemediates resistance against verticillium wilt., 2019, 20: 857–876.
[52] Meyers B C, Morgante M, Michelmore R W. TIR-X and TIR-NBS proteins: two new families related to disease resistance TIR-NBS-LRR proteins encoded inand other plant genomes., 2002, 32: 77–92.
[53] Wu L, Hickson I D. The Bloom’s syndrome helicase suppresses crossing over during homologous recombination., 2003, 426: 870–874.
[54] Lakatos L, Csorba T, Pantaleo V, Chapman E J, Carrington J C, Liu Y P, Dolja V V, Calvino L F, López-Moya J J, Burgyán J. Small RNA binding is a common strategy to suppress RNA silencing by several viral suppressors., 2006, 25: 2768–2780.
[55] López C E, Zuluaga A P, Cooke R, Delseny M, Tohme J, Verdie V. Isolation of resistance gene candidates (RGCs) and characterization of an RGC cluster in cassava., 2003, 269: 658–671.
[56] Wu S, Lau K H, Cao Q H, Hamilton J P, Sun H H, Zhou C X, Eserman L A, Gemenet D C, Olukolu B A, Wang H Y, Crisovan E, Godden G T, Jiao C, Wang X, Kitavi M, anrique-Carpintero N, Vaillancourt B, Wiegert-Rininger K, Yang X S, Bao K, Schaff J, Wolfgang J K, Gruneberg A K, Ghislain M, Ma D, Jiang J M, Mwanga R O M, Leebens-Mack J, Lachlan J M, Coin G, Yencho G C, Buell C R, Fei Z J. Genome sequences of two diploid wild relatives of cultivated sweetpotato reveal targets for genetic improvement., 2018, 9: 4580.
Discovery and analysis of NBS-LRR gene family in sweet potato genome
HUANG Xiao-Fang1,2,**, BI Chu-Yun1,2,**, SHI Yuan-Yuan2, HU Yun-Zhuo3, ZHOU Li-Xiang4, LIANG Cai-Xiao4, HUANG Bi-Fang4, XU Ming1,2, LIN Shi-Qiang1,4,*, and CHEN Xuan-Yang1,2,5,*
1Key Laboratory of Crop Biotechnology, Fujian Agriculture and Forestry University, Fujian Province Universities, Fuzhou 350002, Fujian, China;2College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;3College of Plant Protection, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;4College of Life Science, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;5Key Laboratory of Genetics, Breeding and Multiple Application of Crops, Ministry of Education, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China
The NBS-LRR gene families possess the most abundant resistance genes in plants. Members of the NBS-LRR gene families contain nucleotide-binding site (NBS) and leucine-leucine repeat (LRR) domains. The genome of sweet potato () cultivar has been sequenced but the genes have not been annotated yet. In this study, we predicted the exons of sweet potato genome and obtained the proteins sequences, which were then used to identify and analyze genes of NBS-LRR family. There were 379 genes within NBS-LRR family, amounting to 0.212% of the total genes of sweet potato. The number of the N type, NL type, CNL type, TNL type and PN type was 120, 103, 133, 22, and 1, respectively. All of the chromosomes had NBS-LRR family genes but varied in number and 60.9% of them were clustered. NBS-LRR genes included 15 conservative domains and the genes were conservative within N terminal domain. The results provide references for further studies on the function of NBS-LRR family genes and resistance breeding of sweet potato.
; NBS-LRR;gene; gene family; bioinformatics
10.3724/SP.J.1006.2020.94163
本研究由福建省科技重大专项子专题(2017NZ0002-2)资助。
This study was supported by the Fujian Provincial Department of Science & Technology (CN)(2017NZ0002-2).
林世强, E-mail: linshiqiang@fafu.edu.cn, Tel: 059183789367; 陈选阳, E-mail: cxy@fafu.edu.cn, Tel: 059183789483
**同等贡献(Contributed equally to this work)
黄小芳, E-mail: 1102718600@qq.com, Tel: 059183789483; 毕楚韵, E-mail: 494028227@qq.com, Tel: 059183789483
2019-11-01;
2020-04-15;
2020-04-26.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200426.1548.024.html

