小黑麦NAC转录因子基因TwNAC01克隆及生物信息学分析
王萌,任丽彤,凌悦铭,谷海涛,孔广超*
(1 绿洲生态农业兵团重点实验室/石河子大学农学院,新疆 石河子 832003;2 石河子大学化学化工学院,新疆 石河子 832003)
转录因子(transcription factors,TFs)是一类与真核基因启动子区域顺式作用元件发生特异性作用的蛋白质[1],具有调节目的基因在特定时空表达的功能[2],在调控高等植物生长发育、形态建成、植物应对非生物胁迫以及生物应激反应等方面具有重要作用[3]。NAC转录因子是植物特有的一类转录因子,是NAM(No apical meristem)、ATAF(Arabidopsis transcription activation factor)和CUC(Cup-shaped cotyledon)的总称,其包含3个超家族[4]。首个NAC转录因子是从矮牵牛中克隆得到的[5],因矮牵牛中的NAM基因和拟南芥ATAF1/2和CUC2基因编码蛋白的N端均含有1段相同的氨基酸序列,因而取首字母命名为NAC[6]。目前,在拟南芥、水稻、小麦等物种中已相继发现了多个NAC转录因子[7-9]。NAC转录因子家族典型的特点是在其N端有1个由150个左右氨基酸组成NAC保守结构域,而C端则具有多样性,是转录激活或抑制功能域,主要在调节植物发育以及识别环境刺激等方面发挥作用[10]。NAC转录因子在植物抵御逆境胁迫方面具有重要作用,在高盐或干旱的条件下,拟南芥中NAC基因ANAC019表达量均上调[11]。过量表达ATAF1基因的拟南芥会出现明显的矮化和开花延迟现象,同时转ATAF1基因的拟南芥对干旱的耐性提高[12]。水稻中OsNAC10主要在根和花序中优势表达,并能被ABA、干旱和高盐诱导表达[13]。然而,迄今为止尚未见到在六倍体小黑麦有关NAC转录因子研究的公开报道。
小黑麦(×TriticaleWittmack)由小麦属(Triticum)和黑麦属(Secale)经属间有性杂交和染色体加倍培育而成的一种新型异源多倍体作物[14],不但保持了小麦籽粒产量高和品质优良的特性,还结合了黑麦抗逆性强的优势,是粮、饲兼用的一种新作物[15-16]。在研究[17-18]和生产实践中发现,六倍体小黑麦表现出较强的抗旱性,因此挖掘其抗旱基因对于理解小黑麦抗逆境机制以及基因工程育种将具有重要意义。
1 材料与方法
1.1 实验材料及处理
六倍体小黑麦品种新小黑麦3号[19],由石河子大学麦类作物研究所选育并提供。将新小黑麦3号种子清洗消毒后种植于石河子大学农学院实验站干旱胁迫池中,通过滴管技术控制灌溉量。实验设置3次重复。在小黑麦田间抽穗后对干旱胁迫区停止灌水,对照区正常进行灌溉,并为防止降雨对干旱胁迫效应的影响,在干旱胁迫区搭建遮雨棚,阻断降水。在开花期选取生育进程基本一致的植株进行标记。待干旱胁迫区小黑麦植株呈现叶片卷曲、土壤含水量、叶片游离脯氨酸、电导率均较对照明显升高时,采集干旱胁迫和正常灌溉下的小黑麦根、茎、旗叶和幼粒(开花后22 d),分别于液氮中迅速冷激后保存于-80 ℃冰箱备用,以供提取RNA进行基因克隆以及基因表达特点分析。
1.2 总RNA提取及cDNA反转录
以上述小黑麦根、茎、旗叶以及幼粒为材料,利用abm(广州)生物科技公司Hipure HP Plant RNA Mini Kit试剂盒提取小黑麦各组织总RNA,并经琼脂糖凝胶电泳及超微量紫外-可见光分光光度计(NanoDrop,ND-1000,USA)检测质量合格后,采用宝日医生物技术(北京)有限公司Prime ScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time)进行反转录合成cDNA。
1.3 基因全长cDNA序列克隆
在本实验室前期试验的基础上,以干旱胁迫后的小黑麦经RNA-Seq测序获得的1条显著上调表达的长度为708 bp的Unigene c51971的测序结果为基础,采用Primer Premier 5.0分别设计3′RACE引物:C065-1TGGGGGACCAGCAGACCGCGATC;C065-2TCGTCGGCGCTGCTGAGCCCTTC;5′RACE引物:B086-1CGGTCTTGTTGAGGTC;B086-2CTTGGGGGTGAGGTAGT,B086-3:GGTGGAACCGGAAGCC,以上述旗叶中cDNA为模板,经RACE扩增获得3′端和5′端序列,利用生物信息学软件CAP3进行全长拼接获得1 624 bp序列,在NCBI中预测基因的起始和终止密码子位置,获得ORF全长为1 052 bp。以拼接到的ORF全长序列为基础,设计全长扩增引物(上游:5′-TCAGATCTTCCACATGTTGG-3′;下游:5′-ATGTCGGACGTGACGGCGGTG-3′),以上述旗叶中cDNA为模板进行PCR扩增。扩增程序为:94 ℃,3 min;94 ℃,30 s;55 ℃,30 s;72 ℃,80 s;35 个循环,72 ℃,5 min。PCR产物经1.0%琼脂糖凝胶电泳后回收纯化,连接到pMD19-T Vector载体上转化TOPO10,经蓝、白斑筛选与PCR 鉴定、测序。
1.4 生物信息学分析
对已测序的序列采用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)在线查找序列的ORF开放阅读框和编码区CDS,并采用在线生物学软件Protparam(http://www.expasy.ch/tools/protparam.html)对该基因编码的氨基酸序列理化性质进行分析。利用ExPASy Prot Scale(http://web.expasy.org/protscale/)预测蛋白的疏水性。利用(https://www.ncbi.nlm.nih.gov/)进行超家族分析。通过DNAMAN软件将所克隆基因的氨基酸序列与同源性较高的NAC基因氨基酸序列进行多重比较分析。通过NCBI(http://www.ncbi.nlm.nih.gov/)的Blastn和Blastp搜索核苷酸和蛋白质数据库,进行序列同源性分析。利用TMHMM Server(http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜区域,借助TargetP 1.1 Server(http://www.cbs.dtu.dk/services/TargetP/)进行亚细胞定位。使用MEGA 5.0 软件采用相邻连接法(neighbor-joining,NJ)绘制系统发育树。利用 Signal(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽,利用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat)预测蛋白的二级结构。利用SWISS-MODEL(http://swissmodel.expasy.org/)构建该基因预测蛋白质三维结构模型。
1.5 基因表达特点检测
为明确该基因在小黑麦不同组织部位以及干旱胁迫下的表达特点,以开花期后22 d干旱胁迫和对照区小黑麦根、茎、旗叶和幼粒cDNA为模板,以所克隆基因的CDS序列(避开保守域)为基础、采用Oligo 7.0软件设计实时荧光定量引物(上游引物5′-CGTCGGCATGAAGAAGACGC-3′,下游引物5′-AGTCCTTGTCGAACACCCGG-3′),并以小麦Actin为内参基因进行实时定量PCR检测。采用TIANGEN公司SuperReal PreMix Plus(SYBR Green)试剂盒,以10 μL反应体系(2x SuperReal PreMixPlus 6 μL,上游引物0.25 μL,下游引物0.25 μL,cDNA模板1 μL,以RNase-free ddH2O补至10 μL),在Roche Light-Cycler480R仪进行qPCR扩增反应。qPCR扩增程序如下:预变性95 ℃,15 min;变性95 ℃,10 s;退火61 ℃,30 s;扩增72 ℃,30 s;40个循环。每个样品及内参基因做3次重复取平均值,采用2-△△Ct(Livak)法计算基因相对表达量[20]。
2 结果与分析
2.1 提取的总RNA质量
所提取的小黑麦根、茎、旗叶以及幼粒总RNA,经1.0%琼脂糖凝胶电泳检测,其28S与18S核糖体RNA条带清晰可见,且亮度比基本呈2∶1(图1),可见所提取的总RNA完整性较好,无DNA污染。经超微量紫外-可见光分光光度计检测,所提RNA的OD260/280均在1.8~2.0之间,无明显污染。

泳道1~4分别表示根、茎、旗叶、幼粒。图1 小黑麦总RNA电泳图
2.2 小黑麦基因全长cDNA
通过RACE技术分别获得的5′RACE序列为224 bp、3′RACE序列为557 bp。经在线拼接获得1条全长1 624 bp的序列。利用ORF Finder查找到的ORF全长为1 059 bp。以该ORF(1 059 bp)为基础通过PCR扩增获得了1条清晰的条带(图2)。将所获得的基因片段回收(图3左)并克隆到大肠杆菌中(图3右)、经测序结果表明实验获得了完整的1 059 bp序列CDS。
M—2 000 bp DNA markerⅡ;1~3为重复。图2 小黑麦基因全长cDNA序列PCR扩增

M—2 000 bp DNA markerⅡ;左为胶回收,1~5为重复;右为菌液PCR,1~6为重复。图3 基因回收及大肠杆菌菌液PCR
2.3 生物信息学分析
2.3.1 保守结构域
通过NCBI在线分析,该全长cDNA预测蛋白由352个氨基酸组成,其中第20~148个氨基酸序列之间有1个由129个连续氨基酸组成的结构域。该结构域与NAC转录因子家族NAM亚家族的保守结构域(PPGFRFHPTDEEVVTHYLTPKAVNNAFSCLVIADVDLNKTEPWDLPGKAKMGEKEWYFFVHKD RKYPTGTRTNRATEKGYWKATGKDKEIFRGKGRDAV LVGMKKTLVFYTGRAPRGDKTPYVMHEYRL)完全相同(图4)。

虚线方框标记部分为NAM超家族域。图4 预测蛋白保守域预测
通过DNAMAN软件将所克隆小黑麦TwNAC01氨基酸序列与NCBI中已知具有NAC保守结构域蛋白进行多序列比较,发现TwNAC01氨基酸序列与山羊草中含有NAC结构域的蛋白XP 020161331同源性达到了95%以上,且与具有NAC保守域的大麦蛋白CBZ41151、KAE8777325及KAE8777329同源性也达到83.26%。尤其是在其N端第20~148个氨基酸之间有着高度一致的NAC保守的氨基酸序列(图5),在其C端第175~187、197~239、305~327个氨基酸分别有AAKNAPPPMAPAA、FLDVDDFLNNPDLLNNADLPMLMDSPSGADDFAGASSSTSS AA、AASSSALLSPSLGFDAGALAGAD 3个转录激活区。据此,可确认所克隆基因为小黑麦NAC基因,将其命名为TwNAC01,并在GenBank注册,登录号为MG736919。

蓝色表示50%相似度,红色表示75%相似度,黑色表示100%相似度,红色框部分为保守氨基酸序列,绿色框部分为转录激活区。图5 TwNAC01预测氨基酸序列比对
2.3.2 系统进化
通过MEGA 5.0中的ClustalW比对,采用相邻连接法(Neighbor-joining,NJ)绘制了小黑麦TwNAC01预测蛋白与NCBI中相似性较高蛋白的系统进化树,结果显示小黑麦TwNAC01预测预测蛋白与山羊草中含有NAC结构域的蛋白XP020161331同源性最高(图6),其次是与大麦中的NAC转录因子CBZ41151、KAE8777325以及GRAB2 蛋白(KAE8777329)同源性也较高,再次是小麦中的GRAB2蛋白(CAA09372)与粗山羊草中的含有NAC结构域的蛋白(XP020182284、XP020166616、XP020166615、XP020166614),同源性再低一点的还有大麦GRAB2蛋白(KAE8782198)以及渐尖二型花(Dichantheliumoligosanthes)含有NAC结构域的蛋白(OEL23749)以及高粱中的含有NAC结构域的蛋白(XP002461233),与其同源性最远的是小麦中含有NAC结构域的蛋白(AVG22584)。由此推断该小黑麦TwNAC01预测蛋白就是NAC转录因子。
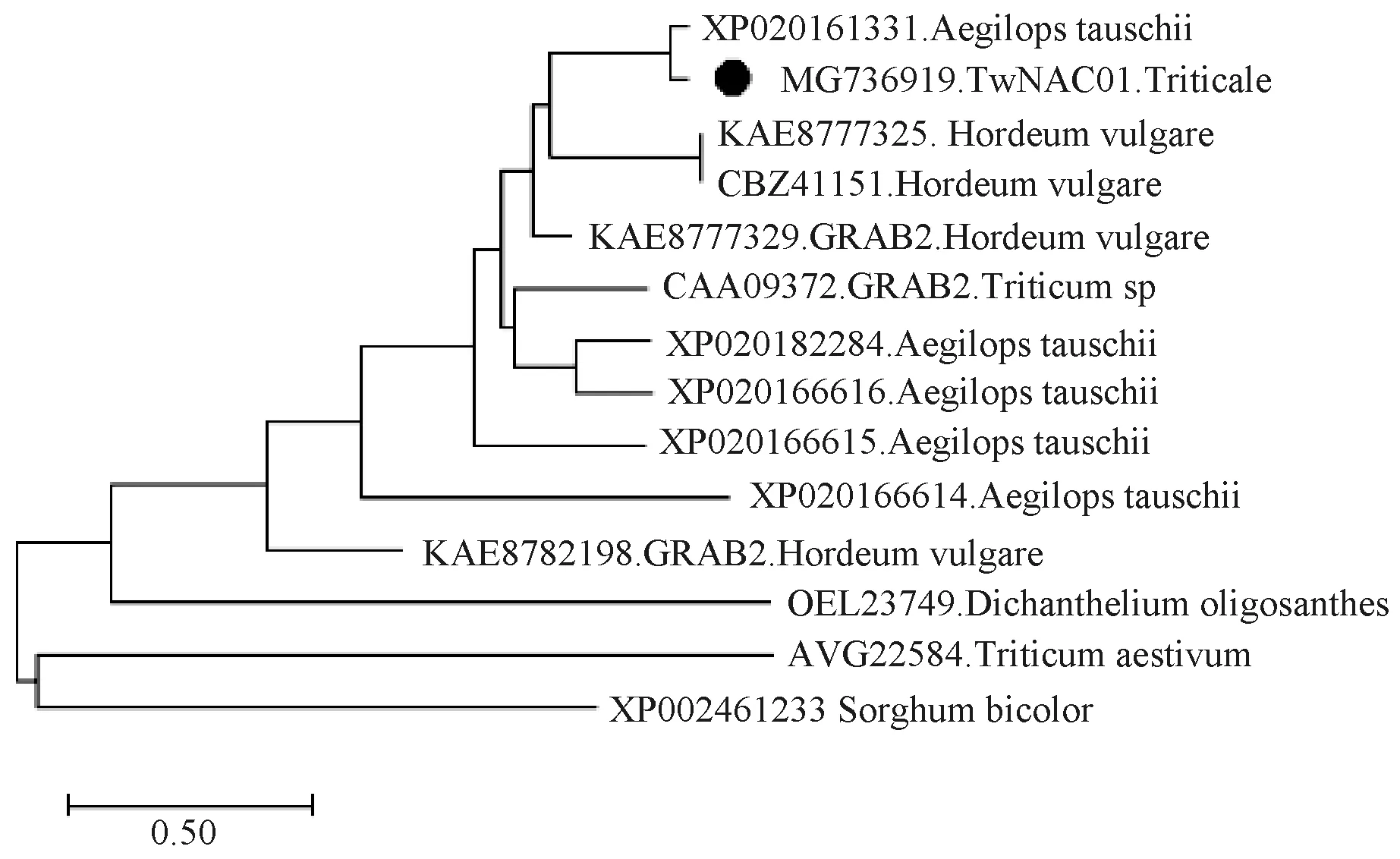
图6 TwNAC01预测蛋白系统进化树
2.3.3 蛋白质理化性质
采用Protparam预测到该基因的蛋白分子式为C1722H2642N464O522S19,相对分子质量为38 805.86,理论等电点为5.44,正/负电荷残基总数分别为46、37,消光系数46 996,在280 nm波长处吸光度值为1.211。
采用Protparam预测到该蛋白中含有碱性氨基酸共45个,其中21个精氨酸(占6.0%)、16个赖氨酸(占4.5%)、8个组氨酸(占2.3%);包含酸性氨基酸共85个,其中28个天冬氨酸(占8.0%)、18个谷氨酸(占5.1%)、36个丙氨酸(占10.2%)、3个半胱氨酸(占0.9%),故此蛋白为偏中性。同时预测到该蛋白的不稳定指数为45.06,脂肪指数为63.27。根据Guruprasad方法判定该蛋白质不稳定。利用ExPASy Prot Scale在线预测该蛋白总平均疏水系数-0.494,为亲水性蛋白(图7)。
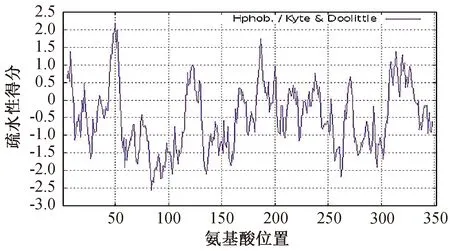
亲水、疏水性分别用负值、正值表示。图7 TwNAC01预测蛋白亲水/疏水性
2.3.4 跨膜区和信号肽
利用TMHMM Server和Signal对TwNAC01预测蛋白的跨膜区和信号肽特点预测表明,该蛋白信号肽S值在1~61个氨基酸之间,预测值为0.096,其中最大值在第54个氨基酸,预测值为0.121,其中剪切位点(C)在第62个氨基酸,最大值为0.113,综合剪切位点(Y值)在第62个氨基酸,最大值为0.107,SP信号肽显示NO。因此,可推断该蛋白中不含信号肽,无蛋白运转结构。TMHMM分析结果也显示TwNAC01预测蛋白不存在跨膜结构,属于非膜蛋白及非分泌蛋白。
2.3.5 亚细胞定位
利用在线软件TargetP 1.1 Server对小黑麦TwNAC01基因的氨基酸序列亚细胞定位分析表明(表1),该序列叶绿体转运肽(CTP)分值0.090、线粒体转运肽(MTP)分值0.104和信号肽(SP)分值0.171,这些分值均较小,而预测为其他类型的分值为0.887,目的蛋白的分泌途径为“_”型,即该蛋白定位在除叶绿体、线粒体、信号肽外的其它细胞器上。同时结合软件ProtComop分析确定小黑麦TwNAC01蛋白定位在细胞核中预测值为8.26、细胞膜预测值为1.01、细胞外预测值为0.04、线粒体预测值为0.01、叶绿体预测值为0.11、液泡预测值为0.48,可见,定位于细胞核中的预测值最高。综上分析,可断定该蛋白亚细胞定位在细胞核中。

表1 亚细胞定位分值
2.3.6 二级结构
利用SOPMA对小黑麦TwNAC01预测蛋白的二级结构预测表明,TwNAC01预测蛋白中α-螺旋(H)、β-折叠(E)、无规则卷曲(C)组成分别占12.78%、16.19%、71.03%含量极高,故认为该小黑麦NAC蛋白属于C型(图8)。

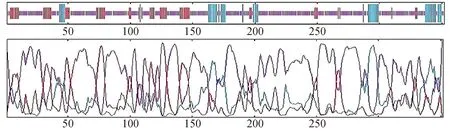
H为α 螺旋(蓝色);E为延伸链(红色);C为无规则卷曲(黄色)。图8 TwNAC01基因编码蛋白的二级结构预测
2.3.7 三级结构
利用SWISS-MODEL对小黑麦TwNAC01蛋白的三级结构模拟显示,小黑麦TwNAC01蛋白与水稻中NAC1模型3ulx.1.A蛋白一致性达到52.87%,由空间立体结构可以看出TwNAC01蛋白主要由α-螺旋和无规则卷曲(C)组成,且无规则卷曲含量高(图9)。

图9 小黑麦TwNAC01蛋白三级结构
2.4 TwNAC01表达特点
为了明确TwNAC01基因在小黑麦不同器官间以及干旱胁迫前后的表达特点,对开花后22 d时的干旱胁迫和对照新小黑麦3号的根、茎、旗叶和幼粒中该基因表达量分别进行了检测。结果如图10所示,在干旱胁迫下,小黑麦TwNAC01基因在开花后22 d 时的根、茎、旗叶以及幼粒的中表达量均较对照明显上升,其中在根和幼粒中表达量较对照呈极显著上调表达;同时该基因在茎和叶中的表达量明显低于根与幼粒。可见,小黑麦TwNAC01基因在受到干旱胁迫时会显著上调。

采用LSD-T法进行显著性检验结果;*、**分别表示干旱处理和对照差异在0.05、0.01水平上显著。图10 干旱胁迫下TwNAC01基因在小黑麦不同组织器官的表达量
3 讨论
NAC是目前所知的植物所特有、成员数量最多的转录因子家族之一[14-15,21-22],不但参与花器官发育、叶片衰老以及植物次生壁合成[16,23],还参与植物应对非生物胁迫反应[10,21,24]。本研究结合RNA-seq和RACE技术克隆了六倍体小黑麦NAC转录因子基因TwNAC01,在GenBank中注册为MG736919。该基因编码区全长1 059 bp,预测编码352个氨基酸。TwNAC01预测蛋白的第20~148氨基酸间有1个由129个连续氨基酸组成的NAM亚家族的保守结构域。同时该基因预测氨基酸序列与山羊草中包含NAC结构域的蛋白XP020161331(https://www.ncbi.nlm.nih.gov/protein/XP_020161331)同源性可达到95%以上,与大麦NAC转录因子(CBZ41151、KAE8777325以及KAE8777329)同源性达83%。CBZ41151是大麦中参与植物细胞次生壁合成、叶片衰老、根系发育、种子萌发有关,同时受ABA和MeJA诱导上调表达的大麦NAC转录因子[25],蛋白KAE8777325是大麦NAC转录因子(https://www.ncbi.nlm.nih.gov/protein/KAE8777325)。KAE8777329蛋白大麦中GRAB2蛋白(https://www.ncbi.nlm.nih.gov/protein/KAE8777329),与其同源性较高的蛋白CAA09372也是小麦NAC转录因子(GRAB2)[26]与粗山羊草中的含有NAC结构域的蛋白(XP020182284(https://www.ncbi.nlm.nih.gov/protein/XP_020182284)、XP020166616(https://www.ncbi.nlm.nih.gov/protein/XP_020166616)、XP020166615(https://www.ncbi.nlm.nih.gov/protein/XP_020166615)、XP020166614(https://www.ncbi.nlm.nih.gov/protein/XP_020166614))。由此推断该小黑麦TwNAC01预测蛋白就是NAC转录因子。
自首个NAC转录因子基因在矮牵牛中被克隆后[27],关于NAC基因表达模式与功能的研究相继报道,如小麦NAC基因TaNAC2和TaNAC67过量表达可明显提高拟南芥对干旱、盐和低温胁迫耐性[28];过量表达小麦NAC基因(TaNAC2a、TANAC4a、TaNAC6、TaNAC7、TaNAC13和TaNTL5)能明显提高烟草对干旱的耐性[29]、在拟南芥中过表达ATAF1基因后,其植株蒸腾速率降低、耐旱性能提高[30];过表达OsNAP的水稻在营养生长期水分损失率更低,对盐、干旱和低温胁迫的耐性增强,同时对外源ABA响应敏感性增加[31]。本研究所克隆的小黑麦NAC基因TwNAC01在小黑麦受到干旱胁迫后表达量显著增强,特别是在根和幼粒中表达量升高显著。在本实验室前期的转录组测序中也发现该基因在干旱胁迫后表达量较对照提高4倍以上。这与研究[29-30,32]发现NAC基因表达受干旱诱导明显升高的结论相一致。同时本研究发现该基因在开花后22 d的小黑麦根部表达量高于旗叶和茎部,这也与小麦基因TaNAC4在根中较叶片和茎中表达量高的规律相一致[33]。这表明所克隆的小黑麦基因TwNAC01受干旱胁迫诱导,并在小黑麦响应干旱胁迫反应中具有重要作用。
通过生物信息学分析,预测到的TwNAC01蛋白定位在细胞核中。这在我们的后继研究中也已经通过生物学实验手段验证,该蛋白确定定位于烟草表皮细胞核中。生物信息学预测TwNAC01蛋白属于亲水性蛋白,无跨膜结构,与康美玲等[34]研究结果一致。
NAC转录因子在植物对干旱胁迫反应中的机理研究也已有了较大进展,这为理解植物抗旱机制与NAC转录因子生物功能提供了理论依据,例如,干旱诱导水稻保卫细胞中NAC转录因子调控OsSRO1c基因的过量表达,从而促进了保卫细胞中H2O2积累、减少完全开放气孔的数量[35]、减少可腾水分散失[36]。水稻中SNAC3转录因子靶基因具有活性氧清除功能,过量表达SNAC3能增加活性氧清除基因的表达[37]。此外,NAC蛋白的核心结合区还调控多个与胁迫相关的基因,增强植物的耐旱性[38]。这些研究均为TwNAC01转录因子的具体作用机理和小黑麦耐旱机制研究奠定了基础。

