基于Illumina Hiseq 4000高通量测序平台的暗色唇鲮线粒体基因组特征分析
韩耀全 黄励 朱琼华 吴伟军 何安尤 施军 李育森
摘要:[目的]研究暗色唇鲮(Semilabeo obsodrus)的分子遗传学特性,为暗色唇鲮种质资源遗传多样性分析和分子标记研究提供基础数据。[方法]利用高通量第二代测序技术,构建暗色唇鲮线粒体基因组全序列,分析序列的结构特征。[结果]暗色唇鲮的线粒体基因组全序列长度16598~16599bp,GC含量42.65%~42.7%,共含有13个蛋白编码基因、22个tRNA、2个rRNA(12s-rRNA和16S-rRNA)以及1个控制区。线粒体基因组除ND6外,GC-skew值均为负值,COXI、ND3以及ND6的AT-skew值为负值,其余基因的AT-skew值均为正值。暗色唇鲮的13个蛋白编码基因氨基酸总长为3794aa,偏好密码子(RSCU>2)分别是终止密码子Arg(CGA)、Gly(GGA)、Leu(CTA)、Pro(CCA)、Ser(TCA)以及终止密码子Stp(TAA)。暗色唇鲮(唇鲮属Semilabeo)与泉水鱼PJeudogyrinocheilusprochilus的亲缘关系最近,和卷口鱼Ptychidiojordani、直口鲮Rectorisposehensis、异华鲮Parasinilabeoassimilis存在较高的亲缘关系。[结论]暗色唇鲮的线粒体基因组结构和基因排列顺序与鱼类基因组典型结构和排列一致。
关键词:暗色唇鲮;线粒体基因组;结构特征;系统发育
中图分类号:Q 173;Q959.46+8文献标志码:A 文章编号:1008-0384(1020)02-0130-10
0 引言
(研究意义)暗色唇鲮(Semilabeo o℃ums Lin,1981)系鲤形目Cypriniformes鲤科Cyprinidea野鲮亚科Labeoninae唇鲮属Semilabeo Peters鱼类,地方名猪嘴鱼、假没六鱼、马鼻勾等。仅分布于珠江水域及红河水域干支流,包括我国广西、贵州、云南及越南部分地区,是优质、上等经济鱼类,已被列为《中国濒危动物红皮书》易危和《世界自然保护联盟(IUCN)》濒危物种,受威胁程度较高。随着保护生物多样性意识的提高,珍稀唇鲮属鱼类的资源保护和增殖研究工作受到重视。近年来,线粒体基因组研究技术日趋成熟,其结构简单、编码区域高度保守、重组率低、拷贝数高等特征使其逐渐成为研究鱼类群体遗传结构、系统进化关系和开展鱼类分子标记的首选载体。通过研究暗色唇鲮的线粒体基因组序列结构特征,可为暗色唇鲮种质资源多样性分析和分子标记研究提供基础数据。(前人研究进展)唇鲮在驯养繁殖等方面已取得了丰富的研究成果,暗色唇鲮在驯养繁殖方面的研究工作也相继开展并取得少量成果,但研究报告仅见关于暗色唇鱼精子冷冻保存研究及表型形态分析,或仅为相关鱼类志的收录介绍,或仅是对暗色唇鲮濒危程度的分析、水域渔业资源调查的捕获记录等。线粒体基因组作为研究DNA复制和转录的良好模型,在遗传和进化领域分子标记具有较高应用价值,近年来被广泛应用于生物学的许多领域。司从利等对暗色唇鲮亲缘关系较近的泉水鱼Pseudogyrinocheilus prochilus开展了线粒体方面的相关研究。(本研究切入点)关于暗色唇鲮分子遗传学的研究成果较少,目前未见其线粒体基因组特征相关研究。(拟解决的关键问题)本研究利用高通量第二代测序技术进行测序,组装与注释暗色唇鲮线粒体基因组序列,构建系统发育进化树,并分析其序列结构特征及相关参数。
1 材料与方法
1.1 试验材料
根据暗色唇鲮的分类鉴定依据,分别随机选取在云南省曲靖市红水河上游支流(YN)、贵州省黔西南州红水河上游支流(GZ)、广西壮族自治区河池市柳江上游支流(GX)采集获得的鲜活暗色唇鲮中各1例,测量表型性状及相关数据后,采集尾鳍并用去离子水冲洗,置于无水乙醇中-20℃冰柜保存备测。
1.2全基因组DNA提取
3例暗色唇鲮全基因组DNA的提取依据北京康为世纪生物科技有限公司的海洋动物基因组DNA提取试剂盒(CW2089)说明流程进行。提取后的DNA采用l%琼脂凝胶电泳检测基因组DNA的纯度和完整性,Qubit.0Flurometer(Life Technologies,CA,USA)检测样品浓度。
1.3 全基因组二代测序
检测合格的DNA样品分别采用Covaris超聲波破碎仪随机分割成长度为350bp左右的片段,构建小片段基因组DNA测序文库,文库构建流程参照NEB Next Ultra DNA Library Prep Kit for Illumina(NEB,USA)文库构建试剂盒说明书进行。文库构建完成后以qPCR方法和Agilent 2100Bioanalyzer进行质控。对质检合格的DNA文库采用Illumina Hiseq4000高通量测序平台进行测序,测序策略为PEl50(Pair-End 150),每例样品测序数据量不少于2Gb.
1.4 线粒体基因组的组装与注释
1.4.1线粒体基因组组装 Illumina HiSeq 4000测序平台所得原始下机序列,称之为Raw reads,通过去低质量序列、去接头污染等过程完成数据处理得到高质量的序列,称之为Clean reads.去低质量序列的判断原则:当任一测序read中N含量超过该read碱基数的10%时,去除此Paired reads;当任一测序read中含有的低质量(Q≤5)碱基数超过该条read碱基数的50%时,去除此Paired reads.后续所有分析都是基于Clean reads进行。根据序列的overlap关系和插入片段的大小关系,采用SPAdes v.3.5.0软件(http://cab.spbuxu/software/spades/)对高通量测序的短片段序列(Clean reads)进行拼接组装,最终得到完整的暗色唇鲮线粒体基因组序列。序列结果SQN文件已上传至NCBI数据库,YN、GZ及GX暗色唇鲮的GeneBank登录号分别为:MN229747、MN229748、MN229749.
1.4.2线粒体基因组注释利用DOGMA (http://dogma.ccbb.utexas.edu/)和ORF Finder(https://www.ncbi.nlm.nih.gov/orffmder/)对线粒体基因组进行注释。对注释的初步结果,运用Blastn和Blastp(https://blast.ncbi.nlm.nih.gov/Blast.cgi)的方法与已报道的近缘物种线粒体基因组编码蛋白和rRNA进行比对,验证结果的准确性并进行修正。tRNA的注释采用tRNAsean-SE2.0(http://lowelab.ucsc.edu/tRNAscan-SE/)和ARWEN(Versionl.2http://mbio-serv2.mbioekol.lu.se/ARWEN/)进行,舍去长度不合理和结构不完整的tRNA,并生成tRNA二级结构图。GC偏移率(GC-skew)和AT偏移率(AT-skew)分别采用如下公式计算:GC-skew=(G-C)/(G+C),AT-skew=(A-T)/(A+T)。相对同义密码子使用度(RSCU:Relative synonymous codonusage)计算参照Sharp PM等的公式进行分析。
1.5系统进化树构建
目前已公开发布的野鲮亚科(Labeoninae)鱼类约有26个属近300个种。研究以NCBI(https://www.nebi.nlm.nih.gov/)上已发布完整线粒体基因组的野鲮亚科的10个属18个种进行系统进化树构建。选择编码基因的核酸序列和蛋白序列分别采用MUSCLEv.3.8.31(http://www.drive5.com/muscle/)软件进行多物种间单个基因的多序列比对。采用jModelTest2.1.7(https://code.google.com/p/jmodeltest2/)软件对所选序列DNA进行核酸模型测试,AIC最小值为最佳模型。采用RAxML8.1.5(https://sco.h-its.org/exelixis/web/software/raxml/index.html)软件,最大似然法(Maximum Likelihood method,ML法)构建系统发育树,Bootstrap值设置为1000.
2 结果与分析
2.1 线粒体基因组的组成及其特征
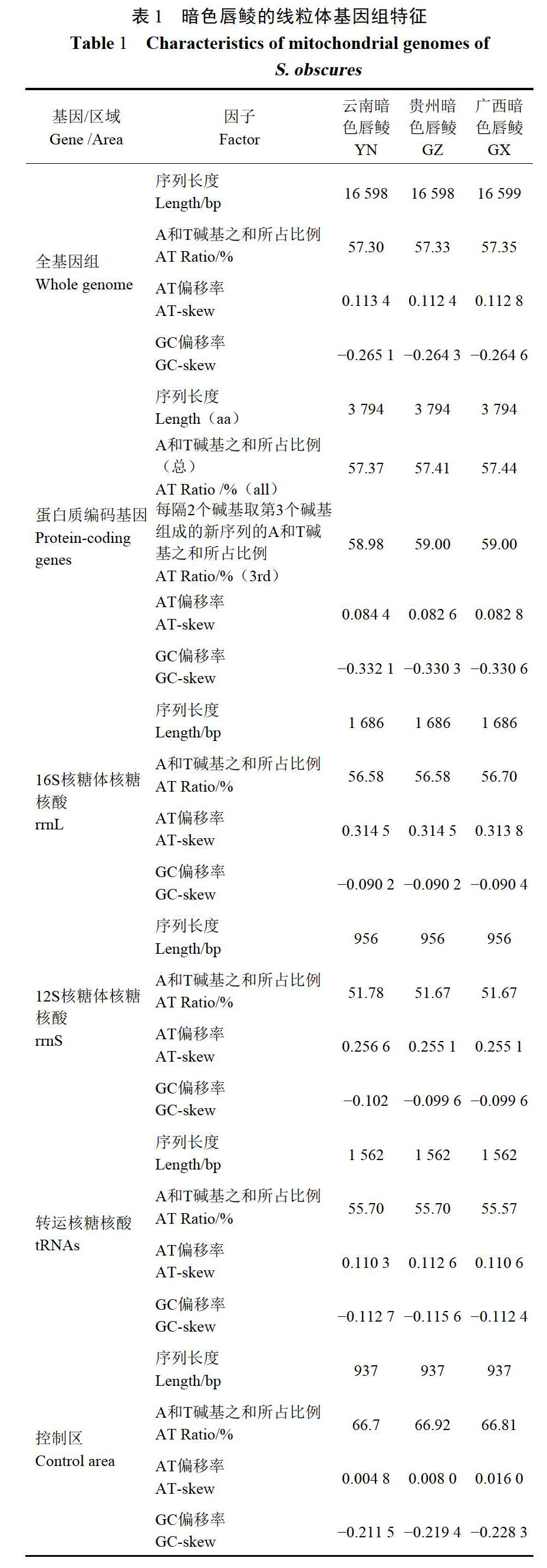
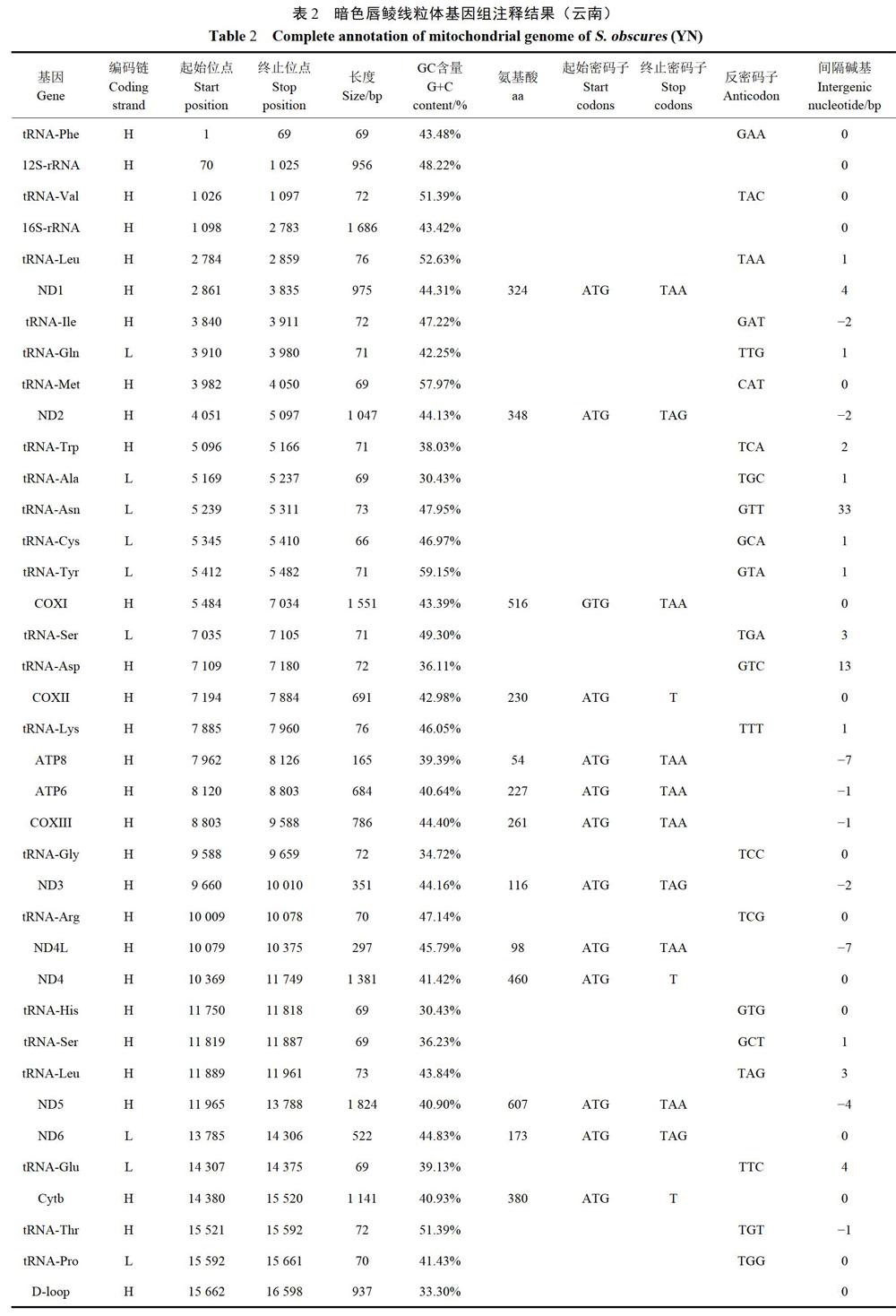
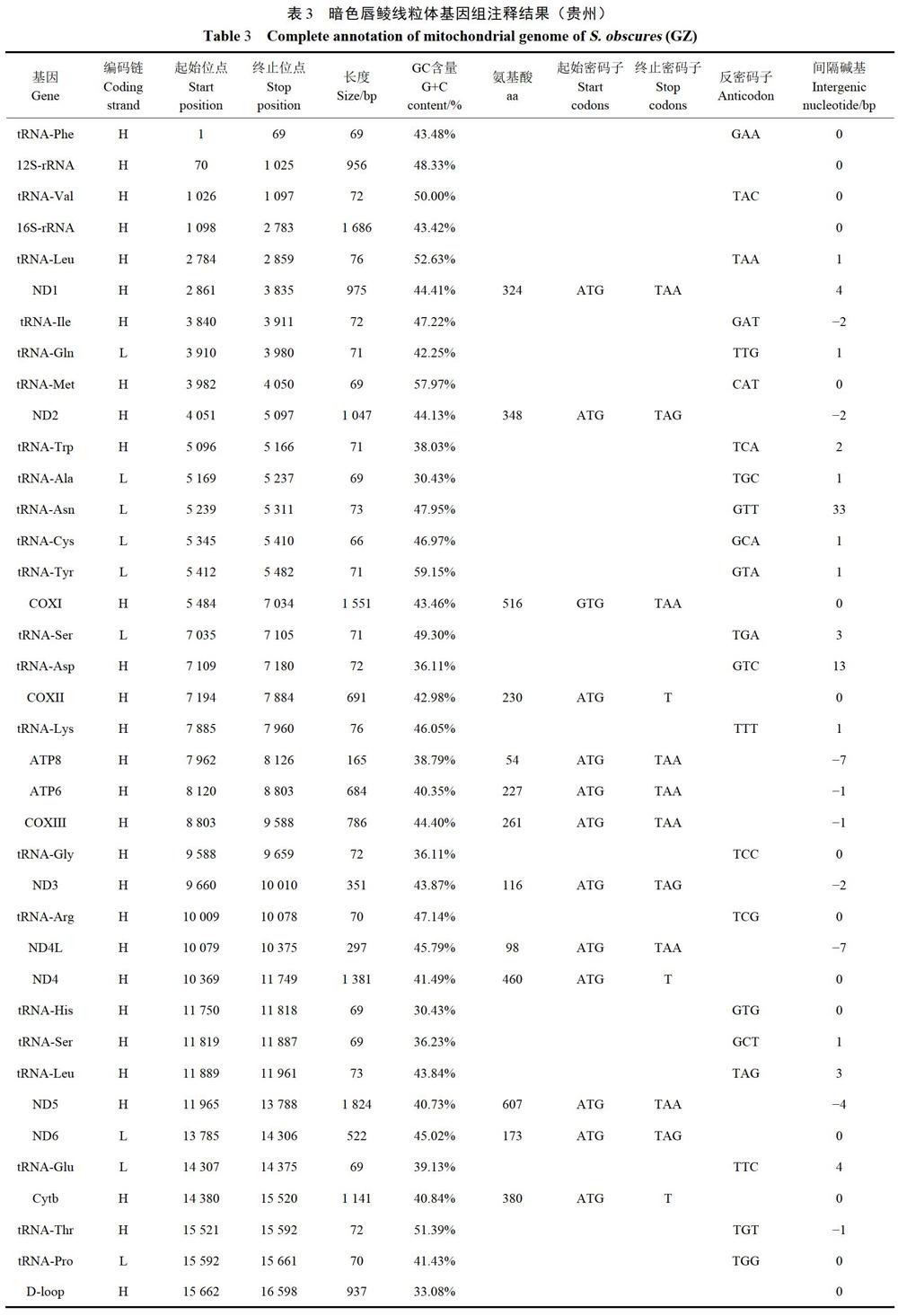
3例暗色唇鲮线粒体全基因组高通量测序,最终组装得到的线粒体基因组全序列长度,以及G碱基与C碱基占总碱基数的比例分别为:16598bp和42.7%(YN)、16598bp和42.66%(GZ)、16599bp和42.65%(GX),均含有13个蛋白编码基因,22个tRNA,2个rRNA(12S-rRNA和16S-rRNA)以及1个控制区。3例物种的线粒体基因组除ND6外,GC-skew值都为负值,COXI、ND3以及ND6的AT-skew为负,其余基因的AT-skew值均为正值(表1-4)。暗色唇鲮22个tRNA长度66~76bp不等,除tRNA-Ser(GCT)外,其他21个tRNA都具有典型的三叶草结构,其线粒体基因组结构和基因排列顺序与大部分的鱼类基因组典型结构和排列一致(图1)。
2.2密码子使用频率(RSCU)
不同生物乃至同一物种不同基因间其同义密码子的使用频率存在差异,具有一定的偏爱性,暗色唇鲮线粒体密码子的使用同样具有偏好性(CodonUsage Bias)。暗色唇鲮13个蛋白编码基因氨基酸总长为3794aa,偏好密码子(RSCU>2)分别是终止密码子Arg(CGA)、Glv(GGA)、Leu(CTA)、Pro(CCA)、Ser(TCA)以及终止密码子Sip(TAA)。暗色唇鲮的偏好密码子使用情况见表5和图2.
2.3系统进化分析
该研究系统发育树的构建核苷酸采用的最优模型是GTR+I+g.进化树结果显示目标物种暗色唇鲮与NCBI已公布的暗色唇鲮Semilabeo ObSCHrHS(NC037408)以及泉水鱼Pseudogyrinocheilusprochilus(NC024588)聚为一支(图3)。物种进化树分析结果显示暗色唇鲮(唇鲮属Semilabeo)与泉水鱼Pseudogyrino-cheilusprochilus的亲缘关系最近,和卷口鱼Ptychidiojordani、直口鲮Rectorisposehensis、异华鲮Parasinilabeoassimilis的亲缘关系较近。该结论与相关鱼类志对暗色唇鲮所属的分类结果相吻合。
3 讨论
3.1 暗色唇鲮的线粒体基因组特征
对暗色唇鲮的线粒体基因组的测序分析显示,3例物种线粒体相似性在99.67%~99.78%,基因组十分保守,在全基因組范围内序列高度相似,这与其他鱼类线粒体基因组的保守性研究结果一致。3例暗色唇鲮蛋白序列、tRNA长度、rRNA长度以及control region区长度均一致。13个蛋白编码基因同大多数硬骨鱼类一样含有ATG和GTG两种起始密码子,COXl基因使用GTG作为起始密码子,而其他12个基因的起始密码子均为ATg.3例暗色唇鲮的终止密码子也是2种,分别为TAA和TAG,其中ND2、ND3、ND6采用TAG为终止密码子,NDl、COXI、ATP8、ATP6、COXIII、ND4L以及ND5这6个基因采用TAA为终止密码子,其余3个基因(COXII,ND4和Cytb)使用不完整的T作为终止密码子,这类含有不完整终止密码子的基因将在转录后加工的过程中加入polyA,从而构成完整的“TAA”终止密码子,线粒体基因组结构和基因排列顺序与大部分典型的脊椎动物鱼类一致。
3.2 不同地域暗色唇鲮的线粒体基因组差异探析
由于线粒体具有高度保守性,COXI、CYTB等基因常用作分子标记用于鱼类物种的鉴定,线粒体基因组序列除了用于物种鉴定外,在种质资源多样性分析方面也应用广泛,该研究测序分析的3个样品分别来自相距甚远的3个省区不同水域,经线粒体全基因组差异比较分析,云南水域和贵州水域样品之间有51个SNP差异,不存在插入和缺失突变,序列相似性99.69%。云南水域和广西水域样品之间存在54个SNP和1个碱基插入缺失,序列相似性99.67%。贵州水域和广西水域样品之间存在37个SNP和1个碱基插入缺失,序列相似性99.78%。该研究3例暗色唇鲮的所有编码基因都存在一定的个体差异,推测暗色唇鲮在不同地理间具有一定的差异性,这些可作为暗色唇鲮种质资源多样性分析的重要分子标记。由于该研究样品稀有,测试案例有限,后续有待对更多不同地理位置的物种进行测序,以期得到更加丰富和科学可靠的分子标记。

