BEAMing ddPCR与Super ARMS检测EGFR酪氨酸激酶抑制剂治疗后非小细胞肺癌患者循环肿瘤DNA EGFR基因突变的比较研究
复旦大学附属肿瘤医院病理科,复旦大学上海医学院肿瘤学系,复旦大学病理研究所,上海 200032
循环肿瘤DNA(circulating tumor DNA,ctDNA)检测优点颇多,近年来备受关注[1]。但由于游离核酸(circulating free DNA,cfDNA)大量来源于正常细胞,且有研究表明,绝大多数肺癌和结直肠癌患者在治疗前ctDNA浓度低于0.5%,因而临床检测中对灵敏度就提出了更高的要求[2-3]。目前临床用于ctDNA检测的主要技术包括微滴数字PCR(droplet digital PCR,ddPCR)、扩增阻碍突变系统(Super amplification refractory mutation system,Super ARMS)、二代测序技术等。BEAMing ddPCR和Super ARMS两种技术分别以0.01%和0.20%的突变丰度为最低检测限显示出较高的灵敏度优势,但目前仍缺乏实际病例对照研究证据。本文旨在探讨BEAMing ddPCR和Super ARMS检测表皮生长因子受体(epidermal growth factor receptor,EGFR)酪氨酸激酶抑制剂(tyrosine kinase inhibitor,TKI)治疗后非小细胞肺癌(non-small cell lung cancer,NSCLC)患者ctDNAEGFR基因突变的灵敏度差异,进而为指导临床ctDNA检测方法的选择提供更多参考。
1 材料和方法
1.1 样本来源
33例为复旦大学附属肿瘤医院2017—2018年接受过EGFR TKI治疗后的NSCLC患者血浆,10例为国家病理质控评价中心(Pathology Quality Control Center,PQCC)室间质评项目模拟血浆(Horizon定制标准品)。
1.2 cfDNA的提取
采用凯杰企业管理(上海)有限公司的QIAamp游离核酸提取试剂盒提取,流程简述如下:将200 μL蛋白酶K、1.6 mL ACL和carrier RNA混合液加入到2.0 mL血浆中,置于60 ℃水浴锅裂解30 min;加入3.6 mL ACB裂解液,置于冰水浴中裂解5 min;利用真空泵负压过滤,用600 μL ACW1、750 μL ACW2及750 μL无水乙醇洗涤;烘干后洗脱并收集cfDNA。
1.3 BEAMing ddPCR
采用SysmexOncoBEAM EGFR试剂盒进行EGFR基因突变检测:①多重PCR,以待测cfDNA和对照品为模板,加入多重PCR反应液,运行PCR程序,98 ℃ 30 s;98 ℃ 10 s,60 ℃ 10 s,72 ℃ 10 s,共25个循环;72 ℃15 s;4 ℃保持温度。② 巢式PCR,以多重PCR产物为模板,加入巢式PCR反应液,运行PCR程序,98 ℃ 30 s;98 ℃ 10 s,60 ℃10 s,72 ℃ 10 s,共5个循环;98 ℃ 10 s,72 ℃ 10 s,共10个循环;72 ℃ 15 s;4 ℃保持温度。③乳液PCR,加入BMX液,乳化珠以及乳液聚合酶,运行P C R程序,9 4 ℃2 min;94 ℃ 10 s,68 ℃ 50 s,70 ℃ 80 s,共3个循环;94 ℃ 10 s,59 ℃ 50 s,72 ℃80 s,共50个循环;72 ℃ 2 min;4 ℃保持温度。④ 裂解与杂交,加入杂交探针,于PCR仪中运行程序70 ℃ 30 s;梯度PCR至24 ℃1 min。⑤ 流式细胞术分析及解读,PBS洗涤杂交产物,采用Cube 6i流式细胞仪进行分析,阳性判读标准为突变率≥阈值(amplicon19和21阈值为0.03%,amplicon 20阈值为0.04%)并且MB圈中的突变珠粒聚集,与阳性对照相似的位置/形状,输入DNA(GE)*突变率≥0.5。
1.4 Super ARMS
采用厦门艾德生物医药科技股份有限公司的人类EGFR突变基因检测试剂盒进行EGFR基因突变检测。流程简述如下:①以待测cfDNA和对照品为模板,加入反应液和酶,混匀加入8联反应条中。② 于实时荧光定量PCR(real-time fluorescence quantitive PCR,RTFQ-PCR)仪中运行程序,95 ℃ 10 min;95 ℃ 40 s,64 ℃ 40 s,72 ℃ 30 s,共15个循环;93 ℃ 40 s;60 ℃ 45 s;72 ℃ 30 s共28个循环收集荧光信号。③阳性判读标准为:ΔCt值<ΔCtCut-off值且内控HEXCt值<19。
1.7 统计学处理
采用GraphPad Prism 6.0软件进行χ2检验和非配对t检验分析,P<0.05为差异有统计学意义。
2 结果
2.1 所有样本检测结果比较
综合33例NSCLC血浆和10例PQCC室间质评项目模拟血浆应用BEAMing ddPCR和Super ARMS检测EGFR 19 del、T790M及L858R突变结果(表1~2),两种方法检测敏感突变EGFR 19 del及L858R结果一致率分别为90.7%(39/43)和97.7%(42/43)。19 del结果不一致的样本数为4例,其中2例ddPCR结果为阳性的样本Super ARMS未检出,另2例Super ARMS结果为阳性的样本ddPCR未检出;L858R结果不一致的样本数为1例,为Super ARMS检出而ddPCR未检出。两种方法检测T790M耐药突变结果一致率为74.4%(32/43),其中10例Super ARMS检测为阳性的样本ddPCR均检出,而有11例ddPCR检测为阳性的样本Super ARMS未检出,其余结果一致。
2.2 33例NSCLC样本检测结果比较
ddPCR和Super ARMS检测33例NSCLC样本中EGFR敏感突变阳性率均为27.3%。而在耐药突变的检测中,ddPCR检出阳性率为42.4%,Super ARMS检出阳性率为27.3%(表1)。
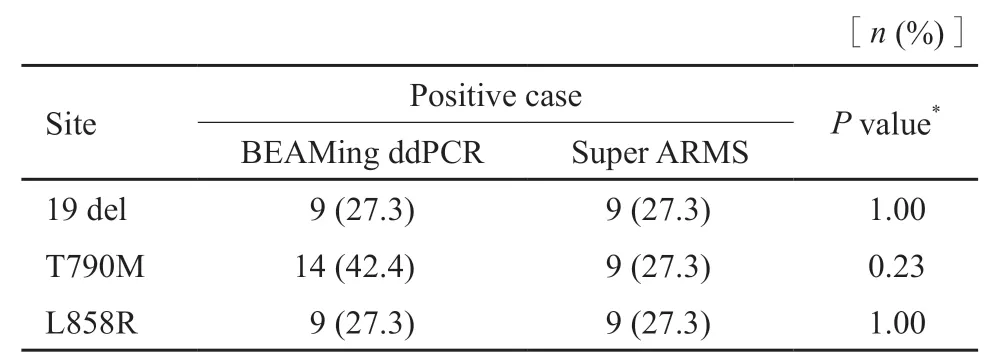
表1 33例NSCLC TKI耐药后血浆样本检测结果Tab.1 Results and positive rates of 33 NSCLC samples
2.3 10例PQCC质控样本检测结果比较
ddPCR和Super ARMS检测10例PQCC质控样本中19 del阳性率均为30.0%(P=1,表2),符合率均为100.0%(3/3);L858R阳性率分别为20.0%和30.0%(P=0.5,表2),符合率分别为66.6%(2/3)和100.0%(3/3)。而在T790M检测中,ddPCR检出阳性率为70.0%,Super ARMS检出阳性率为10.0%(P=0.01,表2),符合率分别为100.0%(7/7)和14.3%(1/7)。
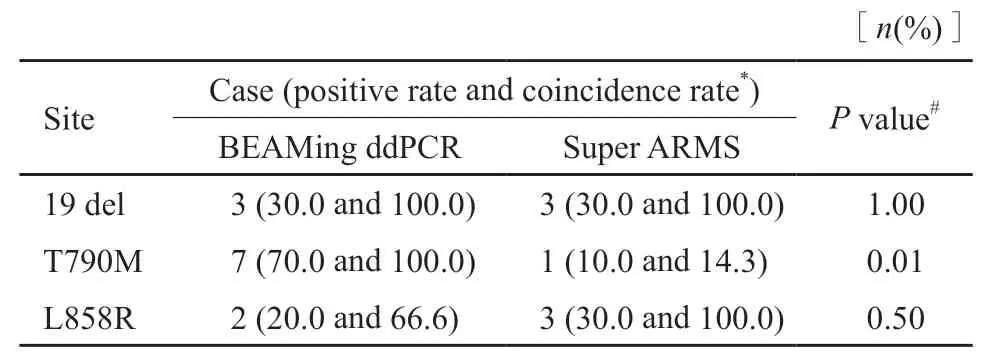
表2 10例PQCC质控样本检测结果及符合率Tab.2 Results and accuracy of 10 standards from PQCC
2.4 两种方法检测T790M耐药突变丰度比较
ddPCR和Super ARMS检出的T790M突变丰度范围分别为0.04%~7.66%和0.05%~7.66%(图1A)。11例ddPCR结果为阳性而Super ARMS未检出的样本平均突变丰度为0.19%,与10例Super ARMS检出的样本平均突变丰度(1.73%)差异有统计学意义(P=0.03,图1B)。
2.5 NSCLC EGFR TKI耐药后T790M丰度分布
14例临床样本T790M突变丰度分布结果见图2。在0.01%~0.20%、0.20%~1.00%和>1.00%各区间的分布分别为42.9%、14.2%和42.9%。

图1 BEAMing ddPCR法和Super ARMS法检测T790M突变丰度的比较Fig.1 Comparison of mutant abundance of T790M detected by BEAMing ddPCR and Super ARMS

图2 NSCLC EGFR TKI治疗后ctDNA EGFR T790M突变丰度在各区间的分布Fig.2 Distribution of ctDNA EGFR T790M mutant abundance of NSCLC with EGFR TKI resistance
3 讨 论
EGFR基因是NSCLC患者TKI治疗的重要靶点,而TKI治疗导致EGFR基因第20号外显子T790M位点突变是患者产生耐药的最重要机制,并且针对T790M的第三代EGFR靶向药物奥希替尼(AZD9291)已经获得美国食品药品管理局(Food and Drug Administration,FDA)/中国国家药品监督管理局(National Medical Products Administration,NMPA)批准用于阳性的耐药患者,因此对TKI治疗后对T790M位点突变的检测非常重要[4],如何准确、快速地检测患者的基因突变情况已成为临床检测的重要工作。
目前瑞士Roche公司的Cobas以及德国Qiagen公司的ARMS方法已经获得FDA批准用于肿瘤组织或外周血ctDNAEGFR基因突变检测,国内厦门艾德生物医药科技股份有限公司的Super ARMS方法通过了NMPA的批准用于外周血中ctDNAEGFR基因突变的检测[5]。然而这些方法都是基于RTFQ-PCR的技术,必须依赖标准曲线对核酸分子进行半定量,因而具有局限性。数字PCR技术结合了极限稀释、PCR及泊松分布的原理,具有对核酸分子进行绝对定量且无需标准品的特点,因此被称为第三代PCR技术,它可以转换指数模拟信号,并提供一个适用于统计分析的线性数字信号输出。研究表明,数字PCR在EGFR基因突变检测中灵敏度与特异性优于传统RTFQ-PCR[6]。目前数字PCR平台主要包括美国Bio-Rad公司QX200、美国Thermo公司QuantStudio 3D数字PCR以及日本Sysmex公司BEAMing数字PCR。其中,BEAMing ddPCR技术融合了基于磁珠的油包水乳液扩增和等位基因特异性杂交的方法对突变分子信号进行放大,进一步采用流式细胞术分析,灵敏度可达0.01%。Toledo等[7]已采用该技术动态监测KRAS野生型转移性结直肠癌患者在接受西妥昔单抗治疗后ctDNA中KRAS、NRAS、BRAF及PIK3CA基因的动态变化,以无偏倚和前瞻性的方式提供了液态活检在转移性结直肠癌患者耐药监测应用中的证据。本文比较BEAMing ddPCR与Super ARMS方法在TKI治疗后NSCLC患者ctDNAEGFR基因突变检测的灵敏度差异,并分析T790M突变丰度分布,以指导临床ctDNA检测方法的选择。
本研究两种方法检测19 del、T790M和L858R突变结果一致率分别为90.7%(39/43)、74.4%(32/43)和97.7%(42/43)。4例19 del结果不一致的样本中有2例样本突变丰度分别为0.09%和0.12%,突变丰度均低于Super ARMS检测下限。另2例Super ARMS结果为阳性而ddPCR结果为阴性的原因则可能是ddPCR探针设计位点差异导致的,从探针设计位点分布和数量上分析,ddPCR检测19 del突变所设计的探针靶点为6个(EGFRexon19 2235-2249D、2236-2250D、2237-2255Dt、2239-2248Dc、2240-2254D和2240-2257D),而Super ARMS为19个,ddPCR中2240-2257D位点与Super ARMS中相对应的探针靶点(2240-2251D和2240-2254D)不完全相同,其余ddPCR探针靶点,Super ARMS均已包括并且还增加了12个特异性靶点。总体而言,两种方法在EGFR敏感突变19 del和L858R的阳性检出率上相似。而在T790M耐药突变检测中,与Super ARMS结果相比,10例PQCC样本的ddPCR结果与标准结果符合率更高,说明ddPCR具有较高的灵敏度和特异度。进一步分析所有T790M突变丰度发现,虽然Super ARMS检测到1例0.05%的突变,但该方法在12例高于0.2%的突变中仍有3例被检测为阴性,提示Super ARMS在1%以下低频突变检测中可能存在结果不稳定性。
既往研究[8-9]采用测序或RTFQ-PCR方法检测EGFR基因突变,结果表明,约50%的NSCLC患者经TKI治疗后会产生T790M耐药突变。本研究收集的33例TKI治疗后NSCLC患者血浆ctDNA用ddPCR检测T790M耐药突变有14例(42.4%),与上述文献报道相似但略低,这一差异可能是由于样本总量偏少,不能完全反映真实世界EGFR耐药突变的概率,另外,本研究所有样本为临床检测的剩余样本,血浆量为1~2 mL,投入量的减少也可能影响阳性率。关于T790M耐药突变丰度的分布,本研究结果提示SuperARMS临床检测T790M耐药突变可能出现较多假阴性结果,但对于小于0.20%的T790M低频突变患者与EGFR三代靶向药物疗效的关系仍需进一步研究。
综上所述,NSCLC患者TKI治疗后ctDNAEGFRT790M低频突变比率较高,BEAMing ddPCR比Super ARMS可检测更多T790M耐药患者,具有更高的灵敏度,可能为更多患者选择最有效的靶向治疗方案提供参考。

