基于psbA-trnH 序列对药用黄芪及其混伪品的鉴别
耿雅萍,马艳珠,王 芳,解潇冬,张鹏飞,刘亚令
(1.山西农业大学生命科学学院,山西太谷030801;2.山西农业大学园艺学院,山西太谷030801)
黄芪原名黄耆,属多年生草本植物,始载于《神农本草经》,其药用历史悠久,是传统中药体系中的重要药材[1]。《中华人民共和国药典》(2015 版第一部)中规定,正品药用黄芪为豆科黄芪属植物蒙古黄芪(Astragalus membranaceus(Fisch.)Bge.var.mongholicus(Bge.)Hsiao)或膜荚黄芪(Astragalus membranaceus(Fisch.)Bge.)的干燥根[2]。近年来,随着药用黄芪野生量的迅速减少,该种被列为渐危种[3]。目前,市场上多以栽培黄芪为主,但较为混杂,除了正品的药用黄芪外,还存在着许多混伪品,常见的有紫花苜蓿(Medicago sativa L.)、蜀葵(Althaea rosea(Linn.)Cavan.)、锦鸡儿(Caragana sinica(Buc'hoz)Rehder.)、蓝花棘豆(Oxytropis coerulea(Pall.)DC.)等。这些植物药用部位的外形特征与药用黄芪十分相似,但在临床应用方面,其药效相差甚远,其中部分混伪品甚至还具有一定的毒性,严重影响中医药的疗效与用药安全[4-5]。因此,急需一种简便高效的手段对黄芪及其混伪品进行准确鉴别,以确保药品质量和临床用药安全。
传统的植物鉴定方法是对其表型如株高、叶片大小、数量等进行观察,但由于形态观察极易混淆,且极易受到人为主观因素、实际经验以及外界环境、季节性的影响,因此,不能准确充分地鉴定出物种间的进化关系[6-7]。而相较于传统的鉴定方法,DNA条形码(DNA barcoding)是目前发展较快的能够简便有效地鉴定动植物物种的一项分子生物学技术,自2003 年由加拿大分类学家HEBERT 等[8]首次提出后,受到了各国分类学家的广泛关注。其原理是利用一个或几个变异程度高、系统进化信息丰富、引物设计区保守、扩增容易、标准规范且相对较短的DNA 片段作为标记对目标物种进行有效鉴定,具有通用性强、操作简便、鉴定准确、用时少等优点[9-11]。目前,在植物物种鉴定方面,常用的DNA 条形码为叶绿体序列psbA-trnH、trnL-trnF、matK、rbcL 以及核糖体序列ITS、ITS2 等。这些DNA 条形码能够简便有效地鉴定中药材基原物种,并为中药材的溯源、种质资源的评价、保护以及可持续开发利用提供数据支撑和物质基础。其中,psbA-trnH 序列是位于编码光合系统Ⅱ反应中心DI 蛋白的psbA 基因和编码tRNA 组氨酸的trnH 基因之间的一段非编码序列[12]。叶绿体基因组中psbA-trnH 序列在非编码区中功能上限制少,显示的变异位点多,因此,易设计通用引物。同时,与编码区相比,该片段可以提供更多的系统发育信息[13]。
本试验以药用黄芪蒙古黄芪和膜荚黄芪为研究对象,利用psbA-trnH 通用引物对其进行序列分析及对各序列进行差异性分析,观察其变异位点,探讨其遗传变异程度,并从GenBank 上下载的药用黄芪混伪品(紫花苜蓿、蜀葵、锦鸡儿)的psbA-trnH序列为对照,构建其系统发育树,研究其对药用黄芪及其混伪品的鉴定能力,从而为药用黄芪优良种质资源的选择、保护、评价以及分类提供一定的科学依据,为黄芪的产业化发展及可持续性开发利用提供物质基础。
1 材料和方法
1.1 试验材料
供试材料为来自山西浑源和内蒙古包头的蒙古黄芪各5 份以及来自山西浑源和吉林新兴林场的膜荚黄芪各5 份(表1)。另外从GenBank 数据库中下载了6 份黄芪混伪品,分别为2 份紫花苜蓿、2 份蜀葵、2 份锦鸡儿(表2)。

表1 黄芪样品信息

表2 混伪品样品信息
1.2 试验方法
1.2.1 黄芪样本的DNA 提取 试验采用CTAB 法提取黄芪的DNA,所得DNA 用1%的琼脂糖凝胶电泳进行质量检测,并用核酸蛋白测定仪进行纯度检测后,放于-20 ℃冰箱中保存备用。
1.2.2 PCR 扩增及测序 本试验扩增体系为30 μL,其中,1 μL DNA 模板,1 μL psbA 引物(5′-GTTATG CATGAACGTAATGCTC-3′),1 μL trnH 引物(5′-CGCGCATGGTGGATTCACAATCC-3′),15 μL Taq PCR MasterMix(+dye),加ddH2O 至30 μL。PCR 反应扩增程序为:95 ℃预变性5 min;94 ℃变性45 s,55 ℃复性40 s,72 ℃延伸1 min,33 个循环;72 ℃补平1 min,4 ℃保存。利用1%琼脂糖凝胶电泳法检测PCR 产物,后将扩增成功的PCR 产物送至上海美吉常规测序公司进行双向测序。
1.3 数据分析
利用CExpress 序列拼接软件对测序结果进行人工拼接校对,并去除两端引物区及低质量区,得到一条完整的psbA-trnH 序列;利用EditSeq 软件对20 个样本psbA-trnH 序列的碱基含量及序列特征进行统计分析;通过DNAman 软件对本试验所有样品的20 条psbA-trnH 序列进行差异性比对,并分析其变异位点;通过MEGA 7[14]软件基于K2-P(Kimura 2-Parameter)距离模型,计算黄芪种内、居群间的遗传距离,构建Neighbor-Joining(NJ)系统发育树,并用bootstrap1000 次检验各分支置信度,评价分支系统发育关系的可靠性。
2 结果与分析
2.1 PCR 产物检测
利用1%琼脂糖凝胶电泳对PCR 扩增产物进行检测,从图1 可以看出,扩增的条带亮且清晰,无其他杂带,蒙古黄芪与膜荚黄芪样品psbA-trnH 序列的PCR 产物长度在500 bp 附近,符合测序要求,可用于后续分析。
2.2 黄芪psbA-trnH 序列特征及差异性分析
利用DNAstar 软件计算黄芪psbA-trnH 序列中的G+C 含量及总长度(表3),结果表明,G+C含量为26.10%~27.88%,序列长度在345~378 bp,其中,M-JX5 的psbA-trnH 序列最短,为345 bp;M-JX1 的psbA-trnH 序列最长,为378 bp。不同居群药用黄芪psbA-trnH 序列的G+C 含量不同,居群G-SH、G-NB、M-SH、M-JX 的G+C 平均含量分别27.35%、26.76%、26.85%、27.48%。其中,G-NB(26.76%)与M-JX(27.48%)的G+C 含量相差最大,表明这2 个居群之间的亲缘关系较远。对20 份药用黄芪psbA-trnH 序列进行比对差异分析,结果表明,20 份药用黄芪样品中有337 个保守位点,保守率为95.74%;有13 个变异位点,变异率为3.69%(表4)。

表3 黄芪psbA-trnH 序列中G+C 含量及序列长度
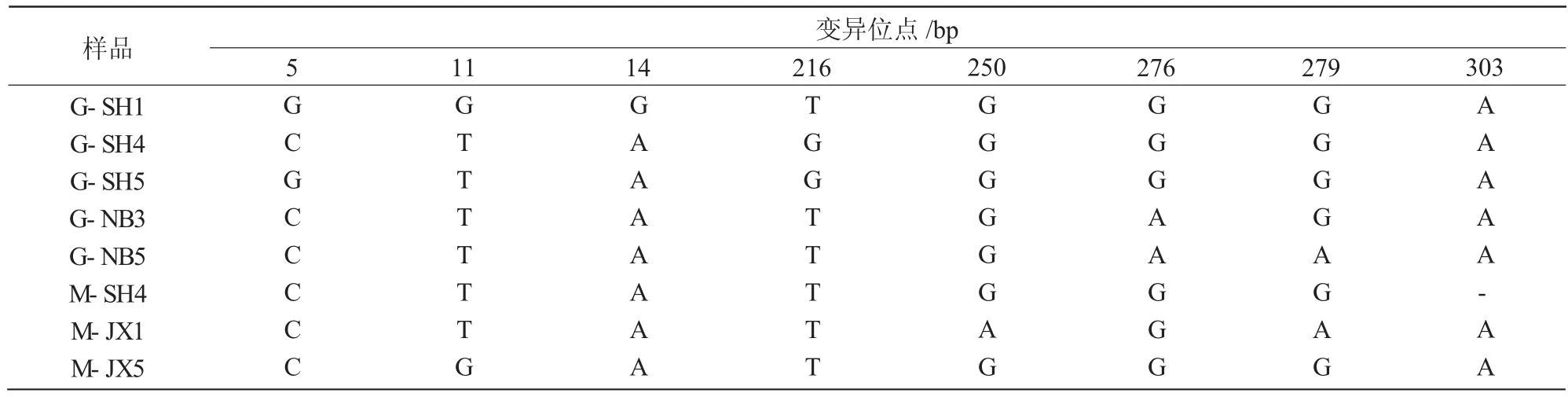
表4 黄芪psbA-trnH 序列变异位点
2.3 黄芪psbA-trnH 序列的遗传距离分析

表5 黄芪各居群之间的遗传距离
利用MEGA 7 软件计算黄芪各居群之间的遗传距离和黄芪种内个体间的遗传距离(表5、图2)。黄芪各居群之间的遗传距离为0.002~0.009,其中,居群G-SH 与M-JX 之间遗传距离最大,为0.009;居群G-NB 与M-SH 之间遗传距离最小,为0.002。黄芪种内遗传距离范围为0.000~0.023,其中,个体M-JX1 与G-SH5 之间遗传距离最大,为0.023。
2.4 基于psbA-trnH 序列的黄芪及其外类群系统发育树分析
本试验以紫花苜蓿、蜀葵、锦鸡儿作为外类群,采用邻接法构建系统发育树对蒙古黄芪和膜荚黄芪样品以及外类群进行聚类分析(图3)。结果显示,将其分成了三大支,其中,蒙古黄芪与膜荚黄芪为一大支,紫花苜蓿和锦鸡儿为一大支,蜀葵单独为一大支,其自展支持率各达100%。在药用黄芪的一大支中,内蒙古包头的蒙古黄芪(G-NB1~5)、山西浑源的8 个样品(4 个蒙古黄芪与4 个膜荚黄芪)以及辽宁新兴林场的膜荚黄芪M-JX2、M-JX3 聚为一支,自展支持率达64%,山西浑源的G-SH5、M-SH1 与吉林新兴林场的M-JX1、M-JX5 聚为一支,自展支持率达61%。
3 结论与讨论
近年来,DNA 条形码技术已成为国内外研究人员在生物物种鉴定及遗传多样性分析方面的重要手段之一。在药用植物鉴定方面,psbA-trnH 被作为ITS2 序列的辅助序列,可有效用于药用植物的鉴定[15]。万如等[16]通过对21 份材料进行psbA-trnH序列分析发现,枸杞属植物的航天诱变种与普通栽培种各聚为一支,得出psbA-trnH 可以用于亲缘关系较远的枸杞属植物研究。韩正洲等[17]通过对芦根及其混伪品进行psbA-trnH 序列分析发现,芦根及其混伪品分成了两大支,同时各分支均有较高的自展率。吴海燕[18]研究表明,利用叶绿体psbA-trnH 基因在序列和结构上相对保守,能够有效并鉴别甘肃各地的黄芪和红芪,有利于保障黄芪的用药安全和临床疗效。徐子欣等[19]通过对不同居群的巴戟天进行psbA-trnH 和rDNA ITS 序列分析,得出psbAtrnH 基因能够广泛应用于地理居群较近的物种之间的研究。张宏意等[20]对不同产地不同个体的何首乌进行了psbA-trnH 序列分析,结果显示,各居群何首乌的亲缘关系大体上符合地域差异且其遗传变异显著。本试验利用叶绿体基因组psbA-trnH 序列分析山西浑源的蒙古黄芪和膜荚黄芪、内蒙古包头的蒙古黄芪、吉林新兴林场的膜荚黄芪,以黄芪混伪品(紫花苜蓿、蜀葵、锦鸡儿)为对照,利用NJ法构建系统发育树。结果表明,药用黄芪及其混伪品之间存在有较大程度的变异。药用黄芪聚为一大支,紫花苜蓿与锦鸡儿聚为一支,蜀葵单独聚为一支,且自展支持率各达100%,表明利用psbA-trnH序列可以有效鉴别黄芪及其混伪品。此外,根据聚类结果得到药用黄芪与紫花苜蓿、锦鸡儿的亲缘关系较近,与蜀葵的亲缘关系较远。此结果与崔占虎等[4]利用ITS 序列对黄芪与其混伪品的鉴定结果一致。
不同物种psbA-trnH 序列的G+C 不同。G+C含量的差异是反映物种亲缘关系远近的遗传性特征,亲缘关系越近,G+C 含量差异较小;亲缘关系越远,G+C 含量差异较大[21]。王丽芝等[22]对8 种川芎种质及藁本、辽藁本之间G+C 含量进行了比较分析,结果表明,藁本、辽藁本与川芎间G+C 含量相近,表明彼此间亲缘关系较近。周文才等[23]基于ITS 序列鉴定三叶木通与白木通,结果表明,二者具有相似的G+C 含量,验证了其很近的亲缘关系。本试验通过计算各居群蒙古黄芪与膜荚黄芪psbAtrnH 序列的G+C 含量,得出来自吉林的膜荚黄芪M-JX(27.48%) 与来自内蒙古包头的蒙古黄芪G-NB(26.76%)之间的含量差异最大,因此,这2 个居群之间的亲缘关系较远,与其居群间的遗传距离结果一致,即居群M-JX 与G-NB 的遗传距离最大,为0.023。
本试验通过分析药用黄芪psbA-trnH 序列的G+C 含量、遗传距离、保守率、变异率等指标,以混伪品为对照构建系统发育树,表明可以利用psbAtrnH 序列鉴别药用黄芪及其混伪品。此结果为药用黄芪及其混伪品的鉴别提供了分子水平依据,为药用黄芪优良种质资源的保护、评价以及分类提供了一定的数据支撑。

