蛋白质的核酸适配体筛选及应用的研究进展
赵丽萍 杨歌 张小敏 屈锋


摘 要 核酸适配体是通过指数富集配体系统进化(SELEX)筛选方法获得的,能够与靶标分子高亲和力和特异性结合的单链DNA或RNA。蛋白质是一种非常重要的生物功能大分子,迄今为止,已经开发了许多SELEX技术,用于蛋白质的适配体的筛选。目前,适配体的筛选优化方法主要集中在提高筛选效率、降低筛选成本,以及提升适配体性能等方面,从而获得可与靶标分子以高亲和力和高特异性结合的适配体。适配体与靶标分子亲和力的表征是关键的筛选步骤,用于判定所筛选的适配体是否可以满足后续应用需求。本文归纳总结了2016年以来蛋白质靶标的适配体筛选的研究进展,对核酸适配体筛选过程中有关核酸库的优化方法和筛选方法的改进、新筛选方法的开发、核酸适配体的应用等方面的研究进行了评述。
关键词 核酸适配体; 蛋白质; 筛选; 指数富集配体系统进化; 评述
1 引 言
核酸适配体是指通过指数富集的配体系统进化技术(Systematic evolution of ligands by exponential enrichment,SELEX)在体外筛选获得的可与靶标高特异性和高亲和力结合的单链DNA(ssDNA)或RNA。核酸适配体的靶标范围广,包括蛋白质、多肽、小分子、细胞、细菌,甚至组织。核酸适配体可通过化学方法合成,与抗体相比,核酸适配体具有成本低、易修饰、稳定性好等特点。此外,核酸适配体有较好的组织渗透性,易進入细胞,且无免疫原性。近年来,以蛋白质为靶标的核酸适配体在分子生物学、生物技术、生物医学等领域广受关注。
核酸适配体是通过体外筛选获得的,故可根据特定应用环境或靶标分子特征进行筛选。1990年,Tuerk和Gold[1]首次采用SELEX技术从随机序列中筛选获得T4 DNA聚合酶的RNA配体序列,并预测通过SELEX技术可筛选获得任何蛋白质或其它目标分子的高亲和力配体。同年,Ellington和Szostak[2]通过亲和色谱柱方法筛选获得具有特异性结合于有机染料的RNA配体序列,并将其定义为“适配体”。1992年,Toole等[3]筛选获得具有抑制人凝血酶催化作用的首个DNA适配体。核酸适配体的筛选过程主要包括随机寡核酸库的建立、核酸库与靶标的相互作用、靶标-核酸复合物的分离、次级库的形成(通过聚合酶链式反应(PCR)进行扩增),再重复以上筛选步骤。不同的筛选方法,重复筛选3次不同,一般需4~20轮的筛选。最后对筛选得到的核酸序列进行测序,获得与靶标分子以高亲和力特异性结合的适配体序列[4]。
尽管以蛋白质为靶标的核酸适配体应用前景广泛,但其筛选过程复杂,一般需要十多轮筛选,耗费大量人力、财力和时间,并且,筛选所得的核酸适配体还可能存在亲和力不高、特异性不好等问题,影响其后续应用。因此,适配体的筛选仍是目前适配体研究应用亟待解决的问题。本研究组在之前的综述[4]中,归纳总结了2015年之前的研究工作。近年来,对SELEX技术的改进和发展的相关研究主要集中在核酸库的优化和筛选方法的改进、适配体的结构优化和计算机辅助筛选等方面。本文综述了2016年以来,以蛋白质为靶标的核酸适配体的筛选过程中核酸库的优化方法和筛选方法的改进、新筛选方法的开发、适配体亲和力表征方法以及核酸适配体的应用等工作。
2 核酸库的设计与优化
基于对RNA分子可形成功能性空间结构的认识,早期的SELEX技术中优先使用RNA随机文库[5]。随后的研究结果表明,单链DNA(ssDNA)能够形成比RNA更复杂的三级结构,且ssDNA的化学性质更稳定、易合成,筛选过程中不需要额外的逆转录步骤,相比于RNA适配体筛选更节省时间和成本[6]。目前,超过85%的适配体筛选使用了ssDNA随机文库[7,8]。
大多数适配体体外筛选使用ssDNA或RNA的随机文库。然而,它们在体外相对稳定,但在体内则很容易被核酸酶降解,通常在10 s内就会被破坏。此外,小片段的ssDNA或RNA可提供的分子结构简单,相互作用位点有限; 并且,当其进入生物体体内时,循环时间短,可被肾脏快速过滤。考虑到ssDNA与RNA的结构多样性[9]、与靶标的结合能力以及在体内的稳定性与功能等因素,初始核酸库的设计与优化是SELEX的重点考虑问题之一。
2.1 核酸库长度
筛选所用的核酸库一般是按照核酸合成的标准方法进行合成。通常在两端分别设计两段固定的引物序列,中间区域由4个核苷酸随机排序组成,核苷酸数为N。理论上,每个合成的核酸库应有4N个不同的序列,序列的长度为引物-N-引物。核酸库中N的选择主要由经验决定,并因实际合成技术的限制,核酸库所含的实际的序列数约为1014~1015条[10]。较短的随机区域(N<20)可确保合成得到完全覆盖的序列空间(即每条随机序列至少有1个分子),但可能会因序列较短而使核酸分子的结构多样性减少。另一方面,较长的随机区域(N>40)则可能提供更多的序列的三维结构与结构多样性,但仅能覆盖序列空间的一小部分(理论上应该存在的序列不能全部出现)[11]。目前,已经筛选报道的有短至8个核苷酸[12]和长至228个核苷酸的适配体序列[13],而大多数筛选仍使用随机区域长度N为20~60 nt之间的核酸库。
不同长度的核酸库可同时用于蛋白质靶标的适配体筛选。Lee等[14]采用双库策略(Double library (DL) SELEX),将不同长度的两个核酸库(16 nt 与20 nt)同时用于筛选VEGF16的ssDNA适配体。两个核酸库的部分碱基序列互补。通过DL-SELEX,获得一对可结合在蛋白不同位点上的适配体。
此外,也可对次级库的核酸长度进行裁剪,缩短次级库长度以简化适配体序列并提高其亲和力。He等[15]通过截短(Truncation)SELEX方法筛选了人红细胞生成素α的核酸适配体In27。经首轮筛选得到次级核酸库后,对此次级库进行截短,以构建由所有可能的核苷酸序列截短组成的步进型文库,重复以上步骤,直至获得具有更高亲和力的最短适配体序列。所获得的适配体In27没有原始的茎环部分,但仍保留了对靶标的亲和力。
核酸库的设计与优化还包括镜像筛选(Spiegelmer SELEX)[43~46]以及嵌合库(Chimeric SELEX)的设计与使用[47]等。此外,还应考虑核酸库中引物序列的设计[48,49],或结合生物信息学分析指导核酸库的设计与合成。而且,使用修饰的核酸库进行适配体筛选时,必须考虑由非天然核苷酸可能引起的化学毒性和免疫原性,以提高修饰适配体的生物相容性。
3 复合物分离
分离核酸与靶标形成的复合物是SELEX的关键步骤之一。基于复合物分离技术开发的SELEX方法主要包括硝化纤维素膜过滤SELEX、微珠/磁珠SELEX、微柱SELEX、毛细管电泳(CE)SELEX、微流控自由流电泳SELEX、微流控芯片SELEX、SPR-SELEX、微阵列SELEX、AFM-SELEX等,表1列举了2016年至今蛋白质靶的适配体筛选所用的基于分离的SELEX方法。
3.1 CE-SELEX
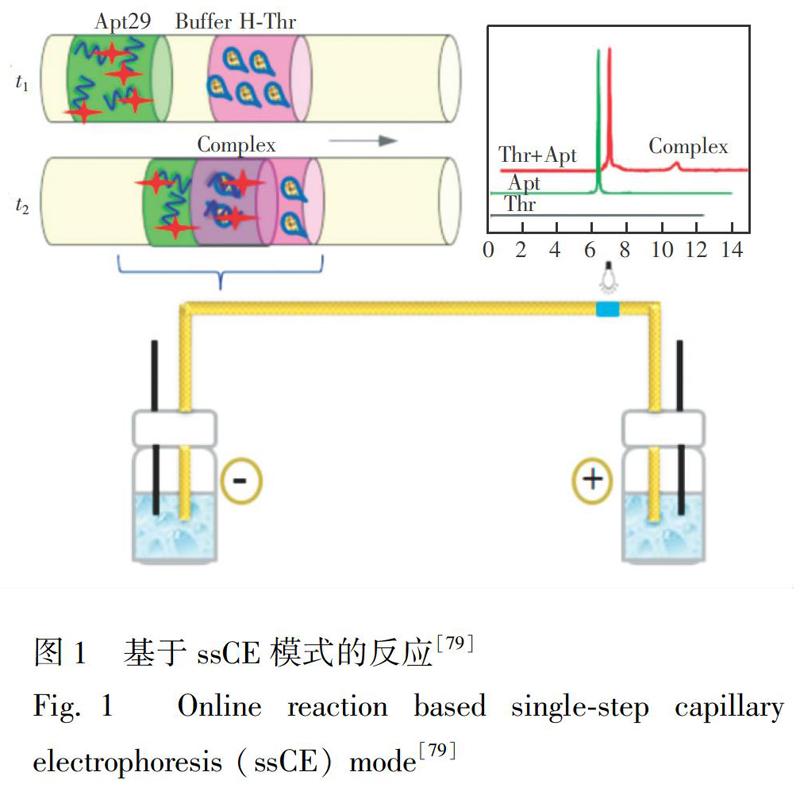
CE-SELEX是快速、高效筛选核酸适配体的方法之一。在CE-SELEX中,通常将核酸库与靶蛋白孵育后,进行CE分离分析。高压电场下,未结合的核酸序列与核酸-靶蛋白复合物因迁移速率不同而分离。在毛细管的出口端收集复合物,经PCR扩增、纯化,生成次级库,再进行下一轮筛选。通常CE只需1~4轮筛选即可获得高亲和力和特异性的适配体[102]。在CE-SELEX中,靶蛋白与适配体的结合发生在自由溶液中,避免了对靶蛋白或核酸库的固定,同时可降低非特异性结合,且二者相互作用不受空间位阻影响。CE-SELEX方法可进一步改进为平衡混合物的非平衡毛细管电泳(NECEEM)、平衡混合物的平衡毛细管电泳(ECEEM)及Non-SELEX等系列筛选方法[4]。本研究组建立了两种新的CE-SELEX模式: 同步竞争结合筛选方法(Synchronized competition CE-SELEX,scCE-SELEX)和一步快速CE-SELEX(Single-step CE-SELEX,ssCE-SELEX)。Yang等[86]建立了双靶蛋白的scCE-SELEX方法,将两种靶蛋白A、B与核酸库同时孵育,二者竞争结合达到动态平衡状态后,将混合物注入CE中进行分析。两种靶蛋白与核酸库结合分别形成两种复合物,二者具有不同的迁移速率,能够与未结合的核酸库完全分离。在同步竞争结合筛选过程中,蛋白A和蛋白B彼此为反筛靶,通过竞争效应为对方增加筛选压力,显著提高了筛选效率,仅需一轮筛选即可得到高亲和力(KD值在nmol/L级)和特异性良好的适配体。该方法扩展了高效筛选核酸适配体的CE-SELEX方法的应用。Zhu等[79,81,82]建立了一种基于在线反应的ssCE-SELEX模式,将核酸序列与靶蛋白的混合、孵育、核酸序列与靶蛋白-核酸复合物的分离、检测以及复合物的收集在一步CE运行中完成(图1),并对影响复合物生成的关键因素(如孵育缓冲溶液、靶蛋白与核酸序列的比例等)及影响靶蛋白-核酸复合物分离与收集的因素(电压、温度、进样时间等)进行了考察。进一步利用该模式成功筛选了人凝血酶以及牛乳铁蛋白的适配体。此外,还利用此模式对6种蛋白进行了适配体筛选的初步实验,结果表明,ssCE-SELEX模式对蛋白靶标核酸适配体的筛选具有普适性。近期,本研究组在Biotechnology Advance期刊发表了多功能CE用于核酸适配体筛选的综述[102],对近年来CE技术在核酸适配体筛选方面的应用进行了全面地归纳和总结。Krylov研究组[85]提出了理想过滤式毛细管电泳法(Ideal-filter capillary electrophoresis,IFCE),即在生理pH值和生理离子强度(pH 7,150 mmol/L NaCl)时,可使游离的核酸序列与靶蛋白-核酸复合物反向移动,分离效率高达109。同时,较高的离子强度可抑制靶标与核酸序列的非特异性结合,故一步筛选可获得高亲和力适配体序列。
3.2 微流控SELEX
微流控技术在复合物分离方面也有明显优势[103,104]。2005年,Hybarger等[105]首次将微流控芯片技术运用到SELEX。微流控SELEX主要有两种方式: 将靶分子固定在磁珠上,用核酸库与磁珠孵育[106],在微通道内外加上磁铁,形成高强度的局域磁场梯度,精确控制大量的微磁珠; 或用溶胶-凝胶包封靶分子,再将核酸库加入包封的溶胶-凝胶中[107],通过将这些目标分子包封在凝胶液滴中,在微流控芯片内进行分离。与CE-SELEX相似,使用微流控分离所需的样品量非常少,可严格控制筛选条件,加速适配体进化[108]。如筛选过程中通过控制流速,在靶标损失最小的情况下去除弱结合的或非特异性结合的核酸序列[8],通过1~6轮筛选可获得高亲和力適配体[106]。
Hong等[92]开发了一种基于磁珠的多功能微流控筛选平台,该芯片将筛选芯片和定量芯片结合。将BSA(反筛靶标)包被的磁珠和粘蛋白1(MUC1)包被的磁珠分别注入到筛选芯片反筛区域和正筛区域。将初始ssDNA库首先注入筛选芯片的反筛区域,在此区域中,一部分核酸库与BSA结合; 未结合的核酸库流入正筛区域,与MUC1结合,从而减少了非特异性吸附。同时,可通过倒置荧光显微镜观察荧光信号的变化监测整个筛选过程。收集磁珠上与目标蛋白结合的核酸序列进行扩增生成次级库,用于下一轮筛选。重复上述步骤,进行多轮筛选,并将每个次级库注入定量评价芯片,通过荧光强度实时评价每轮筛选的富集程度。由于次级库可特异性地结合微通道中的MUC1包被的磁珠,当每轮富集的荧光强度不再明显增加时,停止筛选。该筛选平台可在流动的环境中同时进行正筛和反筛,并可通过连续洗涤来施加高强度筛选条件。与常规孵育方法相比,该筛选方法明显减少了核酸的非特异性结合,还可消除弱结合的核酸。通过2轮筛选,获得3条与MUC1高亲和力(KD值分别为44.8、22.4和65.0 nmol/L)和特异性结合的适配体序列,并被用于癌细胞的标记和外泌体捕获。
利用荧光显微镜可监测微流控SELEX过程,但因观测区域的限制,荧光强度的均匀采集比较困难。Xu研究组[88]提出了PMM(Protein microarray integrated into microfluidic chip)-SELEX方法,通过静电吸附将蛋白固定在芯片上,制备蛋白质微阵列芯片,嵌入SELEX微流控芯片中,并注入核酸库。与靶标蛋白结合的核酸序列被保留在蛋白质芯片上,未结合的序列流出。通过荧光芯片扫描仪对保留在蛋白质芯片上的核酸序列进行均匀成像,以监测筛选过程中蛋白与核酸的结合。以乳铁蛋白为靶标制备了靶蛋白芯片,以BSA、酪蛋白、乳球蛋白等为反筛靶制备了反筛蛋白芯片,将两块蛋白芯片同时嵌入到SELEX微流控芯片,可同时进行正筛和反筛。通过7轮筛选得到了KD值为nmol/L级的适配体。
在微流控SELEX中,无法在芯片上直接完成核酸序列的扩增,使得與核酸结合的靶标包被磁珠可能在后续扩增与筛选过程中因移液和转移而丢失。Sinha等[87]设计了一个小型的自动化微流控SELEX平台,它集样品的输送、孵育及核酸扩增于一体,用非靶蛋白包被磁珠进行1轮反筛后,用靶蛋白进行5轮正筛。筛选过程可以在一个芯片中连续进行,6轮筛选只需8 h。微流控中的PCR过程由自制的温度控制模块进行,利用Arduino电路和温度传感器进行实时温度检测,实现了芯片上的PCR温度循环。每次循环后,收集PCR产物以琼脂糖凝胶电泳确认筛选过程。利用此平台,筛选了3种与心血管病相关的蛋白标志物的适配体。
微流控SELEX在实际筛选时有所不足,如样品的注入量不稳定、微通道需要进行表面修饰提高亲水性,并且还没有商品化的微流控设备等。
3.3 原子力显微镜(AFM) SELEX
原子力显微镜(AFM)是可在分子水平上精确检测分子间亲和力、相互作用和识别特征的方法[109]。AFM灵活的悬臂末端的尖头,可以检测样品表面和悬臂之间的微弱力。当尖端接触到样品表面或与样品表面靠近接触时,悬臂的挠度被反射到悬臂背面的激光束记录下来[110,111]。利用AFM方法可以动态检测样品表面与悬臂梁之间的作用力,并用于高亲和力适配体的筛选,即AFM-SELEX。
Yusuke等[112]用AFM-SELEX通过3轮筛选得到了高亲和力的凝血酶适配体(KD=200 nmol/L)。将链霉亲和素固定在悬臂顶端,再结合生物素化的ssDNA库,而将凝血酶固定在金片上。当悬臂末端与金片接触时,抗凝血酶的DNA适配体会与金片上的凝血酶结合,形成的DNA-凝血酶复合物滞留在金片上,得到高亲和力的DNA适配体。Takenaka等[100]提出了一种新型AFM-SELEX方法。通过T和A碱基配对作用将核酸库(含有35 mer腺嘌呤(A35))固定在金芯片(含10 mer胸腺嘧啶(T10))上,将人血清白蛋白(HSA)固定在悬臂上。当HSA与核酸分子的相互作用大于A-T间的相互作用时,该核酸分子会因与蛋白的结合而使悬臂发生变化,基于悬臂变化可进行高亲和结合的适配体筛选。这个过程中,连接分子间的相互作用(A-T间作用)在减少或控制筛选轮数方面起着关键作用。通过4轮筛选,得到了HSA的特异性适配体。虽然AFM SELEX方法筛选轮次较少,3~4轮内即可得到高亲和力的适配体。但是,该方法存在AFM仪器设备昂贵、需要对靶标和适配体进行固定,以及初始库容量较小等局限。
3.4 其它SELEX
分子印迹聚合物具有识别模板分子功能,也可用于适配体筛选。Liu研究组[67]开发了一种基于聚糖印迹磁性纳米颗粒(MNPs)的SELEX方法,有效地筛选到与糖蛋白特异性结合的适配体。采用硼酸盐亲和可控定向表面印迹方法合成了多糖印迹MNPs,以此多糖印迹MNPs为亲和界面与靶标糖蛋白结合,再以靶标糖蛋白结合的MNPs为亲和底物筛选适配体。多糖印迹的MNPs对糖蛋白表现出较高的亲和力和特异性,因此可优先结合目标糖蛋白,同时排除不需要的物质。以RNase B和转铁蛋白两种具有代表性的糖蛋白作为靶蛋白,通过3轮筛选得到对两种靶蛋白具有高亲和力和特异性的适配体。该方法具有亲和度高、速度快、避免阴性筛选等优点。
4 非纯化靶蛋白的适配体筛选
4.1 全细胞蛋白的适配体筛选(Cell-SELEX)
目前报道的适配体筛选多使用重组蛋白或具有稳定构象的纯化蛋白,但当靶蛋白处于修饰状态或其结合域可能被掩蔽时,不能很好地识别核酸分子[113]。例如,虽然EGFRⅧ蛋白的RNA适配体可以结合在大肠杆菌中表达的重组EGFRⅧ蛋白,但不能识别真核细胞表面表达的全长EGFRⅧ蛋白[114]。
Cell-SELEX是将整个细胞作为靶标,由于细胞表面复杂,包含了大量的蛋白,因此,可针对多种靶标分离出相应的适配体。全细胞筛选具有以下优点: 不需要预先了解靶标分子构象; 细胞表面分子呈现自然构象和分布; 可避免纯化过程中靶标结构的破坏; 还具有筛选未知分子适配体的潜力[115~118]。
Haghighi等[98]以淋巴球抗原(CD20)蛋白过表达的HEK293T细胞为靶标,经过11轮筛选得到KD为nmol/L级的CD20适配体。该适配体被用于细胞的特异性识别,有望替代抗体用于免疫缺陷、自身免疫性疾病、白血病和淋巴瘤的诊断和治疗。He等[119]报道了两种由Cell-SELEX筛选获得的适配体,它们可与紫杉醇耐药卵巢癌细胞系(A2780T)表面的两种不同的糖蛋白特异性结合。这两种核酸适配体对目标细胞均有较高的选择性和较强的亲和力,并且适配体与靶细胞的结合不依赖于二价离子,对血清中的核酸酶有较强的耐受性,可用于人血清中耐药卵巢癌细胞的检测。Bing等[120]经5轮筛选获得了碱性磷酸酶(Alkaline phosphatase,AP)二聚体的适配体BG2,该适配体可结合碱性磷酸酶二聚体而非其单体,可用于细胞和组织上蛋白质二聚体的原位检测。
Cell-SELEX可以解决真实细胞表面蛋白的适配体筛选,但很难直接筛选到细胞表面表达量较少的蛋白的适配体。此外,还需要去除死细胞(如利用流式细胞仪分选以及基于微珠的方法等)以减少非特异性结合的干扰; 全细胞的靶标组成复杂而且需要多轮筛选才能获得高亲和力和高选择性的适配体。筛选过程中通常需要反筛步骤,以提高适配体的特异性,如筛选某肿瘤细胞的肿瘤标志物的核酸适配体时,需要以其它肿瘤细胞或正常细胞为反筛靶。与单一靶标的适配体筛选方法相比,全细胞表面靶蛋白的鉴定是Cell-SELEX筛选的难点,可将与适配体特异性结合的靶标提取后,通过质谱的方法对靶蛋白进行鉴定。但是,由于某些靶蛋白含量低、疏水性强、不易提取等,使得其分离、鉴定和表征较为困难,真正的全细胞蛋白靶的适配体筛选还难以实现。
4.2 体内蛋白的适配体筛选(In-vivo SELEX)
體外筛选的适配体可能因不具有组织通透性而无法在体内发挥作用。体内筛选(In-vivo SELEX)是指利用疾病相关模型的小鼠直接进行体内筛选。首先,将修饰后具有抗核酸酶性能的核酸库通过静脉注射到荷瘤小鼠中,收集目标靶器官/组织,提取结合的核酸序列后在体外进行扩增得到次级库。将次级库注入动物体内,重复进行体内筛选。筛选时,可使用荧光分子层析成像(FMT)对深埋器官中的肿瘤进行成像,观察核酸序列在体内的结合和迁移。体内筛选策略用于鉴定原位定位结合肿瘤的适配体,具有药物靶向递送和有效设计诊断试剂的潜力。Chen等[121]通过In-vivo SELEX鉴定了前列腺癌骨转移小鼠模型中的骨靶向适配体,筛选获得Toll样受体4(TLR4)阻断适配体,有望用于急性中风治疗[122]。Mai等[123]在淋巴瘤小鼠模型中筛选了对淋巴瘤骨髓细胞具有高特异性的ssDNA适配体。
体内筛选具有原位和实时筛选的优点,但筛选的适配体具有明显的个体差异,因此,即便筛选成功也在很大程度上限制了其普适性应用。如果使用修饰的核酸,还需考虑聚合酶扩增修饰核酸的能力限制。此外,体内结合的核酸序列的提取过程更加复杂,增加了筛选难度和成本。
5 基于生物信息学的SELEX(In-silico SELEX)
In-silico SELEX是一种基于计算机技术的非实验筛选和优化适配体的新方法,可为适配体筛选的3个关键过程提供计算解决方案,包括: 增强型初始库的设计、序列的富集、高亲和适配体的鉴定。In-silico SELEX主要包括两个步骤: (1)根据二级结构设计增强型的初始库。对核酸库的分析表明,高亲和力的适配体的热力学参数与其他序列存在差异。例如,GTP适配体二级结构的自由能明显低于相同长度的其它随机序列。因此,在初始核酸库的设计中可以减少具有简单茎环结构或微分支结构的序列,同时,最大化稳定低能结构,以形成增强型的初始库,用于下一步筛选。(2)通过分子对接计算进行筛选。不同的分子对接工具可用于识别与靶标结合的序列。Zhang等[124]开发了改良版DOVIS包,可用于RNA库的高通量虚拟筛选。根据分子对接分析的数据(如结合自由能和适配体的平衡解离常数),选择适配体候选序列进一步研究。
Yokoyama等[95]运用G4启动子衍生的适配体筛选技术(G4PAS)与In-silico maturation (ISM)技术,通过计算机辅助定向进化成功筛选了肝细胞生长因子(HGF)的适配体,并进一步通过ISM方法将HGF适配体的特异性提高了45倍。Ahirwar等[96]设计了人雌激素反应元素(ERE)的RNA类似物作为初始文库,筛选了雌激素受体α(ERα)的类适配体。首先,对单链RNA类似物进行建模,并使用AutoDock Vina、HADDOCK和PatchDock软件完成与靶标的对接,判断其作为ERα适配体的可能性。进一步通过等温滴定量热法测定ERα-RNA相互作用的热力学参数,并验证了计算机模拟预测结果。最后获得Ka=1.02±0.1×108 mol/L的RNA序列为ERα-适配体ERaptR4,并通过细胞化学和固相免疫分析证实了ERaptR4适配体的靶向特异性。Bavi等[97]筛选了上皮细胞粘附蛋白(EpCAM)的15-mer RNA适配体。首先,生成初始库序列,并模拟其全部序列的3D结构,通过分子对接、分子动力学模拟以及结合自由能计算筛选适配体,并估算其亲和力。进一步通过流式细胞术和荧光显微镜验证了该RNA适配体可结合癌细胞表面过表达的EpCAM。
6 蛋白质适配体的应用
6.1 疾病相关蛋白的检测
蛋白质的适配体可作为检测探针用于各种传感检测。基于适配体的光学检测、电化学检测以及结合各种纳米材料的检测方法都可以显著提高检测灵敏度[125]。Díaz-Fernández等[65]经过8轮筛选得到了特异性识别前列腺特异性抗原(PSA)糖基化部分的适配体,将此适配体用于夹心式电化学传感器的设计可以检测人血清中的PSA(检出限为0.66 ng/mL)。该传感器检测的前列腺增生患者血清的结果低于标准ELISA方法的检测结果,表明该传感器可精确检测到含量更低的具有明显糖基结构的PSA。所设计的传感器可为临床前列腺特异性抗原检测提供新的方法,有望改良现有临床检测方法,获得更精准的检测结果,减少不必要的前列腺癌活检。Ruan研究组[126]以甲胎蛋白特异性结合的适配体修饰的CdTe量子点(QDs)为供体,以抗AFP抗体的金纳米粒(AuNPs)为受体,基于FRET原理,实现快速灵敏检测血清中的AFP(线性范围为0.5~45 ng/mL,检出限为400 pg/mL)。随着越来越多的生物标志物的适配体被筛选报道,所建立的各种传感器方法可以方便地用于多种生物标志物的检测,并且有望用于相关疾病的更好诊断,甚至现场检测。
6.2 生物成像
适配体荧光探针具有肿瘤穿透性好、生物相容性好、信噪比低和分辨率高的优点[127],在生物成像中的应用越来越广泛。特别是以活细胞为筛选靶标的多种具有特殊生物学功能适配体的出现,为基于适配体的荧光探针在生物成像领域的应用奠定了基础,也提供了优异的分子工具[128]。Bavi等[97]通过In-silico SELEX筛选了EpCAM的RNA适配体,利用荧光显微成像观察到所筛选的RNA适配体可特异性地与EpCAM阳性细胞结合,而不与EpCAM阴性细胞系结合。此实验结果表明,RNA适配体分子小,对肿瘤生物标志物EpCAM具有良好的选择性和高亲和力,有望用于新型分子成像和靶向治疗。
6.3 治疗药物
由于适配体具有可与小分子药物和蛋白配体竞争结合靶标的能力[129],被认为具有潜在的药物性能,可用于疾病治疗。此外,适配体还具有激活目标受体的功能,或可作为载体向目标细胞或组织递送治疗药物[130]。例如,适配体有作为抗病毒药物的潜力。有研究表明,针对gp120的合成衍生物的RNA适配体可中和HIV-1[131]。此外,RIG-I的RNA适配体可以抑制新城疫病毒(NDV)、疱疹性口炎病毒(VSV)和流感病毒的复制[129]。
6.4 生物标记物的发现
与疾病相关的新生物标志物的发现有助于疾病的早期发现和及时治疗。生物标志物还可用于靶向给药及细胞成像等,开发新型生物标志物及其识别探针在医学研究和临床检测领域十分必要,也有强烈的市场需求。目前,抗体是最常用的识别分子。与抗体相比,适配体有可能可以根据病变细胞表面未知膜蛋白的分子差异,将病变细胞与正常细胞区分开来,因此适配体在发现新的生物标志物方面有巨大的潜力。采用Cell-SELEX方法进行适配体筛选,能够发现新的肿瘤生物标志物,而不需要事先对肿瘤生物标志物进行了解[132]。先构建针对靶细胞的核酸库,然后根据核酸库的结合活性对不同蛋白进行分离,最后,通过质谱分析鉴定蛋白。例如,Berezovski等[133]在未成熟和成熟树突状细胞中分别发现了6个和3个生物标志物。Jia等[134]通过Cell SELEX和基于适配体的蛋白的纯化策略,鉴定出CD109作为鼻咽癌的潜在生物标志物,用于早期诊断和靶向治疗。
7 总结与展望
自1990年SELEX技术被提出以来,核酸适配体的研究取得了长足进步,被认为是具有发展前景的新型分子探针,所筛选的核酸适配体数量增加迅速。近年来,随着对传统SELEX筛选方法的改进以及高通量测序和计算机辅助设计等新技术的快速发展,核酸适配体的筛选效率已显著提高(如筛选轮次显著减少、筛选周期明显缩短以及适配体的亲和力提高等),但仍未有自动化、标准化的适配体筛选程序和方法。亲和力的测定方法虽多,但没有统一的标准或界限,用于判定筛选的适配体是否可以满足后续应用的要求。不同的筛选方法测定的亲和力难以对应,甚至在数量级上有很大差异,也缺少可将不同方法测定的亲和力之间进行转换的标尺。特别是,有效好用的适配体(如凝血酶等靶标分子的“明星”適配体)还十分有限,远不能满足实际应用的需求。因此,适配体筛选的机理和高效筛选方法还有待深入研究,适配体所面临的这些问题也将会转化为促进适配体发展和应用的研究动力。
References
1 Tuerk C,Gold L. Science,1990,249 (4968): 505-510
2 Ellington A D,Szostak,J W. Nature,1990,346: 818-822
3 Bock L C,Griffin L C,Latham J A,Vermaas E H,Toole J J. Nature,1992,355: 564-566
4 YANG Ge,WEI Qiang,ZHAO Xin-Ying,QU Feng. Chinese Journal of Chromatography,2016,34(4): 370-381
杨 歌,魏 强,赵新颖,屈 锋. 色谱,2016,34(4): 370-381
5 Gilbert W. Nature,1986,319: 618
6 Wang T,Chen C,Larcher L M,Barrero R A,Veedu R N. Biotechnol. Adv.,2019,37(1): 28-50
7 McKeague M,DeRosa M C. J. Nucleic Acids,2012: 748913
8 Bayat P,Nosrati R,Alibolandi M,Rafatpanah H,Abnous K,Khedri M,Ramezani M. Biochimie,2018,154: 132-155
9 Blind M,Blank M. Mol. Ther. Nucleic Acids,2015,4: e223
10 Knight R,Yarus M. Nucleic Acids Res.,2003,31(6): e30
11 Velez T E,Singh J,Xiao Y,Allen E C,Wong O Y,Chandra M,Kwon S C,Silverman S K. ACS Comb. Sci.,2012,(14): 680-687
12 Kwon Y S,Raston N H A,Gu M B. Chem. Commun.,2014,(50): 40-42
13 Li Y F,Geyer C R,Sen D. Biochemistry,1996,(35): 6911-6922
14 Lee K H,Zeng H. Anal. Bioanal. Chem.,2017,409(21): 5081-5089
15 He X,Guo L,He J,Xu H,Xie J. Anal. Chem.,2017,89(12): 6559-6566
16 Griffin L C,Tidmarsh G F,Bock L C,Toole J J,Leung L L K. Blood,1993,81(12): 3271-3276
17 Pagratis N C,Bell C,Chang Y F,Jennings S,Fitzwater T,Jellinek D,Dang C. Nat. Biotechnol.,1997,15(1): 68-73
18 Matsunaga K,Kimoto M,Hanson C,Sanford M,Young H A,Hirao I. Sci. Rep.,2015,5: 18478
19 Shoara A A,Reinstein O,Borhani O A,Martin T R,Slavkovic S,Churcher Z R,Johnson P E. Biochimie,2018,145: 137-144
20 Dunn M R,Jimenez R M,Chaput J C. Nat. Rev. Chem.,2017,1(10): 0076
21 Zhou J H,Rossi J. Nat. Rev. Drug Discov.,2017,16(3): 181-202
22 Hollenstein M,Hipolito C J,Lam C H,Perrin D M. Nucleic Acids Res.,2009,37(5): 1638-1649
23 Davydova A,Vorobyeva M,Bashmakova E,Vorobjev P,Krasheninina O,Tupikin A,Venyaminova A. Anal. Biochem.,2019,570: 43-50
24 Liu M,Yin Q,Brennan J D,Li Y. Biochimie,2018,145: 151-157
25 Aldag J,Persson T,Hartmann R. Int. J. Mol. Sci.,2018,19(12): 3883-3398
26 Kratschmer C,Levy M. Nucleic Acid Ther.,2017,27(6): 335-344
27 Rthlisberger P,Hollenstein M. Adv. Drug Delivery Rev.,2018,134: 3-21
28 Levi-Acobas F,Katolik A,Rthlisberger P,Cokelaer T,Sarac I,Damha M J,Hollenstein M. Org. Biomol. Chem.,2019,17(35): 8083-8087
29 Kamatkar N,Levy M,Hébert J M. Mol. Ther. Nucleic Acids,2019,17: 530-539
30 Kimoto M,Nakamura M,Hirao I. Nucleic Acids Res.,2016,44(15): 7487-7494
31 Wang H,Lam C H,Li X,West D L,Yang X. Biochimie,2018,145: 125-130
32 Gawande B N,Rohloff J C,Carter J D,von Carlowitz I,Zhang C,Schneider D J,Janjic N. Proc. Natl. Acad. Sci. USA,2017,114(11): 2898-2903
33 Chen T J,Hongdilokkul N,Liu Z X,Adhikary R,Tsuen S S,Romesberg F E. Nat. Chem.,2016,8(6): 557-563
34 Thirunavukarasu D,Chen T J,Liu Z X,Hongdilokkul N,Romesberg F E. J. Am. Chem. Soc.,2017,139(8): 2892-2895
35 Liu Z X,Chen T J,Romesberg F E. Chem. Sci.,2017,8(12): 8179-8182
36 Chen T J,Romesberg F E. Angew. Chem. Int. Edit.,2017,56(45): 14046-14051
37 Taylor A I,Pinheiro V B,Smola M J,Morgunov A S,Peak-Chew S,Cozens C,Weeks K M,Herdewijn P,Holliger P. Nature,2015,518(7539): 427-430
38 Larsen A C,Dunn M R,Hatch A,Sau S P,Youngbull C,Chaput J C. Nat. Commun.,2016,7: 11235
39 Kimoto M,Yamashige R,Matsunaga K,Yokoyama S,Hirao I. Nat. Biotechnol.,2013,31: 453-457
40 Matsunaga K,Kimoto M,Hirao I. J. Am. Chem. Soc.,2017,139(1): 324-334
41 Zhang L Q,Yang Z Y,Trinh T L,Teng I T,Wang S,Bradley K M,Hoshika S,Wu Q F,Cansiz S,Rowold D J,McLendon C,Kim M S,Wu Y,Cui C,Liu Y,Hou W J,Stewart K,Wan S,Liu C,Benner S A,Tan W H. Angew. Chem. Int. Edit.,2016,55(40): 12372-12375
42 Golden M C,Collins B D,Willis M C,Koch T H. J. Biotechnol.,2000,81(2-3): 167-178
43 Maasch C,Buchner K,Eulberg D,Vonhoff S,Klussmann S. Nucleic Acids Symp. Ser.,2008: 61-62
44 Wang Z M,Xu W L,Liu L,Zhu T F. Nat. Chem.,2016,8(7): 698-704
45 Pech A,Achenbach J,Jahnz M,Schulzchen S,Jarosch F,Bordusa F,Klussmann S. Nucleic Acids Res.,2017,45(7): 3997-4005
46 Vater A,Klussmann S. Curr. Opin. Drug Discov. Devel.,2003,6(2): 253-261
47 Kanwar J R,Roy K,Kanwar R K. Crit. Rev. Biochem. Mol. Biol.,2011,46(6): 459-477
48 Yang D K,Chou C F,Chen L C. RSC Adv.,2018,8(34): 19067-19074
49 Tsao S M,Lai J C,Horng H E,Liu T C,Hong C Y. Sci. Rep.,2017,7: 45478
50 Sypabekova M,Bekmurzayeva A,Wang R,Li Y,Nogues C,Kanayeva D. Tuberculosis,2017,104: 70-78
51 Rose K M,Ferreira-Bravo I A,Li M,Craigie R,Ditzler M A,Holliger P,DeStefano J J. ACS Chem. Biol.,2019,14(10): 2166-2175
52 Yang C,Wang Y,Ge M H,Fu Y J,Hao R,Islam K,Naranmandura H. Biomater. Sci.,2019,7(3): 938-950
53 Shubham S,Hoinka J,Banerjee S,Swanson E,Dillard J A,Lennemann N J,Przytycka T M,Maury W,Nilsen-Hamilton M. Sci. Rep.,2018,8: 12373
54 Li K,Xiu C L,Gao L M,Shi M,Zhai Y. J. South Med. Univ.,2016,36(12): 1592-1598
55 Alshaer W,Ababneh N,Hatmal M,Izmirli H,Choukeife M,Shraim A,Awidi A. PloS one,2017,12(12): 189558-189571
56 Donkor D A,Bhakta V,Eltringham-Smith L J,Stafford A R,Weitz J I,Sheffield W P. Sci. Rep.,2017,7: 2102
57 Yazdian-Robati R,Ramezani M,Khedri M,Ansari N,Abnous K,Taghdisi S M. Microchim. Acta,2017,184(10): 4029-4035
58 Leblebici P,Leirs K,Spasic D,Lammertyn J. Anal. Chim. Acta,2019,1053: 70-80
59 Zhou W,Zhang Y,Zeng Y,Peng M,Li H,Sun S,Liu J. Biochimie,2018,151: 150-158
60 Fukasawa K,Higashimoto Y,Ando Y,Motomiya Y. Ther. Apher. Dial.,2018,22(1): 61-66
61 Hu Y,Li X,An Y,Duan J,Yang X D. Oncotarget,2018,9(42): 26605-26616
62 Nazari M,Gargari S,L M,Sahebghadam Lotfi A,Rassaee M J,Taheri R A. Biochemistry,2019,58(18): 2373-2383
63 Alshaer W,Ababneh N,Hatmal M,Izmirli H,Choukeife M,Shraim A,Awidi A. PloS One,2017,12(12): e0189558
64 Bhardwaj J,Chaudhary N,Kim H,Jang J. Anal. Chim. Acta,2019,1064: 94-103
65 Díaz-Fernández A,Miranda-Castro R,de-los-Santos-lvarez N,Rodríguez E F,Lobo-Castaón M J. Biosens. Bioelectron.,2019,128: 83-90
66 Yu F,Li H,Sun W,Zhao Y,Xu D,He F. Talanta,2019,193: 110-117
67 Ma Y,Li X,Li W,Liu Z. ACS Appl. Mater. Interfaces,2018,10(47): 40918-40926
68 Liu M,Yin Q,Chang Y,Zhang Q,Brennan J D,Li Y. Angew. Chem. Int. Edit.,2019,58(24): 8013-8017
69 Tucker W,Kinghorn A,Fraser L,Cheung Y W,Tanner J. Int. J. Mol. Sci.,2018,19(3): 763-778
70 Wang C,Du X,Xie T,Li H. J. Sep. Sci.,2019,42(23): 3571-3578
71 Sedighian H,Halabian R,Amani J,Heiat M,Taheri R A,Fooladi A A I. Anal. Biochem.,2018,548: 69-77
72 Bayat P,Taghdisi S M,Rafatpanah H,Abnous K,Ramezani M. Talanta,2019,194: 399-405
73 Sedighian H,Halabian R,Amani J,Heiat M,Amin M,Fooladi A A I. J. Biotech.,2018,286: 45-55
74 Moreno M,Fernández-Algar M,Fernández-Chamorro J,Ramajo J,Martínez-Salas E,Briones C. Molecules,2019,24(7): 1213-1229
75 Eissa S,Zourob M. Biosens. Bioelectron.,2017,91: 169-174
76 Heiat M,Ranjbar R,Latifi A M,Rasaee M J. Peptides,2016,82: 101-108
77 Chonco L,Fernández G,Kalhapure R,Hernáiz M J,Garcia-Oliva C,Gonzalez V M,Parboosing R. Nucleic Acid Ther.,2018,28(4): 242-251
78 Stuart C H,Riley K R,Boyacioglu O,Herpai D M,Debinski W,Qasem S,Gmeiner W H. Mol. Ther. Nucl. Acids,2016,5: 386-395
79 Zhu C,Wang X,Li L,Hao C,Hu Y,Rizvi A S,Qu F. Biochem. Biophys. Res. Commun.,2018,506(1): 169-175
80 Lisi S,Fiore E,Scarano S,Pascale E,Boehman Y,Duconge F,Ravelet C. Anal. Chim. Acta,2018,1038: 173-181
81 Zhu C,Li L,Yang G,Fang S,Liu M,Ghulam M,Qu F. Anal. Chim. Acta,2019,1070: 112-122
82 Zhu C,Li L,Yang G,Irfan M,Wang Z,Fang S,Qu F. Talanta,2019,205: 120088
83 Wakui K,Yoshitomi T,Yamaguchi A,Tsuchida M,Saito S,Shibukawa M,Yoshimoto K. Mol. Ther. Nucl. Acids,2019,16: 348-359
84 Wakui K,Abe A,Yoshitomi T,Furusho H,Yoshimoto K. Anal. Sci.,2019,35(5): 585-588
85 Le A T,Krylova S M,Kanoatov M,Desai S,Krylov S N. Angew. Chem. Int. Edit.,2018,58(9): 2739-2743
86 Yang G,Feng Q,Abstract. BCEIA,2017
87 Sinha A,Gopinathan P,Chung Y D,Lin H Y,Li,K H,Ma H P,Lee G B. Biosens. Bioelectron.,2018,122: 104-112
88 Liu X,Li H,Jia W,Chen Z,Xu D. Lab Chip,2017,17(1): 178-185
89 Park J W,Lee S J,Ren S,Lee S,Kim S,Laurell T. Sci. Rep.,2016,6: 27121
90 Olsen T R,Tapia-Alveal C,Yang K A,Zhang X,Pereira L J,Farmakidis N,Lin Q. J. Electrochem. Soc.,2017,164(5): B3122-B3129
91 Kim J,Olsen T R,Zhu J,Hilton J P,Yang K A,Pei R,Lin Q. Sci. Rep.,2016,6: 26139
92 Hong S L,Wan Y T,Tang M,Pang D W,Zhang Z L. Anal. Chem.,2017,89(12): 6535-6542
93 Olsen T,Zhu J,Kim J,Pei R,Stojanovic M N,Lin Q. SLAS Technol.,2017,22(1): 63-72
94 Kinghorn A B,Dirkzwager R M,Liang S,Cheung Y W,Fraser L A,Shiu S C C,Tanner J A. Anal. Chem.,2016,88(14): 6981-6985|
95 Yokoyama T,Tsukakoshi K,Yoshida W,Saito T,Teramoto K,Savory N,Ikebukuro K. Biotech. Bioengeer.,2017,114(10): 2196-2203
96 Ahirwar R,Nahar S,Aggarwal S,Ramachandran S,Maiti S,Nahar P. Sci. Rep.,2016,6: 21285
97 Bavi R,Liu Z,Han Z,Zhang H,Gu Y. Biochem. Biophys. Res. Commun.,2019,509(4): 937-942
98 Haghighi M,Khanahmad H,Palizban A. Molecules,2018,23(4): 715-728
99 Fafińska J,Czech A,Sitz T,Ignatova Z,Hahn U. Nucleic Acid Ther.,2018,28(6): 326-334
100 Takenaka M,Okumura Y,Amino T,Miyachi Y,Ogino C,Kondo A. Bioorg. Med. Chem. Lett.,2017,27(4): 954-957
101 Nafissi-varcheh N,Kazemi B,Aboofazeli R,Shahhosseini S,Tabarzad M. Iran. J. Pharm. Res.,2017,16(2): 734-741
102 Zhu C,Yang G,Ghulam M,Li L,Qu F. Biotechnol. Adv.,2019: 107432
103 Lenshof A,Laurell T. Chem. Soc. Rev.,2010,39(3): 1203-1217
104 Pappas D,Wang K. Anal. Chim. Acta,2007,601(1): 26-35
105 Hybarger G,Bynum J,Williams R F. Anal. Bioanal. Chem.,2005,384(1): 191-198
106 Cho M,Xiao Y,Nie J,Stewart R,Csordas A T,Oh S S,Soh H T. Proc. Natl. Acad. Sci. USA,2010,107(35): 15373-15378
107 Bae H,Ren S,Kang J,Kim M,Jiang Y,Jin M M,Kim S. Nucleic Acid Ther.,2013,23(6): 443-449
108 Qian J,Lou X,Zhang Y,Xiao Y,Soh H T. Anal. Chem.,2009,81(13): 5490-5495
109 Jiang Y,Zhu C,Ling L,Wan L,Fang X,Bai C. Anal. Chem.,2003,75(9): 2112-2116
110 Allison D P,Hinterdorfer P,Han W. Curr. Opin. Biotech.,2002,13(1): 47-51
111 Zlatanova J,Lindsay S M,Leuba S H. Prog. Biophys. Mol. Biol.,2000,74(1-2): 37-61
112 Miyachi Y,Shimizu N,Ogino C,Kondo A. Nucleic Acids Res.,2009,38(4): 21-21
113 Ye M,Hu J,Peng M,Liu J,Liu J,Liu H,Tan W. Int. J. Mol. Sci.,2012,13(3): 3341-3353
114 Liu Y,Kuan C T,Mi J,Zhang X,Clary B M,Bigner D D,Sullenger B A. Biol. Chem.,2009,390(2): 137-144
115 Sefah K,Shangguan D,Xiong X,Odonoghue M B,Tan W. Nat. Protoc.,2010,5(6): 1169-1185
116 Blank M,Weinschenk T,Priemer M,Schluesener H. J. Biol. Chem.,2001,276(11): 16464-16468
117 Shangguan D,Cao Z,Meng L,Mallikaratchy P,Sefah K,Wang H,Tan W. J. Proteome Res.,2008,7(5): 2133-2139
118 Mallikaratchy P,Tang Z,Kwame S,Meng L,Shangguan D,Tan W. Mol. Cell. Proteomics,2007,6(12): 2230-2238
119 He J,Wang J,Zhang N,Shen L,Wang L,Xiao X,Shangguan D. Talanta,2019,194: 437-445
120 Bing T,Shen L,Wang J,Wang L,Liu X,Zhang N,Xiao X,Shangguan D. Adv. Sci.,2019,6(11): 1900143
121 Chen L,He W,Jiang H,Wu L,Xiong W,Li B,Zhou Z,Qian Y. Int. J. Nanomed.,2019,14: 149-159
122 Fernández G,Moraga A,Cuartero M I,García-Culebras A,Pea-Martínez C,Pradillo J M,Hernández-Jiménez M,Sacristán S,Ayuso M I,Gonzalo-Gobernado R. Mol. Ther.,2018,26: 2047-2059
123 Mai J,Li X,Zhang G,Huang Y,Xu R,Shen Q,Lokesh G L,Thiviyanathan V,Chen L,Liu H. Mol. Pharm.,2018,15: 1814-1825
124 Zhang S,Kumar K,Jiang X,Wallqvist A,Reifman J. BMC Bioinformatics,2008,9(1): 126
125 Ma H,Liu J,Ali M M,Mahmood M A I,Labanieh L,Lu M,Wan Y. Chem. Soc. Rev.,2015,44(5): 1240-1256
126 Zhou L,Ji F,Zhang T,Wang F,Li Y,Yu Z,Ruan B. Talanta,2019,197: 444-450
127 Opazo F,Levy M,Byrom M,Schfer C,Geisler C,Groemer T W,Rizzoli S O. Nat. Methods,2012,9(10): 938-939
128 HUANG Zi-Ke,LIU Chao,FU Qiang-Qiang,LI Jin,ZOU Jian-Mei,XIE Si-Tao,QIU Li-Ping. Chinese J. Appl. Chem.,2018,35(1): 28-39
黃子珂,刘 超,付强强,李 进,邹建梅,谢斯滔,邱丽萍. 应用化学,2018,35(1): 28-39
129 Hwang S Y,Sun H Y,Lee K H,Oh B H,Cha Y J,Kim B H,Yoo J Y. Nucleic Acids Res.,2012,40: 2724-2733
130 Zhang Y,Lai B S,Juhas M. Molecules,2019,24(5): 941-963
131 Mufhandu H T,Gray E S,Madiga M C,Tumba N,Alexandre K B,Khoza T,Wibmer C K,Moore P L,Morris L,Khati M. J. Virol.,2012,86: 4989-4999
132 Kunii T,Ogura S I,Mie M,Kobatake E. Analyst,2011,136: 1310-1312
133 Berezovski M V,Lechmann M,Musheev M U,Mak T W,Krylov S N. J. Am. Chem. Soc.,2008,130(28): 9137-9143
134 Jia W,Ren C,Wang L,Zhu B,Jia W,Gao M,Zeng F,Zeng L,Xia X,Zhang X,Fu T,Li S,Du C,Jiang X,Chen Y,Tan W,Zhao Z,Liu W. Oncotarget,2016,7: 55328-55342

