基于TCGA数据库肺腺癌RNAs构建ceRNA网络的综合分析
唐怀慧 王忠帅 邵茜茜

摘要:目的 基于TCGA数据库挖掘肺腺癌差异表达的RNAs,通过ceRNA网络的综合分析并预测其与患者预后的相关性。方法 利用TCGA数据库下载肺腺癌的表达数据,使用R软件“edgeR”包初步筛选差异表达的RNAs,进一步构建肺腺癌的lncRNA-miRNA -mRNA的ceRNA网络,对ceRNA网络中的mRNAs进行了K-M生存分析。结果 筛选的差异表达的RNAs中有3个mRNA(ANLN、IGFBP1、TFAP2A)、4个lncRNA (AC015923.1、FGF12-AS2、LINC00211、MED4-AS1)、2个miRNA(hsa-mir-31、hsa-mir-490),均与预后相关。其中hsa-mir-31预后价值最高(P<0.001),LINC00461和has-mir-139为关键节点的ceRNA调控网络。结论 本次筛选出的3个mRNA、4个lncRNA和2个miRNA可作为肺腺癌的新候选预后因子,且LINC00461和has-mir-139是肺腺癌ceRNA 中两个重要的调控网络节点。
关键词:TCGA;肺腺癌;ceRNA网络;预后
Abstract:Objective To explore the differentially expressed RNAs of lung adenocarcinoma based on TCGA database, comprehensively analyze and predict the correlation with the prognosis of patients through ceRNA network.Methods The TCGA database was used to download the expression data of lung adenocarcinoma, Use the R software "edgeR" package to preliminarily screen differentially expressed RNAs to further construct the ceRNA network of lncRNA-miRNA-mRNA of lung adenocarcinoma, K-M survival analysis was performed on the mRNAs in the ceRNA network. Results The screened differentially expressed RNAs included 3 mRNA(ANLN, IGFBP1, TFAP2A), 4 lncRNA(AC015923.1, FGF12-AS2, LINC00211, MED4-AS1), and 2 miRNA(hsa-mir-31, hsa- mir-490), both related to prognosis. Among them, hsa-mir-31 has the highest prognostic value (P<0.001), and LINC00461 and has-mir-139 are key node ceRNA regulatory networks.Conclusion The 3 mRNA, 4 lncRNA and 2 miRNA selected this time can be used as new prognostic factors for lung adenocarcinoma, and LINC00461 and has-mir-139 are two important regulatory network nodes in ceRNA of lung adenocarcinoma.
肺腺癌(lung adenocarcinoma)是癌癥死亡的重要原因,目前其发病率和死亡率仍在不断加[1]。男性肺癌患者发病率和死亡率在所有恶性肿瘤中占第1位,女性占第2位[2]。非小细胞肺癌(NSCLC)是肺癌中最常见的类型,其中以腺癌和鳞癌最常见,并且腺癌已经超过了鳞癌[3]。越来越多的研究证实,lncRNA可以作为miRNA和mRNA的竞争平台,在乳腺癌、胃癌、肝癌、肺癌及结直肠癌等多种恶性肿瘤的细胞周期和细胞死亡调控中起着重要的调节作用,影响肿瘤的侵袭和迁移,从而在肿瘤的发生、发展过程中发挥重要作用[4]。癌症基因组图谱(TCGA)是一个公共资助的项目,该项目可提供公开的癌症数据集,以帮助改进诊断方法、治疗标准,并最终预防癌症[5]。本研究利用高通量测序技术和生物信息学分析方法对肺腺癌的表达数据进行分析,为肺腺癌患者筛选新候选预后因子,以期为肺腺癌预后判断提供新的思路。
1资料与方法
1.1资料来源 利用TCGA数据库(https://portal.gdc.cancer.gov/)获取594例肺腺癌患者的mRNAs、lncRNAs和miRNAs表达数据(包括癌组织535例,癌旁正常组织59例),并收集患者的临床信息。基因表达量用于差异基因分析和构建ceRNA网络,患者的临床信息用于生存分析。
1.2方法
1.2.1筛选差异基因 基于TCGA数据库,利用R软件“TCGA biolinks”包获取肺腺癌的表达数据,perl语言提取Ensembl数据库鉴定了的lncRNAs和mRNAs。利用R软件“edgeR”包,除去所有表达量低的RNA(平均读数≤2)。设置差异倍数(fold change,FC)≥2,差异的显著性(fdr:调整P值)<0.01。将肺腺癌组织的表达数据与正常组织的表达数据进行差异分析,并利用R软件“gplots”包绘制火山图。
1.2.2 构建lncRNA-miRNA-mRNA的ceRNA网络 提取筛选出的差异表达RNAs,利用miRcode(http://www.mircode.org/)数据库预测差异lncRNAs和差异miRNAs之间的相互作用。miRDB(http://mirdb.org/),mirTarBase(http://www.mirbase.org/),miRanda(http://www.microrna.org/ microrna/home.do)和Target Scan(http://www.targetscan.org)数据库用于检索差异miRNAs的靶基因mRNAs。利用R软件“VennDiagram”包绘制Venny图,并利用Cytoscape v3.7.1构建肺腺癌 lncRNA-miRNA-mRNA的ceRNA网络。
1.2.3 生存分析 基于TCGA数据库下载的肺腺癌患者的临床信息,提取有效的生存状态及生存时间,通过构建Lasso回归模型和Cox风险回归分析,利用R软件“survival”包进行K-M生存分析,设置P<0.05。筛选出的差异RNAs认为与预后具有相关性。
2结果
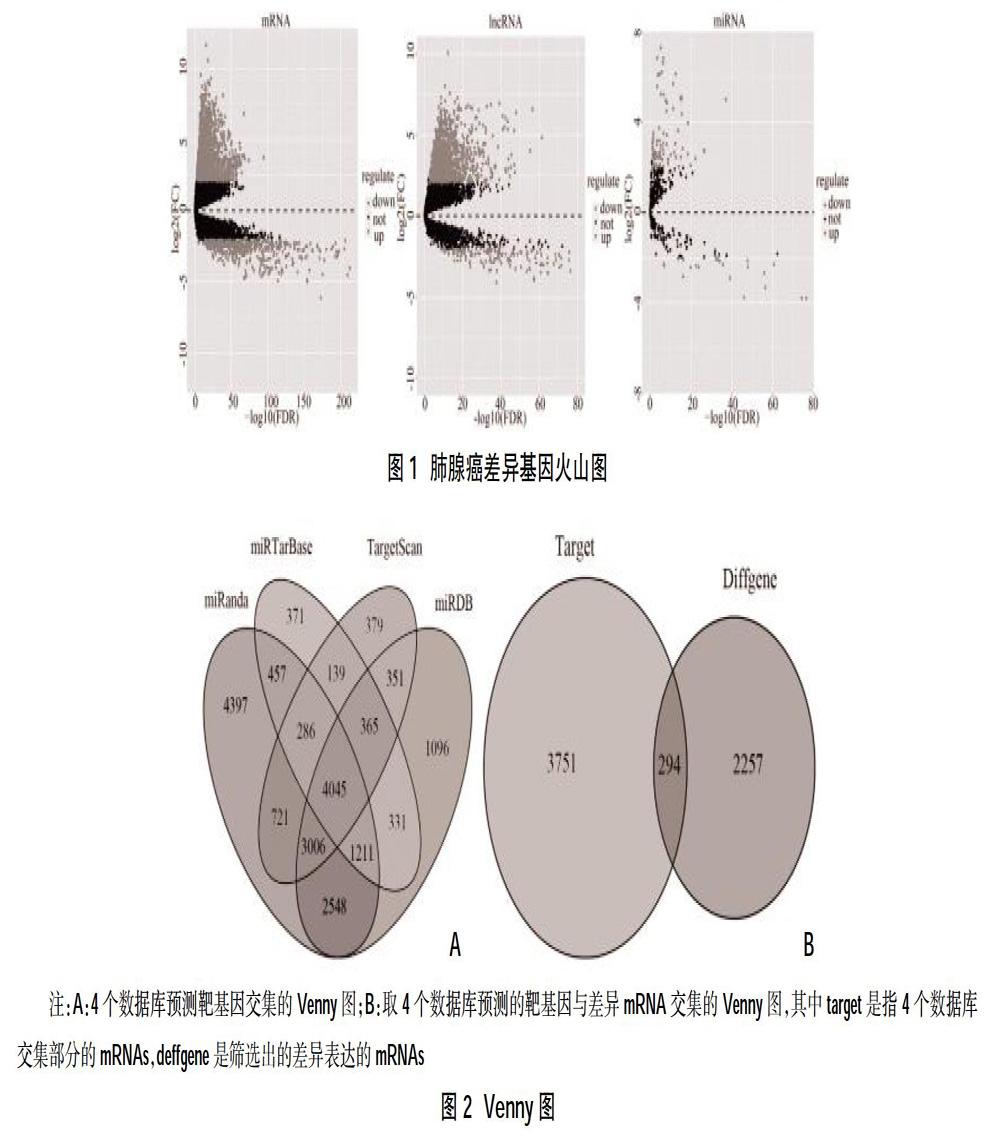
2.1差异表达的RNAs 共筛选出了2551个差异表达的mRNA,其中高表达的2033个,低表达518个;1359个差异表达的lncRNA,高表达的1185个,低表达的174个;99个差异表达的miRNA,高表达的78个,低表达21个。利用R软件“gplots”包绘制火山图,见图1 。
2.2肺腺癌 lncRNA-miRNA-mRNA ceRNA网络 通过miRcode数据库查找与差异表达的miRNA相关的差异lncRNA有100个,而与100个差异lncRNA对应的差异miRNA有43个,筛选出四个数据库共有的基因有4045个,见图2A。并与提取出的差异表达mRNA取交集,最终获得与肺腺癌有关的差异mRNA有294个,见图2B。对筛选出的100个lncRNA、43个miRNA和294个mRNA基因,利用Cytoscape v3.7.1绘制lncRNA-miRNA-mRNA的ceRNA网络,见3A、图3B,并提取了以LINC00461和has-mir-139为关键节点的核心子网络,见图3C、图3D。
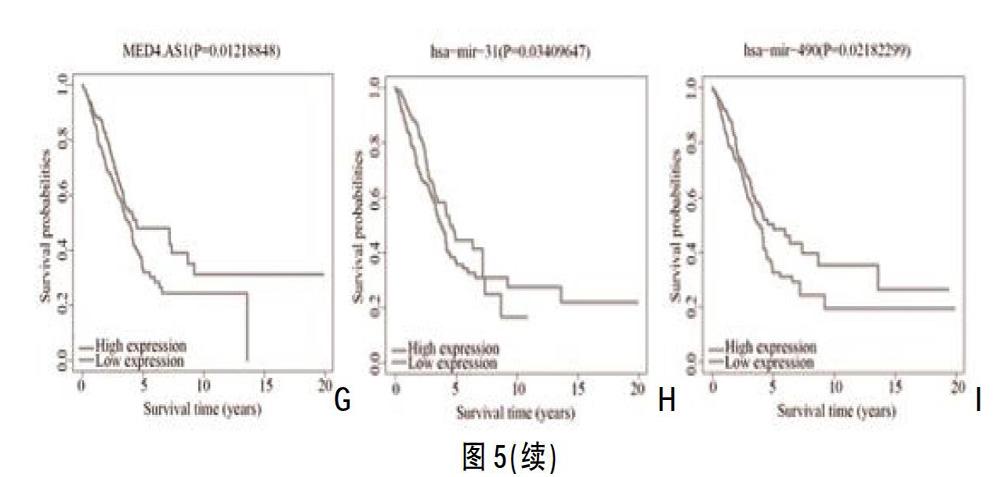
2.3生存分析 ①单因素Cox风险回归分析共筛选出对肺腺癌预后可能具有影响作用的有144个mRNA,29个lncRNA,5个miRNA。②采用LASSO回归鉴定预后相关的RNAs,Lasso回归模型拟合共筛选出7个mRNA,18个lncRNA和5个miRNA。③多因素Cox风险回归分析共发现3个mRNA:ANLN、IGFBP1、TFAP2A;7个lncRNA:AC015923.1、AC017048.3、FGF12-AS2、HOXA11-AS、LINC00211、LINC00319、MED4-AS1;3个miRNA:hsa-mir-142、hsa-mir-31、hsa-mir-490,可能与预后相关,其中hsa-mir-31具有强的预后价值,见图4。④Kaplan-Meier生存分析共发现3个mRNAs(ANLN,IGFBP1,TFAP2A),4个lncRNAs(AC015923.1,FGF12-AS2,LINC00211,MED4-AS1)和2个miRNAs(hsa-mir-31,hsa-mir-490)具有预后性,见图5。
3讨论
随着医疗技术的不断发展与创新,近年来对肺腺癌的治疗已取得了较大进展,肺癌患者的生存率有了一定的改善,但其治疗的疗效仍不尽如人意,肺腺癌患者的预后仍然很差,其5年总体生存率仍低于15%[6]。因此,尋找新的肺癌治疗策略已成为当前研究的热点。近年来高通量测序技术和生物信息学分析方法的不断完善,开辟了研究肺腺癌的新思路和新方法。TCGA是一个公共数据库,包含有关癌症中癌基因表达的全面数据集。本研究从TCGA数据库中获取了肺腺癌的表达数据,用以构建lncRNA- miRNA-mRNA的ceRNA网络,并分析参与ceRNA网络中的RNAs的预后相关性。
目前,越来越多的研究已经证明ceRNA涉及多种的肿瘤发生发展过程,其在该过程中具有重要生物学功能。如ceRNA网络的构建揭示了直肠腺癌中潜在的lncRNA生物标志物[7]。FOXO1 3'UTR通过调节miRNA活性在抑制乳腺癌细胞转移中起ceRNA的作用[8]。长的非编码RNA Unigene56159通过在肝细胞癌中充当miR-140-5p的ceRNA来促进上皮-间质转化[9]。随着对ceRNA网络的不断深入开发和挖掘,本研究前期发现已不能将miRNA,lncRNA或mRNA视为在肿瘤中的独立影响元素。在对miRNA的表达进行调控之后,由miRNA调节的靶基因mRNA的表达也随之改变,最终导致了多种疾病(包括肿瘤在内)的发生。由于癌症的发生和发展机制过于错综复杂,单基因或单一途径的作用已非常有限。因此,系统构建和分析ceRNA网络可以提供更有针对性的研究领域和揭示癌症潜在机制的新视角。
本研究共筛选出2551个差异表达的mRNA,1359个差异表达的lncRNA和99个差异表达的miRNA,通过生物信息学数据库筛选出与差异表达的miRNA相关的差异lncRNA有100个,而与100个差异lncRNA对应的差异miRNA有43个,检索和提取出与43个差异miRNA相关的差异mRNAs有294个,构建了肺腺癌的ceRNA网络,发现了以LINC00461和has-mir-139为关键节点的ceRNA调控子网络。通过对ceRNA网络中涉及的差异RNAs进行单因素Cox风险回归分析、构建LASSO回归模型以及多因素Cox风险回归分析,共发现3个mRNA,7个lncRNA和3个miRNA可能具有预后相关性,最终通过K-M生存分析绘制生存曲线发现3种mRNA ,4种lncRNA,2种miRNA具有预后相关性,且hsa-mir-31显示出最大的预后价值。
总之,通过构建肺腺癌的lncRNA-miRNA-mRNA的ceRNA网络,本次筛选出的3个mRNA、4个lncRNA和2个miRNA可作为肺腺癌的新候选预后因子,且LINC00461和has-mir-139是肺腺癌ceRNA 中两个重要的调控网络节点。
参考文献:
[1]Liu H,Zhou G,Fu X,et al.Long noncoding RNA TUG1 is a diagnostic factor in lung adenocarcinoma and suppresses apoptosis via epigenetically silencing of BAX[J].Oncotarget,2017(8):101899-101910.
[2]Torre LA,Bray F,Siegel RL,et al.Global Cancer Statistics,2012[J].CA Cancer J Clin,2015,65(2):87-108.
[3]Yang J,Lin J,Liu T,et al.Analysis of lncRNA expression profiles in non-small cell lung cancers (NSCLC) and their clinical subtypes[J].Lung Cancer,2014,85(2):110-115.
[4]Chen X,Chen Z,Yu S,et al.Long noncoding RNA LINC01234 functions as a competing endogenous RNA to regulate CBFB expression by sponging miR-204-5p in gastric cancer[J].Clin Cancer Res,2018,24(8):2002-2014.
[5]Tomczak K,Czerwińska P,Wiznerowicz M.Review The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge[J].Contemp Oncol(Pozn),2015,19(1A):A68-A77.
[6]Wakeam E,Acuna SA,Leighl NB,et al.Surgery Versus Chemotherapy and Radiotherapy For Early and Locally Advanced Small Cell Lung Cancer:A Propensity-Matched Analysis of Survival[J].Lung Cancer,2017(109):78-88.
[7]Zhang Z,Wang S,Ji D,et al.Construction of a ceRNA network reveals potential lncRNA biomarkers in rectal adenocarcinoma[J].Oncology Reports,2018,39(5):2101-2113.
[8]Yang J,Li T,Gao C,et al.FOXO1 3′UTR functions as a ceRNA in repressing the metastases of breast cancer cells via regulating miRNA activity[J].FEBS Letters,2014,588(17):3218-3224.
[9]Lv J,Fan HX,Zhao XP,et al.Long non-coding RNA Unigene56159 promotes epithelial–mesenchymal transition by acting as a ceRNA of miR-140-5p in hepatocellular carcinoma cells[J].Cancer Letters,2016,382(2):166-175.
收稿日期:2019-12-05;修回日期:2019-12-16
編辑/成森

