基于半监督多尺度特征分解网络的CTA图像冠状动脉分割
赵凤军 张涵 陈一兵 贺小伟 宋小磊

摘要:CT血管造影圖像(CT angiography,CTA)已成为心血管疾病筛查、诊断和治疗的重要依据,从图像中分割提取出冠状动脉对于形象展示冠脉解剖结构、分析评估血流情况和疾病精准诊断有重要意义。针对CTA图像特点,该文提出了一种只需少量标记数据的半监督深度学习方法进行冠脉分割。由于不同层面图像中冠脉血管直径大小不一,该文构建了包含编解码模块的多尺度网络来有效分割不同尺寸冠脉,其中,编码模块中加入跳跃连接以获取更丰富的特征信息,而解码阶段加入密集连接模块来避免网络过深产生的梯度消失。在测试时,首先,使用卷积神经网络识别包含冠脉的切片,然后,在筛选出的包含冠脉切片中使用前述训练网络进行冠脉分割,以进一步减少由于半监督学习中标记过少而产生的过拟合现象。实验使用了28个心脏病病人的CTA数据,冠脉三维结构分割结果的灵敏度、特异性、准确率、阳性预测值、阴性预测值与Dice系数分别为86.86%,99.79%,99.99%,66.12%,99.99%,0.721 6,优于其他对比方法,证明该文提出的多尺度特征分解网络能够有效完成冠状动脉分割任务。
关键词:冠状动脉分割;多尺度特征分解网络;深度学习;半监督学习;CTA图像
中图分类号:TP39
DOI:10.16152/j.cnki.xdxbzr.2020-04-007开放科学(资源服务)标识码(OSID):
Coronary artery segmentation from CTA images using asemi-supervised learning method with a multi-scaledecomposition network
ZHAO Fengjun1,2, ZHANG Han1,2, CHEN Yibing1,2,HE Xiaowei1,2, SONG Xiaolei1,2
(1.School of Information Science and Technology, Northwest University, Xi′an 710127, China;
2.Xi′an Key Lab of Radiomics and Intelligent Perception, Northwest University, Xi′an 710127, China)
Abstract: CT angiography (CTA) has been frequently used for screening, diagnosis and treatment of cardiovascular diseases, and segmentation of coronary arteries from 2D CTA slices can aid in visualization and analysis of coronary anatomy, assessment of blood flow and precise diagnosis. According to the scenery of CTA segmentation, a semi-supervised deep learning method which only required a small amount of tag data is proposed. To adapt the varied diameters of vessels at different image slices, a multi-scale networks with encoder and decoder modules is defined, whereas in encoder module skip connections were employed to capture more features and in decoder stage dense block were added to avoid gradient vanishing caused by too deep network. To overcome the overfitting phenomenon due to too few tagging in semi-supervised learning, in the test phase, the slices that contained coronary artery pixels were firstly selected by a convolutional neural network. The selected slices were then passed through previous mentioned training network to achieve coronary artery segmentation. The experiment used CTA data from 28 heart patients. We finally achieve the 3D result of coronary artery segmentation in CTA images. The sensitivity, specificity, accuracy, positive predictive value, negative predictive value and Dice coefficient are 86.86%, 99.79%, 99.99%, 66.12%, 99.99%, and 0.721 6, which are better than the contrasted methods. The experiments have demonstrated the effectiveness of the proposed multi-scale spatial decomposition network for segmentation of coronary artery.
Key words: coronary artery segmentation; multi-scale spatial decomposition network; deep learning; semi-supervised learning; computed tomography angiography
冠状动脉疾病是最常见的心脏病类型,也是目前导致人类死亡的重要原因之一。当冠状动脉产生狭窄或被阻塞时,流向心肌的血流量减少有可能导致心肌缺氧,从而会引起心绞痛、心肌梗死以及心律失常等致命后果[1]。
近年来,冠状动脉CT血管造影(CT angiography,CTA)由于无创、费用低和多视角等的特点,已成为心血管疾病筛查、诊断和治疗的重要依据。在CTA断层图像中准确分割提取出冠状动脉血管,对于形象展示冠脉解剖结构、建模评估血流情况、疾病精准诊断和治疗决策有重要意义。然而,临床医生手动跟踪冠脉结构异常较为困难,人工分析也非常耗时,且诊断易受主观因素影响。因此,CTA图像中冠状动脉的精准自动分割具有重要的实际意义和临床价值。
现有的冠脉分割方法主要包括主动轮廓模型、区域生长法、最小路径法、匹配滤波法以及基于机器学习的方法等[2-5]。近几年,越来越多的机器学习算法被应用于血管分割领域,即将其看作像素分类问题,判断每个像素是否为血管[6-8]。然而,传统机器学习方法需要将自行设计的特征与分类器相结合,而特征设计的复杂性与不同分类器的选择是应用传统机器学习方法的一大挑战。深度学习算法则可以通过端到端学习,直接从输入图像中输出期望结果,尤其适用于拥有海量数据的医学图像分割场景。Liskowski等人首次在视网膜血管的分割中使用深度卷积神经网络(convolutional neural networks,CNN)[9],此后CNN在各类血管分割任务中得到了广泛的应用;Wu等人通过主成分分析和最近邻检索来训练CNN模型预测局部血管概率图,并将结果映射到概率追踪框架中来提取整个血管树[10];Prentasic等人将CNN应用于光学相干断层成像血管造影中进行血管分割[11];Maji等人提出了集合CNN用于血管分割[12],首先训练多个CNN来分割血管和非血管区域,然后求出多个CNN响应的平均值。
尽管深度学习方法在血管分割中取得了一定成果,其用于心臟CTA图像的冠脉分割仍存在巨大挑战。第一,冠脉血管结构复杂,直径尺寸大小不一,且存在由于心脏跳动产生的运动伪影;第二,冠脉分割方法大多以冠脉血管与背景区域的强度差别为基础,而心脏CTA图像中存在一些与冠状动脉强度相似的组织器官,比如心脏腔室、肺动静脉、骨骼等,因此冠脉分割过程中容易将与冠脉强度相似的组织器官一并分割出来,造成过分割现象;第三,难以获取大量标签数据,深度学习中网络模型的训练往往需要大量标记数据,而专家手工分割血管耗时耗力,且存在观察者误差,因此很难拥有大量冠状动脉血管标签。
针对上述挑战,本文提出一种半监督多尺度特征分解网络(multi-scale spatial decomposition network,MSDNet)实现心脏CTA图像中冠状动脉的三维结构分割。本文方法的主要贡献在于:
1)针对不同层面图像中冠脉血管直径大小不一,构建了改进的多尺度特征分解网络,包括多尺度模块、跳跃连接以及密集连接模块。通过引入空洞卷积设计了多尺度模块,以捕获血管的多尺度信息;编码阶段加入跳跃连接获得更丰富的特征信息;解码阶段加入密集连接模块避免由于网络过深而产生的梯度消失现象。
2)设计有监督损失函数和无监督损失函数组成总损失函数。结合大量无标签数据进行冠脉分割模型训练,节省了专家手工分割的标签资源。
3)为了减少在测试数据的背景切片上进行冠脉分割而产生过分割现象,本文提出基于一种带有注意力机制的卷积神经网络用于冠脉血管切片的识别,将含有血管的切片识别出来后再进行冠状动脉分割。
1 相关工作
1.1 分解表征学习
学习与任务相关的良好特征是机器学习的长久目标[13]。一般来说,如果特征对于特定任务的有效性是可以解释的,那么便被认为是好的表征。在深度学习研究中,近年来提出一种新的学习方法被称为分解(factorized)学习,也叫作解纠缠(disentangled)学习,该方法在医学图像分析中极具潜力[14]。分解学习的优势在于:一方面,可以保留与主要任务不直接相关的信息,而且可以将分解得到的独立因素作为后续任务的输入;另一方面,由于不同因素之间是相互独立的,且这些因素表示了数据不同方面的特征。因此该方法为特征的可解释性提供了依据。
现有研究者提出变分自编码机(variational auto encoder,VAE)[15]和生成对抗网络(generative adversarial networks,GAN)[16]来实现这种分解表征学习。Biffi等人使用VAE学习潜在向量来完成三维心脏分割[17],训练出可用于疾病诊断的心脏形状模型。Mathieu等人将VAE和GAN思想进行结合,分解学习已知因素和其他因素[18]。在医学图像任务中,获取与解剖结构相关的空间信息和与成像模态相关的信息均至关重要。本文将分解表征学习用于医学图像中,将原始图像特征分解为与解剖结构相关的空间图谱(“解剖因素”)和与模态信息相关的高维向量(“模态因素”),从而对原始图像进行更好的表征。
1.2 半监督学习
将深度学习应用于医学图像时,面临的巨大挑战包括数据收集过程繁琐,以及对数据进行标记较为困难。因此,在医学图像处理中半监督学习的研究获得极大关注。通过半监督学习,我们将有标签数据和无标签数据结合起来训练,有效利用大量无标签数据的信息,提高分割精度。
GAN是目前较为流行的半监督深度学习算法,该网络包括生成器和鉴别器,生成器希望生成的假样本能够骗过鉴别器,而鉴别器则希望尽可能将原始样本和生成样本区分开来。生成器和鉴别器这两个网络完全独立,单独交替迭代训练,二者不断博弈,直至生成器和鉴别器无法提升自己,便得到了较好的模型。Nie等人采用对抗学习和置信度网络来进行半监督分割[19],Zhang等人提出深度对抗网络,利用未标记图像训练分割网络模型[20]。Luc等人提出的方法中鉴别器对生成器产生的分割掩膜进行分类[21],而Souly等人提出的方法中生成器用于增加数据集的大小,鉴别器用来执行分割[22]。
2 方 法
2.1 方法概述
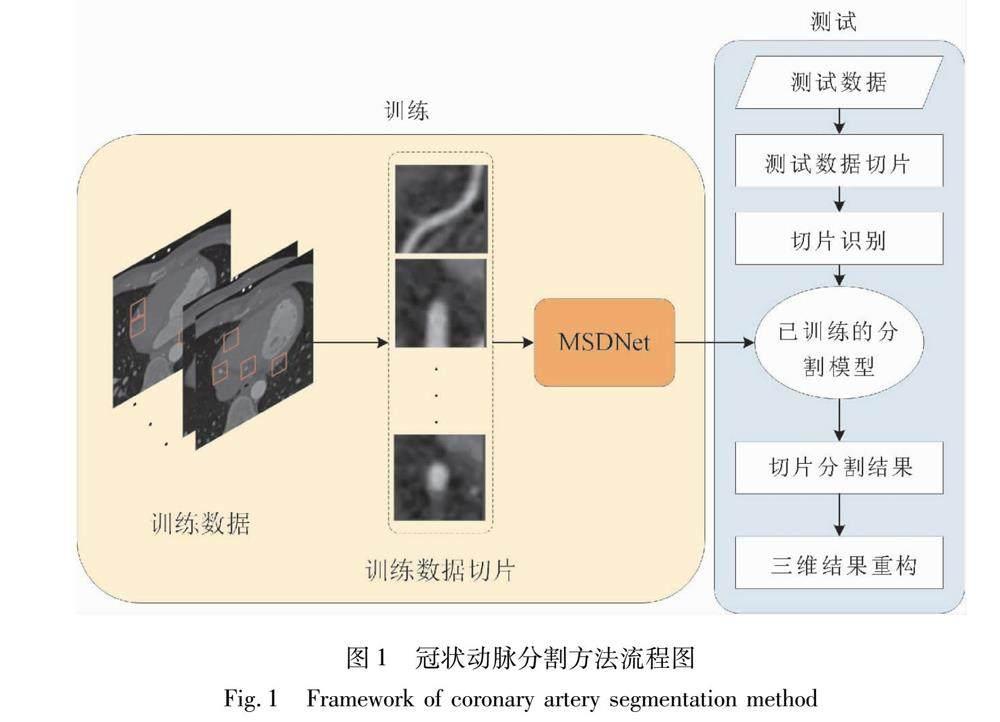
冠状动脉分割方法流程如图1所示,由训练阶段和测试阶段两部分组成。训练过程中,提取训练数据的二维切片送入MSDNet中进行分割网络的训练;测试过程中,首先,对测试数据中含有血管像素的切片进行识别,然后,将这些冠脉血管切片送入已训练好的MSDNet模型中得到二维切片的分割结果,最后,按原图尺寸将分割结果进行三维重组得出冠状动脉分割三维结果。
2.2 分割网络结构及改进
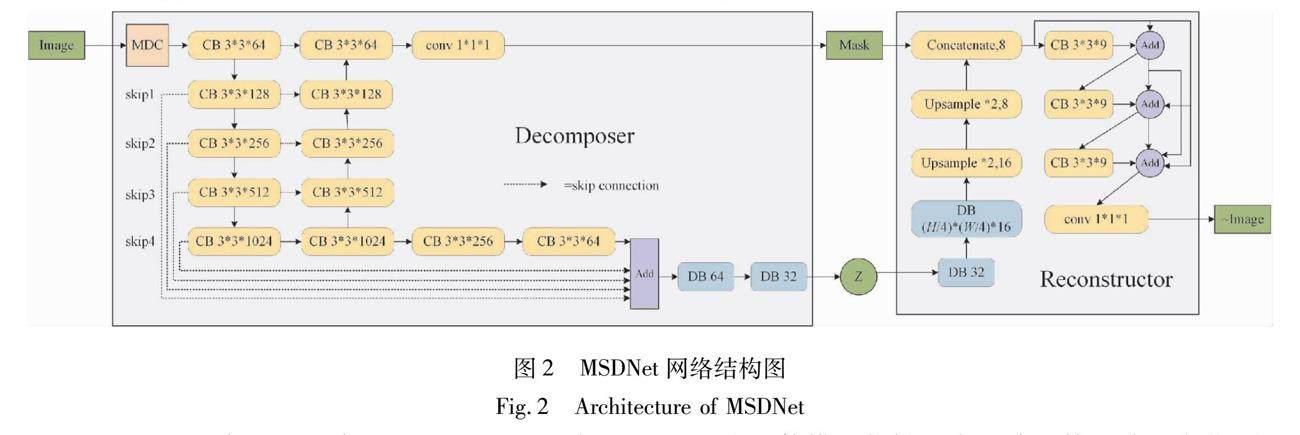
本文提出的MSDNet实现了特征分解表征,将与冠脉空间结构相关的信息和像素强度相关信息(与成像模态相关)分开学习。MSDNet由两个相互连接的神经网络组成,分别为“分解器”网络(decomposer)和“重构器”网络(reconstructor)。“分解器”即为编码过程,其将输入的二维图像(切片数据)分解为两个部分:一个类似于U-Net结构的网络将原图生成二进制掩膜形式的冠脉血管的空间图谱(Mask),另一个通过多层卷积产生包含周围结构和像素强度相关信息的潜在矢量表示(Z)。掩膜是一种输出像素与输入像素一一对应的图像,具有空间性,而潜在特征向量的作用是学习血管周围的一些拓扑结构以及必要的强度信息,允许多对多映射。解剖特征(Mask)和高维向量(Z)结合起来作为“重构器”的输入,并希望合成的重构图像与原图尽可能相似,因此“重构器”即为解码过程。如果成功分解,二元掩膜可以作为重构冠脉血管位置的重要指南。
图2为MSDNet的网络结构图,CB表示卷积模块, DB表示密集连接模块,MDC为多尺度空洞卷积模块,虚线表示跳跃连接部分。分解器的最后一层通过激活函数sigmoid输出血管的分割掩膜(Mask),從而得到原图的分割结果。模型的空间图谱包含下采样的图像信息,然后通过一系列的卷积和全连通层来获得潜在向量Z,最后的输出通过sigmoid函数,得到有边界范围的潜在向量Z。在重构器中使用了3个密集连接块对原图进行重构。
Mask和Z并不是明确独立的,所以,训练过程中模型可以存储所有需要的信息作为重构器的输入。本文在重构器的空间特征输入上应用了一个阶跃函数(即阈值)对前向输出中的掩膜进行二值化。在反向传播期间存储原始值并绕过阶跃函数,然后在原始的非二值掩膜上进行更新。掩膜的二值化只发生在输入端,二值化结果没有送入鉴别器。
MSDNet的改进主要包括以下3个方面:第一,为了使模型能够适应冠脉血管尺寸的变化,在分解器阶段定义了多尺度模块来获取冠脉血管的多尺度特征;第二,在高维特征Z的获取网络中添加多层跳跃连接,将浅层信息融入到深层;第三,在重构网络中,引入密集连接模块来获取更加丰富的冠脉血管信息。
2.2.1 多尺度空洞卷积模块 冠脉血管的一个显著特点是血管直径变化较大,这就要求冠脉血管分割算法能获取多尺度的血管信息。然而,目前大多数基于CNN的血管分割方法都缺乏获取多尺度特征的模块。
本文基于Inception模块的思想[23]。设计了一个基于并行结构的多尺度卷积(multiscale dilated convolution,MDC)模块。与Inception模块相比,本文的模块使用了空洞卷积,避免了大卷积核的使用。使用空洞卷积代替大卷积核可以有效减少参数量,提高网络的非线性表达能力。空洞卷积是一种带有空洞率r的卷积,它可以在不增加滤波器参数的情况下有效地扩大滤波器的视野。空洞率为r的空洞卷积在相邻滤波器值之间插入了r-1个零,因此,卷积核为k×k的滤波器便被扩大为[k+(k-1)(r-1)]×[k+(k-1)(r-1)]。通过调整滤波器中不同的空洞率r,便可获得不同的视野。本文使用不同的空洞率实现网络中的MDC模块来获得图像的多尺度信息。
MDC模块由4个滤波器组成,3个3×3的空洞卷积,空洞率r分别为1,2,3,和一个空洞率r=3,卷积核为1×1的空洞卷积。通过不同的视野并行获取多尺度信息后,再将4个通道连接起来。考虑到血管属于小目标,因此在多次卷积操作后引入多尺度模块不如在原图输入后便引入多尺度模块。
2.2.2 跳跃连接 本文的分解器网络基于U-Net结构,将原图分解为空间特征Mask和高维特征Z。空间特征获取中,使用跳跃连接将下采样层与上采样层相连,而高维特征通过多次卷积、下采样来实现。考虑到一次下采样后可能会丢失大量的细节信息,在高维特征的获取过程中加入了长跳跃连接,如图2中skip1,skip2,skip3,skip4为本文网络中新添加的跳跃连接部分。在跳跃连接中,从低层到高层的连接均使用池化、卷积,最后相加完成。skip1对输入尺寸32×32×64的特征进行最大池化操作,其中,池化窗口7×7,步长为7;skip2对输入尺寸16×16×128的特征向量先进行卷积,卷积核为3×3,通道为64,接着进行最大池化,池化窗口4×4,步长为4;skip3对输入尺寸8×8×256的特征向量先进行卷积,再进行最大池化,卷积核为3×3,通道为64,池化窗口2×2,步长为2;skip4对尺寸为4×4×512的特征向量进行两次3×3卷积,通道数分别为256和64,最后将这些跳跃连接结果与对应尺寸的高维特征求和。跳跃连接可以将浅层的信息与更深层的信息结合,获得更丰富的血管特征信息,适合于冠状动脉血管分割任务。
2.2.3 密集连接模块 网络的深度是获得较好分割结果的重要因素,对于冠脉血管分割任务,更深层次的网络意味着提取的语义信息更丰富,因此分割结果会更好。然而,过深的网络在训练过程中会产生梯度消失现象,导致损失函数难以收敛。为了解决此问题,研究者们提出了密集连接网络[24],其增强了特征的有效传递,同时也能缓解网络过深带来的副作用。具体方法是将前面所有层的特征映射结合起来作为后面各层的输入,公式表示为:Xl=Hl([X0,X1,…,Xl-1]),其中,[X0,X1,…,Xl-1]表示将0到l-1层的输出特征做通道的合并。本文针对网络过深的情况使用了密集连接模块,如图2重构器部分所示。
2.2.4 分割网络损失函数
本文使用f和g分别表示分解器和重构器。输入切片表示为Xi,经过分解器f,分解得到掩膜M和16维的向量Z,这个过程表示为:f(Xi)={fM(Xi),fZ(Xi)}={M, Z}。 重构器g将分解结果映射回原图, 表示为g(fM(Xi),fZ(Xi))。
在半监督学习中,数据来源有两组,一组是有标签数据集SL={Xi,Mi},i=1,2,…,N,其中,Xi表示输入数据,Mi表示与其相对应的标签,i表示有标签数据的数量;另一组为无标签数据集SU={Xj},j=1,2,…,Q,其仅包含数据Xj而没有标签,j表示无标签数据的数量。训练过程中的损失函数由3类不同的损失函数组成,分别为重构损失函数、有监督损失函数和对抗损失函数。
1)重构损失函数:计算重构图1与原图之间的误差得到重构损失函数,计算公式为
Lrec(f,g)=EX[||X-g(f(X))||1]。
2)有监督损失函数:根据部分有标签数据,设计了两个有监督损失函数。
a)分割结果与标签之间的Dice系数值作为第一个有监督损失函数,表示为
LM(f)=EX[Dice(MX,fM(X))]。
b)将标签与分解器得到的高维向量Z一起送入重构器2中得到重构图,然后计算重构图2与原图之间的平均绝对值误差作为第二个有监督损失函数,表示为
LI(f,g)=EX[||X-g(MX,fZ(X))||1]。
3)对抗损失函数:网络的训练过程包含生成对抗的思想,重构图1看作关于原图的伪图片,解剖特征Mask即分割结果可以作为冠脉标签的伪图片。根据生成对抗思想,本文定义的两个对抗损失函数如下:
a)重构图1与原图进行比较,鉴别器为DX,则对抗损失AI为
AI(f,g,DM)=EX[DX(g(f(X)))2+(DX(X)-1)2],
b)分割结果与冠脉标签进行比较,鉴别器为DM,则对抗损失AM为
AM(f)=EX,M[DM(fM(X))2+(DM(M)-1)2]。
网络训练过程中,上述损失函数分别用于有标签数据和无标签数据中,有标签数据集的总损失函数LossL表示为
LossL=λ1LM(f)+λ2AM(f,DM)+λ3Lrec(f,g)+λ4LI(f,g)+λ5AI(f,g,DX),
无标签数据集的总损失函数LossU表示为
LossU=λ2AM(f,DM)+λ3Lrec(f,g)+λ5AI(f,g,DX)。
其中,λ表示各项的参数。
2.3 冠脉血管切片识别网络
2.3.1 测试过程介绍 训练完成的MSDNet模型只能用于分割尺寸大小为64×64的二维图像。为了使训练好的MSDNet模型能实现三维冠脉分割,首先,在测试数据中进行64×64的切片(patch)提取,具體方法是从左上角取一个64×64的滑动窗口,移动步长设置为S,实验中S取64,依次覆盖整个图像,则此移动窗口的每一步都产生一个对应的patch;接着,将顺序采样得到的patch送入血管切片分类网络中,识别出含有冠脉像素的切片;然后,再将含有冠脉的patch送入训练好的MSDNet中,得到血管切片的二维分割结果;最后,将切片级的分割结果按照原图中切片位置索引以及原图大小组合起来,从而得到CTA图像的三维冠状动脉分割结果。
2.3.2 识别网络结构及改进 为了优化送入分割网络的数据并减少过分割现象,测试过程中使用带有注意力机制的卷积神经网络用于冠脉血管切片的识别,判断出含有冠脉血管像素的二维切片。
冠脉血管切片识别网络由卷积层和全连接层组成。其中,输入图片的尺寸为64×64×1,卷积层分为4层,每一层均由卷积、Relu和最大池化组成。第1层对原图进行卷积,卷积核尺寸为3×3,滤波器个数为64,最大池化的池化核是2×2,步长为2,且采用有填充的池化(图片外围填充像素0);第2层卷积中滤波器个数为128,其他均和第一层一致;第3层包含了两次卷积,卷积核尺寸是3×3,滤波器数量均为256,两次卷积完成后进行最大池化,池化核为2×2,步长为2,且为有填充的池化;第4层包含两次卷积,卷积核为3×3,滤波器个数均为512,两次卷积完成后进行最大池化,池化核为2×2,步长为1,且为无填充的池化。卷积层完成之后,连接的是3个全连接层,前两个全连接层的通道数是4 096,第3层根据分类类别而定,此处实现的是二分类,因此最终的通道数为2,最后的全连接层使用激活函数sigmoid输出二分类结果。
为了使分类结果更加准确,本文在每个卷积后加入注意力机制(convolutional block attention module,CBAM模块)[25],且在损失函数中会加入L2正则化项来减少过拟合。网络结构和参数分别如表1所示。
3 实 验
3.1 数据集和参数设置
3.1.1 数据集 本文使用的心脏CTA数据来自西门子双源CT扫描仪(SOMATOM Definition Flash),带有心电触发与对比度增强,管电压为120 kVp,管电流为55 mAs。总共的28个心脏病人CTA图像中14个处于心脏收缩期,14个处于心脏舒张期,图像前两维尺寸为512×512,第3维在197~276之间不等,灰度值在0~4 000之间。所有心脏CTA图像的冠脉体素金标准均由两位资深心脏病专家使用商用软件MITK手动标注,将图像体素标记为冠状动脉血管目标体素或非冠脉的背景体素。采集到的CTA图像经过重采样,每个体素对应0.5mm×0.5mm×0.5mm的真实空间。实验中随机选取10个数据作为训练集,3个数据作为验证集,剩余15个作为测试集。
3.1.2 实验参数设置 冠脉分割的训练过程中迭代次数(epoch)设置为100,训练停止条件为验证集数据的Dice系数值连续十次迭代没有升高。使用随机梯度下降(SGD)优化器,学习率固定为0.01,每次训练的批量大小设置为32。训练数据由两部分组成,有标签数据和无标签数据。有标签数据的损失函数由LM,AM,Lrec,LI,AI组成,系数分别设置为10,10,5,5,5;无标签数据的损失函数由AM,Lrec,AI组成,系数分别设置为10,5,5。
冠脉切片识别实验中迭代次数设置为150,训练完150个epoch则训练停止,使用Adam优化器,初始学习率为0.000 1,每隔两个epoch学习率下降5%,每次训练的批量大小设置为32。
3.2 实验评价指标
分割性能通过与专家手工分割的结果进行对比来评估。定量分析分割算法性能时,通常使用灵敏度、特异性、准确率、阳性预测值、阴性预测值以及Dice系数作为计算性能指标。其计算公式均与真阳(true positive,TP)、真阴(true negative,TN)、假阳(false positive,FP)和假阴(false negative,FN)相关。真阳表示被判定为正样本实际上也是正样本,真阴表示被判定为负样本实际也是负样本,假阳表示被判定为正样本实际是负样本,假阴表示被判定为负样本实际是正样本。
灵敏度(sensitivity)表示正确判断正样本的比率,计算公式为
Sensitivity=TPTP+FN。
特異性(specificity)表示正确判断负样本的比率,计算公式为
Sepcificity=TNTN+FP。
准确率(accuracy)表示所有样本中被正确识别的比率,计算公式为
Accuracy=TP+TNTP+FN+TN+FP。
阳性预测值(positive predictive value,PPV)表示预测出的正样本中实际为正样本的比率,计算公式为
PPV=TPTP+FP。
阴性预测值(negative predictive value,NPV)表示预测出的负样本中实际为负样本的比率,计算公式为
NPV=TNTN+FN。
Dice系数是一种集合相似度度量函数,通常用于计算两个样本的相似度(值范围为[0,1]),其计算公式为
Dice=2TPFP+FN+2TP
在实验阶段,本文通过上述评价指标,将提出的分割方法与改进前的方法以及其他分割方法进行性能比较。
3.3 二维切片分割结果及分析
本文使用空间分解网络(SDNet)作为分割结果的评价基准[26]。该网络原本用于MRI图像上心肌的分割,本文对其网络结构进行改进后用于CTA数据中冠状动脉的分割。为了验证本文提出改进模块的有效性,本节实验以测试数据二维切片分割结果的平均Dice系数作为评价标准,每组实验进行5次并将其平均值作为最终结果。
3.3.1 改进模块有效性验证 MSDNet在原网络基础上添加了3个改进模块,分别为跳跃连接、多尺度模块以及密集连接模块,为了验证其有效性,分别进行了如表2所示的8组实验,第1组实验为原始网络,第2到4组实验分别增添一种改进模块,第5到8组实验分别添加两到三种改进模块。
表2中看出,实验2到4分别添加不同改进模块后,Dice系数值相比原网络均有不同程度的提高,证明MSDNet中每一个改进模块对于冠状动脉的分割都是有效的。最终在原网络基础上同时添加3个改进模块可以得到最优的分割结果,如实验8中结果所示。
3.3.2 半监督方法有效性验证 为了定量的评估半监督方法的有效性,本实验通过调整有标签数据和无标签数据的比例,来验证无标签数据在网络训练中的有效性。实验分别在原始网络和几种改进网络上进行模型训练,然后对测试集中二维切片的分割结果进行比较。表3中第一列表示不同的网络结构,改进一表示在原始网络基础上添加多尺度模块,改进二表示在原始网络基础上添加跳跃连接,改进三在原始网络基础上添加密集连接模块,MSDNet为本文提出的网络(包含3个改进模块)。训练过程中保持无标签数据量(4 448张或0张)不变,依次减少有标签数据的数量,如表3第1行所示,第2到5列分别表示有标签数据量为4 448张,2 224张,1 112张,556张切片。表3中,斜线(/)左边的结果是无标签数据4 448张的Dice系数值,右边的结果是无标签数据0张的Dice系数值。
从表3中看出,无论是否使用无标签数据,分割结果的Dice系数均随着有标签数据量的减少而逐渐降低,而使用无标签数据参与训练后的结果(左边)普遍高于不使用无标签数据(右边)的结果。当训练过程中使用4 448张无标签数据时,即使有标签数据仅剩下556张切片,分割结果的Dice系数仍然可以达到0.5以上,而此时不使用无标签数据的结果在0.3或0.4左右,这表明无标签数据在网络训练过程中发挥了极大作用,特别是在有标签数据量较少的时候,无标签数据对模型的优化作用更加明显。另外,与原始网络和其他3种改进网络相比,本章提出的MSDNet网络在不同数据量的实验中,均取得了最高的Dice系数值。
3.4 三维分割结果与现有方法进行比较
上面实验对改进模块以及无标签数据有效性的证明均建立在测试数据的二维切片上。为了验证本文方法在三维分割结果上的有效性,我们对测试数据的二维切片进行三维重组得到冠状动脉的三维分割结果。同时,实验将本文提出的冠状动脉分割方法MSDNet和现有的一些分割方法进行比较,对比方法包括Vesselness滤波法[27],双高斯滤波法(Bi-Gauss)[28],U-Net网络[29],带有多层短连接的全卷积网络(FCNRes)[30], 以及SDNet网络[25]。 因为Vesselness和Bi-Gauss均为图像增强的方法, 所以使用不同的阈值进行后处理会得到不同的分割结果。 图4(c)和(d)所示二者的分割结果均是根据ROC曲线得到最优阈值处理的结果。 由分割结果看出, 这两种滤波方法可以分出冠脉的主干血管, 而对于一些较细小的血管却难以分割出来, 而且在主动脉入口部位产生了较大的错分现象, 其中, Bi-Guass滤波方法的错分在两者间更严重一些, 因此, 其分割指标相对更低一些, 如表4中第二三行所示。FCNRes网络采用全卷积方式,加入残差模块建立了更深的网络,分割结果如图4(e)所示,此方法能将冠脉主干及部分细小血管分割出来,然而容易将过多背景像素错分。U-Net是现在最流行的医学图像分割网络之一,本实验构建了三维U-Net网络,并将其用于CTA数据中的冠状动脉分割,分割结果如图4(f)所示,可以看出,U-Net网络不仅能分出冠脉主干,还能分出部分细小的血管,然而分出的细小血管会存在间断现象且容易将血管远端漏分。MSDNet在SDNet网络的基础上进行了改进,增加了跳跃连接、多尺度模块和密集连接模块,二者的冠脉分割结果如图4中(g)和(h)所示。可以看出,SDNet网络分割的冠脉结构存在部分血管间断现象,而本文的改进方法对其进行了改善,得到了更为连贯的冠脉结构。图4(b)为冠脉的金标准,通过与其进行比较可以看出,本文的方法不仅能将冠脉主干分割出来,而且能够分割出更多的细小血管,且冠脉结构更为连贯。
表4中给出了几种方法的冠脉分割结果在准确率(Acc)、灵敏度(Sen)、特异性(Sep)、阳性预测值(PPV)、阴性预测值(NPV)以及Dice系数上的结果。可以看出,与其他方法相比,本文提出的MSDNet网络在准确率、灵敏度、阳性预测值以及Dice系数上均取得最高值。其中,灵敏度(86.86%)最高反映出本文的方法能更多地将冠脉分割出来,Dice系数(0.721 6)最高表示本文分割的冠脉结构与金标准更为接近。
4 结 语
本文提出了用于心脏CTA图像中冠状动脉分割的一种半监督多尺度特征分解网络的深度学习方法。在心脏CTA图像中,针对心脏内外与冠脉强度相似的组织器官对分割结果的影响,本文设计含有注意力机制的卷积神经网络对实验数据中的二维切片进行二分类,识别出含有冠状动脉血管像素的切片;针对标签数据稀少的挑战,本文使用半监督多尺度特征分解网络用于冠状动脉分割。该网络包括3个方面的改进:①多尺度模块;②跳跃连接;③密集连接模块。本文方法最终实现了CTA图像中冠状动脉的三维分割,分割结果优于实验中的其他对比方法。实验证明了本文的多尺度特征分解网络在冠状动脉分割上的优越性,具有较大的临床应用潜力。
参考文献:
[1] ZHOU C, CHAN H P, CHUGHTAI A, et al. Automated coronary artery tree extraction in coronary CT angiography using a multiscale enhancement and dynamic balloon tracking (MSCAR-DBT) method[J]. Computerized Medical Imaging and Graphics, 2012, 36(1):1-10.
[2] ZHAO F J, CHEN Y R, HOU Y Q, et al. Segmentation of blood vessels using rule-based and machine-learning-based methods: A review[J]. Multimedia Systems, 2019, 25(2):109-18.
[3] 楊栋. 面向CTA图像的冠脉血管分割算法研究和血管狭窄度分析[D]. 杭州:浙江大学,2015.
[4] 王宇慧. 基于主动形状模型的医学图像分割方法研究[D]. 西安:西北大学,2015.
[5] TAYEBI R M , SULAIMAN P S B , WIRZA R , et al. Coronary artery segmentation in angiograms with pattern recognition techniques:A survey[C]∥2013 International Conference on Advanced Computer Science Applications and Technologies (ACSAT). IEEE, 2013.
[6] YOU X, PENG Q, YUAN Y, et al. Segmentation of retinal blood vessels using the radial projection and semi-supervised approach[J]. Pattern Recognition, 2011, 44(10):2314-24.
[7] 朱承璋,向遥,邹北骥,等.基于分类回归树和AdaBoost的眼底图像视网膜血管分割[J].计算机辅助设计与图形学学报,2014,26(3):445-451.
ZHU C Z,XIANG Y,ZOU B J,et al.Retinal vessel segmentation in fundus images using CART and AdaBoost[J].Journal of Computer-Aided Design & Computer Graphics, 2014,26(3):445-451.
[8] SCHNEIDER M, HIRSCH S, WEBER B, et al. Joint 3-D vessel segmentation and centerline extraction using oblique Hough forests with steerable filters[J]. Medical Image Analysis, 2015, 19(1):220-249.
[9] LISKOWSKI P, KRAWIEC K. Segmenting retinal blood vessels with deep neural networks[J]. IEEE Transactions on Medical Imaging, 2016, 35(11):2369-2380.
[10]WU A, XU Z Y, GAO M C,et al. Deep vessel tracking: A generalized probabilistic approach via deep learning[C]∥2016 IEEE 13th International Symposium on Biomedical Imaging (ISBI). IEEE, 2016.
[11]PRENTASIC P, HEISLER M, MAMMO Z, et al. Segmentation of the foveal microvasculature using deep learning networks[J]. Journal of Biomedical Optics, 2016, 21(7):075008.
[12]MAJI D, SANTARA A, MITRA P, et al. Ensemble of deep convolutional neural networks for learning to detect retinal vessels in fundus images[EB/OL].2016:arXiv:1603.04833[cs.LG].https:∥arxiv.org/abs/1603.04833.
[13]BENGIO Y, COURVILLE A, VINCENT P. Representation learning: A review and new perspectives[EB/OL].2012:arXiv:1206.5538[cs.LG].https:∥arxiv.org/abs/1206.5538.
[14]CHEN X, DUAN Y, HOUTHOOFT R, et al. InfoGAN: Interpretable representation learning by information maximizing generative adversarial nets[EB/OL].2016 :arXiv:1606.03657 [cs.LG].https:∥arxiv.org/abs/1606.03657.
[15]KINGMA D P, WELLING M. Auto-encoding variational Bayes[EB/OL].2013:arXiv:1312.6114 [cs.LG].https:∥arxiv.org/abs/1312.6114.
[16]GOODFELLOW I J, POUGET-ABADIE J, MIRZA M, et al. Generative adversarial networks[EB/OL].2014: arXiv:1406.2661 [cs.LG].https:∥arxiv.org/abs/1406.2661.
[17]BIFFI C, OKTAY O, TARRONI G, et al. Learning interpretable anatomical features through deep generative models: Application to cardiac remodeling[M]∥Medical Image Computing and Computer Assisted Intervention - MICCAI 2018. Cham: Springer International Publishing, 2018: 464-471.
[18]MATHIEU M, ZHAO J, SPRECHMANN P, et al. Disentangling factors of variation in deep representations using adversarial training[EB/OL].2016: arXiv:1611.03383 [cs.LG].https:∥arxiv.org/abs/1611.03383.
[19]NIE D, GAO Y Z, WANG L, et al. ASDNet: Attention based semi-supervised deep networks for medical image segmentation[M]∥Medical Image Computing and Computer Assisted Intervention - MICCAI 2018. Cham: Springer International Publishing, 2018: 370-378.
[20]ZHANG Y Z, YANG L, CHEN J X, et al. Deep adversarial networks for biomedical image segmentation utilizing unannotated images[M]∥Medical Image Computing and Computer Assisted Intervention - MICCAI 2017. Cham: Springer International Publishing, 2017: 408-416.
[21]LUC P, COUPRIE C, CHINTALA S, et al. Semantic segmentation using adversarial networks[EB/OL].2016: arXiv:1611.08408 [cs.CV].https:∥arxiv.org/abs/1611.08408.
[22]SOULY N, SPAMPINATO C, SHAH M. Semi and weakly supervised semantic segmentation using generative adversarial network[EB/OL].2017:arXiv:1703.09695 [cs.CV].https:∥arxiv.org/abs/1703.09695.
[23]SZEGEDY C, LIU W, JIA Y Q, et al. Going deeper with convolutions[EB/OL].2014: arXiv:1409.4842[cs.CV].https:∥arxiv.org/abs/1409.4842.
[24]HUANG G, LIU Z, MAATEN L V D, et al. Densely connected convolutional networks[C]∥Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR).Honolulu:IEEE,2017,2261-2269.
[25]WOO S, PARK J, LEE J Y, et al. CBAM: Convolutional block attention module[M]∥Computer Vision - ECCV 2018. Cham: Springer International Publishing, 2018: 3-19.
[26]CHARTSIAS A, JOYCE T, PAPANASTASIOU G, et al. Factorised spatial representation learning: Application in semi-supervised myocardial segmentation[EB/OL].2018: arxiv:1803.07031 [cs.CV]:https:∥arxiv.org/abs/1803.07031.
[27]SATO Y, NAKAJIMA S, SHIRAGA N, et al. Three-dimensional multi-scale line filter for segmentation and visualization of curvilinear structures in medical images[J].Medical Image Analysis, 1998, 2(2):143-168.
[28]XIAO C Y, STARING M, WANG Y N, et al. Multiscale Bi-Gaussian filter for adjacent curvilinear structures detection with application to vasculature images[J]. IEEE Transactions on Image Processing, 2013,22(1):174-188.
[29]RONNEBERGER O, FISCHER P, BROX T. U-net: Convolutional networks for biomedical image segmentation[M]∥Lecture Notes in Computer Science. Cham: Springer International Publishing, 2015: 234-241.
[30]HE K, ZHANG X, REN S, et al. Deep residual learning for image recognition[EB/OL].2015:arXiv:1512.03385 [cs.CV].https:∥arxiv.org/abs/1512.03385.
(編 辑 李 静)

