低氮条件下响应接种根瘤菌的大豆产量相关性状遗传位点及基因挖掘
霍晓波,李喜焕,杜 汇,孔佑宾,李文龙,张彩英
(河北农业大学 教育部华北作物种质资源研究与利用重点实验室,河北 保定 071001)
大豆是人类食用油与植物蛋白重要来源,在国民经济中占有不可替代的地位。同时由于大豆可通过与根瘤菌互作进行共生固氮,因而又是养地作物,在农作物轮作倒茬中具有重要作用[1]。据统计,大豆籽粒中的生物固氮量在肥沃与贫瘠土壤条件下可分别达到25%~50%和80%~90%。因此,根瘤固氮成为提高大豆产量、减少化肥使用、维持农业可持续发展重要途径[2-4]。然而,大豆根瘤固氮属于多基因控制数量性状[5],其对大豆产量的作用机制亦同样复杂。
全基因组关联分析和连锁分析是进行基因定位的重要方法。Prabhu等以自然群体为研究材料,定位到与固氮量、氮含量以及C/N显著关联SNP 60个[6];Kamfwa等通过259份菜豆自然群体的叶绿素含量、根瘤个数、地上部生物重、地上部氮百分含量等固氮相关性状,结合基因型数据(5 398个SNP),关联到40个显著关联SNP,分布于9条染色体[7];Zhou等以302份重测序大豆野生品种、地方品种和育成品种为材料,关联到控制含油量、株高、茸毛等13个农艺性状的230个QTL[8];Sonah等以139个大豆品种为材料,发掘了与花色、种脐颜色、株高、籽粒油分和蛋白等8个性状显著关联的25个遗传位点[9];Fang等鉴定了809份大豆品种的84个农艺性状,检测到245个显著关联SNP位点,其中95个遗传位点之间存在互作[10];Zhang等鉴定了314份大豆的镰刀菌抗性,发掘了12个遗传位点,分布于8条染色体[11];Lee等测定了621份大豆籽粒蛋白、脂肪、以及主要氨基酸含量,结合基因型数据(34 014个SNP),发掘了17个显著关联位点[12];Tanya等[13]利用大豆RIL群体定位到5个与根瘤个数相关QTL,7个与根瘤鲜重、干重相关QTL, 2个与固氮酶活性相关QTL;Nicola´s等[14]以 F2:3群体为材料,定位到多个大豆固氮相关QTL;Santos等[15]定位到2个地上部干重相关QTL、3个根瘤个数相关QTL、1个单个根瘤干重相关QTL;Hwang等[16]定位到与植株地上部酰脲及全氮含量相关QTL;Ray等[17]利用大豆自然群体关联到53个与地上部酰脲含量相关位点。由此可见,针对大豆发育早期固氮相关性状已有较多研究,而关于低氮条件下接种根瘤菌后的大豆成熟期产量相关性状QTL与候选基因研究较少。
鉴于此,本研究以前期构建的大豆自然群体和RIL分离群体为材料,采用关联分析与连锁分析2种方法,在低氮条件下接种根瘤菌对大豆成熟期的株高、主茎节数、分枝数、单株荚数、单株粒数、单株粒重和百粒重等产量相关性状进行QTL定位,寻找控制产量相关性状遗传位点及候选基因,为接种根瘤菌大豆产量相关性状遗传基础解析、大豆品种固氮能力改良的分子育种提供选择标记和候选基因。
1 材料与方法
1.1 试验材料
1.1.1 供试品种 以前期构建的大豆自然群体和RIL群体为研究材料,自然群体包含155份大豆种质资源,包括育成品种(68份)、品系(56份)和地方品种(31份);RIL群体以高固氮品种‘郑92116’和低固氮品种‘辽豆14’为杂交亲本,包含154个F6:10家系。
1.1.2 供试根瘤菌菌株 采用优势快生型费氏中 华 根 瘤 菌[Sinorhizobium(Ensifer) fredii]CCBAU45436为供试菌株,由中国农业大学根瘤菌研究中心提供[18-19]。
1.2 试验方法
1.2.1 大豆RIL和自然群体种植与管理 2018年6月将大豆自然群体与RIL群体及其亲本播种于河北农业大学育种中心低氮池。低氮池底部和四周用水泥加固,池内土壤肥力为有机质9.10 g/kg,碱解氮22.18 mg/kg,速效磷5.72 mg/kg,速效钾133.54 mg/kg。随机区组设计,2次重复。首先将氯气消毒后的15粒种子均匀播种在1.5 m行长的犁沟中,随后将50 mL制备好的CCBAU45436菌液均匀喷洒在种子上,覆土[20]。待大豆出苗后定苗,每周浇1次无氮B&D营养液[21]。
1.2.2 大豆RIL群体与自然群体产量相关性状鉴定 待大豆成熟后,每行选取具有代表性的连续5株进行产量相关性状测定,包括株高(PH)、底荚高(LPH)、主茎节数(NN)、分枝数(BN)、单株荚数(PN)、单株粒数(SN)、单株粒重(SW)和百粒重(HSW)等。其鉴定方法参考国家大豆区域试验考种标准。
1.2.3 大豆群体产量相关性状数据分析 利用SPSS statistics V19.0分析大豆产量相关性状均值、标准差、最大值、最小值、变异系数、偏度和峰度等,同时计算各性状间的相关系数。
1.2.4 大豆群体产量相关性状关联与连锁分析 大豆自然群体的群体结构分析见已发表论文[22]。利用上述大豆自然群体各产量相关性状表型值,结合群体基因型,利用GAPIT V2软件包中的MLM模型进行关联分析[23-24];同时利用Icimapping V4.1中的BIP功能,对大豆RIL群体产量相关性状进行加性和上位性QTL检测,其中加性QTL步长为0.1 cM,LOD值2.5,上位性QTL步长5 cM,LOD值4.5。
1.2.5 大豆产量相关性状候选基因预测 基于Soybase参考基因组Glyma.Wm82.a1版本(https://www.soybase.org),筛选显著关联SNP衰减距离内的候选基因,并结合Soynet(https://www.inetbio.org/soynet/)分析候选基因间的互作网络[25]。
2 结果与分析
2.1 大豆产量相关性状遗传变异分析
2.1.1 大豆自然群体产量相关性状遗传变异 分析供试大豆自然群体在低氮条件下接种根瘤菌的产量相关性状,结果发现(表1),株高、主茎节数、分枝数、单株荚数、单株粒数等8个产量相关性状在品种间的差异均达到极显著,变异系数分布在15.32%~49.03%,最大的为分枝数(49.03%),其次是单株荚数(40.10%),最小的为主茎节数(15.32%),说明供试大豆群体产量相关性状存在广泛遗传变异。同时,各产量相关性状表型呈连续的正态分布,属于多基因控制数量性状。
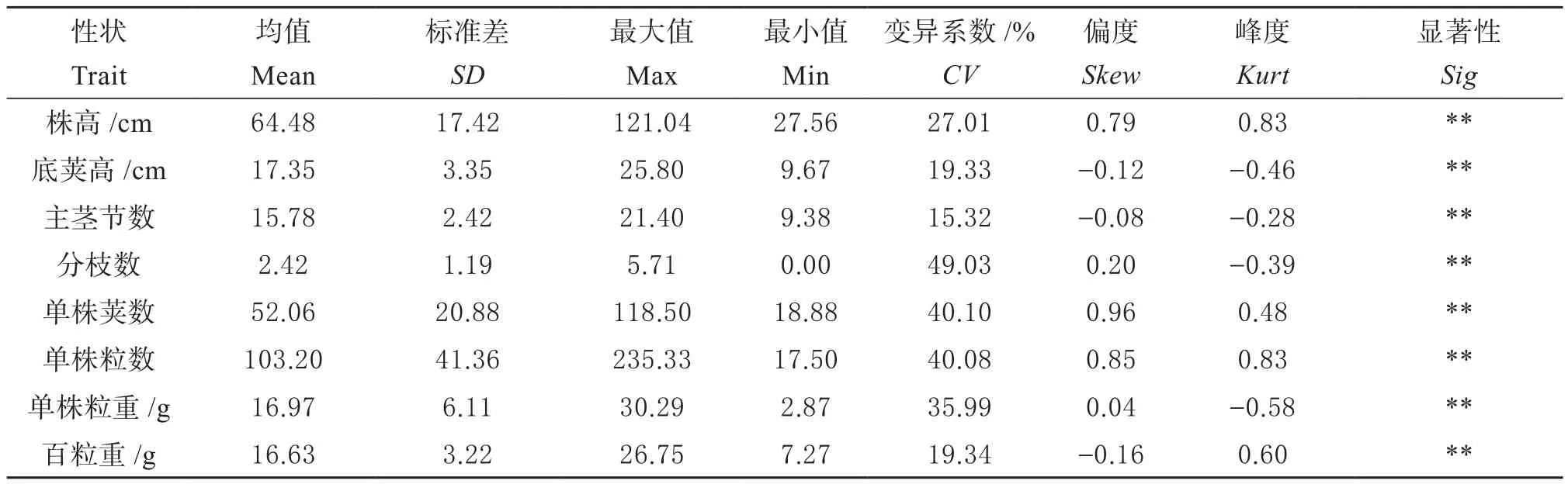
表1 大豆自然群体产量相关性状遗传变异Table 1 Genetic variation of yield-related traits in soybean natural population
2.1.2 大豆RIL群体及其亲本产量相关性状遗传变异 分析供试大豆RIL群体及其亲本在低氮条件下接种根瘤菌的产量相关性状,结果发现(表2),RIL亲本间存在较大遗传差异,‘郑92116’的株高、主茎节数、分枝数、单株荚数、单株粒数等各产量相关性状均高于辽豆14。进一步分析RIL群体产量相关性状发现,株系间的差异达到极显著,变异系数在10.87%~34.02%之间,其中以分枝数和单株荚数的变异系数最高,说明供试RIL群体各产量相关性状存在较大遗传变异。同时,各相关性状表型也呈连续的正态分布。另外还发现,RIL群体部分株系表现正向或负向超亲优势。

表2 大豆RIL群体产量相关性状遗传变异Table 2 Genetic variation of yield related traits in soybean RIL population
2.2 大豆产量相关性状相关性分析
分别分析自然群体和RIL群体8个产量相关性状间的相关系数,结果发现(表3),多个产量相关性状间存在显著(极显著)相关,其中RIL群体的单株荚数、单株粒数、单株粒重间的相关系数高达0.85~0.93,株高与主茎节数间的相关系数也高达0.85;供试自然群体中以株高与主茎节数间的相关系数最高(r=0.78),其次单株荚数与单株粒重(r=0.73)、单株粒数(r=0.68)相关系数较高。这些显著或极显著的相关系数一方面说明各产量性状间的紧密联系,另一方面也为关联/连锁位点的一因多效解释提供了依据。
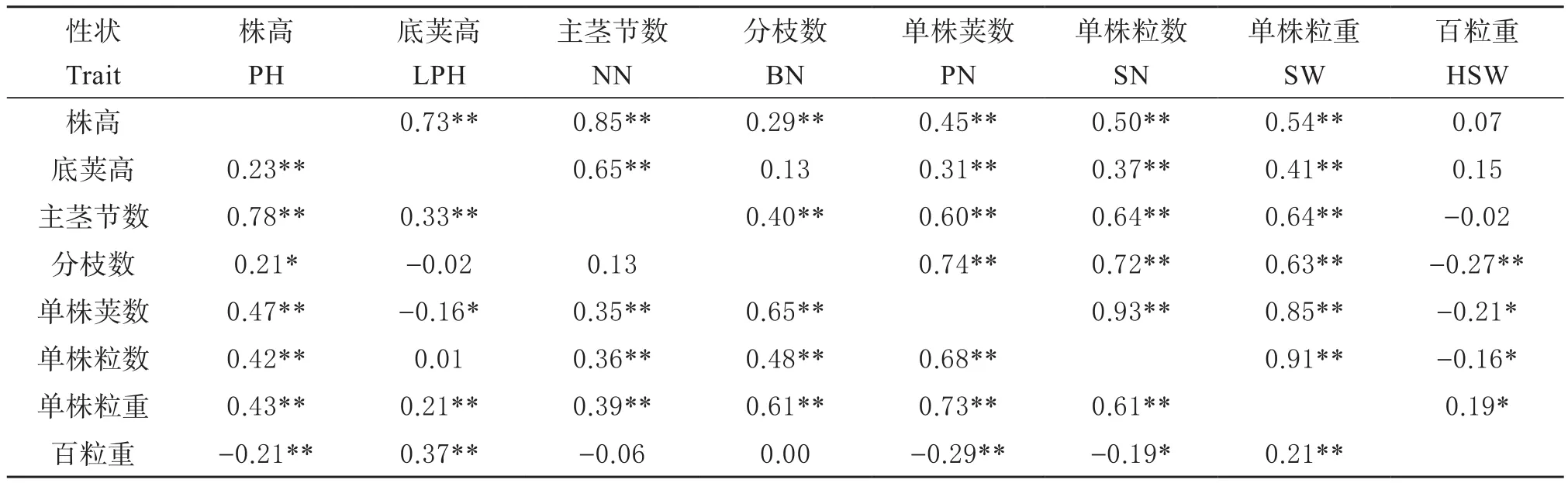
表3 供试大豆群体各产量相关性状间的相关性分析Table 3 Correlation analysis of yield related traits in two soybean populations
2.3 大豆自然群体产量相关性状全基因组关联分析
利用自然群体产量相关性状表型值,结合群体基因型进行关联分析,结果发现(图1),有59个SNP与产量相关性状显著关联,分布于10条染色体上,其中与株高关联的SNP最多,共18个;其次为主茎节数(17个SNP)、分枝数(11个SNP),与单株荚数显著关联的SNP 4个、与单株粒重显著关联的SNP 2个、与百粒重显著关联的SNP 1个。同时发现,株高与主茎节数有17个共关联的SNP,分别为2号染色体ss715581250、6号染色体ss715593820和ss715593829、7号染色体ss715598582和8号染色体ss715602540等。
进一步分析上述关联位点发现,19号染色体与株高和主茎节数显著关联的ss715635425与已报道Dt1基因(Glyma19g37890)相距仅19 kb,而Dt1已被证实可同时控制大豆开花期、株高、产量等多个重要产量相关性状;同时6号染色体与株高和主茎节数显著关联的位点ss715593820和ss715593829恰好位于已报道的开花期基因QNE1(Glyma06g22240)和E1(Glyma06g23040)之间[26]。另外还发现2个一因多效SNP:15号染色体ss715622869同时与分枝数、单株荚数和单株粒重关联,19号染色体ss715635361同时与株高、主茎节数和底荚高关联。

图1 大豆自然群体产量相关性状关联分析(阈值以红线标出)Fig.1 Association analysis results of yield related traits in soybean natural population(The threshold was indicated with red line)
2.4 大豆RIL群体产量相关性状连锁分析
2.4.1 产量相关性状加性QTL分析 对供试RIL群体产量相关性状进行QTL分析发现(表4),有23个加性QTL与产量性状显著相关,分布于8条染色体,在23个QTL中,6号和19号染色体QTL最多(7个、5个),其次为10号染色体(4个);这些QTL的表型贡献率为5.90%~32.87%,其中有11个QTL的表型贡献率超过10%。控制单株荚数的qPN-C2位于Gm06的91.7 cM、qPN-O位于Gm10的36.7 cM;控制单株粒数的qSN-C2位于Gm06的91.7 cM、qSN-O位于Gm10的36.7 cM、qSN-L位于Gm19的45.5 cM;控制单株粒重qSW-C2位于Gm06的91.7 cM、qSW-O位于Gm10的39.8 cM、qSW-L位于Gm19的45.5 cM;控制百粒重的qHSW-N位于Gm03的141.0 cM、qHSW-O位于Gm10的37.0 cM。对表型贡献率分别为9.18%~17.42%、6.31%~20.35%、5.95%~24.60%和7.27%~9.45%。发掘到控制底荚高QTL 4个,株高、主茎节数、分枝数各3个。
进一步分析发现,在6号染色体上定位到1个同时控制株高、主茎节数、单株荚数、单株粒重和单株粒数的QTL,位于ss715594069-ss715593914之间,表型贡献率17.42%~24.60%,增效基因来自‘郑92116’;在19号染色体上定位到1个同时控制株高与主茎节数的QTL,位于ss715635545-ss715635383之间,贡献率为25.90%~32.87%,以及1个同时控制单株粒数和单株粒重的QTL,位于ss715635619-ss715635545之间,贡献率为6.31%~7.67%,2个QTL相邻,上述两个QTL的增效基因均来自‘辽豆14’;在10号染色体上定位到1个同时控制单株荚数和单株粒数的QTL(qPN-O、qSN-O),位于ss715607504-ss715607483之间,且与控制百粒重的QTLqHSW-Oss715607483-ss715607458相 邻,解释表型变异7.27%~11.76%。另外,在13号染色体上还定位到1个同时控制株高和底荚高QTL,位于ss715616007-ss715616040之间,表型贡献率8.88%~11.27%。

表4 大豆RIL群体产量相关性状加性QTL分析Table 4 The additive QTLs of yield related traits in soybean RIL population
2.4.2 产量相关性上位性QTL分析 分析RIL群体产量相关性状上位性QTL发现(表5),共定位到13对影响单株粒数、单株荚数、百粒重等性状上位性QTL,分布于13条染色体上,其中3对单株粒数上位性QTL中有2对效应值为负,表现出重组型大于亲本型,1对效应值为正,表现出亲本型大于重组型;2对单株荚数上位性QTL效应值为负,表现出重组型大于亲本型。进一步分析还发现,7号染色体ss715596707-ss715597345和12号染色体ss715611920-ss715611545上位性QTL同时影响单株粒数和单株荚数,属于一因多效上位性QTL。
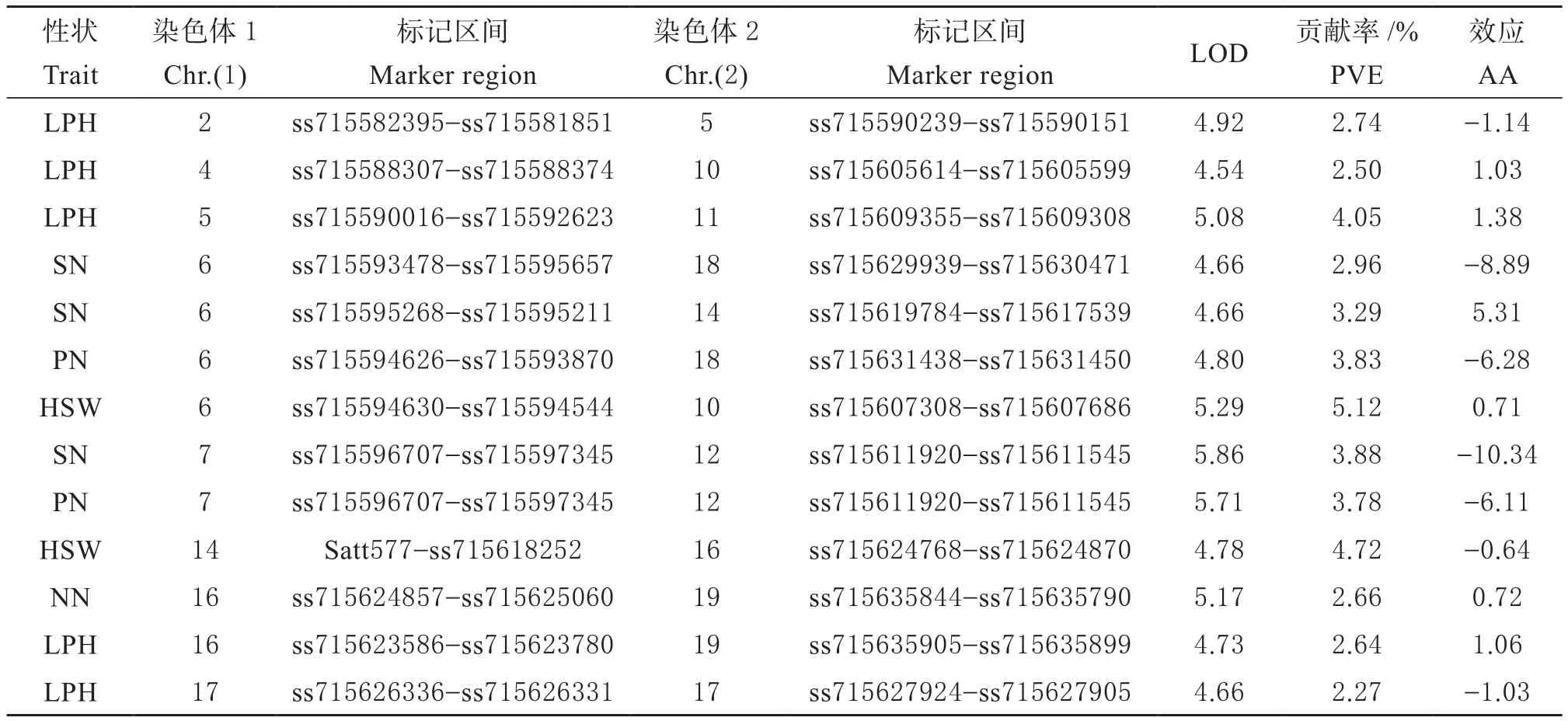
表5 大豆RIL群体产量相关性状上位性QTL分析Table 5 The epistatic QTL of yield related traits in soybean RIL population
2.5 大豆产量相关性状候选基因发掘
综合分析自然群体和RIL群体定位结果,发现6号和19号染色体存在产量相关性状一致性QTL,在其附近寻找候选基因,6号染色体有7个,包含磷酸丙酮酸激酶、转移酶、PCF转录因子和发育调节因子基因等;19号染色体有16个,包含Dt1(Glyma19g37890)、氧化还原酶、亮氨酸重复受体激酶、结瘤素、铁硫蛋白基因等(表6)。
为进一步明确上述候选基因间的内在联系,利用Soynet在线工具分析上述基因发现(图2),21个基因参与互作网络,说明其相互之间存在直接或间接联系(除Glyma19g39370、Glyma19g37430外)。在21个参与网络的候选基因中,存在直接互作关系且处于网络核心位置的有2对,其中之一是位于19号染色体的Glyma19g38520(氧化还原酶基因)和Glyma19g37500(氧化还原酶基因),另一对为该染色体的Glyma19g38450(氨基酸连接酶基因)和Glyma19g37930(铁硫蛋白基因)。

表6 大豆6号与19号染色体产量相关性状候选基因Table 6 Candidate genes associated with yield related traits on Gm06 and Gm19

续表:
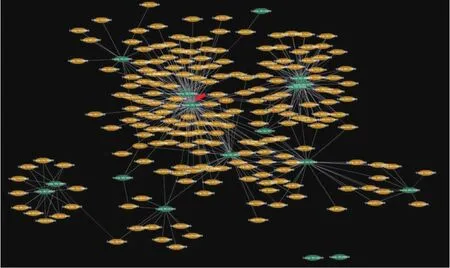
图2 大豆6号与19号染色体候选基因的互作网络(节点中心以红色箭头标出)Fig.2 The network of candidate genes on Gm06 and Gm19(The node center was pointed with red arrow)
3 结论与讨论
本研究采用关联与连锁分析方法对低氮条件下接种根瘤菌的大豆株高、主茎节数、分枝数、单株荚数、单株粒数、单株粒重和百粒重等产量相关性状进行QTL研究。通过关联分析发掘到响应根瘤菌接种的产量相关性状SNP 59个,利用连锁分析获得23个加性QTL和13对上位性QTL,其中控制株高、主茎节数等产量相关性状的一致性QTL主要位于6号和19号染色体上,在自然群体和分离群体均被定位,并在其附近筛选到23个候选基因。
利用RIL群体与自然群体进行连锁和关联分析是目前寻找植物目标性状QTL重要研究手段,已有学者综合运用2种方法寻找控制植物性状QTL与基因,且效果较好[27]。本研究分别利用这2种方法获得了多个与大豆产量相关性状QTL。为进一步寻找2种方法共定位的一致性QTL,本研究对比了连锁定位与关联分析SNP,结果发现,在6号与19号染色体存在较好的一致性位点,其中6号染色体的22.27 mb~28.32 mb(参考基因组WM82.a1v1)区间存在1个可同时控制株高、主茎节数、单株荚数、单株粒数和单株粒重QTL,其两侧连锁标记为ss715593914和ss715594069,同时在该染色体19.54 mb位置关联到1个与株高和主茎节数显著相关标记ss715593829,二者物理距离相距较近(2.73 mb)。在19号染色体定位到1个同时控制株高、主茎节数QTL,该QTL两侧标记为ss715635383和ss715635545,位于参考基因组(WM82.a1v1)44.64 mb和45.81 mb位置,其间包含Dt1位点,并且包含自然群体关联分析中的4个同时影响株高和主茎节数SNP(ss715635477、ss715635494、ss715635506和ss715635548)。另外,RIL群体中还定位到1个与上述QTL相邻,控制单株粒重和粒数的一因多效QTL(ss715635545-ss715635619),物理位置为45.81 mb~46.76 mb,该区间也包含自然群体中2个同时与株高和主茎节数关联的一因多效SNP(ss715635548和ss715635553)。
结合前期我们利用相同自然群体在温室和大田两种环境下对苗期结瘤固氮相关性状遗传位点及基因的研究结果[22],发现成熟期产量相关性状与苗期结瘤固氮相关性状部分遗传位点存在一致性,如在6号染色体上关联到的控制底荚高QTL(qLPH-C2),该位点距离本研究前期检测到的控制苗期根系鲜重、干重的Locus14仅108 kb;同时在该染色体定位到的成熟期控制分枝数QTL(qBN-C2)距离上述Locus14仅694 kb,并与控制苗期地上部总氮含量的Locus19相距331 kb。另外,13号染色体定位到的成株期控制株高、底荚高QTL(qPH-F、qLPH-F)距离苗期控制地上部干重QTL(qSDW-F-2)仅625 kb[22]。上述一致性位点的发现,不仅说明了本研究结果的可靠性,也为解析大豆苗期结瘤固氮与成熟期产量性状相关性提供了理论依据。
关于大豆产量相关性状QTL报道目前已有很多,但多是基于自然土壤条件下研究结果,本研究在低氮条件下接种根瘤菌研究大豆产量相关性状QTL,结果发现部分QTL与前人报道及Soybase公布QTL存在较好一致性,如本研究在19号染色体关联到的多个与株高、主茎节数显著相关SNP,其中ss715635425标记与已报道的株高位点Plantheight 1-g25和Plant height 3-g5定位在同一位点,ss715635454、ss715635477、ss715635494和ss715635506位点则分别与已报道的株高位点Plant height 3-g2,Plant height 3-g6,Plant height 3-g7和Plant height 3-g3共定位,该QTL区间包含大豆开花期和株高Dt1基因[28-29],并且Hwang等利用大豆RIL群体在低氮条件下接种根瘤菌也定位到Dt1,故推测Dt1对低氮接菌条件下的大豆产量相关性状也同样具有重要作用[30]。同时,本研究定位到的6号染色体控制株高QTLqPH-C2与Soybase公布QTL的株高位点Plant height 10-1和Plant height 23-2一致[31-32];13号染色体的株高 QTLqPH-F与已报道的株高位点mqPlant height-006和Plant height 39-1一致[33-34];6号染色体控制单株荚数QTLqPN-C2与已有单株荚数位点Pod number 2-g4一致[35];10号染色体的单株荚数QTL qPN-O与已报道的单株荚数位点Pod number 8-3一致[36];19号染色体的单株粒数QTLqSN-L与已有单株粒数位点Seed number 5-1一致[36];6号染色体控制单株粒数QTL qSN-C2区间含有已报道的单株粒数候选基因Glyma06g28570[37],这些结果充分说明了本研究结果的可靠性。更重要的是,除上述与前人报道一致性的QTL外,本研究还定位/关联到多个前人未报道的新QTL,这些QTL可能是大豆在低氮条件下接种根瘤菌特有的产量相关性状位点,目前课题组正在针对这些位点展开后续研究,以期为大豆根瘤固氮相关基因克隆及生物固氮效率改良奠定物质基础。

