乏情和发情初产母猪下丘脑–垂体–卵巢轴中lincRNAs表达谱比较分析
任巧玲,张家庆,陆东锋,王璟,陈俊峰,马强,白献晓,郭红霞,高彬文,邢宝松
研究报告
乏情和发情初产母猪下丘脑–垂体–卵巢轴中lincRNAs表达谱比较分析
任巧玲1,张家庆1,陆东锋2,王璟1,陈俊峰1,马强1,白献晓1,郭红霞1,高彬文1,邢宝松1
1. 河南省农业科学院畜牧兽医研究所,河南省畜禽繁育与营养调控重点实验室,郑州 450002 2. 河南花花牛实业总公司,郑州 450000
初产母猪断奶后能否正常发情对养猪生产影响重大,也是初产母猪被淘汰的主要原因。本研究以乏情和发情初产母猪为研究对象,首次利用RNA-seq技术对其下丘脑-垂体-卵巢轴中的基因间长链非编码RNAs(long intergenic noncoding RNAs, lincRNAs)进行筛选比较,得到lincRNAs的表达图谱,并对其特征和功能进行了初步分析。结果显示,在乏情和发情初产母猪下丘脑–垂体–卵巢轴中鉴定得到3519个lincRNAs,以发情组为对照共有17个lincRNAs存在差异表达,其中12个表达上调,5个表达下调(FC≥2,<0.05)。选择4个差异表达的lincRNAs经qRT-PCR验证,其表达水平与测序结果基本一致。对这17个差异表达的lincRNAs进行GO分析、KEGG通路分析及lincRNA-mRNA共表达网络分析,发现这些lincRNAs主要与猪卵母细胞减数分裂成熟、卵巢细胞分化及颗粒细胞凋亡等生殖活动相关。本研究结果丰富了猪lincRNAs数据资源,为进一步深入研究初产母猪的生殖机能提供了理论依据。
lincRNAs;乏情;发情;初产母猪;下丘脑–垂体–卵巢轴
初产母猪乏情或发情延迟已成为养猪生产中的一个常见难题。大长二元母猪作为当今瘦肉型商品猪生产的主要母本,其乏情问题对生产的影响重大。造成初产母猪乏情的主要原因有遗传、环境、营养和管理水平等因素,随着规模化猪场管理水平的提高及动物营养研究的深入,非遗传因素已得到了很好的改善。近年来,一些学者从基因遗传方面对母猪情期控制进行了一些有意义的探索。研究发现在发情和乏情母猪的下丘脑–垂体–卵巢轴中许多生殖激素基因的表达发生了明显的改变,如、、、、和等在乏情母猪体内显著下调,这些基因均与母猪的发情等生殖活动密切相关[1,2]。Kong等[2]报导在发情和乏情全同胞初产母猪的垂体和卵巢中均检测到的表达量较高,且存在显著差异(<0.01),并证实了该基因通过调控卵泡细胞的发育平衡来最终调控断奶后初产母的发情行为。另外,研究表明由编码的NKB与其受体NK3R结合能够促进GnRH的释放进而诱导动物初情期的启动,当发生突变时,导致低促性腺激素综合症的发生,动物初情期启动失败[3~5]。下丘脑–垂体–卵巢轴(hypothalamic- pituitary-ovarian axis, HPOA)是一个完整而协调的神经内分泌系统,它的每个环节均有其独特的神经内分泌功能,并且互相调节、互相影响,在哺乳动物的生殖活动中起着主要的调节作用,也是研究动物生殖问题的关键切入点。
lincRNA (long intergenic noncodingRNA, lincRNA)是一类由蛋白编码基因间的非编码序列转录且转录本长度超过200 nt的功能性RNA分子[6]。随着高通量RNA-seq技术的发展,不同物种中越来越多的lincRNAs被鉴定,至今在哺乳动物基因组中已发现3500多种lincRNAs,多项研究已经证实一些lincRNAs在不同的生物过程中发挥着重要作用,如表观遗传调控[7]、多能性维持和基因转录调控[8]、X染色体失活[9]及生殖调控[10,11]等。与mRNA相比,lincRNA的表达水平在组织间变化更大,许多lincRNAs优先在大脑和睾丸组织中表达[6]。lincRNAs的组织特异性表达表明其具有发育和细胞类型特异性功能[12]。目前lincRNAs在初产母猪乏情和发情状态下丘脑–垂体–卵巢轴中的表达及功能还鲜有报导。为了探索lincRNAs在初产母猪下丘脑-垂体-卵巢中的表达情况及对生殖活动的影响,本研究对乏情和发情初产母猪下丘脑–垂体–卵巢轴中的lincRNAs进行全基因组筛选比较,并对其特征和功能进行了初步分析,为进一步深入研究初产母猪的生殖机能提供了理论依据。
1 材料与方法
1.1 实验动物和样品制备
试验猪为体况和年龄基本一致的半同胞初产大长二元母猪,由河南鄢陵天鹏牧业有限公司提供。在同等条件下饲养在同一圈舍内,每天用同一头公猪试情1~2次,选取断奶后7 d有明显发情反应的3头视为正常发情母猪(发情组),屠宰后分别取下丘脑、垂体和卵巢,用生理盐水冲洗后,立即放入液氮中保存。选取公猪试情到21 d仍无任何发情反应的3头视为乏情母猪(乏情组),屠宰后分别取下丘脑、垂体和卵巢,处理方法同发情母猪。
1.2 样品总RNA提取
采用RNAiso Plus (日本TaKaRa公司)试剂盒分别提取乏情组3头母猪和发情组3头母猪的下丘脑、垂体和卵巢组织总RNA,用Nano-Drop ND-2000紫外分光光度计(美国Thermo Scientific公司)分析浓度,用Agilent BioAnalyzer 2100 (加拿大Agilent Technologies公司)进行质量检测,所有样品要求:260 nm/280 nm>2.1,RIN>8.0。分别取等量乏情和发情母猪的下丘脑、垂体、卵巢质检合格的RNA混合成6个样品池。
1.3 cDNA文库构建和RNA-seq
分别从6个混池中取大约20 µg RNA,用带有Oligo(dT)的磁珠(无锡百迈格生物科技有限公司)富集PolyA+RNA,并加入RNA断裂试剂(美国Ambion公司)使之片段化。用六碱基随机引物和M-MuLV逆转录酶(上海生工生物工程股份有限公司)合成第一链cDNA,用DNA聚合酶Ⅰ、RNase H和dNTPs (上海生工生物工程股份有限公司)合成第二链cDNA。依照Illumina Hiseq2500测序平台的要求,对构建的6个cDNA文库依次进行末端修复、3′末端加A、连接接头后纯化、PCR扩增富集、定量,质检符合要求后上机测序。测序工作由上海欧易生物医学科技有限公司完成。
1.4 序列比对和转录本构建
6个文库测序获得的Raw reads进行质控处理获得Clean reads,用Tophat (v2.0.14)软件把每个样本的Clean reads比对到猪参考基因组(11.1) (表1)。用Cufflinks (v2.2.1)把比对上的片段拼接成转录本(GTF格式),把来自乏情组和发情组的6个Cufflinks拼接转录本用Cufflinks软件包中的Cuffmerge融合成1个非冗余转录组,后续用这个非冗余转录组来鉴定novel lincRNAs。

表1 6个cDNA文库测序质量和比对信息统计分析
1.5 lincRNAs鉴定
乏情和发情母猪下丘脑、垂体、卵巢融合非冗余转录组中的lincRNAs通过以下步骤鉴定[13,14]: (1)用Cuffcompare把融合转录本与Ensembl数据库中的已知注释比对,去除可能的已知转录本;(2)过滤掉单外显子和长度小于200 bp的转录本;(3)只挑选离Ensembl中注释的猪的任何编码基因或持家的非编基因至少500 bp远的转录本;(4)分别用蛋白编码潜力计算软件CNCI和CPAT预测所筛选转录本的编码潜力,二者取交集即为lincRNAs (图1)。
1.6 差异表达的lincRNAs和差异表达的mRNAs筛选
利用DESeq软件(http://bioconductor.org/packages/ release/bioc/html/DESeq.html)对各样本的counts数目进行标准化处理(采用baseMean值来估算表达量),计算差异倍数,并采用NB (负二项分布检验的方式)对reads数进行差异显著性检验,筛选<0.05且Fold Change>2的为差异表达的lincRNAs和mRNAs。lincRNAs和mRNAs表达量计算用FPKM法,即每百万片段中来自某一基因每千碱基长度的片段数目。
1.7 lincRNAs功能预测
对于差异表达的17个lincRNAs,计算每个lincRNA与蛋白编码基因之间的Pearson相关系数,选取<0.05,||>0.8的基因,即为与之共表达的编码基因。使用 DAVID软件在线对共表达蛋白编码基因进行功能富集和通路分析,用超几何检验,找出与整个基因组背景相比,共表达基因显著富集的GO条目和KEGG通路。
1.8 lincRNA-mRNA共表达网络分析
为了鉴定差异表达的lincRNAs和差异表达的mRNAs之间的相互作用,在lincRNAs和mRNAs进行Pearson相关性分析的基础上构建了lincRNA- mRNA共表达网络。即先选取<0.05,||>0.8关系对,然后用Cytoscape软件来构建lincRNA-mRNA共表达网络。

图1 猪lincRNAs鉴定流程
1.9 差异表达lincRNAs qRT-PCR验证
分别选取2个表达上调和2个表达下调的lincRNAs进行qRT-PCR验证(每个组织重复3次)。RNA为实验室测序时提取冻存的RNA,即乏情母猪(3个下丘脑样品、3个垂体样品、3个卵巢样品)和发情母猪(3个下丘脑样品、3个垂体样品、3个卵巢样品)的总RNA。用RevertAid Strand cDNA Synthesis Kit反转录试剂盒(美国Thermo Scientific公司)合成cDNA,根据SYBR Premix Ex TaqTM(日本TaKaRa公司)说明书进行qRT-PCR验证。以为内参基因,引物由武汉赛维尔生物科技有限公司设计、合成,引物序列见表2。用2–ΔΔCt法计算lincRNAs的相对表达量。检验对相对表达量进行统计分析,数据表示为“平均数±标准差(Mean± SD),<0.05表示差异显著,<0.01表示差异极显著。
2 结果与分析
2.1 转录本组装及lincRNAs鉴定
为了鉴定乏情和发情初产母猪下丘脑–垂体–卵巢轴中的lincRNAs,首先用Illumina Hiseq2500对乏情和发情初产母猪的下丘脑、垂体和卵巢组织进行了转录组测序,去除接头及低质量序列后,平均每个样本得到52,400,881条clean reads,平均有42,364,859条比对到了猪的参考基因组(11.1),平均比对率为80.85% (表1)。利用Cufflinks重建了每个样本的转录组,并用Cuffmerge软件将乏情和发情母猪的下丘脑、垂体、卵巢的重建转录组融合成1套无冗余的转录组,共有55,734条转录本,利用CNCI软件预测得到3754条lincRNAs,利用CPAT软件预测得到4220条lincRNAs,二者的交集为3519条lincRNAs (图1)。这些lincRNAs分布在除Y染色体以外的所有染色体上(图2)。
2.2 lincRNAs结构特征分析
以前的研究已经表明人和小鼠的lincRNAs与其蛋白编码基因在结构上有很大的差异,如转录本长度、外显子长度和外显子数量等。为了鉴定本研究分析的3519个lincRNAs是否也具有这些特征,将这些lincRNAs与Ensembl数据库中所注释的猪mRNAs (更新于2019年6月,共有48,413个mRNAs)进行了比较。结果发现lincRNAs的这些特征与以往的研究十分类似[15,16]:转录本平均长度(1797 bp)明显小于mRNA (3551 bp) (图3A),外显子平均长度(571 bp)大于mRNA (285 bp) (图3B),外显子平均个数(3.1个)小于mRNA (12个) (图3C)。

表2 引物序列信息

图2 lincRNAs在染色体上的分布

图3 lincRNAs和mRNAs转录本长度、外显子长度和外显子个数比较
A:转录本长度比较;B:外显子长度比较;C:外显子个数比较。
2.3 差异表达lincRNAs和差异表达mRNAs的表达谱
为探讨lincRNAs的功能,利用DEseq软件对乏情组和发情组下丘脑–垂体–卵巢轴中的lincRNAs进行差异表达分析。共有17个lincRNAs存在差异表达,以发情组为对照组,其中12个上调表达,5个下调表达(FC≥2,<0.05) (表3)。这17个lincRNAs中,TCONS_00035245上调倍数最高(log2FC值为15.665),TCONS_00032971下调倍数最高(log2FC值为16.210)。散点图(图4A)揭示了这17个lincRNAs在两组间的变化情况,聚类分析热图(图5A)揭示了这17个lincRNAs在不同样本间表达模式的关系,即乏情组的3种组织聚为一类,发情组的3种组织聚为一类,在两组间都是垂体和卵巢的表达模式更为相似,聚为一小类。
同时,对mRNAs差异表达分析显示,共有72 mRNAs存在差异表达,以发情组为对照,其中的35个上调,37 个下调(FC≥2,<0.05)。在一个组(乏情组或发情组)平均表达量较高,且在另一组表达量大于1 (乏情组或发情组)的17个mRNAs具体情况如表4所示。同样,散点图(图4B)揭示了这72个mRNAs在两组间的变化情况,聚类分析热图(图5B)揭示了这72个mRNAs在不同样本间表达模式的关系,该模式关系与上述的17个lincRNAs相似。
2.4 差异表达lincRNAs验证
为了验证RNA-seq结果的可靠性,选取4个差异表达的lincRNAs进行验证(图6)。RNA-seq生物信息学分析筛选的17个差异表达lincRNAs均为与对照组相比在下丘脑、垂体、卵巢3种组织中表达趋势一致的lincRNAs。qRT-PCR验证结果显示,TCONS_00012288在下丘脑(图6A)有上调趋势(>0.05),TCONS_00001098在卵巢(图6C)有下调趋势(>0.05),但RNA-seq结果为TCONS_00012288表达下调,TCONS_00001098表达上调。除上述两种情况外,其他所有lincRNAs在下丘脑、垂体和卵巢中的结果均与RNA-seq相同,即上调的lincRNAs与对照组相比,在下丘脑、垂体和卵巢中均为上调的趋势,下调的lincRNAs与对照组相比,在下丘脑、垂体、卵巢中均为下调的趋势。
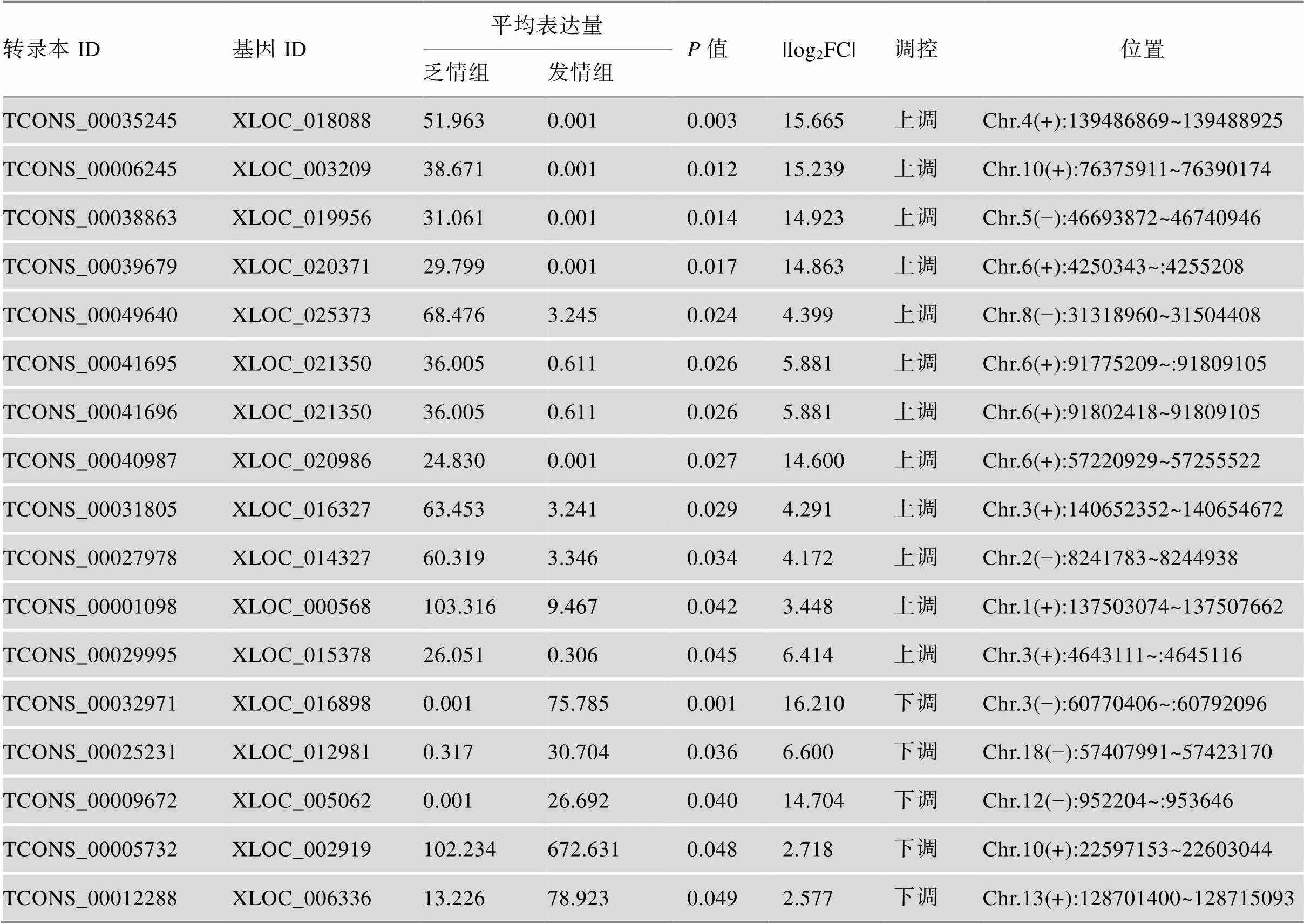
表3 17个差异表达lincRNAs相关信息
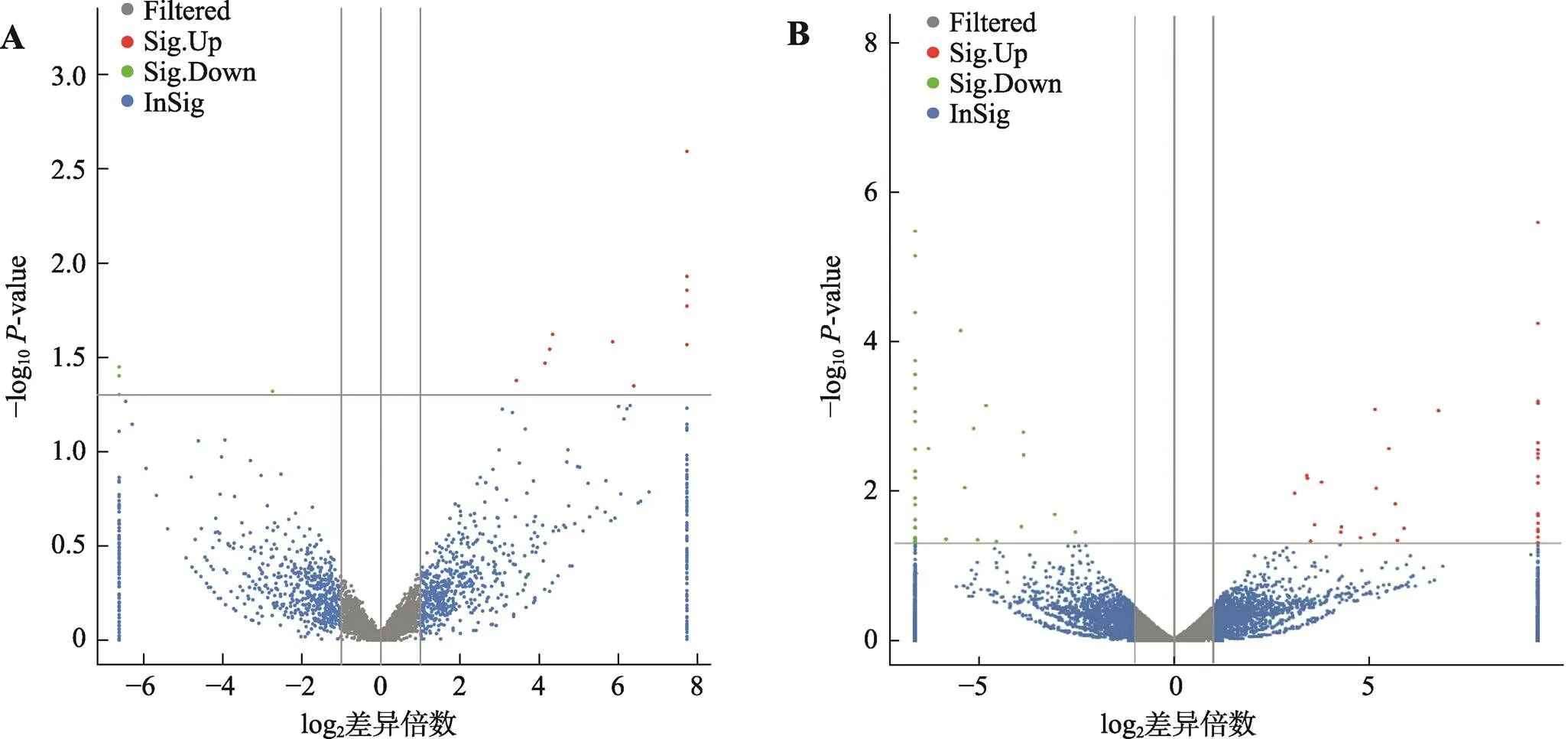
图4 差异表达lincRNAs和mRNAs散点图
A:差异表达lincRNAs散点图;B:差异表达mRNAs散点图。

图5 差异表达lincRNAs和mRNAs聚类分析热图
A:差异表达lincRNAs聚类分析热图;B:差异表达 mRNAs聚类分析热图。

表4 17个差异表达的mRNAs相关信息

图6 差异表达lincRNAs的qRT-PCR验证
A:差异表达lincRNAs在下丘脑中的相对表达量;B:差异表达lincRNAs在垂体中的相对表达量;C:差异表达lincRNAs在卵巢中的相对表达量。*表示<0.05,有统计学差异;**表示<0.01,差异显著。
2.5 GO富集和KEGG通路分析
为了预测差异表达lincRNAs的功能,对17个差异表达的lincRNAs经Pearson相关分析共得到256个共表达基因。这些基因前30富集的GO条目如图7所示。GO分析表明与lincRNAs共表达基因最富集条目为正调控NF-kappa转录因子的活动(生物学过程,GO:0051092)、蛋白磷酸酶1(细胞组分,GO:00164)和细胞周期蛋白依赖性蛋白丝氨酸/苏氨酸激酶活性(分子功能,GO:000469)。此外,KEGG通路分析表明,与lincRNAs共表达基因主要与疾病的发生发展、抗原加工递呈、T细胞受体信号通路和细胞粘附分子等通路相关(图8)。
2.6 lincRNA-mRNA共表达网络分析
17个差异表达的lincRNAs和72个差异表达的mRNAs,在分析其存在共表达关系的基础上,构建了lincRNA-mRNA共表达网络(图9)。在这个网络中共包括89个节点和274个关系对,其中有174个正调控关系对,100个负调控关系对。此外,分析还显示,1个mRNA可能与1~11个lincRNAs相关,而1个lincRNA则与3~33个mRNAs相关。
3 讨论
近年来研究表明,越来越多的lincRNAs参与哺乳动物的生殖调控,如lincRNA-介导哺乳动物X染色体失活[9],lincRNA-在黄体形成和妊娠维持中起重要作用[17],lincRNA-和与人类的胎盘和胎儿发育密切相关[18]。与人类和其他哺乳动物相比,lincRNAs是如何调控母猪生殖活动的还未见报道[19,20]。本研究首次对乏情和发情初产母猪下丘脑–垂体–卵巢轴中的lincRNAs进行了鉴定和分析,系统研究了乏情和发情初产母猪下丘脑–垂体–卵巢轴中差异表达lincRNAs和mRNAs的比较图谱,并对其生物学功能进行了初步的探索,为深入研究lincRNAs在初产母猪情期启动中的作用提供分子生物学参考,也为深入阐明初产母猪复杂的生殖机制提供了新的分子标记方向。
本研究共鉴定出3519条lincRNAs,这些lincRNAs的转录本平均长度为1797 bp,外显子平均长度为571 bp,外显子平均个数为3.1,分布在除Y染色体以外的所有染色体上。与蛋白编码转录本相比较,这些lincRNAs具有较短的转录本长度、较少的外显子个数和较长的外显子长度,这些特征与以往的研究报导一致[15,16]。在这些lincRNAs中共有17个存在差异表达,以发情组为对照其中12个上调,5个下调。同时也对mRNAs的表达情况进行了分析,发现共有72个mRNAs存在差异表达,以发情组为对照其中35个上调表达,37个下调表达。
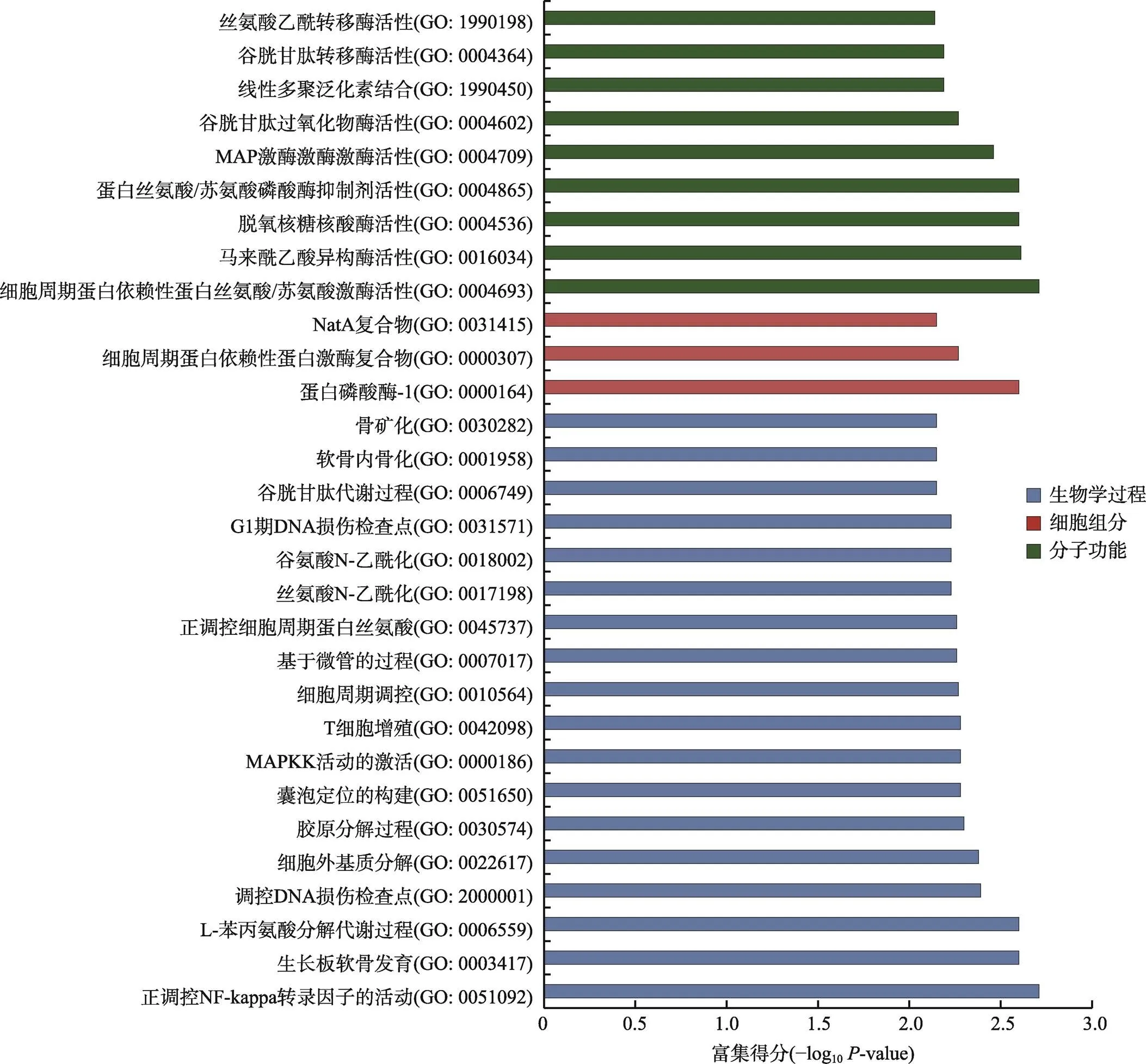
图7 共表达基因最富集的前30个GO条目
GO:ID代表该功能在GO数据库中的编号。
lincRNAs通常不编码蛋白质,而是通过与染色体或蛋白质结合来实现自身的功能,特别是与某些转录因子的结合。最近的研究已经表明lincRNAs的生物学功能可以通过与之共表达的基因来预测。为了进一步探讨差异表达lincRNAs对初产母猪情期启动等生殖活动的影响,对与之共表达的256个基因进行了GO富集和KEGG通路分析。对发情母猪而言,共表达基因富集到的MAP激酶活性(GO:0004709)、细胞周期蛋白依赖性蛋白丝氨酸/苏氨酸激酶活性(GO:0004693)、细胞周期蛋白依赖性蛋白激酶复合物(GO:0000307)和胶原分解过程(GO:0030574)等GO条目都直接或间接与母猪发情生物学机制相关。动物发情最显著的生理特征是卵巢上的卵泡发育成熟并排卵,在发情期卵母细胞受到促性腺激素的刺激后恢复第一次减数分裂,在这一过程中卵母细胞发生了一系列的变化,如染色体聚集、核仁解体、生发泡破裂(GVBD)、纺锤体组装和排出第一极体(PB1)[21~24]。成熟促进因子(maturation promoting factor, MPF)在哺乳动物卵母细胞减数分裂成熟过程中起着关键性的调控作用,它发挥活性的催化亚基就是由细胞周期蛋白依赖性蛋白丝氨酸/苏氨酸激酶构成[25,26]。在猪卵丘卵母细胞复合体培养过程中抑制MPF的活性,卵母细胞减数分裂成熟显著受阻,同时MPF活性还影响卵母细胞减数分裂成熟过程中母源基因的表达[27]。在发育不良的猪卵母细胞内,MPF催化亚基和调节亚基的mRNA和蛋白表达水平都显著降低[28]。这说明MPF的高水平表达及正常激活在猪卵母细胞成熟过程中起关键作用。丝裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)是一类广泛存在于真核细胞中的丝氨酸/苏氨酸蛋白激酶,在卵母细胞减数分裂成熟过程中也具有重要作用。研究发现,MAPK调控卵母细胞减数分裂过程中微管和染色质的行为,在小鼠卵母细胞减数分裂时,即使MPF的活性被抑制,MAPK依然能够诱导卵母细胞染色质凝聚和纺锤体形成[29,30]。在猪卵母细胞减数分裂成熟过程中,用MAPK专用抑制剂抑制MAPK的活性,染色体分离、第一极体排出和MⅡ期纺锤体的形成都被抑制[31]。另外,GO条目胶原分解过程(GO:0030574)与发情动物的排卵行为密切相关。在蛋白分解酶的作用下,排卵前卵泡顶端胶原分解和细胞死亡是卵泡即将破裂排卵的标志。因此推测,对发情母猪而言,差异表达lincRNAs主要是通过调控卵母细胞的减数分裂成熟过程来影响母猪的生殖活动。对乏情母猪而言,最值得关注的GO条目是正调控NF-kB转录因子的活性(GO:0051092),该条目在分子功能模块最为富集。NF-kB作为信号传导通路的中枢,参与许多生物过程的调控,如炎症和免疫反应、细胞增殖和凋亡、细胞分化等[32~35]。NF-kB异常活化可调节与细胞增殖和凋亡有关的基因转录,从而调控细胞的增殖和凋亡,并且已有研究证实NF-kB具有促进细胞凋亡的作用,NF-kB亚单位的种类及数量在细胞凋亡中起着决定性的作用[36,37]。研究表明NF-kB直接参与猪卵巢细胞的增殖和分泌活动,NF-kB的两个亚基P65和P50均有抑制猪卵巢颗粒细胞分化的作用,且P50在过表达时,促进了颗粒细胞的凋亡[38,39]。因此推测,差异表达的lincRNAs可能通过异常活化NF-kB信号通路,抑制了卵巢细胞的分化,促进了颗粒细胞的凋亡,引起了卵泡闭锁,最终导致母猪乏情。
Pathway:ID代表该通路在KEGG数据库中的编号。
蓝色节点代表差异表达lincRNAs,黄色节点代表差异表达mRNAs,绿线代表正相关,红线代表负相关。
本研究利用lincRNA-mRNA共表达网络分析来确定差异表达的lincRNAs和差异表达的mRNAs之间的关系。在这个lincRNA-mRNA共表达网络中,基因关联到的lincRNAs (11个)最多,在人体内该基因编码的蛋白NIBP参与膜泡运输过程,增强TNTα诱导的NF-kB活性,直接与IKK和MAP3K14相互作用,参与NF-kB信号通路的经典激活和替代激活[40,41],NIBP在进化过程中高度保守,而猪又拥有与人类极为相似的基因特征,因此,推测差异表达的lincRNAs通过调控基因的表达来参与NF-kB信号通路的激活过程,从而影响母猪的生殖活动。另外,该网络与TCONS_00009672共表达的基因与卵母细胞的减数分裂成熟密切相关。是一个休眠的母源mRNA,在卵母细胞成熟过程中被招集并翻译来调控组蛋白的去乙酰化,与成熟相关的组蛋白乙酰化水平降低对卵母细胞减数分裂成熟正常进行及染色体准确分离至关重要,被SiRNA耗尽的卵母细胞出现了严重的染色体错排、着丝粒–微管连接不当、SAC功能受损、胞质分裂缺陷和MⅡ非整倍体发生率增加[42,43]。因此,在猪下丘脑–垂体–卵巢轴中的鉴定TCONS_ 00009672很有可能也是通过调节基因来实现对卵母细胞成熟发育的影响,从而影响母猪的发情活动,详尽的机理还得进一步的挖掘探讨。
本研究通过RNA-seq技术分析了乏情和发情初产母猪下丘脑–垂体–卵巢轴中lincRNAs的表达图谱,并对差异表达lincRNAs的功能进行初步分析,发现这些lincRNAs主要与卵母细胞的减数分裂成熟、卵巢细胞分化及颗粒细胞凋亡等生殖活动相关,但与重要生殖激素相关的生物学过程并未预测到,存在测序没有生物学重复,预测到的差异表达lincRNAs并不全面所致的可能。
[1] Zhou DS, Zhuo Y, Che LQ, Lin Y, Fang ZF, Wu D. Nutrient restriction induces failure of reproductive function and molecular changes in hypothalamus-pituitary-gonadal axis in postpubertal gilts., 2014, 41(7): 4733–4742.
[2] Kong LJ, Wang AG, Fu JL, Lai CH, Wang XF, Lin HC. Peroxisome proliferator-activated recptor γ is involved in weaning to estrus of primiparous sows by regulating the expression of hormone genes in hypothalamus-pituitary- ovary axis., 2007, 20(3): 340–350.
[3] Topaloglu AK, Reimann F, Guclu M, Yalin AS, Kotan LD, Porter KM, Serin A, Mungan NO, Cook JS, Ozbek MN, Imamoglu S, Akalin NS, Yuksel B, Rahily SO, Semple RKandmutations in familial hypogonadotropic hypogonadism reveal a key role for Neurokinin B in the central control of reproduction., 2009, 41(3):354–358.
[4] Lehman MN, Coolen LM, Goodman RL. Minireview: Kisspeptin/neurokinin B/dynorphin (KNDy) cells of the arcuate nucleus: A central node in the control of Gonadotropin-Releasing hormone secretion., 2010, 151(8): 3479–3489.
[5] Song S, Cui P, Wang YY, Jiang SD, Zhang XR, Fang FG, Li SB. Expression of neurokinin B in hypothalamus, pituitary and ovary of rats., 2015, 43(6): 7–14, 20.宋爽, 崔培, 王尧尧, 蒋书东, 章孝荣, 方富贵, 李书宝. 神经激肽B在大鼠下丘脑–垂体–卵巢轴上的表达. 西北农林科技大学(自然科学版), 2015, 43(6): 7–14, 20.
[6] Ulitsky I, Bartel DP. LincRNAs: genomics, evolution, and mechanisms., 2013, 154(1): 26–46.
[7] Popadin K, Gutierrez-Arcelus M, Dermitzakis ET, Antonarakis SE. Genetic and epigenetic regulation of human lincRNA gene expression., 2013, 93(6): 1015–1026.
[8] Guttman M, Donaghey J, Carey BW, Garber M, Grenier JK, Munson G, Young G, Lucas AB, Ach R, Bruhn L, Yang XP, Amit I, Meissner A, Regev A, Regev A, Rinn JL, Root DE, Lander ES. LincRNAs act in the circuitry controlling pluripotency and differentiation., 2012, 477(7364): 295–300.
[9] Augui S, Nora EP, Heard E. Regulation of X-chromosome inactivation by the X-inactivation centre., 2011, 12(6): 429–442.
[10] Li JY, Gao ZL, Wang XY, Liu HB, Zhang Y, Liu ZH. Identification and functional analysis of long intergenic noncoding RNA genes in porcine pre-implantation embryonic development., 2016, 6: 38333.
[11] Muys BR, Lorenzi JCC, Zanette DL, Bueno RdBLe, Araújo LFd, Dinarte-Santos AR, Alves CP, Ramão A, Molfetta GAd, Vidal DO, Silva WA Jr. Placenta-enriched LincRNAsanddecrease migration and invasion potential of JEG-3 cell line., 2016, 11(3): e0151560.
[12] Amin V, Harris RA, Onuchic V, Jackson AR, Charnecki T, Paithankar S, Lakshmi Subramanian SL, Riehle K, Coarfa C, Milosavljevic A. Epigenomic footprints across 111 reference epigenomes reveal tissue-specific epigenetic regulation of lincRNAs., 2015, 6: 6370– 6380.
[13] Zhou C, Li S, Deng LL, Guan Y, Chen DK, Yuan XK, Xia TR, He XL, Shan YW, Li CC. Transcriptome analysis reveals long intergenic noncoding RNAs contributed to growth and meat quality differences between yorkshire and wannanhua pig., 2017, 8(8): E203.
[14] Zhao YH. Identification and functional analysis of lincRNA based on multi-tissue transcriptome data in mice[Dissertation]. Beijing Institute of Genomics, Chinese Academy of Sciences, 2015.赵宇慧. 基于多组织转录组数据的小鼠lincRNA的鉴定和功能分析[学位论文]. 中国科学院北京基因组研究所, 2015.
[15] Tang ZL, Wu Y, Yang YL, Yang YCT, Wang ZS, Yuan JP, Yang Y, Hua CJ, Fan XH, Niu GG, Zhang YB, Lu ZJ, Li K. Comprehensive analysis of long non-coding RNAs highlights their spatio-temporal expression patterns and evolutional conservation in Sus scrofa., 2017, 7: 43166.
[16] Li JY, Gao ZL, Wang XY, Liu HB, Zhang Y, Liu ZH. Identification and functional analysis of long intergenic noncoding RNA genes in porcine pre-implantation embryonic development., 2016, 6: 38333.
[17] Liu KS, Li TP, Ton H, Mao XD, Chen YJ. Advances of long noncoding RNAs-mediated regulation in reproduction., 2018, 131(2): 226–234.
[18] Muys BR, Lorenzi JCC, Zanette DL, De Barros Lima E Bueno R,De Araujo LF, Dinarte-Santos AR, Alves CP, Ramao A, De Molfetta GA, Vidal DO, Silva WA. Placenta-enriched LincRNAsanddecrease migration and invasion potential of JEG-3 cell line., 2016, 11(3): e0151560.
[19] Cabili MN, Trapnell C, Goff L, Koziol M, Tazon-Vega B, Regev A, Rinn JL. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses.,2011, 25(18): 1915–1927.
[20] Lv J, Liu H, Yu SH, Liu HB, Cui W, Gao Y, Zheng T, Qin G, Guo J, Zeng TB, Han ZB, Zhang Y, Wu Q. Identification of 4438 novel lincRNAs involved in mouse pre-implantation embryonic development., 2015, 290(2): 685–697.
[21] Zhang JY, Lv S, Niu HM, Lei AM. Research progress on the asymmetric division in mammalian oocytes., 2018, 40(4): 279–291.张俊玉, 吕珊, 牛慧敏, 雷安民. 哺乳动物卵母细胞不对称分裂的研究进展. 遗传, 2018, 40(4): 279–291.
[22] Zhu XL, Qi ST, Liu J, Chen L, Zhang C, Yang SW, OuYang YC, Hou Y, Schatten H, Song YL, Xing FQ, Sun QY. Synaptotagmin1 is required for spindle stability and metaphase-to-anaphase transition in mouse oocytes, 2012, 11(4): 818–826.
[23] Solc P, Schultz RM, Motlik J. Prophase I arrest and progression to metaphase I in mouse oocytes: comparison of resumption of meiosis and recovery from G2-arrest in somatic cells., 2010, 16(9): 654–664.
[24] Yang F, Zhang BY, Feng GD, Xiang W, Ma YX, Chen H, Chu MX, Wang PQ. A Mechanistic review of how hypoxic mircroenvironment regulates mammalian ovulation., 2016, 38(2): 109–117.杨芳, 张宝云, 冯光德, 向伟, 马云霞, 陈航, 储明星, 王凭青. 卵巢低氧微环境调节哺乳动物排卵的分子机制. 遗传, 2016, 38(2): 109–117.
[25] Nanassy L, Lee K, Javor A, Machaty Z. 316 Changes in MPF activity in porcine oocytes following activation by various methods., 2006, 18(2): 265– 265.
[26] Faerge I, Terry B, Kalous J, Wahl P, Lessl M, Ottesen JL, Hyttel P, Grøndahl C. Resumption of meiosis induced by meiosis-activating sterol has a different signal transduction pathway than spontaneous resumption of meiosis in denuded mouse oocytes cultured., 2001, 65(6): 1751–1758.
[27] Zhang DX, Park WJ, Sun SC, Xu YN, Li YH, Cui XS, Kim NH. Regulation of maternal gene expression by MEK/MAPK and MPF signaling in porcine oocytes duringmeiotic maturation., 2011, 57(1): 48–56.
[28] Zhang DX, Cui XS, Kim NH. Molecular characterization and polyadenylation-regulated expression of cyclin B1 and Cdc2 in porcine oocytes and early parthenotes., 2010, 77(1): 38–50.
[29] Liang CG, Su YQ, Fan HY, Schatten H, Sun QY. Mechanisms regulating oocyte meiotic resumption: roles of mitogen-activated protein kinase., 2007, 21(9): 2037–2055.
[30] Lee SE, Kim JH, Kim NH. Inactivation of MAPK affects centrosome assembly, but not actin filament assembly, in mouse oocytes maturing., 2007, 74(7): 904–911.
[31] Lee J, Miyano T, Moor RM. Localisation of phosphorylated MAP kinase during the transition from meiosis I to meiosis II in pig oocytes., 2000, 8(2): 119–125.
[32] Lan W, Petznick A, Heryati S, Rifada M, Tong L. Nuclear factor-κB: central regulator in ocular surface inflammation and diseases., 2012, 10(3): 137–148.
[33] Siomek A. NF-κB signaling pathway and free radical impact., 2012, 59(3): 323–331.
[34] Wullaert A, Bonnet MC, Pasparakis M. NF-κB in the regulation of epithelial homeostasis and inflammation., 2011, 21(1): 146–158.
[35] Ling XQ, Wang JK. Techniques for assaying the activity of transcription factor NF-κB., 2013, 35(5): 551–570.凌小倩, 王进科. 转录因子NF-κB活性检测技术. 遗传, 2013, 35(5): 551–570.
[36] Campbell KJ, Rocha S, Perkins ND. Active repression of antiapoptotic gene expression by RelA(p65) NF-kappa B., 2004, 13(6): 853–865.
[37] Ma JJ, Ren ZJ, Ma Y, Xu L, Zhao Y, Zheng CG, Fang YH, Xue T, Sun BL, Xiao WH. Targeted knockdown of EGR-1 inhibits IL-8 production and IL-8-mediated invasion of prostate cancer cells through suppressing EGR-1/NF-κB synergy., 2009, 284(50): 34600–34606.
[38] Pavlová S, Klucska K, Vašíček D, Ryban L,Harrath AH, Alwasel SH, Sivotkin AV. The involvement of SIRT1 and transcription factor NF-κB (p50/p65) in regulation of porcine ovarian cell function., 2013, 140(3–4): 180–188.
[39] Sirotkin AV, Dekanová P, Harrath AH, Alwasel SH, Vašíček D. Interrelationships between sirtuin1 and transcription factors P53 and NF-κB(P50/P65) in the control of ovarian cell apoptosis and proliferation., 2014, 358(4): 627–632.
[40] Cai HQ, Zhang YY, Pypaert M, Walker L, Ferro-Novick S. Mutants indisrupt traffic from the early endosome to the late Golgi., 2005, 171(5): 823–833.
[41] Hu WH, Pendergast JS, Mo XM, Brambilla R, Bracchi- Ricard V, Li F, Walters WM, Blits B, He L, Shaal SM, Bethea JR.NIBP, a novel NIK and IKKβ-binding protein that enhances NF-κB activation, 2005, 280(32): 29233–29241.
[42] Balboula AZ, Stein P, Schultz RM, Schindler K. Knockdown of RBBP7 unveils a requirement of histone deacetylation for CPC function in mouse oocytes., 2014, 13(4): 600–611.
[43] Balboula AZ, Stein P, Schultz RM, Schindler K. RBBP4 regulates histone deacetylation and bipolar spindle assembly during oocyte maturation in the mouse., 2015, 92(4): 105–117.
Comparison and analysis of lincRNAs expression profile in the hypothalamic-pituitary-ovarian axis of anestrous and estrous primiparous sows
Qiaoling Ren1, Jiaqing Zhang1, Dongfeng Lu2, Jing Wang1, Junfeng Chen1, Qiang Ma1, Xianxiao Bai1, Hongxiao Guo1, Binwen Gao1, Baosong Xing1
The normal estrus in weaned primiparous sows has a great impact on pig production and abnormal estrus is the main reason for the elimination of primiparous sows. In this study, we studied the long intergenic noncoding RNAs (lincRNAs) in the hypothalamic-pituitary-ovarian axis of anestrous and estrous primiparous sows. These long intergenic noncoding RNAs (lincRNAs) were screened and compared through RNA-seq analysis. The expression profiles of lincRNAs were obtained and their characteristics and functions were preliminarily analyzed. There are 3519 novel lincRNAs identified in the hypothalamic-pituitary-ovarian axis of anestrous and estrous primiparous sows. Compared with estrous primiparous sows, 17 differentially expressed lincRNAs were indentified, including 12 up-regulated lincRNAs and 5 down-regulated lincRNAs (FC≥2,<0.05). The four lincRNA transcripts obtained through selection were verified by qRT-PCR, which are consistent with the RNA-seq results. The GO, KEGG pathway, and lincRNA-mRNA co-expression network analysis of these 17 lincRNAs revealed that these lincRNAs were mainly involved in reproductive activities, such as oocyte meiosis mature, ovarian cells differentiation and granulosa cells apoptosis. The results enriched the data resources of pig lincRNAs and provided useful information for further research about the reproductive performance of primiparous sows.
lincRNAs; anestrus; estrus; primiparous sows; hypothalamic-pituitary-ovarian axis
2019-11-14;
2020-02-29
河南省农业科学院科技创新创意项目(编号:2020CX06),河南省重点研发专项(编号:182102110063),河南省财政预算科研专项项目(编号:2019CY015)和河南省农业科学院自主创新项目(编号:2019ZC42)资助[Supported by Science and Technology Innovation Program (No. 2020CX06), Key Special Rresearch and Development Program of Henan Province (No. 182102110063), Henan Province Financial Budget for Scientific Research Program (No. 2019CY015), and the Program for Independent Innovative Research in Henan Academy of Agricultural Sciences (No. 2019ZC42)]
任巧玲,硕士,副研究员,研究方向:猪的育种与营养。E-mail: renql76@163.com
张家庆,博士,助理研究员,研究方向:猪的育种与繁殖。E-mail: zjq8650612@163.com
任巧玲和张家庆为并列第一作者。
邢宝松,博士,副研究员,研究方向:猪的育种与管理。E-mail: baosong@126.com
10.16288/j.yczz.19-347
2020/3/17 10:34:49
URI: http://kns.cnki.net/kcms/detail/11.1913.r.20200316.1144.002.html
(责任编委: 李明洲)

