中国人群中抗结核药物引发肝损伤的易感基因标记研究
周晨希,李沫,怀聪,贺林,秦胜营
研究报告
中国人群中抗结核药物引发肝损伤的易感基因标记研究
周晨希,李沫,怀聪,贺林,秦胜营
上海交通大学Bio-X研究院,上海 200030
为系统性研究中国人群中抗结核药物引发肝损伤的易感基因标记,本研究以41例抗结核药物引发肝损伤的病人和39例健康对照为研究对象,采用Haloplex捕获测序的方法对其基因组中药物代谢、转运和免疫相关通路的109个基因进行靶向测序。用Plink软件对DNA突变位点与肝损伤的发生进行关联分析,以千人基因组计划东亚人群作为对照组,对显著性位点进行验证,并用SIFT和Polyphen2软件对预测显著关联的位点进行功能预测。结果发现rs2011404 (2= 4.6809,=0.0305)是抗结核药物引发的肝损伤的易感基因标记,且rs2011404突变可能引起UGT1A4蛋白的功能障碍。本研究为临床上对抗结核药物的合理用药提供了有益的参考。
抗结核药物;肝损伤;关联分析;
21世纪以来,我国的结核病发病率已降低为本世纪初的一半,但每年新增结核病患者仍有100万之多[1,2]。目前抗结核治疗的标准用药为异烟肼、利福平、吡嗪酰胺联合乙胺丁醇或链霉素等[3]。其中,异烟肼、利福平和吡嗪酰胺均具有潜在的肝毒性[3],其引发的肝脏不良反应发生率约为2.55%~11.9%[4,5],严重时可能会引发肝衰竭导致死亡。抗结核药物所致肝损伤还是导致用药失败、继而诱发结核病耐药的重要原因之一。而抗结核药物引发的肝损伤(anti-tuberculosis drug-induced liver injury, ATLI)具有个体差异明显,难以预测的特点,给病人带来极大的健康危害和经济损失,增加了医疗负担。
现有研究发现遗传因素是抗结核药物引发肝损伤个体间差异的关键因素之一[6]。结核药物导致肝损伤的机制可能有两种:一是药物的异常代谢导致毒性产物的积累,引起胞内的氧化应激反应和线粒体通透性的改变,继而导致肝细胞凋亡或坏死[7];二是药物引起肝脏的免疫调控和炎症反应[8]。因此,本研究系统性地选择了与药物代谢、转运、炎症和免疫相关的109个基因,在抗结核病四联用药(利福平、异烟肼、乙胺丁醇和吡嗪酰胺)引发肝损伤的病人与健康人中进行了关联分析,旨在找出抗结核药物导致肝损伤的易感基因位点,作为潜在的临床预测分子标记。
1 对象与方法
1.1 实验对象
病例组收集自上海肺科医院服用抗结核病四联用药(利福平、异烟肼、乙胺丁醇和吡嗪酰胺)后出现药物性肝损伤(drug-induced liver injury, DILI)的结核病患者。纳入标准:(1)有完整的用药信息和服药前后肝功能信息;(2)按照国际严重药物不良反应联盟(International Serious Adverse Events Consortium, iSAEC)标准评判肝损伤:①丙氨酸氨基转移酶或谷草转氨酶活性高于5倍正常值上限;或②碱性磷酸酶活性高于2倍正常值上限(当没有骨源性原因引起碱性磷酸酶值上升时);或③丙氨酸氨基转移酶活性高于3倍正常值上限并且总胆红素超过2倍正常值上限。(3)因果关系评分量表(roussel uclaf causality assessment method,RUCAM)得分≥7 (极可能或确定为药物引起的肝损伤)[9]。排除标准:同时服用中草药或中成药的患者。病例组最终纳入41例患者,男25例,女16例,年龄19~70岁,平均年龄44.88 (±17.63)岁。
对照组为来自上海新华医院的健康体检者39例。其中,男23例,女15例,年龄22~77岁,平均年龄35.66 (±12.83)岁。
本研究经过上海交通大学Bio-X研究院伦理委员会批准(批准号: M2011003),获得所有参与者的同意并签署知情同意书。
1.2 基因组DNA抽提
采用AxyPrep Blood Genomic DNA Miniprep Kit (Axygen,美国)提取受试者基因组DNA。
1.3 Haloplex捕获测序
针对109个药物相关代谢基因(表1)的外显子、3ʹUTR和5ʹUTR区域,使用Agilent Technologies的在线设计平台SureDesign (www.agilent.com/genomics/ suredesign) 设计Haloplex捕获探针。使用Haloplex Target Enrichment System Kits (Agilent Technologies, 美国) 酶切基因组DNA,与Haloplex探针杂交。经过扩增,纯化后的杂交片段由2100 Bioanalyzer (Agilent,美国)和Qubit dsDNA HS Assay Kit/Qubit 2.0定量分析仪(Life Technology,美国)测定片段大小及浓度。长度在175~625 bp之间的杂交片段为合格目的片段。使用Illumina MiSeq测序平台对合格片段进行测序。使用Agilent SureCall软件对测序结果进行分析,得到突变SNP位点的信息。
1.4 统计学分析
首先对SNP位点进行质量控制,删除缺失率> 10%,哈迪–温伯格遗传平衡检验(Hardy-Weinberg equilibrium, HWE)<10–4的SNP位点。针对每个SNP位点病例组和对照组的等位基因频数进行卡方检验,<0.05为有统计学意义。在验证研究中,将病例与对照组中存在的显著差异位点与其在千人基因组计划东亚人群中的等位基因频数进行卡方检验。统计学分析由PLINK (version1.07, http://www. pngu.mgh.harvard.edu/purcell/plink/)和Epi Info 7完成。
1.5 突变位点功能预测
使用SNP-nexus在线软件(https://www.snp- nexus.org)对显著性差异SNP位点进行PolyPhen2和SIFT蛋白功能预测。使用SWISS-MODEL (https:// swissmodel.expasy.org/)模拟蛋白的结构,并用PyMol软件(Schrödinger, LLC, 美国)进行图像处理。
2 结果与分析
2.1 捕获测序结果
在80例样本中,使用Haloplex测序试剂盒和Miseq平台对109个药物代谢相关基因(表1)进行捕获测序。突变的平均测序深度大于100×,检测到突变位点共5130个。质量控制后,位于57个基因上的228个SNP位点最终纳入关联分析。
2.2 与抗结核药物导致肝损伤相关的风险SNP/SNV位点
在41例患者和39例健康人样本中对SNP/SNV和抗结核药物引发的肝损伤进行关联分析,结果见表2。其中3个位点显示出显著性差异,分别为rs2011404 (OR = 0.1939, 95%= 0.0740– 0.5083,= 0.0004),rs16947 (OR = 6.5620, 95%= 1.4140–30.4500,= 0.0069)和rs338599 (OR = 0.3944, 95%= 0.1646–0.9446,= 0.0330)。为提高统计准确性,将对照组扩大为千人基因组计划中的东亚人群(= 504)对显著性位点进行了验证。结果发现,rs2011404 (2= 4.6809,= 0.0305)依然具有显著性差异,与抗结核药物导致肝损伤显著相关(表3)。
2.3 显著性SNP/SNV突变位点蛋白功能影响预测
用SIFT和PolyPhen2对rs2011404突变的导致的蛋白功能变化进行预测,结果显示rs2011404位置上的T突变为G,可能导致157位的半胱氨酸突变为色氨酸,很有可能为有害的突变(表4)。
用SWISS-MODEL和PyMol对UGT1A4蛋白结构进行同源模拟(图1A),蛋白结构显示rs2011404 (C157W)位于α螺旋上。野生型UGT1A4 157位的半胱氨酸可能会形成二硫键(图1B),而其突变为色氨酸(图1C)后,可能会影响二硫键的形成,进而影响α螺旋的稳定性。
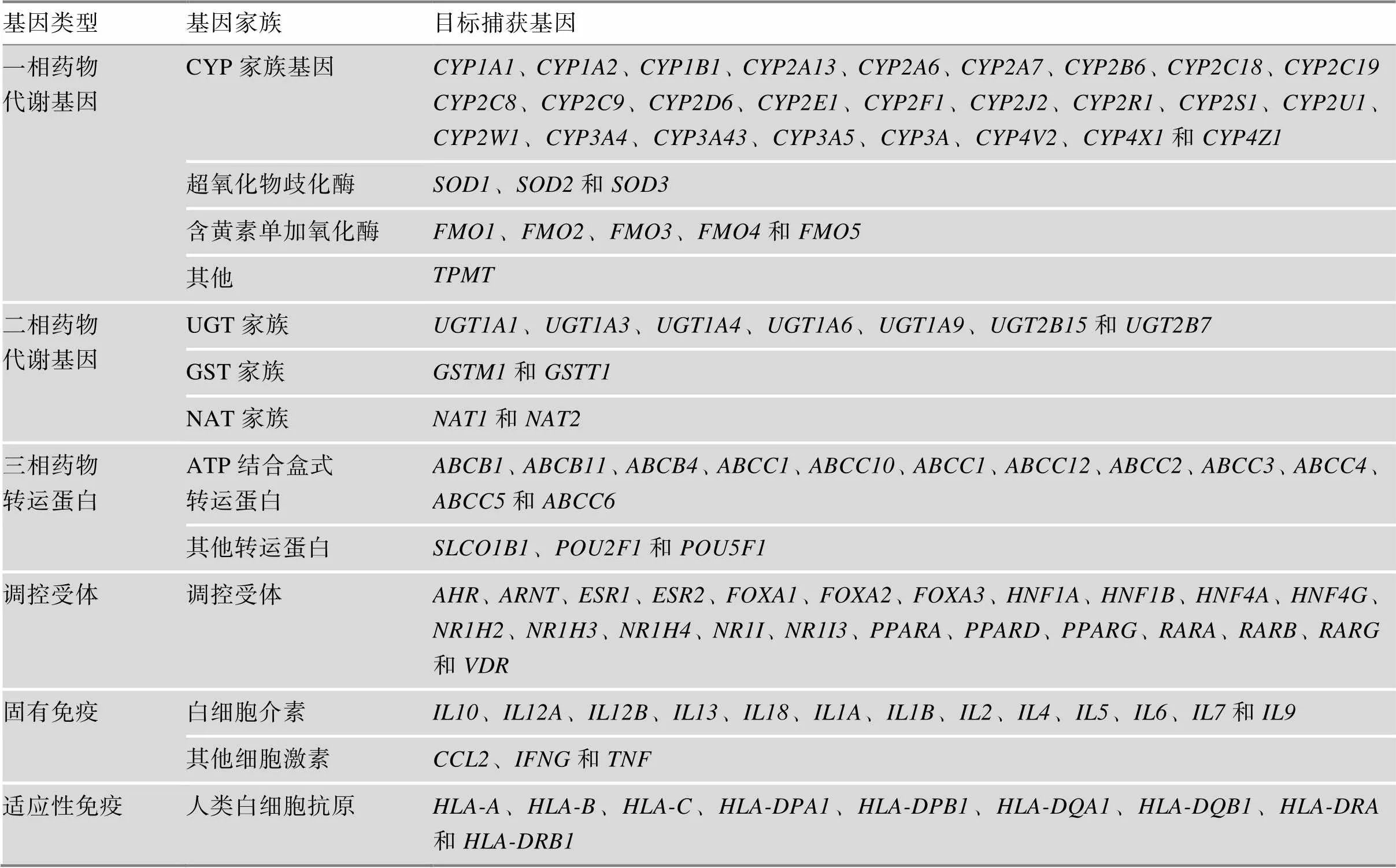
表1 抗结核药物导致肝损伤的109个候选基因
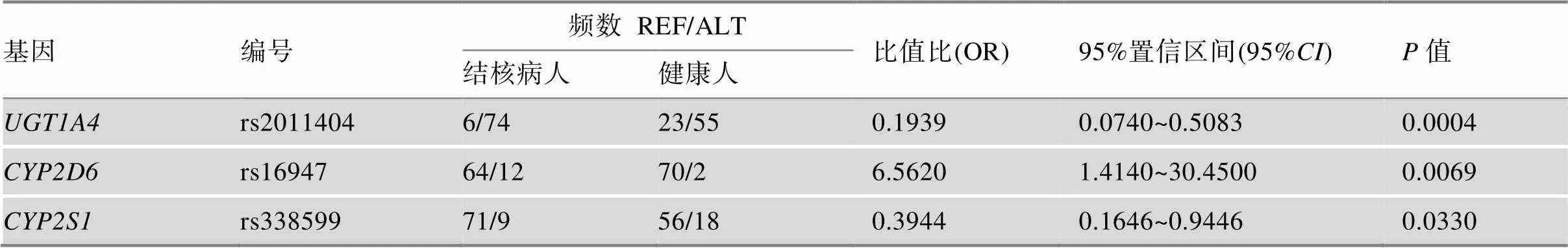
表2 与抗结核药物导致的肝损伤显著相关的SNP/SNV
REF (REF allele)表示原始碱基;ALT (ALT allele)表示突变碱基。
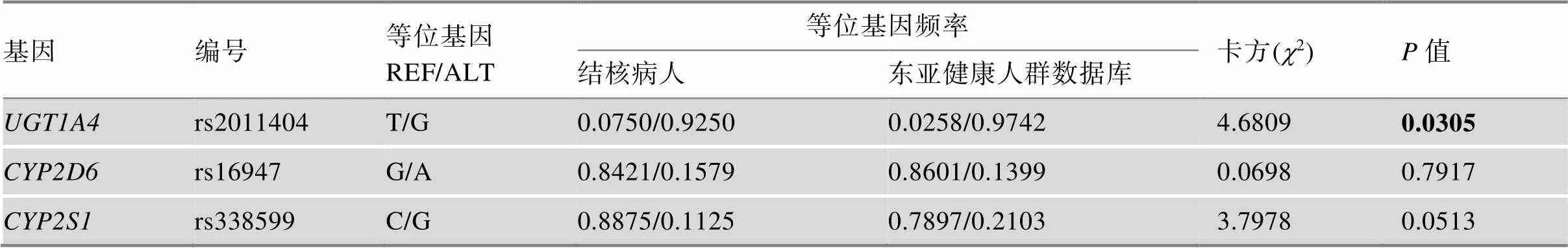
表3 验证与抗结核药物导致肝损伤显著相关的SNP/SNV
REF (REF allele)表示原始碱基;ALT (ALT allele)表示突变碱基。

表4 显著性SNP/SNV突变位点蛋白功能影响预测
−:表示同义突变不适用于PolyPhen2预测。
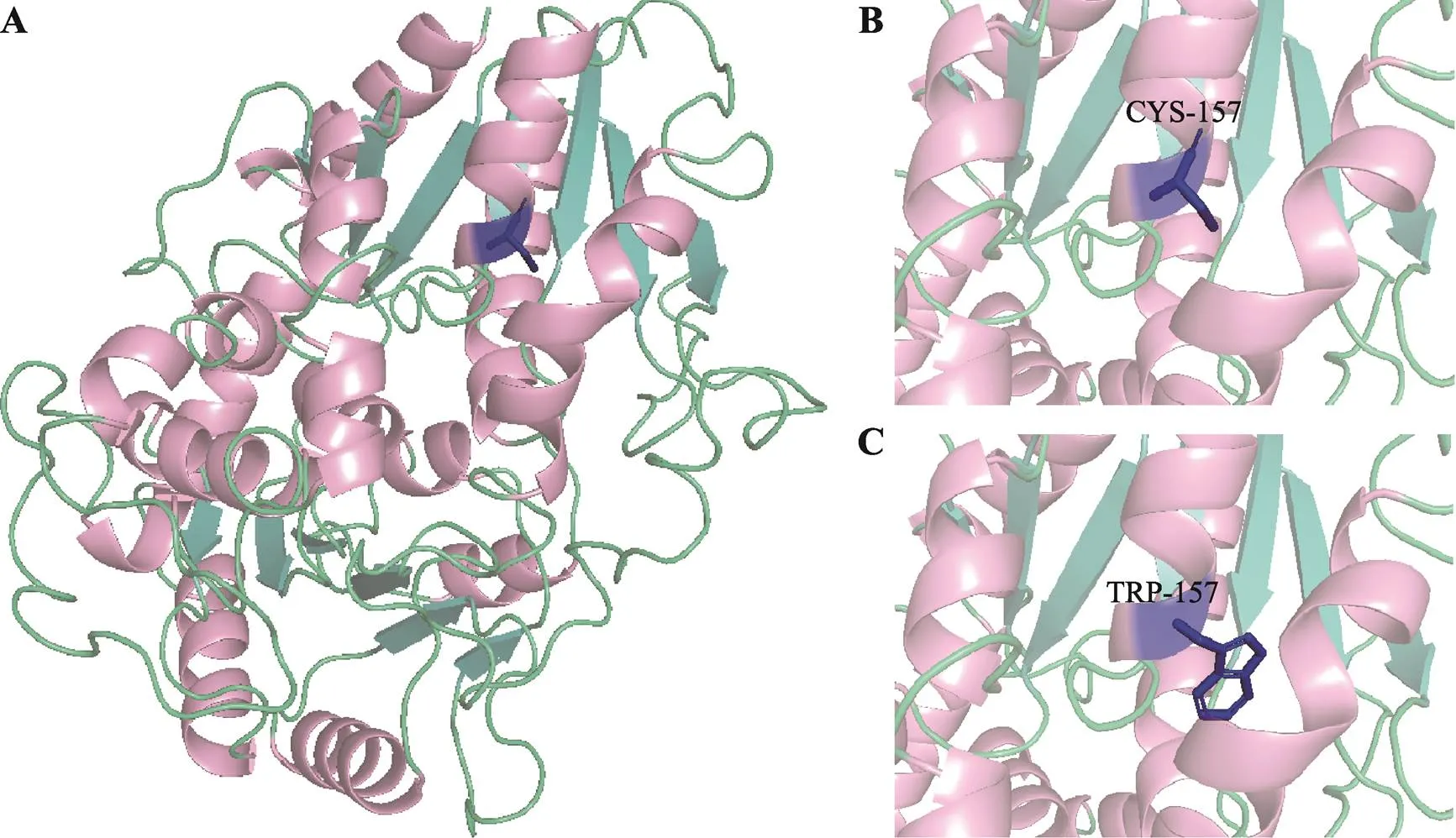
图1 突变蛋白结构分析
A:野生型UGT1A4蛋白结构模拟(粉色部分为α螺旋,青色部分为β折叠,蓝色部位为157位半胱氨酸);B:野生型UGT1A4的157位半胱氨酸局部放大示意图:C:rs2011404突变型UGT1A4的157位色氨酸局部放大示意图。
3 讨论
药物性肝损伤是在药物使用过程中,因药物或其代谢产物产生超敏反应导致的患者肝细胞毒性损伤及肝功能异常,为最常见和严重的药物不良反应(adverse drug reaction, ADR)之一。临床上,药物性肝损伤导致了3%~5%的黄疸症,也是造成急性肝功能衰竭的最主要原因,重者可致死亡[10]。现已知超过1100种药物具有潜在的肝毒性,包括中草药、抗结核药、抗感染药、解热镇痛药,抗肿瘤药等[11]。中国人群中,抗结核药物以异烟肼,利福平和吡嗪酰胺为主引发的肝损伤占所有药物性肝损伤的21.56%,是仅次于中草药的第二大造成药物性肝损伤的原因[12]。
非遗传因素(如性别、年龄、饮酒、肝病史、伴随感染和营养状况等)是抗结核药物引发肝损伤的危险因素,但其在临床上对肝损伤发生的预测作用有限[6]。国内外研究发现一相药物代谢酶[13]、二相药物代谢酶[14][15]、三相药物转运体[16][17]、免疫调节基因和[18]等与抗结核药物引发的肝损伤显著相关。这些研究验证了遗传因素对抗结核药物引发肝损伤的贡献。现有的中国人群中的研究多针对少数候选基因,可能会遗漏潜在的SNP/SNV位点,缺少全面的研究。本研究利用Haloplex捕获测序的方法,捕获与药物代谢、转运、炎症和免疫相关的一、二和三相代谢酶,调控受体,以及固有免疫和适应性免疫家族的共109个基因上的突变位点,旨在系统性地寻找抗结核药物引发肝损伤的潜在遗传标记。
本研究首次发现rs2011404 (471T>G)与抗结核药物引发的肝损伤显著相关。当对照组人群扩大为千人基因组计划中的东亚人群时,此SNP位点依然呈阳性。rs2011404位于基因区段上,此位点上T突变为C或G。其中,T>C不会造成氨基酸的改变,而T>G造成157位的半胱氨酸(C)突变为色氨酸(W)。后续的SIFT和PolyPhen2预测指出,C157W突变极有可能为有害的突变,影响UGT1A4代谢酶的正常功能。(UDP glucuronosyltransferase family 1 member A4)属于(UDP glucuronosyltransferase)家族,编码葡萄糖醛酸转移酶,负责催化一系列外源或内源化合物氨基的葡萄糖醛酸化(N-glucuronidation),属于II相药物代谢酶[19]。Chang等[20]在中国台湾人群中发现,会增加抗结核药物引发的肝损伤的风险,而尚未有研究报道与抗结核药物引发的肝损伤的相关性。Gufford 等[21]和Lee 等[22]证明利福平会诱导的表达,继而影响药物的代谢。与之相反,Cao等[23]发现利福平会抑制UGT1A4的活性。目前尚未发现异烟肼,吡嗪酰胺和乙胺丁醇对UGT酶有抑制作用。本研究发现与抗结核药物引发的肝损伤显著相关,而对于在抗结核药物代谢中的作用机制仍需进一步的实验验证。
rs16947和rs338599在80例患者的关联研究中呈阳性,而在扩大对照组为千人基因组计划东亚人群后未达到统计学意义。这一结果可能是由于本次研究的样本收集困难,样本量较少,而捕获测序后纳入的位点较多导致的,这可能是本研究的一个局限。
综上所述,本研究通过对109个药物代谢、转运、炎症和免疫相关的基因突变位点的系统性研究,发现rs2011404作为抗结核药物引发的肝损伤的潜在基因标记,为临床上对抗结核药物的合理用药提供了有益的参考。
[1] WHO. Global tuberculosis report 2018. 2018.
[2] Zheng W, Ji LD, Xing WH, Tu WW, Xu J. Advances in genome-wide association study of tuberculosis., 2013, 35(7): 823–829.郑伟, 季林丹, 邢文华, 涂巍巍, 徐进, 肺结核全基因组关联研究进展. 遗传, 2013, 35(7): 823–829.
[3] 肺结核诊断和治疗指南(2001年订). 内科急危重症杂志, 2002, 8(4): 225–229.
[4] Xia YY, Zhan SY. Systematic review of anti-tuberculosis drug induced adverse reactions in China., 2007, 30(6): 419–423.夏愔愔, 詹思延, 国内抗结核药物不良反应发生率的综合分析. 中华结核和呼吸杂志, 2007, 30(6): 419–423.
[5] Shang PH, Xia YY, Liu FY, Wang XM, Yuan YL, Hu DY, Tu DH, Chen YX, Deng PY, Cheng SM, Zhou L, Ma Y, Zhu LZ, Gao WW, Wang HY, Chen DF, Yang L, He PP, Wu SS, Tang SW, Lv XZ, Shu Z, Zhang Y, Yang ZR, Chen Y, Li N, Sun F, Li XT, He YJ, Garner P, Zhan SY. Incidence, clinical features and impact on anti-tuberculosis treatment of anti-tuberculosis drug induced liver injury (atli) in china., 2011, 6(7): e21836.
[6] Chen R, Wang J, Zhang Y, Tang SW, Zhan SY. Key factors of susceptibility to anti-tuberculosis drug-induced hepatotoxicity., 2015, 89(6): 883–897.
[7] Russmann S, Kullak-Ublick GA, Grattagliano I. Current concepts of mechanisms in drug-induced hepatotoxicity., 2009, 16(23): 3041–3053.
[8] Metushi I, Uetrecht J, Phillips E. Mechanism of isoniazid- induced hepatotoxicity: Then and now., 2016, 81(6): 1030–1036.
[9] Benichou C, Danan G, Flahault A. Causality assessment of adverse reactions to drugs-ii. An original model for validation of drug causality assessment methods: case reports with positive rechallenge., 1993, 46(11): 1331–1336.
[10] Hoofnagle JH, Björnsson ES. Drug-induced liver injury - types and phenotypes., 2019, 381(3): 264–273.
[11] Yu LC, Mao YM, Chen CW. Guidelines for the management of drug-induced liver injury., 2015, 31(11): 1752–1769.于乐成, 茅益民, 陈成伟. 药物性肝损伤诊治指南. 临床肝胆病杂志, 2015, 31(11): 1752–1769.
[12] Wen ZD, Hu GX, Yang J, Chen ZM. Retrospective analysis of 7018 cases of drug-induced liver injury literature., 2016, 14(12): 64–65.温祝杜, 胡国信, 杨剑, 陈智明. 7018例药物性肝损伤文献回顾性分析. 中外医学研究, 2016, 14(12): 64–65.
[13] Huang YS, Chern HD, Su WJ, Wu JC, Chang SC, Chiang CH, Chang FY, Lee SD. Cytochrome p450 2e1 genotype and the susceptibility to antituberculosis drug-induced hepatitis., 2003, 37(4): 924–930.
[14] Huang YS, Chern HD, Su WJ, Wu JC, Lai SL, Yang SY, Chang FY, Lee SD. Polymorphism of the n-acetyltransferase 2 gene as a susceptibility risk factor for antituberculosis drug-induced hepatitis., 2002, 35(4): 883–889.
[15] Roy B, Chowdhury A, Kundu S, Santra A, Dey B, Chakraborty M, Majumder PP. Increased risk of antituberculosis drug-induced hepatotoxicity in individuals with glutathione s-transferase m1 ‘null’ mutation., 2001, 16(9): 1033–1037.
[16] Yimer G, Ueda N, Habtewold A, Amogne W, Suda A, Riedel KD, Burhenne J, Aderaye G, Lindquist L, MakonnenE, Aklillu E. Pharmacogenetic & pharmacokinetic biomarker for efavirenz based arv and rifampicin based anti-tb drug induced liver injury in tb-hiv infected patients., 2011, 6(12): e27810.
[17] Li LM, Chen L, Deng GH, Tan WT, Dan YJ, Wang RQ, Chen WS. SLCO1B1*15 haplotype is associated with rifampin-induced liver injury., 2012, 6(1): 75–82.
[18] Sharma SK, Balamurugan A, Saha PK, Pandey RM, Mehra NK. Evaluation of clinical and immunogenetic risk factors for the development of hepatotoxicity during antituberculosis treatment., 2002, 166(7): 916–919.
[19] Benoit-Biancamano MO, Adam JP, Bernard O, Court MH, Leblanc MH, Caron P, Guillemette C. A pharmacogenetics study of the human glucuronosyltransferase UGT1A4., 2009, 19(12): 945–954.
[20] Chang JC, Liu EH, Lee CN, Lin YC, Yu MC, Bai KJ, Chen HY. UGT1A1 polymorphisms associated with risk of induced liver disorders by anti-tuberculosis medications., 2012, 16(3): 376–378.
[21] Gufford BT, Robarge JD, Eadon MT, Gao H, Lin H, Liu Y, Desta Z, Skaar TC. Rifampin modulation of xeno- and endobiotic conjugating enzyme mrna expression and associated micrornas in human hepatocytes., 2018, 6(2): e00386.
[22] Lee SY, Lee JY, Kim YM, Kim SK, Oh SJ. Expression of hepatic cytochrome P450s and UDP-glucuronosyltransferases in PXR and CAR double humanized mice treated with rifampicin., 2015, 235(2): 107–115.
[23] Cao L, Greenblatt DJ, Kwara A. Inhibitory effects of selected antituberculosis drugs on common human hepatic cytochrome P450 and UDP-glucuronosyltransferase enzymes., 2017, 45(9): 1035–1043.
Study on hereditary susceptibility genetic markers to anti-tuberculosis drug induced liver injury in Chinese population
Chenxi Zhou, Mo Li, Cong Huai, Lin He, Shengying Qin
To systematically study the susceptible genetic markers for liver injury induced by anti-tuberculosis drugs in the Chinese population, 109 genes related to drug metabolism, transport and immunity were captured by Haloplex capture technique from DNA samples of 41 patients with liver injury induced by anti-tuberculosis drugs and 39 healthy controls, and sequenced completely. Association study was conducted using Plink software. To verify the significant candidate SNPs, thestudywas expanded to the control group from the 1000-person Genome Project of the East Asian population. SIFT and Polyphen2 software were used to predict the functional significance of the associated SNPs. Our results identified thers2011404 (2= 4.6809,= 0.0305) as a susceptible genetic marker for liver injury induced by anti-tuberculosis drugs, and rs2011404 mutation might contribute to UGT1A4 protein dysfunction. This study has provided a potentially useful reference for establishing the precision medicine in rational uses of anti-tuberculosis drugs in the clinic.
anti-tuberculosis drug; liver injury; association study;
2019-11-24;
2019-12-26
国家自然科学基金项目(编号:81773818, 81273596, 30900799, 81671326)和上海市浦江人才计划项目(编号:17PJD020)资助 [Supported by the National Natural Science Foundation of China (Nos. 81773818, 81273596, 30900799, 81671326) and Shanghai Pujiang Program (No. 17PJD020)]
周晨希,硕士,专业方向:生物学。E-mail: chenxizhou@sjtu.edu.cn
秦胜营,研究员,研究方向:药物基因组学与个体化医学。E-mail: chinsir@sjtu.edu.cn
10.16288/j.yczz.19-271
2020/3/11 8:26:00
URI: http://kns.cnki.net/kcms/detail/11.1913.r.20200310.1520.002.html
(责任编委: 张天宇)

