凡纳滨对虾微卫星分子标记的开发及不同养殖家系遗传多态性分析
李强勇 李旻 曾地刚 朱威霖 彭敏 杨春玲 刘青云 赵永贞 陈秀荔 陈晓汉


摘要:【目的】基于第三代高通量測序技术——单分子实时(SMRT)测序开发凡纳滨对虾微卫星分子标记,为凡纳滨对虾的遗传育种研究提供基础资料。【方法】采用SMRT测序对凡纳滨对虾转录组进行测序,经Illumina测序纠错后利用MISA对获得的转录组数据进行分析;以Primer 3.0设计微卫星引物,随机挑选40对微卫星引物进行PCR扩增验证,并选用扩增成功且具有多态性的微卫星引物用于不同养殖家系凡纳滨对虾遗传多态性分析。【结果】凡纳滨对虾转录组SMRT测序共获得51367条非冗余全长转录本序列,鉴定出39674条微卫星序列;微卫星的分布密度为0.232 SSR/kb;以二核苷酸重复微卫星序列分布最多,共有22223条(占56.01%)。随机选取的40对微卫星引物中有26对微卫星引物能成功扩增出特异性条带;微卫星荧光分型结果显示,有16个微卫星位点具有多态性,共获得67个等位基因,平均等位基因数(Na)为4.2个,计算获得的平均观测杂合度(Ho)为0.511,平均期望杂合度(He)为0.451,平均多态性信息含量(PIC)为0.489。在16个微卫星位点中,有2个微卫星位点为低度多态性(PIC<0.25),6个微卫星位点为中度多态性(0.25
关键词: 凡纳滨对虾;单分子实时(SMRT)测序;微卫星分子标记;多态性分析
中图分类号: S917.4 文献标志码: A 文章编号:2095-1191(2020)02-0429-08
Development of microsatellite markers of Litopenaeus vannamei and genetic polymorphism analysis of different cultured families
LI Qiang-yong, LI Min, ZENG Di-gang, ZHU Wei-lin, PENG Min, YANG Chun-ling, LIU Qing-yun, ZHAO Yong-zhen, CHEN Xiu-li, CHEN Xiao-han*
(Guangxi Academy of Fishery Sciences/Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy
Aquaculture, Nanning 530021, China)
Abstract:【Objective】Microsatellite markers of Litopenaeus vannamei based on the third-generation high-throughput sequencing technology, single-molecule real-time (SMRT) sequencing were developed in order to provide basic data for genetic and breeding research of L. vannamei. 【Method】The transcriptome of L. vannamei was sequenced using SMRT. The obtained transcriptome data were corrected by Illumina sequencing, then analyzed by MISA software. The microsatellite primers were designed with Primer 3.0 software, and 40 pairs of microsatellite primers were randomly selected for PCR amplification. Microsatellite primers with successful amplification and polymorphism were selected for genetic polymorphism analysis of L. vannamei in different breeding families. 【Result】In total, 51367 non-redundant full length transcript sequences and 39674 SSR loci were obtained by SMRT sequencing of the L. vannamei transcriptome. The distribution density of microsatellites was 0.232 SSR/kb; the dinucleotide repeat microsatellite sequences were the most distribu-ted, with a total of 22223(56.01%). Of the 40 randomly selected microsatellite primers, 26 pairs of microsatellite primers successfully amplified specific bands. The microsatellite fluorescence phenotyping showed that 16 microsatellite loci were polymorphic. A total of 67 alleles were obtained, and the average number of alleles(Na) was 4.2. The calculated average observed heterozygosity(Ho) was 0.511, the average expected heterozygosity(He) was 0.451,and the average polymorphism information content(PIC) was 0.489. Among the 16 microsatellite loci, 2 loci were low polymorphisms (PIC<0.25), 6 loci were moderate polymorphisms(0.25
Key words: Litopenaeus vannamei; single molecule real-time sequencing; microsatellite markers; polymorphism analysis
Foundation item: Guangxi Innovation Driven Development Project(Guike AA17204088-1); National Fishery Science Data Center Observation and Detection Project of Agricultural Basic Long-term Scientific and Technological Work(ZX08S211664); Science and Technology Plan Project of Guangxi Aquatic Animal Husbandry and Veterinary Bureau (Guiyumuke 201633015); Guangxi Key Laboratory of Genetic Breeding and Healthy Breeding Opening Project (17-A-04-04)
0 引言
【研究意义】凡纳滨对虾(Litopenaeus vannamei)又称为南美白对虾,是世界上养殖最广泛的甲壳类动物(陈锚等,2008)。凡纳滨对虾具有耐高温、抗病力强、出肉率高、营养丰富、生长速度快及盐度适应范围广等特点,现已发展成为对虾养殖的重要对象(王兴强等,2004;黄薇等,2014)。1988年,中国科学院海洋研究所首次引进凡纳滨对虾,并于1992年突破人工繁殖难关,之后凡纳滨对虾在全国范围内广泛养殖,且各地不断从国外引进亲虾用于虾苗生产(王兴强等,2004),如今凡纳滨对虾已成为我国对虾养殖的主导品种。但近年来凡纳滨对虾病害发生频繁,包括白斑综合征、桃拉综合征、传染性皮下及造血组织坏死病和弧菌病等,给其养殖业造成巨大经济损失(Lightner,2011)。我国养殖的凡纳滨对虾主要是从国外引进的品种或品系,且多为杂交后代,遗传背景较复杂,繁育多代后其性状出现分离,表现为群体生长速度减慢、抗病和抗逆能力减弱等(熊建华等,2011)。因此,急需研发出有效的分子标记技术用于凡纳滨对虾种质资源鉴定及分子标记选育等研究工作。【前人研究进展】微卫星(Microsatellite)又称简单重复序列(Simple sequence repeats,SSR),由2~6个核苷酸的串联重复片段构成,是目前应用较广泛的分子标记之一(盛岩等,2002;周大颜等,2019)。因其稳定性和可重复性高,且具有特异性和共显性的特点,现已广泛应用于动植物的生物遗传多样性分析、种质资源鉴定及分子标记等研究领域(孙效文等,2008;张丽颖等,2009;凌士鹏等,2018)。凡纳滨对虾微卫星分子标记在1996年已有报道(Garcia et al.,1996);而Meehan等(2003)最早實现批量开发微卫星分子标记,通过构建基因组DNA文库和使用探针筛选的方法共得包含573个微卫星的173条序列。随着第二代高通量测序技术的发展,已有学者采用高通量测序技术开发凡纳滨对虾微卫星分子标记的研究报道。马宁和曾地刚(2013)运用454高通量测序技术筛选出588条凡纳滨对虾微卫星序列;Yu等(2014)利用Illumina HiSeq 2000测序技术查找凡纳滨对虾SNP位点;杨铭等(2017)采用Illumina测序技术进行凡纳滨对虾转录组测序,结果鉴定获得14767条微卫星序列;Lu等(2018)通过对凡纳滨对虾进行转录组测序,筛选出49个与氨相关的SNPs位点,且进一步鉴定发现有12个SNPs位点与氨耐受性相关;Santos等(2018)通过RNA测序技术(RNA-seq)分析鉴定了与凡纳滨对虾免疫应答及生长性能相关的SNP位点。【本研究切入点】单分子实时(Single-molecule real-time,SMRT)测序是近年来由Pacific Biosciences公司开发的第三代高通量测序技术,为实现全长单分子DNA序列的直接测序提供了平台(Eid et al.,2009)。SMRT测序长度可达20 kb,能对完整的cDNA分子进行测序,而无需片段化或测序后组装(Roberts et al.,2013),但至今未见将SMRT测序应用于凡纳滨对虾微卫星分子标记开发的研究报道。【拟解决的关键问题】利用SMRT测序对凡纳滨对虾全长转录组进行测序分析,从获得的全长RNA序列中筛选微卫星序列并设计微卫星引物,随机挑选微卫星引物对不同养殖家系凡纳滨对虾进行遗传多态性分析,为凡纳滨对虾的遗传育种研究提供基础资料。
1 材料与方法
1. 1 试验材料
供试桂海1号凡纳滨对虾由广西水产科学研究院南美白对虾遗传育种中心提供。取6尾凡纳滨对虾的肌肉、心脏和肝胰腺组织,混合后置于液氮中保存备用。从无特定病原的桂海1号凡纳滨对虾种质库中随机取16个不同养殖家系,对虾平均体长16.0 cm,平均体质量26.8 g/尾,取肌肉组织置于-80 ℃冰箱中保存备用。
1. 2 总RNA提取
按TRIzol LS Reagent试剂盒(Invitrogen,美国)说明从混合的凡纳滨对虾组织中提取总RNA,使用DNase I(Invitrogen,美国)除去基因组DNA。采用NanoDrop 2000(Thermo Scientific,美国)测量总RNA的纯度(OD260/280)、浓度和吸收峰,并以Agilent 2100 Bioanalyser(美国)测定总RNA质量,RIN(RNA integrity number)>7的总RNA用于构建SMRT测序的cDNA文库。
1. 3 SMART测序
使用SMARTer PCR cDNA Synthesis Kit(TaKaRa,日本)将10 μg总RNA反转录合成cDNA,以BluePippinTM系统(Sage Science,美国)构建5个大小分别为<1 kb、1~2 kb、2~3 kb、3~6 kb和>6 kb的cDNA文库。按照PacBio的步骤每个cDNA文库在PacBio RSII平台上使用C4试剂进行SMART测序;同时使用TruSeq RNA试剂盒(Illumina,美国)构建Illumina凡纳滨对虾cDNA文库,在HiSeq2500高通量测序仪上进行测序,测序结果用于SMART测序数据的错误纠正。
1. 4 微卫星序列鉴定及引物设计
对SMRT测序的原始数据进行质控及Illumina测序纠错后获得高质量的全长转录本序列,利用MISA(http://pgrc.ipk-gatersleben.de/misa/)搜寻SSR重复基元的区间。MISA参数设置如下:2-6、3-5、4-5、5-5、6-5,复合微卫星分子标记的最大长度100 bp;然后采用Primer 3.0设计微卫星序列的引物。
1. 5 微卫星引物验证
从批量设计的微卫星引物中随机挑选40对(表1),委托生工生物工程(上海)股份有限公司合成。不同养殖家系对虾肌肉组织分别采用动物基因组提取试剂盒[天根生化科技(北京)有限公司]提取DNA,DNA经1.0%琼脂糖凝胶电泳检查无降解后使用紫外分光光度计测其浓度,以TE(pH 8.0)稀释至50 ng/μL,-20 ℃保存备用。
PCR反应体系50.0 μL:基因组DNA模板20 ng,2×Taq MasterMix 25.0 μL ,正、反向引物(10 μmol/L)各2.0 μL,超纯水补足至50.0 μL。扩增程序:94 ℃预变性1 min;94 ℃ 45 s,58 ℃ 45 s,72 ℃ 45 s,进行35个循环;72 ℃延伸5 min。以2.0%琼脂糖凝胶在1×TAE缓冲液中150 V下电泳30 min,凝胶成像系统拍照。选取PCR扩增成功的微卫星引物在其正向引物5'端标记FAM荧光基团,并对16尾凡纳滨对虾DNA进行PCR扩增。PCR产物经2.0%琼脂糖凝胶电泳检测后送至上海迈浦生物科技有限公司,在ABI 3730测序仪上进行荧光分型。
1. 6 微卫星数据分析
获得不同养殖家系凡纳滨对虾的微卫星分型数据后,使用PopGen32计算每个微卫星位点的等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)及多态信息含量(PIC)。
2 结果与分析
2. 1 凡纳滨对虾微卫星数量及其分布密度
凡纳滨对虾转录组SMRT测序共获得51367条非冗余全长转录本序列(GenBank登录号SRX3267788~ SRX3267801),利用MISA对这些转录本序列进行分析,共获得39674条微卫星序列,分布在29461条转录本序列中,包含微衛星的序列占总序列的57.35%。序列拼接总长度为171322994 bp,微卫星的分布密度为0.232 SSR/kb,平均每4.32 kb出现1条微卫星序列。各核苷酸重复类型的凡纳滨对虾微卫星数量差异明显,其分布密度也存在明显差异。在所鉴定的39674条凡纳滨对虾微卫星序列(表2)中,二核苷酸重复微卫星序列分布最多,共有22223条(占56.01%);三核苷酸重复微卫星序列14781条(占37.26%),四核苷酸重复微卫星序列1947条(占4.91%),六核苷酸重复微卫星序列441条(占1.11%);分布最少的是五核苷酸重复微卫星序列,仅有282条(占0.71%)。
2. 2 凡纳滨对虾微卫星不同重复类型中各核苷酸重复单元的数量与频率
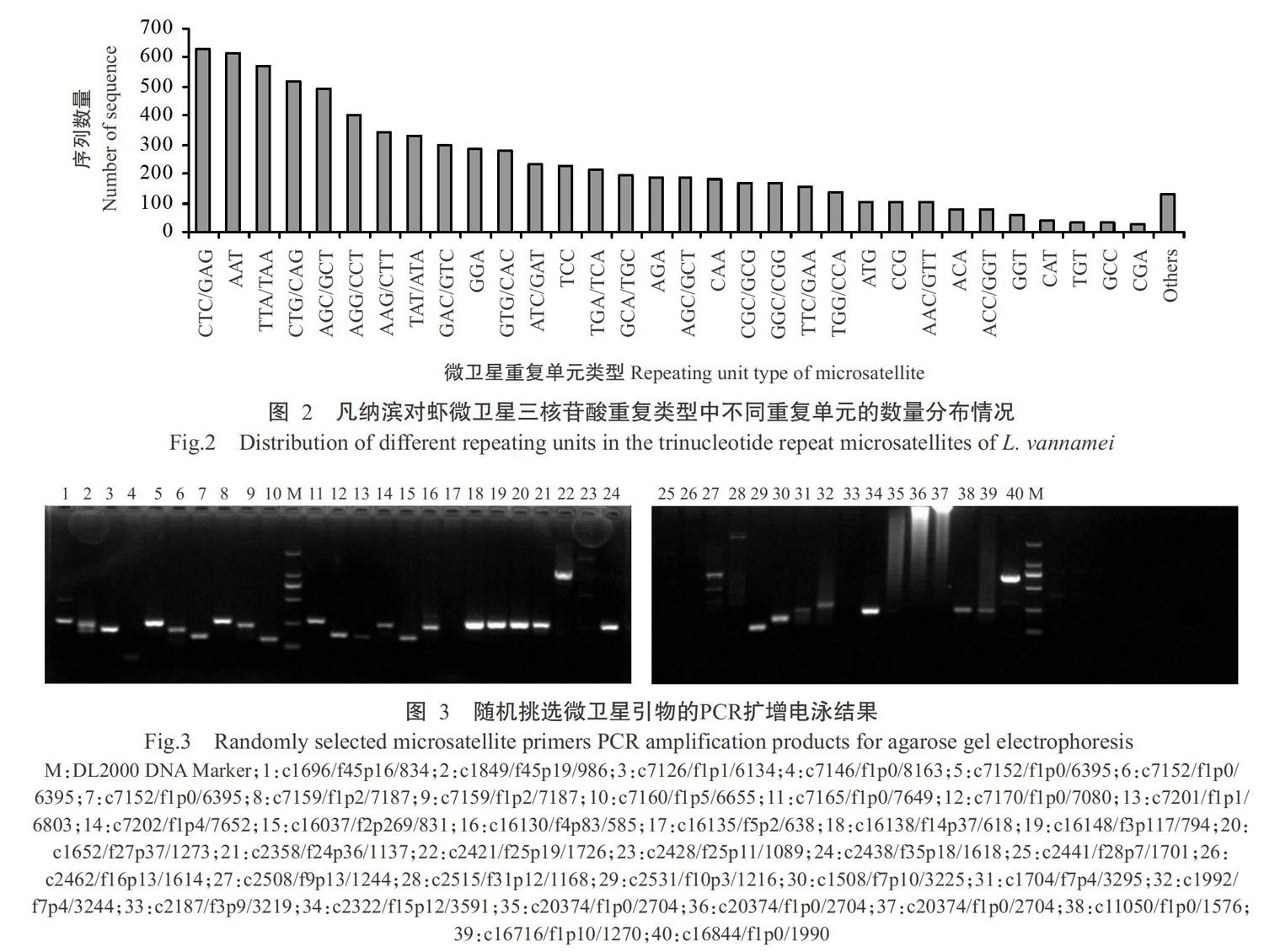
凡纳滨对虾微卫星二核苷酸重复类型中不同重复单元的数量分布情况如图1所示,以含AT/TA重复单元的微卫星序列数量最多,其次是CA/TG重复单元和GA/TC重复单元,AC/GT重复单元和AG/CT重复单元的数量差异不明显。凡纳滨对虾微卫星三核苷酸重复类型中不同重复单元的数量分布情况如图2所示,以含CTC/GAG重复单元的微卫星序列数量最多,其次是AAT、TTA/TAA、CTG/CAG和AGC/GCT等重复单元。
2. 3 微卫星分子标记验证结果
根据鉴定出的39674条微卫星序列,利用Pri-mer 3.0设计39367对微卫星引物。为验证设计的微卫星引物能否通过PCR扩增出微卫星序列,随机选取40对微卫星引物,在桂海1号凡纳滨对虾DNA混池中进行PCR扩增,结果发现有26对微卫星引物能成功扩增出特异性条带(图3),占所选PCR扩增引物数量的65.00%。为进一步验证微卫星的多态性,以筛选出的26对微卫星引物分别对16个不同养殖家系凡纳滨对虾个体DNA进行PCR扩增,PCR扩增产物在ABI 3730测序仪上进行荧光分型(图4),并采用GeneMarker 3.0.0分析分型结果。经微卫星分型后有16个微卫星位点显示出多态性(表3),多态性位点的等位基因数在2~8,共获得67个等位基因,平均Na为4.2个,计算获得的平均Ho为0.511,平均He为0.451,平均PIC为0.489。依据Botstein等(1980)的判断标准:PIC<0.25代表该位点具有低度多态性,0.25
3 讨论
微卫星分子标记对开展水产动物遗传学研究具有重要意义。为开发凡纳滨对虾微卫星分子标记,本研究采用第三代高通量测序技术——SMRT测序对凡纳滨对虾转录组进行测序,结果在获得51367条非冗余全长转录本序列中鉴定出39674条微卫星序列,并设计39367对微卫星引物。Meehan等(2003)通过传统的构建文库和探针筛选仅发现573个凡纳滨对虾微卫星,杨铭等(2017)采用高通量测序技术则鉴定获得14767条凡纳滨对虾微卫星序列。可见,利用高通量测序技术开发微卫星分子标记的优势在于能大规模发现微卫星序列,极大丰富凡纳滨对虾微卫星分子标记的数量。本研究在分析凡纳滨对虾转录组微卫星数量及其分布密度时发现,不同类型重复单元的微卫星数量差异较明显,其分布密度也存在明显差异,其中,二核苷酸重复微卫星序列分布最多,与马宁和曾地刚(2013)、杨铭等(2017)采用高通量测序得到的凡纳滨对虾转录组微卫星结果一致。微卫星的分布密度为0.232 SSR/kb,高于楊铭等(2017)采用第二代高通量测序技术获得的凡纳滨对虾转录组微卫星分布密度(0.215 SSR/kb),究其原因可能是本研究测序获得的RNA序列较长,虽然获得的非冗余全长转录本序列数量(51367条)略少于杨铭等(2017)采用第二代高通量测序技术获得的非冗余全长转录本序列数量(66815条),但相对较长的RNA序列可鉴定出更多微卫星位点。说明采用第三代测序技术开发微卫星分子标记更具优势。
为验证设计的微卫星引物,随机挑选40对微卫星引物对桂海1号凡纳滨对虾DNA混池进行PCR扩增,结果发现有26对微卫星引物扩增成功(占65.00%)。进一步的微卫星分型结果显示,在26对成功扩增的微卫星引物中有16对在不同养殖家系凡纳滨对虾个体DNA的扩增产物中具有多态性,表明本研究开发获得的这些微卫星分子标记有相当高的比例可用于凡纳滨对虾遗传学研究。Na、PIC及遗传杂合度(H)是检测种群遗传多样性的主要指标。本研究挑选的16个微卫星位点的Na在2~9个,平均为4.2个,符合用于遗传多样性评估的微卫星至少具有4个等位基因的标准(Wajid et al.,2014)。Botstein等(1980)认为:PIC<0.25代表该位点具有低度多态性,0.25
H是衡量群体遗传变异的最适参数,是指群体中某个位点上的杂合子频率,主要反映群体的遗传变异程度(Shikano and Taniguchi,2002);PIC与基因突变率有关(Gyapay et al.,1994),因此,通过H和PIC能反映群体内个体遗传变异程度,其数值高低与遗传变异程度呈正相关。本研究中,不同养殖家系凡纳滨对虾的平均Ho为0.511、平均He为0.451,群体的杂合度及遗传多样处于中等水平。杂合子缺失现象的产生可能是研究对象数量有限、近亲杂交或人为干扰等因素导致稀有碱基缺失所致(Antoro et al.,2006)。Hardy-Weinberg平衡检验是评估样本代表性及其基因分型质量的重要工具(Crane et al.,2004),样本群体小、近亲交配、等位基因突变等均可导致所检测的种群基因型偏离Hardy-Weinberg平衡(宋炜等,2017)。本研究所分析的16个微卫星多态性位点在16尾不同养殖家系凡纳滨对虾中,有12个微卫星位点满足Hardy-Weinberg平衡,有4个微卫星点位偏离Hardy-Weinberg平衡,表明不同养殖家系凡纳滨对虾群体遗传结构尚处于相对稳定状态。
SMRT测序是近年来新出现的第三代测序技术,是通过观测单个DNA聚合酶合成过程而实现单分子实时测序的技术。SMRT测序不仅具有测序长度优势,还不存在GC偏好(Roberts et al.,2013),因此开发的微卫星分子标记在基因组中的分布相对较均匀,而有利于构建遗传连锁图谱。本研究首次利用SMRT测序开发凡纳滨对虾微卫星分子标记,有效增加了凡纳滨对虾的可用微卫星分子标记数量,同时证实采用SMRT测序开发微卫星分子标记是一种高效可行的方法。
4 结论
通过对凡纳滨对虾转录组进行SMRT测序、Illumina测序纠错及MISA分析,共获得39674条微卫星序列,从设计的39367对微卫星引物中随机选取40对微卫星引物进行验证,发现有16对微卫星引物显示出多态性,说明采用SMRT测序开发获得的微卫星分子标记有相当高的比例可用于凡纳滨对虾遗传学研究。即采用SMRT测序开发凡纳滨对虾微卫星分子标记是一种简单而高效的途径,有助于开展凡纳滨对虾的遗传育种研究工作。
参考文献:
陈锚,吴长功,相建海,黄皓,刘小林,刘翠红,何建国,孙成波. 2008. 凡纳滨对虾的选育与家系的建立[J]. 海洋科学,32(11):5-8. [Chen M,Wu C G,Xiang J H,Huang H,Liu X L,Liu C H,He J G,Sun C B. 2008. Selective breeding and pedigree foundation of Litopenaeus vannamei[J]. Marine Sciences,32(11):5-8.]
黄薇,李剑锋,张根玉,李永权,施永海,张海明,徐嘉波,骆志强. 2014. 池塘温室南美白对虾、罗氏沼虾三茬轮养技术[J]. 中国水产,(6):69-71. [Huang W,Li J F,Zhang G Y,Li Y Q,Shi Y H,Zhang H M,Xu J B,Luo Z Q. 2014. Three-crop rotation culture of Litopenaeus vannamei and Macrobrachium rosenbergii in pond greenhouse[J]. China Fisheries,(6):69-71.]
凌士鹏,孙萍,林贤锐,沈建生. 2018. 基于SSR标记的桃种质资源遗传多样性研究[J]. 江西农业学报,30(11):14-18. [Ling S P,Sun P,Lin X R,Shen J S. 2018. A study on genetic diversity of peach germplasm resources based on SSR markers[J]. Acta Agriculturae Jiangxi,30(11):14-18.]
马宁,曾地刚. 2013. 凡纳滨对虾微卫星序列的筛选[J]. 西南农业学报,26(6):2629-2633. [Ma N,Zeng D G. 2013. Isolation of microsatellite sequences from Litopenaeus vannamei[J]. Southwest China Journal of Agricultural Sciences,26(6):2629-2633.]
盛巖,郑蔚虹,裴克全,马克平. 2002. 微卫星标记在种群生物学研究中的应用[J]. 植物生态学报,26(S):119-126. [Sheng Y,Zheng W H,Pei K Q,Ma K P. 2002. Applications of micros atellites in population biology[J]. Acta Phytoecologica Sinica,26(S):119-126.]
宋炜,孟永永,蒋科技,赵明,任桂静,张凤英,马凌波. 2017. 棘头梅童鱼七个野生群体遗传多样性的微卫星分析[J]. 水产学报,41(1):31-39. [Song W,Meng Y Y,Jiang K J,Zhao M,Ren G J,Zhang F Y,Ma L B. 2017. Analysis of genetic diversity among seven wild Collichthys lucidus populations by using microsatellite marker[J]. Journal of Fisheries of China,41(1):31-39.]
孙效文,张晓锋,赵莹莹,张研,贾智英,常玉梅,鲁翠云,梁利群. 2008. 水产生物微卫星标记技术研究进展及其应用[J]. 中国水产科学,15(4):689-703. [Sun X W,Zhang X F,Zhao Y Y,Zhang Y,Jia Z Y,Chang Y M,Lu C Y,Liang L Q. 2008. Development and application of microsatellite markers in aquatic species[J]. Journal of Fishery Sciences of China,15(4):689-703.]
王兴强,马甡,董双林. 2004. 凡纳滨对虾生物学及养殖生态学研究进展[J]. 海洋湖沼通报,(4):94-100. [Wang X Q,Ma S,Dong S L. 2004. Studies on the biology and cultural ecology of Litopenaeus vannamei:A review[J]. Transaction of Oceanology and Limnology,(4):94-100.]
熊建华,赵永贞,高永华,谢达祥,张彬,陈晓汉. 2011. 凡纳滨对虾良种培育研究进展[J]. 南方农业学报,42(5):556-561. [Xiong J H,Zhao Y Z,Gao Y H,Xie D X,Zhang B,Chen X H. 2011. Advances in breeding improved varie-ties of Litopenaeus vannamei[J]. Journal of Southern Agriculture,42(5):556-561.]
杨铭,于洋,张晓军,王全超,刘敬文,李富花,相建海. 2017. 基于转录组数据的凡纳滨对虾微卫星标记开发[J]. 海洋科学,41(2):96-102. [Yang M,Yu Y,Zhang X J,Wang Q C,Liu J W,Li F H,Xiang J H. 2017. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp (Litopenaus vannamei)[J]. Marine Sciences,41(2):96-102.]
张丽颖,张忠旭,郝宪斌,姚继攀. 2009. 微卫星标记技术在水稻抗病育种中的应用[J]. 科技信息,(19):767. [Zhang L Y,Zhang Z X,Hao X B,Yao J P. 2009. Application of microsatellite marker technology in rice disease resistance breeding[J]. Science & Technology Information,(19):767.]
周大颜,张志新,黄彩林,招志杰,莫飞龙. 2019. 3种白鲫杂交子代的转录组学分析[J]. 南方农业学报,50(6):1328-1338. [Zhou D Y,Zhang Z X,Huang C L,Zhao Z J,Mo F L. 2019. Transcriptomic analysis of three Carassius auratus cuvieri hybrids[J]. Journal of Southern Agriculture,50(6):1328-1338.
Antoro S,Na-Nakorn U,Koedprang W. 2006. Study of gene-tic diversity of orange-spotted grouper,Epinephelus coioides,from Thailand and Indonesia using microsatellite markers[J]. Marine Biotechnology(New York),8(1):17-26.
Botstein D,White R L,Skolnick M,Davis R W. 1980. Construction genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics,32(3):314-331.
Crane P A,Lewis C J,Kretschmer E J,Miller S J,Spearman W J,DeCicco A L,Lisac M J,Wenburg J K. 2004. Chara-cterization and inheritance of seven microsatellite loci from Dolly Varden,Salvelinus malma,and cross-species amplification in Arctic char,S. alpinus[J]. Conservation Genetics,5:737-741.
Eid J,Fehr A,Gray J,Luong K,Lyle J,Otto G,Peluso P,Rank D,Baybayan P,Bettman B,Bibillo A,Bjornson K,Chaudhuri B,Christians F,Cicero R,Clark S,Dalal R,Dewinter A,Dixon J,Foquet M,Gaertner A,Hardenbol P,Heiner C,Hester K,Holden D,Kearns G,Kong X, Kuse R,Lacroix Y,Lin S,Lundquist P,Ma C,Marks P,Maxham M,Murphy D,Park I,Pham T,Phillips M,Roy J,Sebra R,Shen G,Sorenson J,Tomaney A,Travers K,Trulson M,Vieceli J,Wegener J,Wu D,Yang A,Zaccarin D,Zhao P,Zhong F,Korlach J,Turner S. 2009. Real-time DNA sequencing from single polymerase molecules[J]. Science,323(5910):133-138.
Garcia D K,Dhar A K,Alcivar-Warren A. 1996. Molecular analysis of a RAPD marker (B20) reveals two microsa-tellites and differential mRNA expression in Penaeus vannamei[J]. Molecular Marine Biology and Biotechnology,5(1):71-83.
Gyapay G,Morissette J,Vignal A,Dib C,Fizames C,Millas-seau P,Marc S,Bernardi G,Lathrop M,Weissenbach J. 1994. The 1993-94 Généthon human genetic linkage map[J]. Nature Genetics,7(2):246-339.
Lightner D V. 2011. Virus diseases of farmed shrimp in the Western Hemisphere (the Americas):A review[J]. Journal of Invertebrate Pathology,106(1):110-130.
Lu X,Kong J,Meng X H,Cao B X,Luo K,Dai P,Luan S. 2018. Identification of SNP markers associated with to-lerance to ammonia toxicity by selective genotyping from de novo assembled transcriptome in Litopenaeus vannamei[J]. Fish & Shellfish Immunology,73:158-166.
Meehan D,Xu Z,Zuniga G,Alcivar-Warren A. 2003. High frequency and large number of polymorphic microsate-llites in cultured shrimp,Penaeus (Litopenaeus) vannamei[Crustacea:Decapoda][J]. Marine Biotechnology(New York),5(4):311-330.
Roberts R J,Carneiro M O,Schatz M C. 2013. The advanta-ges of SMRT sequencing[J]. Genome Biology,14(7):405.
Shikano T,Taniguchi N. 2002. Relationships between genetic variation measured by microsatellite DNA markers and a fitness-related trait in the guppy(Poecilia reticulata)[J]. Aquaculture,209(1):77-90.
Santos C A,Andrade S C S,Freitas P D. 2018. Identification of SNPs potentially related to immune responses and growth performance in Litopenaeus vannamei by RNA-seq analyses[J]. PeerJ,6:e5154. doi:10.7717/peerj.5154.
Wajid A,Wasim M,Yaqub T,Firyal S,Tayyab M,Siddique S,Hussain T. 2014. Assessment of genetic diversity in Balochi and Rakhshani sheep breeds of Balochistan using microsatellite DNA markers[J]. The Journal of Animal & Plant Sciences,24(5):1348-1354.
Yu Y,Wei J K,Zhang X J,Liu J W,Liu C Z,Li F H,Xiang J H. 2014. SNP discovery in the transcriptome of white Pacific shrimp Litopenaeus vannamei by next generation sequencing[J]. PLoS One,9(1):e87218.

