不同温度下TMV侵染枯斑三生烟的lncRNA差异表达
贾海燕,宋丽云,徐翔,解屹,张超群,刘天波,赵存孝,申莉莉, 王杰,李莹,王凤龙,杨金广
不同温度下TMV侵染枯斑三生烟的lncRNA差异表达
贾海燕1,宋丽云1,徐翔1,解屹1,张超群2,刘天波3,赵存孝4,申莉莉1, 王杰1,李莹1,王凤龙1,杨金广1
(1中国农业科学院烟草研究所/烟草行业病虫害监测与综合治理重点开放实验室,山东青岛 266101;2江西省烟叶科学研究所,南昌 330029;3中国烟草总公司中南农业试验站,长沙 410128;4甘肃省烟草公司庆阳市公司,甘肃庆阳 745099)
【】筛选不同温度下烟草花叶病毒(,TMV)侵染后枯斑三生烟(var. Samsun NN)差异表达的长链非编码RNA(long non-coding RNA,lncRNA),研究lncRNA在枯斑三生烟抗性反应中的作用。N基因的温度敏感性使枯斑三生烟在25℃时具备对TMV的抗性、在31℃抗性丧失,在这两个温度条件下对枯斑三生烟接种TMV和磷酸盐缓冲盐水(phosphate buffered saline,PBS),48 h后提取系统叶总RNA,构建链特异性文库后进行深度测序。对测序结果进行过滤后利用HTSeq将有效数据与近缘品种TN90(var. TN90)基因组比对,筛选得到lncRNA后利用FPKM法估计lncRNA的表达水平。通过edgeR筛选差异表达lncRNA(differentially expressed lncRNA,DElncRNA),并利用qRT-PCR技术对这一结果进行验证。通过共定位及共表达分析预测DElncRNA的靶基因,通过参考基因组注释、GO和KEGG富集分析研究靶基因的功能。4个处理共12个样本经lncRNA-seq各测得约8 000万条clean reads,共获得4 737条已知lncRNA、40 169条新lncRNA。其中64个lncRNA在不同温度条件下TMV侵染后存在差异表达,qRT-PCR测定结果显示这些lncRNA的测序正确率在80%左右,表明本研究所得测序数据具备较高的可信度。对DElncRNA进行靶基因预测,发现一些基因同时被25℃下调和31℃上调的DElncRNA靶向。靶基因注释功能丰富,主要参与植物抗病、激素和代谢等生理过程。部分可能与激素通路相关的lncRNA,在25℃下TMV侵染时呈现下调趋势,而在31℃下TMV侵染则呈现上调趋势。GO富集分析显示靶基因主要参与构成膜、囊泡等组分,具备钙、钾离子通道抑制剂活性等分子功能,使相应离子得以转运引发随后的反应,同时也参与发病、抗原加工和呈现、细胞分裂素代谢等生理过程。KEGG分析发现靶基因显著富集在植物激素信号转导通路,25℃下调和31℃上调的DElncRNA靶基因同时富集在激素信号传导、ABC运输蛋白、苯丙烷类生物合成等通路。不同温度(25℃和31℃)条件下TMV侵染枯斑三生烟后,长链非编码RNA差异表达,DElncRNA通过作用于激素信号传导、物质转运等过程参与寄主系统获得性抗性反应。研究结果可为揭示植物系统获得性抗性中lncRNA的调控功能以及新型抗病毒技术开发提供依据。
烟草花叶病毒;N基因;枯斑三生烟;长链非编码RNA;系统获得性抗性;深度测序;基因富集分析
0 引言
【研究意义】烟草花叶病毒(,TMV)对主要经济作物生产造成极大危害且难以防治,来源于野生种黏烟草()的N基因对TMV具有抗性[1]。研究长链非编码RNA(long non-coding RNA,lncRNA)在-TMV互作引起的枯斑三生烟(var. Samsun NN)抗性反应中的作用,揭示lncRNA在植物系统获得性抗性中的调控功能,可为新型抗病毒技术开发打下基础。【前人研究进展】非编码RNA即不编码蛋白质的RNA,在RNA水平上行使调控功能。LncRNA是长度超过200 nt的非编码RNA,按其与蛋白质编码基因在基因组上的位置,分为基因间长链非编码RNA(long intergenetic ncRNA,lincRNA)、内含子长链非编码RNA(intronic long ncRNA,incRNA)、反义长非编码RNA(antisense lncRNA)等[2]。LncRNA具有同源性较低、组织特异性高等特点[3]。N基因介导的TMV抗性具备温度敏感性,环境温度低于28℃时含有N基因的烟草在TMV侵染后发生过敏反应(hypersensitive reaction,HR)、建立系统获得性抗性(systemic acquired resistance,SAR),环境温度高于28℃时,N基因对TMV的抗性丧失,TMV在植株体内系统侵染[4-5]。SAR的建立依赖多种激素相互协作或拮抗[6-8],进而引发一系列信号转导级联反应[9-10],其建立标志是病程相关(pathogenicity-related,PR)基因的大量表达[11]。肖万福对感染TMV后烟草差异表达的基因与蛋白质展开研究,发现它们主要富集在光合作用、能量代谢、植物免疫以及与病原物互作等途径[12]。与此同时非编码RNA的调控作用已得到诸多揭示,最近的研究表明许多lncRNA与植物同病原物的互作相关。番茄黄化曲叶病毒(,TYLCV)侵染易感番茄后,2 056个lncRNA参与到植株内的激素信号传导、内质网中的蛋白质加工和植物-病原物互作等多种生物过程中;利用病毒诱导的基因沉默技术降低lncRNA S-slylnc0957的表达后,易感番茄增强了对TYLCV的抗性[13]。此外,寄主lncRNA与病毒产生的小干扰RNA(viral small- interfering RNA,vsRNA)也可能发生相互作用。番茄长链非编码RNA SlLNR1可以通过与TYLCV产生的vsRNA互作来控制病害发展。VsRNA可以诱导易感番茄的沉默,表达下调时番茄叶片呈现发育迟缓和卷曲,而的过表达使TYLCV积累减少[14]。Seo等研究发现,敲除拟南芥长链非编码RNA后表达降低,对丁香假单胞菌(pv.)入侵更敏感;过表达的植物表达也升高,对丁香假单胞菌产生抗性[15]。进一步研究揭示可以解离FIB2/MED19a复合物,从启动子上的激活因子(MED19a)中释放阻遏物FIB2,最终导致的表达增加[16]。LncRNA在寄主植物与病原物互作中发挥着不可或缺的作用,但对于lncRNA在烟草响应病原物胁迫中的作用少有揭示。【本研究切入点】-TMV互作引发寄主抗性反应,此过程中差异表达基因和蛋白的功能已有研究,而关于lncRNA在其中的作用鲜见报道。【拟解决的关键问题】利用长链非编码核糖核酸测序(long non-coding RNA sequencing,lncRNA-seq)技术,分析不同温度(25℃和31℃)TMV侵染后枯斑三生烟系统叶lncRNA的表达情况。确定差异表达lncRNA,注释其功能及富集的相关途径,通过对比预测在抗性反应中起作用的lncRNA及作用途径,探究lncRNA在植物系统获得性抗性中的调控功能。
1 材料与方法
试验于2018年在中国农业科学院烟草研究所烟草行业病虫害监测与综合治理重点开放实验室完成。
1.1 样品准备
供试烟草品种枯斑三生烟,由中国烟草种质资源平台提供;烟草花叶病毒为普通株系(TMV-C),由中国农业科学院烟草研究所植物保护研究中心提供。三生烟生长在昼夜周期为16 h/8 h、25℃或31℃、光合有效辐射为100 μmol·m-2·s-1和相对湿度约为80%的人工气候室中。
1.2 样品处理
以磷酸盐缓冲盐水(phosphate buffered saline,PBS)处理为对照,10 mg·mL-1TMV接种六叶期枯斑三生烟上部第3叶片(图1)。设置4个处理,每个处理包含3个生物学重复,其中处理Ⅰ:TMV+25℃;处理Ⅱ:TMV+31℃;处理Ⅲ:PBS+25℃;处理Ⅳ:PBS+31℃;TMV和PBS处理48 h后,分别将12个样本的上部第2叶片送于北京诺禾致源科技股份有限公司进行lncRNA-seq。
1.3 文库制备与高通量测序
利用TRIzol regent(Invitrogen,美国)对4个处理的12个样品进行总RNA提取,分析样品RNA完整性及是否存在DNA污染,每个样本使用3 μg左右RNA,除去rRNA(ribosome RNA)后逆转录合成cDNA第一条链。链特异性文库与普通建库方法的区别在于将第二链cDNA合成反应缓冲液中的dTTP替换为dUTP,对cDNA末端进行修饰后,利用USER(Uracil-Specific Excision Reagent)酶(New England Biolabs,英国)降解含U的cDNA第二链,从而保留第一条cDNA信息进行PCR扩增并获得文库。LncRNA的来源具备链特异性[3],通过与参考基因组比对,链特异性建库可以明确lncRNA正负链来源,获得的基因信息更准确。
使用Illumina NovaSeq6000 sequencer(Illumina,美国)按读取长度150 bp进行测序,获得原始序列数据(raw reads)后,经测序错误率分布检查、测序数据过滤,得到有效读段(clean reads)用于随后的比对分析。将测序数据上传至NCBI SRA数据库,BioProject号:PRJNA588726。
1.4 序列比对与拼接
由于枯斑三生烟未进行基因组测序,而近缘品种TN90的基因组信息与枯斑三生烟测序数据比对率高、符合要求,故将其作为参考基因组。参考基因组和基因模型注释文件下载自NCBI(ftp://ftp.ncbi.nlm.nih. gov/genomes/all/GCF/000/715/135/GCF_000715135.1_ Ntab-TN90/)。为得到样品各类型基因的表达情况,利用HTSeq(V2.0.4)将clean reads与参考基因组比对、覆盖度分析已知类型基因[17]。使用StringTie(V1.3.1)将各样本比对到参考基因组的reads拼接成转录本[18]。

A:25℃条件下TMV接种前的枯斑三生烟Samsun NN before TMV inoculation at 25℃;B:25℃条件下TMV接种48 h后的枯斑三生烟Samsun NN after 48 hours of TMV inoculation at 25℃;C:31℃条件下TMV接种前的枯斑三生烟Samsun NN before inoculation of TMV at 31℃;D:31℃条件下TMV接种48 h后的枯斑三生烟Samsun NN after 48 hours of TMV inoculation at 31℃。标注为小写字母的图片展示相应枯斑三生烟的接种叶The pictures marked in lowercase letters show the inoculated leaves of the corresponding tobacco
1.5 LncRNA的识别与筛选
利用Cuffmerge(V2.0.2)对拼接得到的转录本进行合并,去除其中链方向不确定的转录本后得到完整转录组信息。从转录组中筛选出外显子数量≥1且长度>200 bp的转录本,用于已知lncRNA(annotated lncRNA,即与数据库注释exon区域有重叠的转录本)的识别和新lncRNA(novel lncRNA)的筛选。
利用Cuffcompare将这些转录本与数据库比对,得到已知lncRNA。对剩余转录本,通过Cuffquant筛选出FPKM(expected number of Fragments Per Kilobase of transcript sequence per Millions base pairs sequenced,每百万fragments中来自某一基因每千碱基长度的fragments数目)≥2的转录本后[19],利用CNCI(coding-non-coding index,V2.0.0)、CPC2(coding potential calculator)、Pfam-scan(V1.3.0)预测其中没有编码潜能的转录本[20-22],取3种工具预测结果的交集作为新lncRNA,而至少被一种软件预测能编码蛋白的转录本作为TUCP(transcripts of uncertain coding potential,编码潜力不确定的转录本)[23]。
1.6 差异表达lncRNA的筛选和聚类分析
通过计算某一lncRNA在不同处理后的FPKM值是否存在差异,判定该lncRNA是否为差异表达lncRNA(differentially-expressed lncRNA,DElncRNA)。通过 StringTie-eB(V1.3.1)定量lncRNA在12个样本中的FPKM值[18],对同一处理的3个重复样本取均值得到lncRNA在该处理下的FPKM信息。
筛选枯斑三生烟在25℃环境中差异表达的lncRNA时,将处理Ⅰ设为试验组、将处理Ⅲ设为对照组(TMV_25_vs_PBS_25),利用edgeR(V3.0.8)[24]筛选DElncRNA:当某一lncRNA对应的|log2(TMV_25_FPKM / PBS_25_FPKM)|>2、且 校正后的值(-adjust)<0.05时,认为该lncRNA在25℃、TMV处理后差异表达。31℃条件下DElncRNA的筛选依据同样方法,以处理Ⅱ为试验组、以处理Ⅳ为对照组(TMV_31_vs_PBS_31)。
为判断不同处理DElncRNA的表达模式,根据DElncRNA在4个处理中的FPKM值,进行表达量层次聚类(hierarchical clustering)分析。
1.7 DElncRNA的实时荧光定量PCR(qRT-PCR)验证
使用Primer Premier(V6.0.0)对各处理DElncRNA进行荧光定量引物设计(表1、表2)。使用ChamQ Universal SYBR qPCR Master Mix(Vazyme,中国)实时荧光定量试剂盒,利用Applied Biosystems 7500 Fast Real-Time PCR System(ABI,美国)进行qRT-PCR反应。总反应体系为20 μL:SYBR qPCR Master Mix 10 μL,PCR正反向引物各0.4 μL,作为模板的逆转录所得cDNA 2 μL,RNase-free water 7.2 μL。扩增条件为95℃预变性30 s;95℃反应10 s和60℃反应30 s,循环40次;95℃反应15 s和60℃反应1 min,95℃反应15 s分析融解曲线。依据融解曲线分析引物的特异性。根据红花烟草肌动蛋白()序列(序列号:GQ339768),设计荧光定量引物(正向引物:5′-TGAGACATTCAACGTTCCGGC-3′,反向引物:5′-GTTAGGTCACGGCCAGCAAG-3′)。以为内参,根据2-ΔΔCt法[25]计算基因相对定量的表达,借助SPSS(V24.0.0)进行数据分析,利用Excel 2016绘制图表,得到DElncRNA表达情况,对测序结果进行验证。
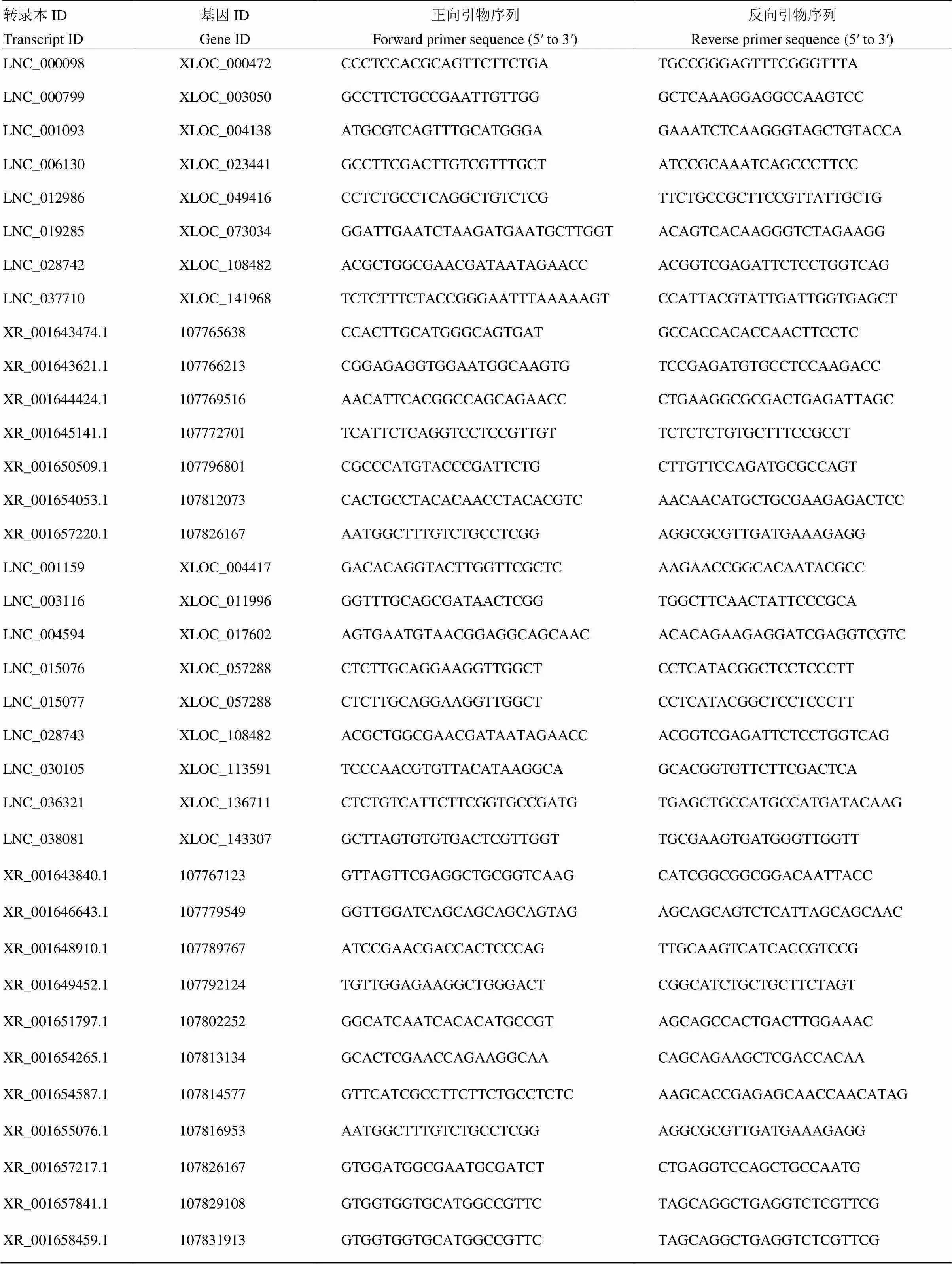
表1 25℃下差异表达lncRNA引物序列
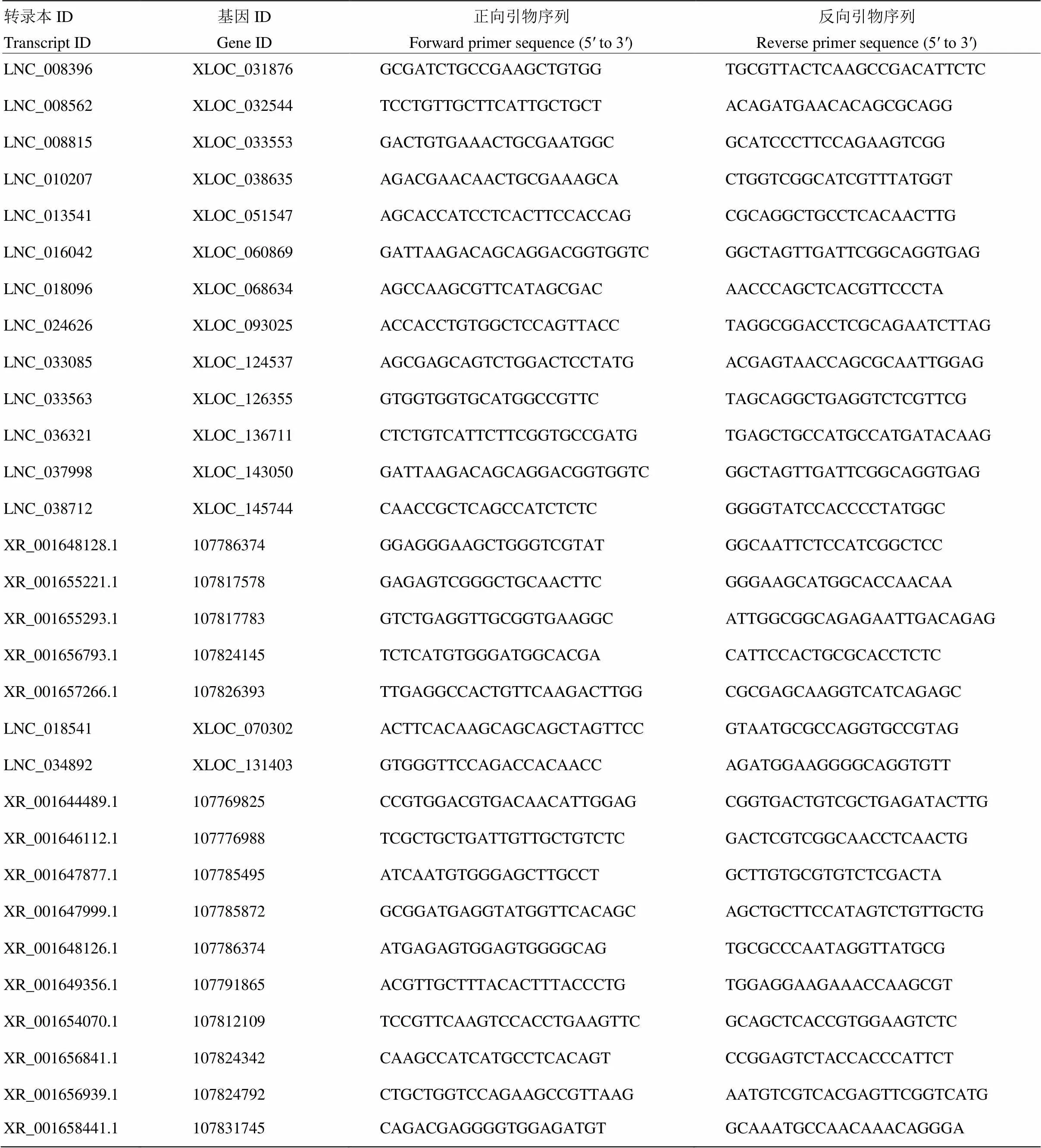
表2 31℃下差异表达lncRNA引物序列
1.8 DELncRNA靶基因预测及注释
LncRNA的调控方式分为作用于相邻靶基因的顺式作用模式,以及在其表达水平上识别其他基因的反式作用模式[26]。DElncRNA顺式作用靶基因通过共定位(co-location)分析预测得到,筛选条件为mRNA位于lncRNA上下游100 kb以内。通过共表达(co-expression)分析预测反式作用靶基因[27],采用Pearson相关系数法研究DElncRNA与mRNA的相关性,相关系数绝对值>0.95(<0.05)时作为反式作用靶基因。研究参考基因组对靶基因的注释,分析其生物学功能。
1.9 GO与KEGG富集分析
对DElncRNA的靶基因分别进行GO(Gene Ontology,基因本体论)和KEGG(Kyoto Encyclopedia of Genes and Genomes,京都基因和基因组百科全书)途经分析,GO富集分析通过GOseq(V3.3.2)实现[28],使用KOBAS(V2.0.0)进行KEGG途径分析[29-30],当<0.05时认为鉴定的GO条目或者KEGG途径被显著富集。
2 结果
2.1 不同温度下TMV侵染的枯斑三生烟系统叶lncRNA测序概述
4个处理12个样本获得clean reads均为8 000万条左右(表3),碱基测序错误率(error rate)最高为0.02%。与参考基因组(total mapped)比对发现,95%以上的clean reads被比对到参考基因组,约46%在参考序列上有多个比对位置(multiple mapped),有唯一比对位置的clean reads(uniquely mapped)占49%左右。
为研究样本基因表达情况,进行已知类型基因定量分析,约36%的clean reads为protein_coding,lncRNA约占1.4%。经过筛选共获得4 737条已知lncRNA,40 169条新lncRNA;其中lincRNA占47.7%,antisense lncRNA较少,为14.8%,剩余37.5%的lncRNA为intronic lncRNA。
密度分布显示已知lncRNA、新lncRNA及mRNA之间存在一定差异。如图2-A所示,已知lncRNA、新lncRNA相对mRNA长度偏短,已知lncRNA长度集中在600 bp,而新lncRNA在300 bp左右。多数新lncRNA有2个外显子,而已知lncRNA多有3个外显子(图2-B)。两种lncRNA的开放阅读框(open reading frame,ORF)长度都在250 bp以下,新lncRNA相对更短(图2-C)。
3种转录本表达水平差异明显(图2-D),mRNA表达量显著高于TUCP和lncRNA,lncRNA较TUCP更低。对同一处理的重复样品取平均值获得该处理FPKM,图2-E显示4种处理表达水平稍有不同,31℃两个处理转录本表达量高于25℃的两个处理,同一温度条件下TMV和PBS处理间基因表达水平差异不大。
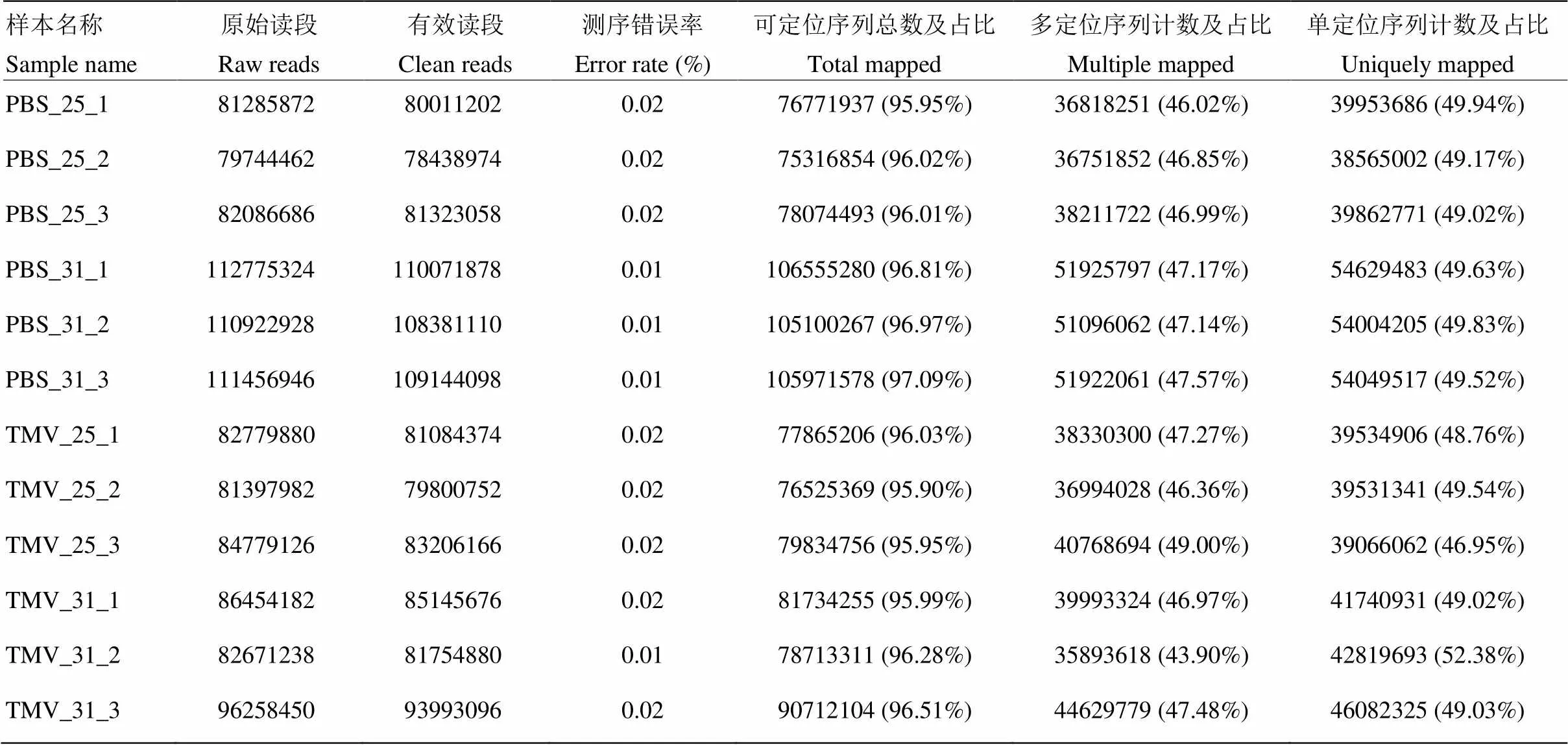
表3 测序数据概览
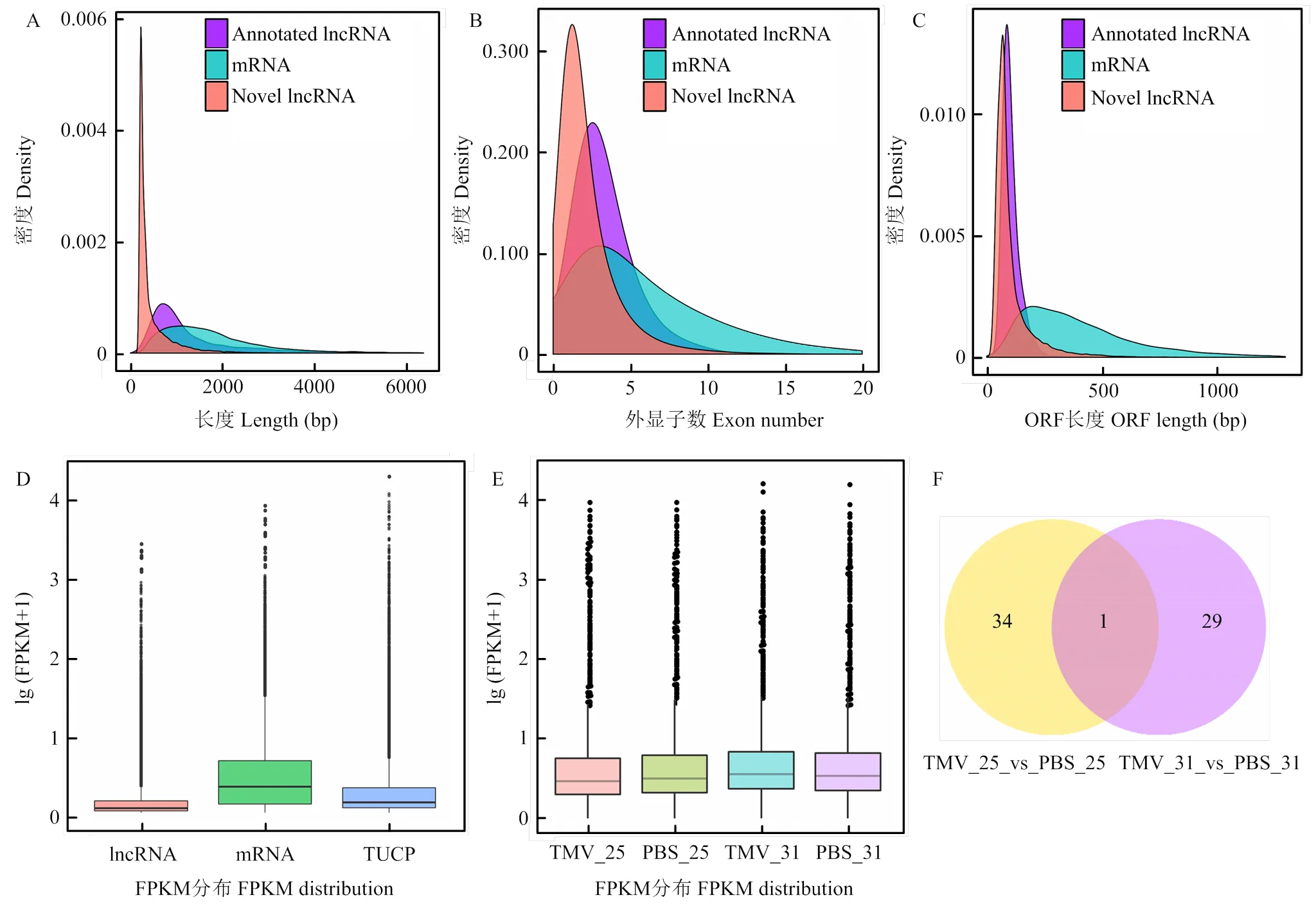
A:lncRNA和mRNA序列长度的密度分布density distribution of sequence lengths of lncRNA and mRNA;B:lncRNA和mRNA外显子个数的密度分布density distribution of exon number of lncRNA and mRNA;C:lncRNA和mRNA ORF长度的密度分布density distribution of ORF length of lncRNA and mRNA;D:不同类型转录本表达水平比较comparison of expression levels of different types of transcripts;E:不同处理转录本表达水平比较comparison of expression levels of different treatments of transcripts;F:25℃和31℃比较组中差异表达lncRNA数目统计The numbers of differentially expressed lncRNAs in the comparison groups of 25℃ and 31℃
2.2 DElncRNA的筛选
为确定系统获得性抗性中相关的lncRNA,以25℃和31℃两个温度条件下PBS处理的枯斑三生烟系统叶为对照,筛选TMV处理后差异表达lncRNA,共测得64个DElncRNA(图2-F)。25℃下TMV处理后15个lncRNA上调表达,20个lncRNA下调表达(TMV_25_vs_PBS_25);18个lncRNA在31℃及TMV侵染的情况下表达上调,12个lncRNA下调(TMV_31_vs_PBS_31)。其中LNC_036321在25℃条件下接种TMV后表达下调,在31℃表达上调。
结合差异表达lncRNA整体分布情况(图3-A、3-B),DElncRNA的表达在不同处理下存在差异。聚类分析(图3-C)将表达模式相同或相近的lncRNA聚成一组,它们可能具有相同功能或参与同一生物学过程。
2.3 DElncRNA的表达验证
为验证lncRNA-seq结果的可靠性,利用qRT-PCR对不同温度(25℃和31℃)TMV接种后三生烟的DElncRNA进行相对表达量测定。如图4-A所示,25℃的15个uDElncRNA(up-regulated DElncRNA)中有11个上调表达,其中LNC_000799、XR_001643621.1显著高表达(<0.05)。对25℃的20个dDElncRNA(down-regulated DElncRNA)定量发现(图4-B)LNC_004594、LNC_038081、XR_001651797.1、XR_001657217.1表达显著下调,共17个与测序结果一致。18个31℃的uDElncRNA中14个表达上调(图5-A),LNC_016042、LNC_024626、LNC_033085、XR_001648128.1显著上调。定量验证的31℃ 12个dDElncRNA中10个lncRNA与lncRNA-seq结果一致(图5-B),XR_001647999.1、XR_001656939.1、XR_001658441.1表达显著降低。80%的DElncRNA的定量结果与lncRNA-seq结果一致,对于不一致的定量结果,除了表达量过低lncRNA(FPKM<1)的测序错误率较高外,与引物设置不合理或者相反链上的基因转录干扰结果也有一定关系[31-32]。
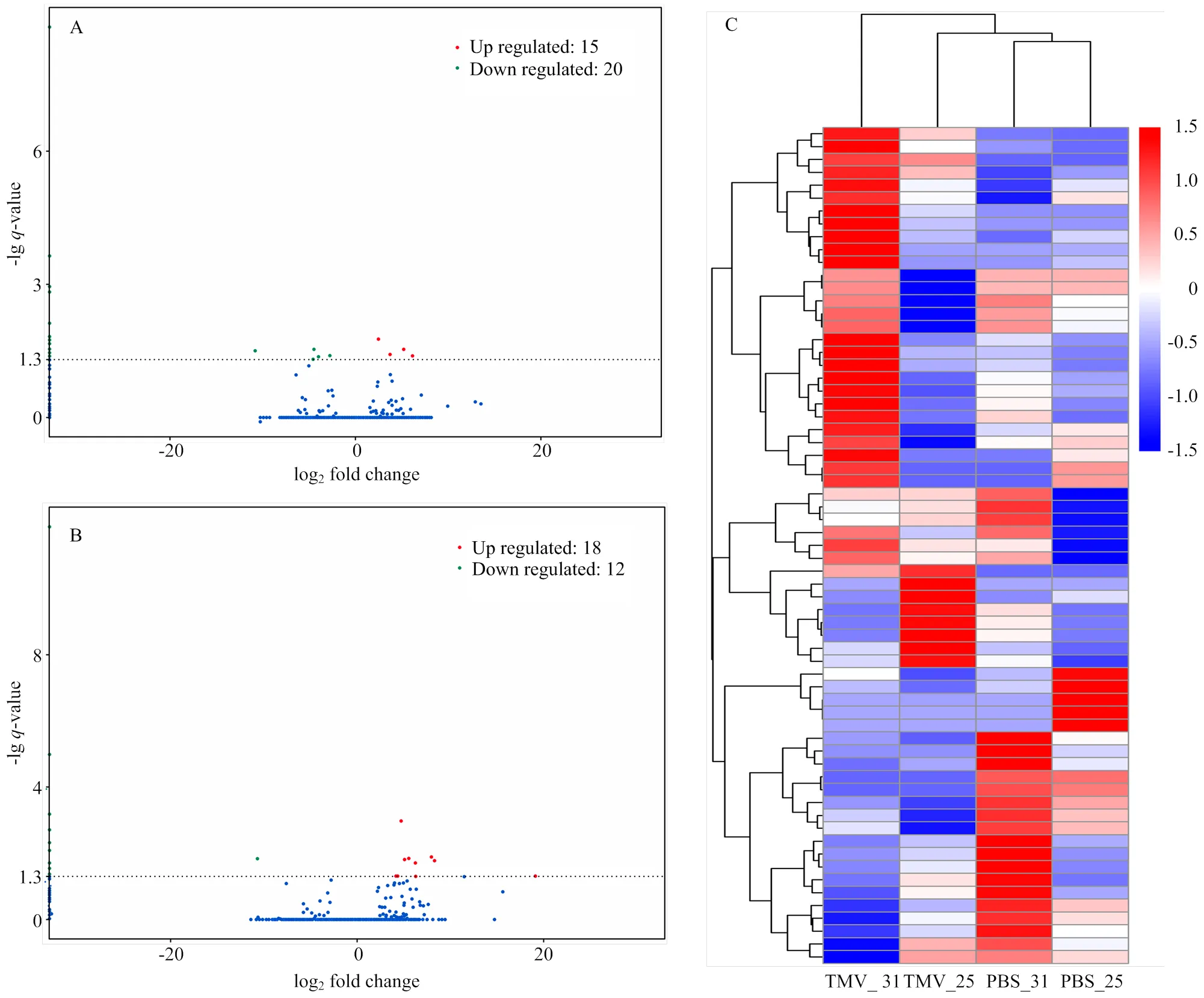
A:25℃差异表达lncRNA火山图,横坐标代表转录本在不同样本中表达倍数变化,纵坐标代表转录本表达量变化差异的统计学显著性,q-value即p-adjust;图中红色点表示有显著性差异表达的上调转录本,蓝色点表示有显著性差异表达的下调转录本volcano plot of differentially expressed lncRNAs at 25℃. The abscissa represents expression fold change in different samples, the ordinate represents the statistical significance of difference in transcript expression, and the q-value is p-adjust. The red dot in the figure indicates the up-regulated transcript with significant differential expression, and the blue dot indicates the down-regulated transcript with significant differential expression;B:31℃差异表达lncRNA火山图volcano plot of differentially expressed lncRNAs at 31℃。C:差异表达lncRNA表达量层次聚类,以 lg(FPKM+1) 值进行聚类,红色代表基因高表达,蓝色表示低表达基因,颜色从红到蓝表示lg(FPKM+1)从大到小DElncRNA expression level hierarchical clustering, clustering was performed with lg(FPKM+1) values, with red representing high expression of genes and blue indicating low expressed genes, colors from red to blue indicate lg(FPKM+1) from large to small
2.4 DElncRNA靶基因预测
通过共定位和共表达分析预测DElncRNA靶基因,lncRNA可能有多个靶基因,同一基因也可能被不同lncRNA靶向。预测得到的DElncRNA靶基因注释功能丰富,除了与光合作用、植物抗性相关外,还与多种植物激素通路密切联系,也作用于TMV侵染、寄主代谢过程等。例如LNC_037710的靶基因LOC107811086,预测编码抗病性蛋白RPP13(resistance pathogen protein 13);XR_001658459.1靶向的LOC107761822,预测编码丝裂原活化蛋白激酶2(mitogen-activated protein kinase 2)[33];XR_001648910.1的靶基因LOC107789767预测编码过氧化物酶9(peroxidase 9)。LNC_006130靶向的LOC107793779预测编码TMV抗性蛋白N(TMV resistance protein N),另一个靶基因LOC107821288预测编码烟草花叶病毒增殖蛋白3(tobamovirus multiplication protein 3)。
值得关注的是,部分可能与激素通路相关的lncRNA在25℃和TMV侵染条件下呈现下调趋势,而在31℃和TMV侵染条件下呈现上调趋势。例如XR_001648910.1(TMV_25_vs_PBS_25)和LNC_008815(TMV_31_vs_PBS_31)同时靶向的LOC107800521,预测编码乙烯响应转录因子ERF003(ethylene-responsive transcription factor ERF003)。两个温度lncRNA的差异表达可能引起靶蛋白相反的积累,进而在25℃的植物抗性建立中发挥作用,而在31℃丧失对TMV的抗性,后续研究中需进一步验证靶基因在不同处理中的表达变化。
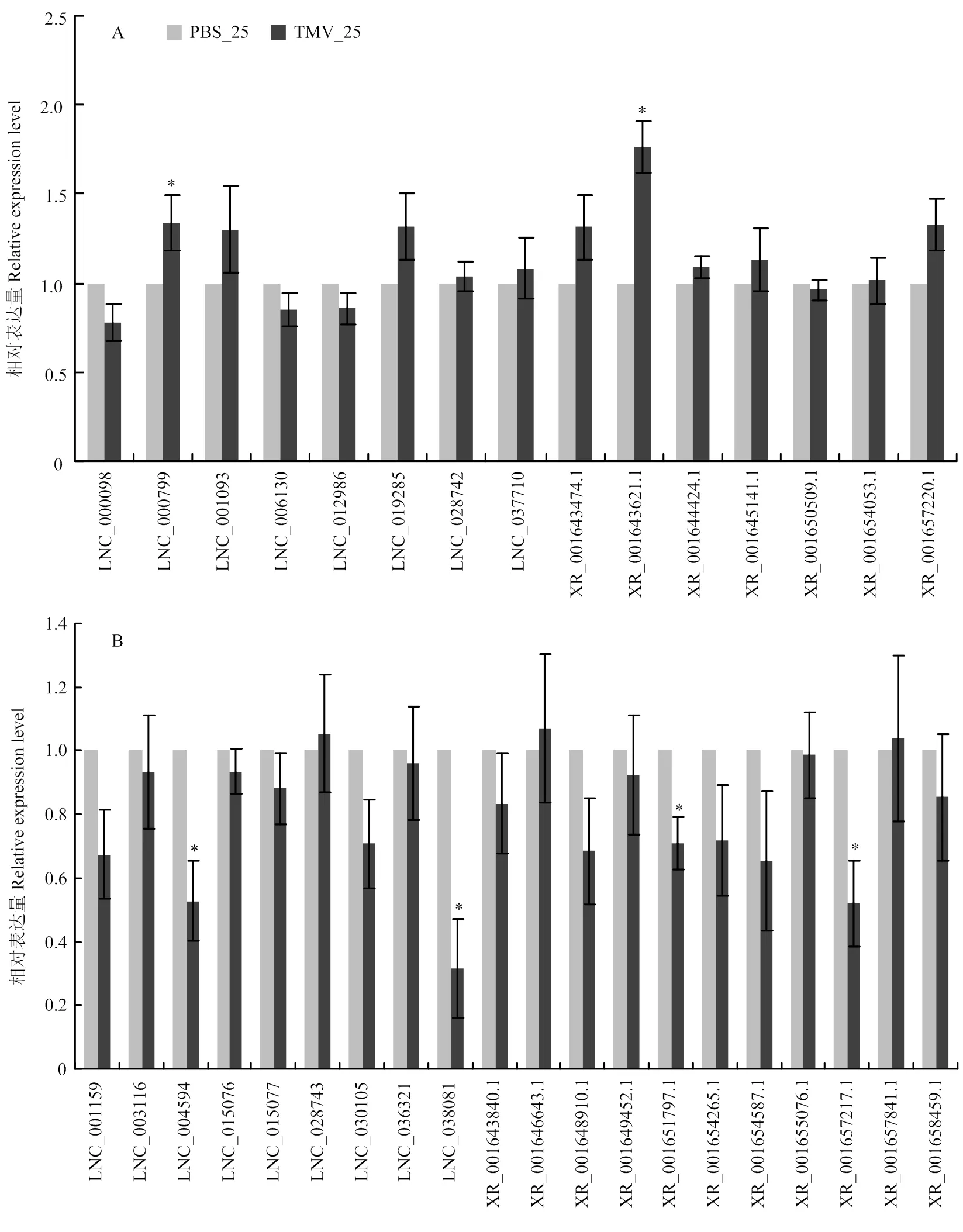
A:25℃和TMV处理下uDElncRNA的相对表达量relative expression of up-regulated DElncRNAs after TMV treatment at 25℃;B:25℃和TMV处理下dDElncRNA的相对表达量relative expression of down-regulated DElncRNAs after TMV treatment at 25℃
误差线表示3组生物学重复的正负标准差,采用检验分析差异显著性Error bars indicate the ±SD of three biological replicates. Student’s-test.
*:<0.1; **:<0.01; ***:<0.001。图5同 The same as Fig. 5
图4 25℃条件下差异表达lncRNA 的qRT-PCR验证
Fig. 4 Validation of differentially expressed lncRNAs at 25℃by qRT-PCR
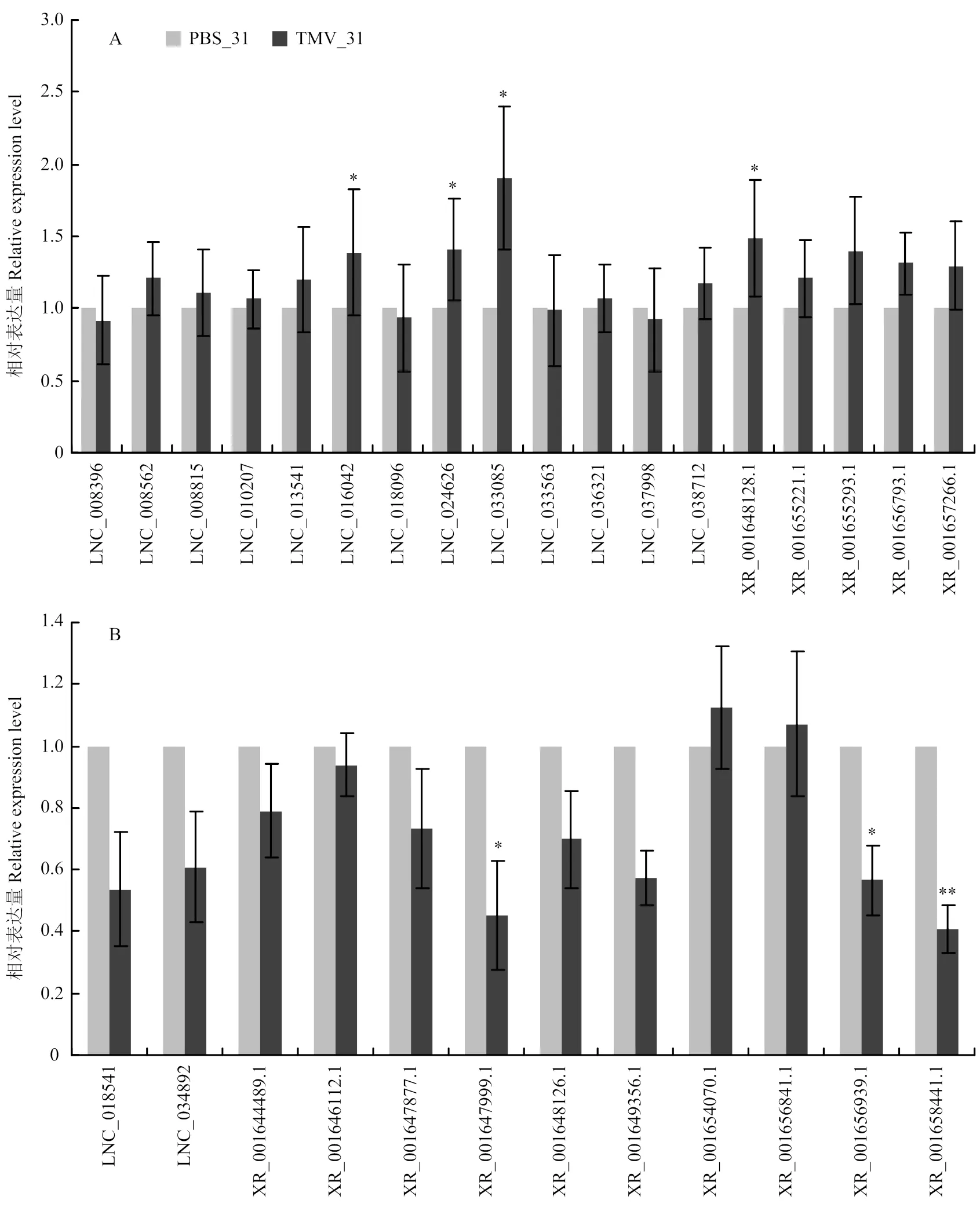
A:31℃和TMV处理下uDElncRNA的相对表达量relative expression of up-regulated DElncRNAs after TMV treatment at 31℃;B:31℃和TMV处理下dDElncRNA的相对表达量relative expression of down-regulated DElncRNAs after TMV treatment at 31℃
2.5 DElncRNA靶基因富集分析
如表4所示,为进一步研究差异表达lncRNA的功能,对其靶基因进行了GO富集分析(<0.05)。结果显示,膜结合的细胞器(GO:0043227)、质膜部分(GO:0044459)等细胞组分被富集,表明膜组织在枯斑三生烟抗病毒应激反应中具有重要作用。分子功能方面,一些靶基因具备转移酶活性、离子通道抑制剂活性等分子功能,使相应物质得以转运,参与后续应激过程。靶基因也参与到化合物代谢、抗原加工代谢等过程。
25℃条件下DElncRNA靶基因富集在色素沉积(GO:0043473)、参与凋亡过程的半胱氨酸型内肽酶活性激活(GO:0006919)等过程,可能与HR形成有关;同时这些靶基因参与蛋白质解聚(GO:0051261)、细胞骨架组织的负调节(GO:0051494)等过程,使细胞膜透性增加、信号分子得以释放。25℃下DElncRNA靶向的基因参与对生长素刺激的反应(GO:0009733),而细胞分裂素代谢过程(GO:00096909)被31℃的靶基因富集。LOC107764060预测被31℃条件下差异表达的lncRNA靶向,其富集在肌醇磷酸代谢过程(GO: 0043647)参与信号转导[34]。两个温度uDElncRNA靶基因都参与到铁载体的合成(GO:0019290)、转运(GO:0015343)、代谢(GO:0009237)等过程,表明铁载体可能在植物应激过程中发挥作用[35]。
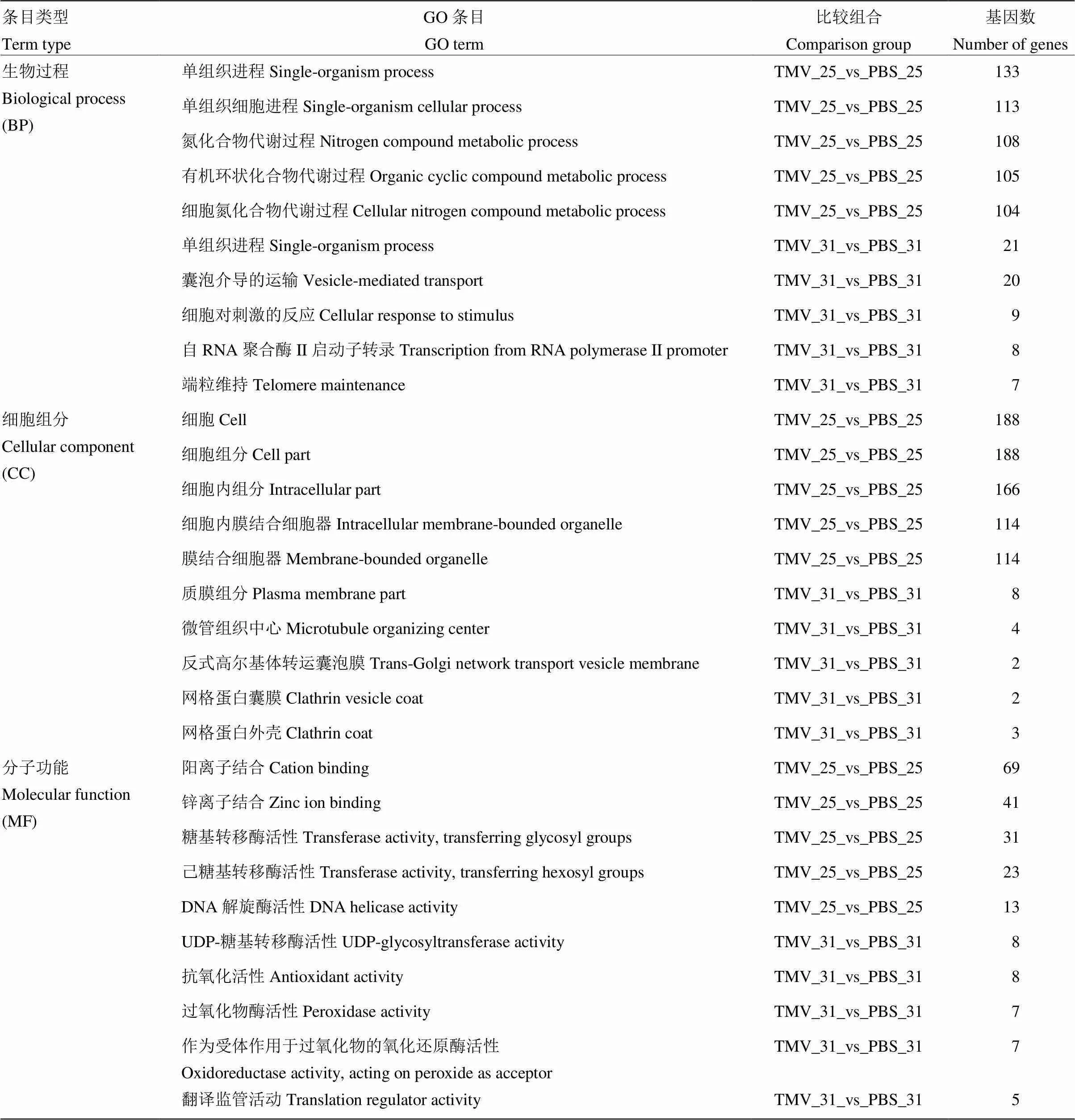
表4 GO条目富集的 DElncRNA靶基因数目
KEGG分析(表5)发现,除22条25℃ dDElncRNA靶基因极显著富集(-adjust<0.05)到植物激素信号传导通路(sly04075)外,31℃ uDElncRNA的15条靶基因也富集(<0.05)到该通路;分析发现除赤霉素外,水杨酸、乙烯、茉莉酸等激素信号通路均得到富集。进一步研究发现植物病原PAMP(pathogen- associated molecular patterns,病原体相关分子模式)触发免疫通路上的CDPK(sly:101261579)节点,同时被这两个分组lncRNA靶向的基因富集,引起HR反应和细胞壁加固。基于25℃ dDElncRNA和31℃ uDElncRNA存在较多相同靶基因,对比分析发现它们共同富集在ABC运输蛋白,缬氨酸、亮氨酸和异亮氨酸降解,苯丙烷类生物合成,以及mRNA监测途径、嘧啶代谢通路。此外,DElncRNA靶基因也参与到二萜生物合成(sly00904)、磷脂酰肌醇信号系统(sly04070)、水泡运输中的SNARE相互作用(sly04130)、酮体的合成和降解(sly00072)等过程。

表5 KEGG 通路富集的DElncRNA靶基因数目比较
3 讨论
LncRNA已被证明参与植物对病毒、细菌、真菌、根结线虫[36-39]等多种病原物的抗性反应,作用于植物激素信号转导、系统抗性诱导、过敏反应等多个过程。系统获得性抗性使植物对多种病原物的入侵具备广谱抗性,是系统获得性抗性的标志基因[11],一定程度上的表达变化揭示系统获得性抗性的建立过程[40]。珊西烟(cv. Xanthi NN)为枯斑三生烟的近缘品种,接种TMV后,珊西烟系统叶SA含量在48 hpi(hours post infection)开始增加、PR1a蛋白在72 hpi开始积累[41]。基于N基因的温度敏感性选定25℃和31℃两个温度条件,在TMV侵染枯斑三生烟48 h后,对其系统叶进行长链非编码RNA测序。
LncRNA-seq发现4 737条已知lncRNA、40 169条新lncRNA表达,其中lincRNA和antisense lncRNA占多数。64个lncRNA在不同温度(25℃和31℃)TMV处理后差异表达,qRT-PCR显示这些DElncRNA上调/下调情况与高通量测序结果基本一致。本研究发现的DElncRNA数目相对于测得的新lncRNA总量而言较少,可能与此时抗性反应较弱有关。DElncRNA在4种处理下的表达模式不同,25℃ TMV处理后15个lncRNA上调表达,20个lncRNA下调表达;18个lncRNA在31℃ TMV处理后高表达,同时12个lncRNA表达降低。对比较得到的DElncRNA顺式作用和反式作用的靶基因展开研究,预测它们编码抗性蛋白、激素信号通路相关蛋白等。部分基因被25℃差异下调lncRNA和31℃差异上调lncRNA同时靶向。考虑到两个温度下枯斑三生烟抗性的差异,认为这些靶基因通过相应lncRNA的调控在烟草抗性反应中发挥作用。靶基因富集的GO条目符合植物感知到病原入侵而启动的一系列应激过程,包括启动离子转运、细胞膜透性改变、激素合成转运等[42]。KEGG途径分析显示两组靶基因都富集到植物激素信号传导,ABC运输蛋白,缬氨酸、亮氨酸和异亮氨酸降解,苯丙烷类生物合成等通路。
DElncRNA富集到水杨酸、茉莉酸、乙烯等多个激素信号通路,水杨酸与HR的形成密切相关[6-7],是重要的内源信号分子,茉莉酸、乙烯也作用于SAR的建立[43-44]。DElncRNA参与调控多种激素通路,使烟草得以建立多重抗性机制,抵御多种病原物的再次入侵[11]。ABC运输蛋白与分子运输过膜有关[45],亮氨酸、异亮氨酸、缬氨酸响应脱落酸介导的渗透胁迫[46],苯丙烷类化合物也被证明与烟草抗病性相关[47],这些通路在两个温度条件下同时富集表明它们可能在枯斑三生烟的抗性反应中发挥作用。
LNC036321在25℃及31℃条件下同时差异表达,它共定位的靶基因LOC107805368预测编码温度诱导的载脂蛋白(temperature-induced lipocalin-1,TIL1),同样富集在激素信号传导通路。进一步的研究发现接种TMV 6 d内LNC_036321与TIL1 mRNA的相对含量呈现此消彼长的趋势,表明二者可能存在负调控关系。LncRNA调控方式多样,依照功能可将其分为4种:吸附RNA或蛋白与之结合的诱饵分子(decoys)、通过改变自身转录调节相关基因表达的信号分子(signals)、结合蛋白形成复合物指导其移动到特定位置的导向分子(guides)以及作为RNA及蛋白等分子结合平台的骨架分子(scaffolds)[27]。LncRNA16397在致病疫霉抗/感番茄品种间差异表达,是它的共定位靶基因,lncRNA16397作为反义转录物调节其表达,使活性氧积累减少、细胞膜损伤减轻,增强番茄对致病疫霉的抗性[38]。正义转录本(sense transcript)的表达水平随正义和反义对之间重叠的增加而显著降低[31]。lncRNA16397作为“信号分子”,对表达进行顺势调控。而LNC_036321与LOC107805368序列相似性不高,考虑到LNC_036321为lincRNA,其更有可能以“诱饵分子”或“导向分子”的方式对LOC107805368进行调控[48]。
载脂蛋白一般在植物对温度等非生物胁迫的应激中发挥作用[49],但SADE等研究发现番茄对病毒抗性/易感载脂蛋白(virus resistant/ susceptible lipocalin,SlVRSLip)与TYLCV抗性相关[50],需要通过进一步的实验明确TIL1是否参与枯斑三生烟对TMV的抗性反应。
本研究仅对接种TMV 48 h的枯斑三生烟系统叶进行了lncRNA测序和分析,此时DElncRNA数目较少,这些lncRNA通过调控植物激素信号传导等过程参与枯斑三生烟抗性反应。为找到更多与系统获得性抗性相关的lncRNA,需要对TMV侵染更长时间的系统叶进行lncRNA测序分析,配合接种叶的lncRNA表达情况,对枯斑三生烟抗性反应中的lncRNA进行更全面深入的分析。
4 结论
不同温度(25℃和31℃)条件下对枯斑三生烟接种TMV后,4 737条已知lncRNA、40 169条新lncRNA在系统叶表达,其中64个lncRNA在不同处理间差异表达。利用qRT-PCR技术验证了DElncRNA的特异性与可靠性,并通过靶基因预测和功能分析,发现这些DElncRNA广泛参与到激素信号传导、物质转运等过程,在枯斑三生烟的抗性反应中发挥作用。研究结果可为-TMV互作过程中关键 lncRNA的筛选和功能研究提供理论支持。
[1] WHITHAM S, DINESH-KUMAR S P, CHOI D, HEHL R, CORR C, BAKER B. The product of theresistance gene: similarity to toll and the interleukin-1 receptor., 1994, 78(6): 1101-1115.
[2] WANG H V, CHEKANOVA J A. Long noncoding RNAs in plants//RAO M R S.. Singapore: Springer, 2017: 133-154.
[3] QUAN M, CHEN J, ZHANG D. Exploring the secrets of long noncoding RNAs., 2015, 16(3): 5467-5496.
[4] RYALS J A, NEUENSCHWANDER U H, WILLITS M G, MOLINA A, STEINER H Y, HUNT M D. Systemic acquired resistance., 1996, 8(10): 1809-1819.
[5] ERICKSON F L, HOLZBERG S, CALDERON‐URREA A, HANDLEY V, AXTELL M, CORR C, BAKER B. The helicase domain of the TMV replicase proteins induces the‐mediated defence response in tobacco., 1999, 18(1): 67-75.
[6] GAFFNEY T, FRIEDRICH L, VERNOOIJ B, NEGROTTO D, NYE G, UKNES S, WARD E, KESSMANN H, RYALS J. Requirement of salicylic acid for the induction of systemic acquired resistance., 1993, 261(5122): 754-756.
[7] MUR L A, KENTON P, LLOYD A J, OUGHAM H, PRATS E. The hypersensitive response; the centenary is upon us but how much do we know?, 2008, 59(3): 501-520.
[8] YANG Y X, AHAMMED G J, WU C, FAN S Y, ZHOU Y H. Crosstalk among jasmonate, salicylate and ethylene signaling pathways in plant disease and immune responses., 2015, 16(5): 450-461.
[9] BAKER B, ZAMBRYSKI P, STASKAWICZ B, DINESH-KUMAR S P. Signaling in plant-microbe interactions., 1997, 276(5313): 726-733.
[10] SMITH H B. Signal transduction in systemic acquired resistance., 2000, 12(2): 179-181.
[11] CUTT J R, KLESSIG D F.:. Vienna: Springer, 1992: 209-243.
[12] 肖万福. 烟草感染花叶病过程中差异表达基因与蛋白质的筛选及应用[D]. 郑州: 河南农业大学, 2017.
XIAO W F. Screening and application of differential expressed genes and proteins in tobacco mosaic disease[D]. Zhengzhou: Henan Agricultural University, 2017. (in Chinese)
[13] WANG J, YANG Y, JIN L, LING X, LIU T, CHEN T, JI Y, YU W, ZHANG B. Re-analysis of long non-coding RNAs and prediction of circRNAs reveal their novel roles in susceptible tomato following TYLCV infection., 2018, 18: 104.
[14] YANG Y, LIU T, SHEN D, WANG J, LING X, HU Z, CHEN T, HU J, HUANG J, YU W, DOU D, WANG M B, ZHANG B. Tomato yellow leaf curl virus intergenic siRNAs target a host long noncoding RNA to modulate disease symptoms., 2019, 15(1): e1007534.
[15] SEO J S, SUN H X, PARK B S, HUANG C H, YEH S D, JUNG C, CHUA N H. ELF18-induced long-noncoding RNA associates with mediator to enhance expression of innate immune response genes in., 2017, 29(5): 1024-1038.
[16] SEO J S, DILOKNAWARIT P, PARK B S, CHUA N H. ELF18- induced long noncoding RNA 1 evicts fibrillarin from mediator subunit to enhance() expression., 2019, 221(4): 2067-2079.
[17] ANDERS S. HTSeq: Analysing high-throughput sequencing data with Python[EB/OL]. https://htseq.readthedocs.io/en/release_0.11.1/.
[18] PERTEA M, PERTEA G M, ANTONESCU C M, CHANG T C, MENDELL J T, SALZBERG S L. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads., 2015, 33(3): 290-295.
[19] TRAPNELL C, WILLIAMS B A, PERTEA G, MORTAZAVI A, KWAN G, VAN BAREN M J, SALZBERG S L, WOLD B J, PACHTER L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation., 2010, 28(5): 511-515.
[20] SUN L, LUO H, BU D, ZHAO G, YU K, ZHANG C, LIU Y, CHEN R, ZHAO Y. Utilizing sequence intrinsic composition to classify protein- coding and long non-coding transcripts., 2013, 41(17): e166.
[21] KANG Y J, YANG D C, KONG L, HOU M, MENG Y Q, WEI L, GAO G. CPC2: a fast and accurate coding potential calculator based on sequence intrinsic features., 2017, 45(Web Server issue): W12-W16.
[22] MISTRY J, BATEMAN A, FINN R D. Predicting active site residue annotations in the Pfam database., 2007, 8: 298.
[23] CABILI M N, TRAPNELL C, GOFF L, KOZIOL M, TAZON-VEGA B, REGEV A, RINN J L. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses., 2011, 25(18): 1915-1927.
[24] ROBINSON M D, MCCARTHY D J, SMYTH G K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data., 2010, 26(1): 139-140.
[25] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod., 2001, 25(4): 402-408.
[26] LI T, ZHANG G, WU P, DUAN L, LI G, LIU Q, WANG J. Dissection of myogenic differentiation signatures in chickens by RNA-Seq analysis., 2018, 9(1): 34.
[27] WANG K C, CHANG H Y. Molecular mechanisms of long noncoding RNAs., 2011, 43(6): 904-914.
[28] YOUNG M D, WAKEFIELD M J, SMYTH G K, OSHLACK A. Gene ontology analysis for RNA-seq: accounting for selection bias., 2010, 11(2): R14.
[29] KANEHISA M, ARAKI M, GOTO S, HATTORI M, HIRAKAWA M, ITOH M, KATAYAMA T, KAWASHIMA S, OKUDA S, TOKIMATSU T, YAMANISHI Y. KEGG for linking genomes to life and the environment., 2007, 36(Database issue): D480-D484.
[30] MAO X, CAI T, OLYARCHUK J G, WEI L. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary., 2005, 21(19): 3787-3793.
[31] OSATO N, SUZUKI Y, IKEO K, GOJOBORI T. Transcriptional interferences innatural antisense transcripts of humans and mice., 2007, 176(2): 1299-1306.
[32] FAGHIHI M A, WAHLESTEDT C. Regulatory roles of natural antisense transcripts., 2009, 10(9): 637-643.
[33] BENJAMINS R, SCHERES B. Auxin: the looping star in plant development., 2008, 59: 443-465.
[34] WILSON M S, LIVERMORE T M, SAIARDI A. Inositol pyrophosphates: between signalling and metabolism., 2013, 452(3): 369-379.
[35] SAHA M, SARKAR S, SARKAR B, SHARMA B K, BHATTACHARJEE S, TRIBEDI P. Microbial siderophores and their potential applications: a review., 2016, 23(5): 3984-3999.
[36] ZHENG Y, DING B, FEI Z, WANG Y. Comprehensive transcriptome analyses reveal tomato plant responses to tobacco rattle virus-based gene silencing vectors., 2017, 7: 9771.
[37] KWENDA S, BIRCH P R, MOLELEKI L N. Genome-wide identification of potato long intergenic noncoding RNAs responsive tosubspeciesinfection., 2016, 17(1): 614.
[38] CUI J, LUAN Y, JIANG N, BAO H, MENG J. Comparative transcriptome analysis between resistant and susceptible tomato allows the identification of lncRNA16397 conferring resistance toby co-expressing glutaredoxin., 2017, 89(3): 577-589.
[39] LI X, XING X, XU S, ZHANG M, WANG Y, WU H, SUN Z, HUO Z, CHEN F, YANG T. Genome-wide identification and functional prediction of tobacco lncRNAs responsive to root-knot nematode stress., 2018, 13(11): e0204506.
[40] 赵长江. 纹枯病菌侵染后水稻防御反应相关基因的表达分析[D]. 福州: 福建农林大学, 2005.
ZHAO C J. Expression analysis of rice defence-related genes after infected by[D]. Fuzhou: Fujian Agriculture and Forestry University, 2005. (in Chinese)
[41] MALAMY J, CARR J P, KLESSIG D F, RASKIN I. Salicylic acid: a likely endogenous signal in the resistance response of tobacco to viral infection., 1990, 250(4983): 1002-1004.
[42] 许志刚. 普通植物病理学. 4版. 北京: 高等教育出版社, 2009: 296-315.
XU Z G.. Beijing: Higher Education Press, 2009: 296-315. (in Chinese)
[43] ZHU F, XI D H, YUAN S, XU F, ZHANG D W, LIN H H. Salicylic acid and jasmonic acid are essential for systemic resistance againstin., 2014, 27(6): 567-577.
[44] VERBERNE M C, HOEKSTRA J, BOL J F, LINTHORST H J. Signaling of systemic acquired resistance in tobacco depends on ethylene perception., 2003, 35(1): 27-32.
[45] BEIS K. Structural basis for the mechanism of ABC transporters., 2015, 43(5): 889-893.
[46] HUANG T, JANDER G. Abscisic acid-regulated protein degradation causes osmotic stress-induced accumulation of branched-chain amino acids in., 2017, 246(4): 737-747.
[47] SHADLE G L, WESLEY S V, KORTH K L, CHEN F, LAMB C, DIXON R A. Phenylpropanoid compounds and disease resistance in transgenic tobacco with altered expression of L-phenylalanine ammonia-lyase., 2003, 64(1): 153-161.
[48] MERCER T R, DINGER M E, MATTICK J S. Long non-coding RNAs: insights into functions., 2009, 10(3): 155-159.
[49] BERTERAME N M, BERTAGNOLI S, CODAZZI V, PORRO D, BRANDUARDI P. Temperature-induced lipocalin (TIL): a shield against stress-inducing environmental shocks in., 2017, 17(6): fox056.
[50] SADE D, EYBISHTZ A, GOROVITS R, SOBOL I, CZOSNEK H. A developmentally regulated lipocalin-like gene is overexpressed in-resistant tomato plants upon virus inoculation, and its silencing abolishes resistance., 2012, 80(3): 273-287.
Differential expression of lncRNAs invar. Samsun NN infected by TMV at different temperatures
JIA HaiYan1, SONG LiYun1, XU Xiang1, XIE Yi1, ZHANG ChaoQun2, LIU TianBo3, ZHAO CunXiao4, SHEN LiLi1, WANG Jie1, LI Ying1, WANG FengLong1, YANG JinGuang1
(1Tobacco Research Institute, Chinese Academy of Agricultural Sciences/Key Laboratory of Tobacco Disease and Pest Monitoring, Controlling & Integrated Management, Qingdao 266101, Shandong;2Tobacco Science Institute of Jiangxi Province, Nanchang 330029;3Central South Agricultural Experimental Station of China National Tobacco Corporation, Changsha 410128;4Qingyang Tobacco Company of Gansu Provincial Company, Qingyang 745099, Gansu)
【】The objective of this study is to screen out the differentially expressed lncRNAs invar. Samsun NN after(TMV) infection at different temperatures, and to investigate the role of identified lncRNAs in Samsun NN’s resistance response.【】The temperature sensitivity of N gene makesSamsun NN have the resistance to TMV at 25℃, but lose it at 31℃. TMV and phosphate buffered saline(PBS) were mechanically inoculated into Samsun NN at 25℃ and 31℃. Total RNAs were extracted from systemic leaves at 48 hpi (hours post infection). Deep sequencing was performed after strand-specific database construction, and the sequencing results were filtered to get clean reads. Usingvar. TN90 as a reference, HTseq was employed to compare obtained reads. LncRNAs were screened out, and their expression levels were estimated by FPKM method. Differentially expressed lncRNAs (DElncRNAs) were then identified by edgeR and verified by qRT-PCR. The target genes of DElncRNAs were predicted by co-localization and co-expression analyses. Gene annotations, GO and KEGG pathways were analyzed for functional prediction of target genes. 【】Altogether, 80 million clean reads were detected for each of 12 samples from 4 treatments by lncRNA-seq. A total of 4 737 annotated lncRNAs and 40 169 novel lncRNAs were obtained. Among them, 64 lncRNAs were differentially expressed after TMV infection at different temperatures. qRT-PCR results showed that the sequencing accuracy of these DElncRNAs was about 80%, which indicated the sequencing data obtained in this study had high reliability. Importantly, some target genes were simultaneously targeted by DElncRNAs that were down-regulated at 25℃ and up-regulated at 31℃. Gene annotations showed that DElncRNAs’ target genes involved in many functions, such as plant resistance, hormone and metabolic pathways. Particularly, some lncRNAs that may be associated with hormone pathways showed a down-regulation trend after TMV infection at 25℃, while up-regulated at 31℃. Furthermore, GO enrichment analysis showed that target genes were mainly involved in the composition of membranes, vesicles, and acted as calcium and potassium ion channel inhibitor activity, so that consequently the corresponding ions could be transported to their sites of action to trigger subsequent reactions. Moreover, these genes were also involved in pathogenesis, antigen processing and presentation, cytokinin metabolism and other physiological processes. The plant hormone signaling pathway was significantly enriched by target genes during KEGG pathway analysis. DElncRNAs related genes down-regulated at 25℃ and up-regulated at 31℃ were simultaneously enriched in pathways such as hormone signaling, ABC transporters, and phenylpropanoid biosynthesis.【】LncRNAs were differentially expressed in Samsun NN after TMV infection at different temperatures (25℃ and 31℃). DElncRNAs participated in host plant systemic acquired resistance by acting on hormone signaling transduction, substance transport and other processes. Taken together, this study lays a foundation for further research on lncRNA’s regulatory function in plant systemic acquired resistance and the development of new strategies to overcome virus invasion into host plant.
(TMV); N gene;var. Samsun NN; long non-coding RNA (lncRNA); systemic acquired resistance; deep sequencing; affinity enrichment analysis

10.3864/j.issn.0578-1752.2020.07.008
2019-09-16;
2019-11-06
烟草绿色防控重大专项(110101601024(LS-04))烟草绿色防控重大专项(110101601024(LS-04))、甘肃省烟草公司科技项目(201862100020016,201862100020017)、湖南省烟草公司科技项目(201743010020088)、江西省烟草公司科技项目(201701002)
贾海燕,E-mail:jiahaiyan1025@163.com。宋丽云,E-mail:jiayoulily2009@126.com。贾海燕和宋丽云为同等贡献作者。通信作者王凤龙,E-mail:wangfenglong@caas.cn。通信作者杨金广,E-mail:yangjinguang@caas.cn
(责任编辑 岳梅)

