人类APOBEC3蛋白的结构与功能分析①
吴小霞 许天委 李国寅
(琼台师范学院,琼台儿童认知与行为发展研究中心,海口 571127)
位于人类22号染色体上的七个APOBEC3(apolipoprotein B mRNA editing enzyme-catalytic polypeptide like3,亦称APOBEC3)蛋白是一个通过控制外源性逆转录病毒复制和内源性逆转录因子转座而广泛参与固有免疫的胞苷脱氨酶家族[1,2],分别是APOBEC3A(简称A3A)、APOBEC3B(简称A3B)、APOBEC3C(简称A3C)、APOBEC3DE(简称A3DE)、APOBEC3F(简称A3F)、APOBEC3G(简称A3G)和APOBEC3H(简称A3H)[1-3]。
脱氨酶活性会引起人类基因组的不稳定和癌症,A3A是目前这一家族中最活跃的[4],也是IAP和MusD以及其他逆转录转座,如LINE-1的有效抑制剂[5]。A3G是该家族中第一个被证实限制逆转录病毒的家庭成员[5]。A3B在正常组织内不表达或表达较低,而在乳腺癌、宫颈癌、膀胱癌、肺癌、卵巢癌等肿瘤组织却过量表达,尽管机制仍不太清楚,但A3B被认为是癌细胞内驱动肿瘤发展的体细胞突变源头[6-9]。A3C抑制病毒和内源性逆转录元件的功能较弱[10,11],但Wittkopp等[10]分析发现A3C在灵长类进化抵御病原体时起非常重要的作用。而A3DE、A3F和A3H在Vif缺陷的细胞限制HIV-1感染,也在Vif成熟的细胞内对病毒基因组进行催化导致亚致死水平的突变[9]。
本文利用生物信息学方法对人类七个APOBEC3蛋白质序列进行理化性质、蛋白质定位等结构和功能的分析,寻找人类APOBEC3蛋白的相似及不同之处,为进一步深入研究和分析人类APOBEC3蛋白的结构与功能的相互关系提供思路。
1 材料与方法
1.1材料 本文中的人类APOBEC3蛋白序列来自NCBI数据库,A3A(检索号:AKE33285),包含199个氨基酸,A3B(检索号:AAH53859)包含251个氨基酸,A3C(检索号:AAH11739)包含190个氨基酸,A3DE(检索号:ACB32248)包含386个氨基酸,A3F(检索号:AAH38808 )包含373个氨基酸,A3G(检索号:AAZ38722 )包含384个氨基酸以及 A3H(检索号:ACK77778)包含154个氨基酸。
1.2方法
1.2.1APOBEC3多重序列比对 进入主页https://www.ebi.ac.uk/Tools/msa/,选择Launch Clustal Omega,输入人类APOBEC3的蛋白序列fasta格式,点击submit。
1.2.2APOBEC3理化性质分析 进入网页http://web.expasy.org,然后点击Proteomics,找到ExPASy数据分析系统中的ProtParam工具,输入NCBI数据库中获得的APOBEC3的蛋白序列,点击computer parameters,从而对APOBEC3的分子量、分子式、酸碱性和稳定性等理化性质进行分析。在网页https://www.expasy.org/proteomics,点击Protsc-ale,使用Protscale工具,输入APOBEC3蛋白序列,对APOBEC3的亲疏水性进行分析。
1.2.3APOBEC3亚细胞定位 进入网页http://psort.hgc.jp/,选择PSORTⅡ工具,输入AOBEC3序列,点击Submit,进行APOBEC3蛋白亚细胞定位预测。
1.2.4APOBEC3结构分析 进入网页使用http://www.cbs.dtu.dk/,点击Prediction Servers,找到TMHMM2.0工具,输入APOBEC3蛋白序列,分析APOBEC3蛋白的跨膜区域。
进入https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl网页,输入APOBEC3蛋白序列,使用SOPMA工具,预测APOBEC3的二级结构及各成分所占比例。进入网页http://pfam.xfam.org/,点击search,输入APOBEC3序列,点击submit,预测APOBEC3蛋白的结构域。进入网页http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index,利用Phyre2工具,进行APOBEC3蛋白质三维结构的预测。
1.2.5APOBEC3蛋白相互作用分析 使进入http://string-db.org/,点击protein by sequence,输入APOBEC3蛋白质序列,选择Homo sapiens物种,点击search,构建与APOBEC3蛋白相互作用的蛋白网络。
1.2.6APOBEC蛋白进化分析 使用MEGA 7.0软件,Neighbor-joining方法设置Boot-strap分析重复数为1 000,构建人类APOBEC3分子进化树。
2 结果
2.1APOBEC3多重序列比对结果 以A3H(ACK77778)作为参考序列,经过Clustal Omega比对运算,结果见表1。
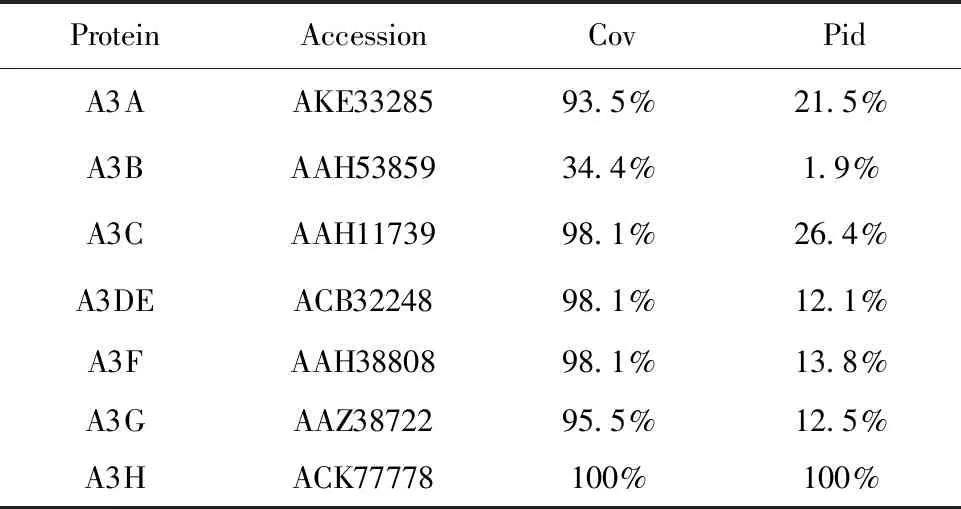
表1 Clustal Omega比对结果Tab.1 Sequence alignment results of clustal omega
2.2理化性质分析 利用ProtParam工具,可以分析人类APOBEC3七个蛋白质的理化性质,结果见表2。可以看出人类A3A、A3DE、A3G和A3H含量最高的氨基酸均为Leu,A3B、A3DE、A3G和A3H为碱性蛋白质,而A3A、A3C和A3F均为弱酸性蛋白质,A3A、A3B和A3F均为稳定蛋白质,而A3C、A3DE、A3G和A3H为不稳定蛋白质。
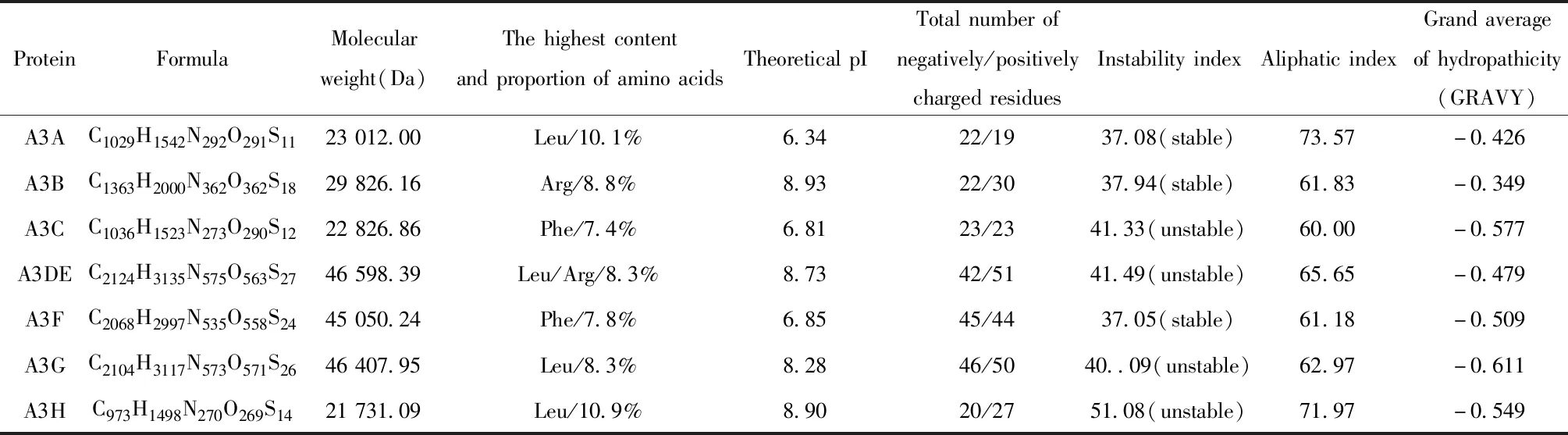
表2 APOBEC3的理化性质Tab.2 Physical and chemical properties of APOBEC3
利用ProtScale在线工具分析APOBEC3的亲疏水性,结果见表3及图1,可以看出APOBEC3七个蛋白质的亲水区域多于疏水区域,所以均为亲水性蛋白质。
2.3亚细胞定位分析 利用PSORTⅡ分析APOBEC3蛋白的亚细胞定位,结果见表4,可看出除A3H可能主要定位于细胞核外,其余APOBEC3可能主要定位于细胞质。
2.4跨膜结构分析 经TMHMM2.0工具预测结果表5和图2所示,A3A、A3B、A3C、A3DE、A3F、A3G及A3H均为不跨膜蛋白。图2中红色细线表示跨膜区域,A3A、A3DE、A3F、A3G及A3H位于膜外(粉色细线)的概率几乎为100%,位于膜内(蓝色细线)和跨膜区域(红色细线)的概率几乎为0,而A3B、A3C位于膜外的概率接近80%。粉色粗线代表多肽链中跨膜区域所在位置,因没有跨膜区域,所以不显示相应标记。

表5 APOBEC3 TMHMM分析结果Tab.5 TMHMM analysis of APOBEC3 protein
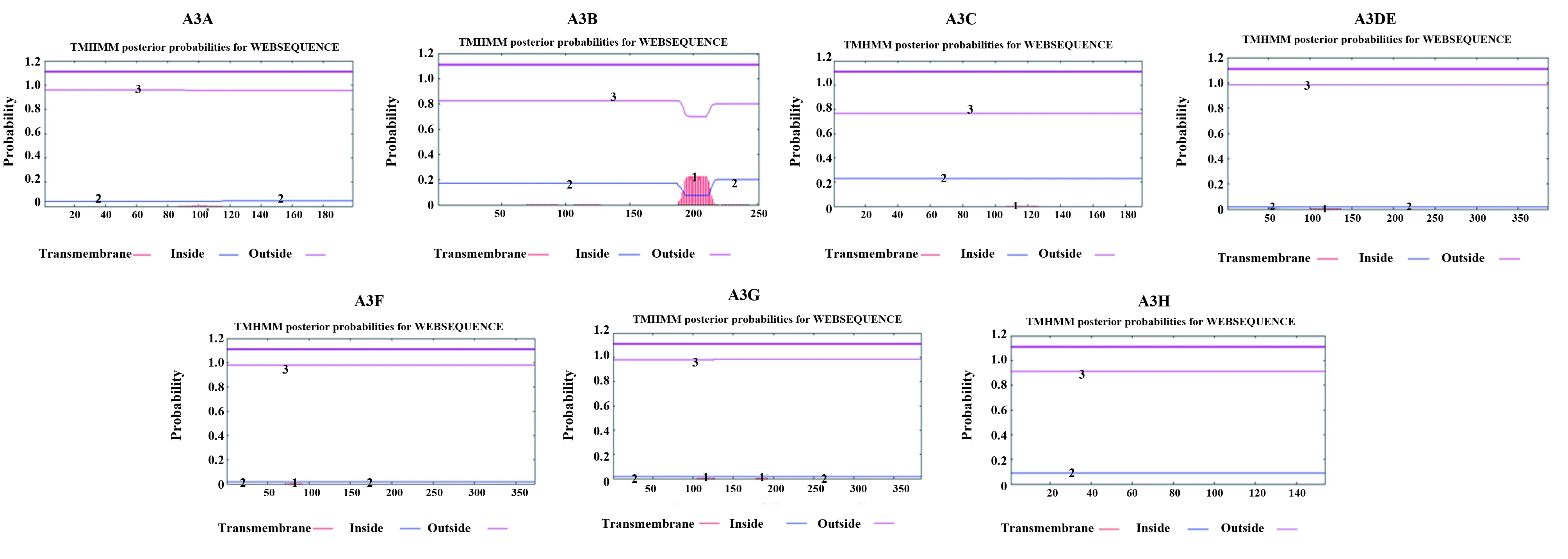
图2 APOBEC3的跨膜结构分析Fig.2 Analysis of transmembrane structure of APOBEC3Note:In the figure,1 is a red thin line for the transmembrane,2 is a blue thin line for the inside,and 3 is a pink thin line for the outside.
2.5空间结构分析 利用 SOPMA工具分析APOBEC3蛋白的二级结构如图3,与相应二级结构占比见表6。由此可见,APOBEC3蛋白的二级结构比例相差不大,结构相似,只有A3Fα螺旋所占比例最高,其余蛋白均为无规则卷曲所占比例最高。
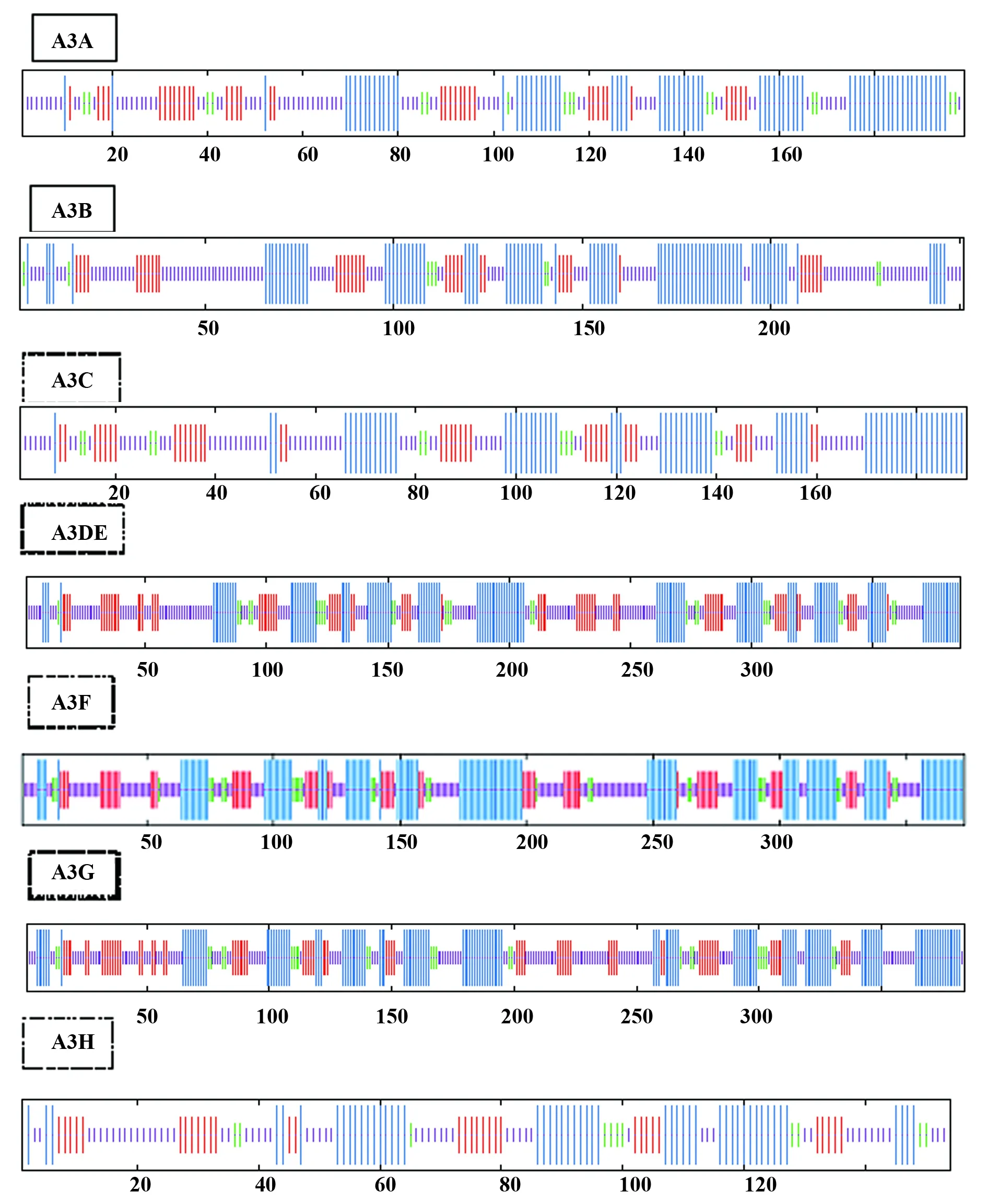
图3 APOBEC3蛋白二级结构分析Fig.3 Secondary structure analysis of APOBEC3 proteinNote:Blue represents the α helix,red represents the Extended strand,green represents the β turn,and purple represents the Random curl.
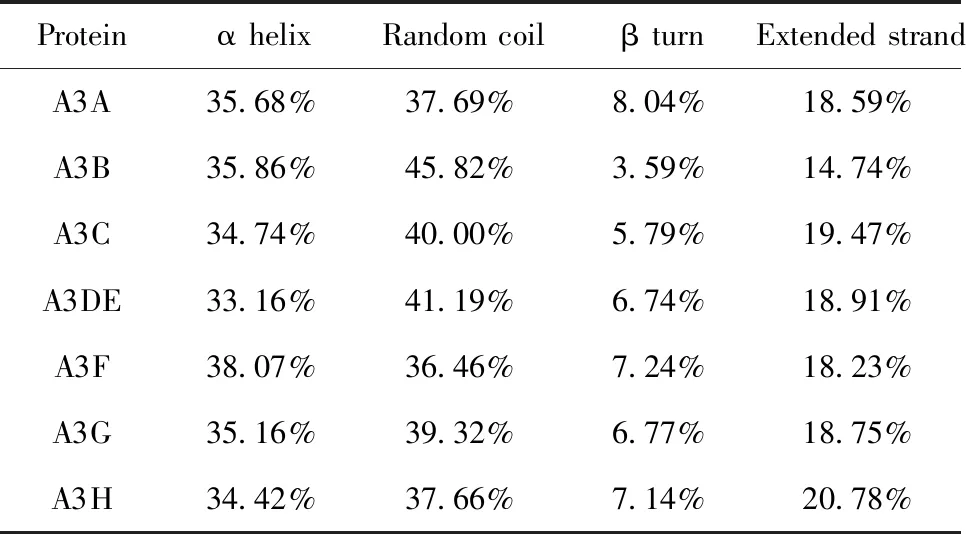
表6 APOBEC3蛋白二级结构类型及占比Tab.6 Secondary structure types and proportions of APOBEC3 protein
利用pfam工具得出,A3A、A3B、A3C、A3DE、A3F和A3G都属于NAD2家族,一个新的AID/APOBEC分支,而A3H属于APOBEC家族;A3A、A3B、A3C、A3H有一个结构域,而A3DE、A3F和A3G有两个结构域,A3F和A3G的结构域相似性极高,具体结果如表7。
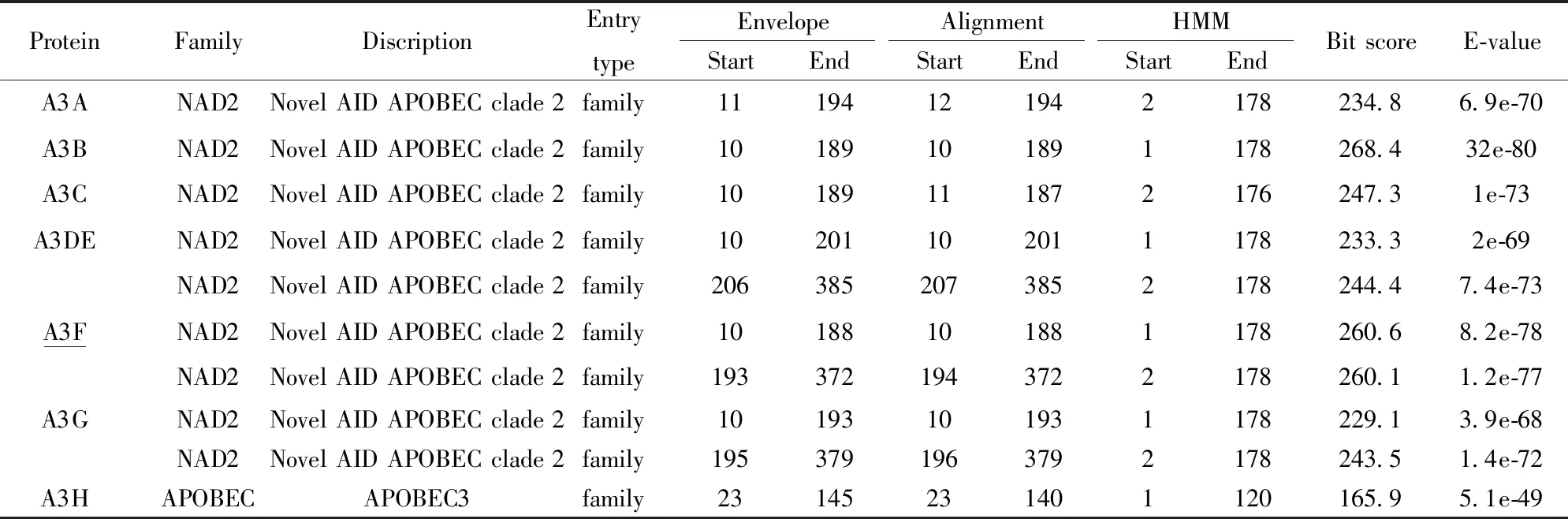
表7 APOBEC3 Pfam-A的重要匹配Tab.7 Signifitant Pfam-A matches of APOBEC3
利用Phyre2工具,进行APOBEC3蛋白质三维结构的预测结果如图4,三维结构中的二级结构预测结果如图5。A3A的模板为c2m65A,为人类A3A的NMR结构,可信度100%,覆盖面为100%;A3B的模板为c5k83C,为灵长类A3G N-结构域的晶体结构,可信度100%,覆盖面为75%;A3C的模板为c3vowB,为具有HIV-1 vif结合界面的人A3C的晶体结构,可信度为100%,覆盖面为98%;A3DE的模板为c5k83C,为灵长类A3G N-结构域的晶体结构,可信度为100%,覆盖面为49%;A3F模板为c5k83C,为灵长类A3G N-结构域的晶体结构,可信度为100%,覆盖面为51%;A3G的模板为 c2kboA,为野生型A3G结构,可信度为100%,覆盖面为50%;A3H的模板为c6b0bE,是人类A3H的晶体结构,可信度100%,覆盖面为90%。

图4 APOBEC3蛋白三维结构Fig.4 Three-dimensional structure of APOBEC3 protein
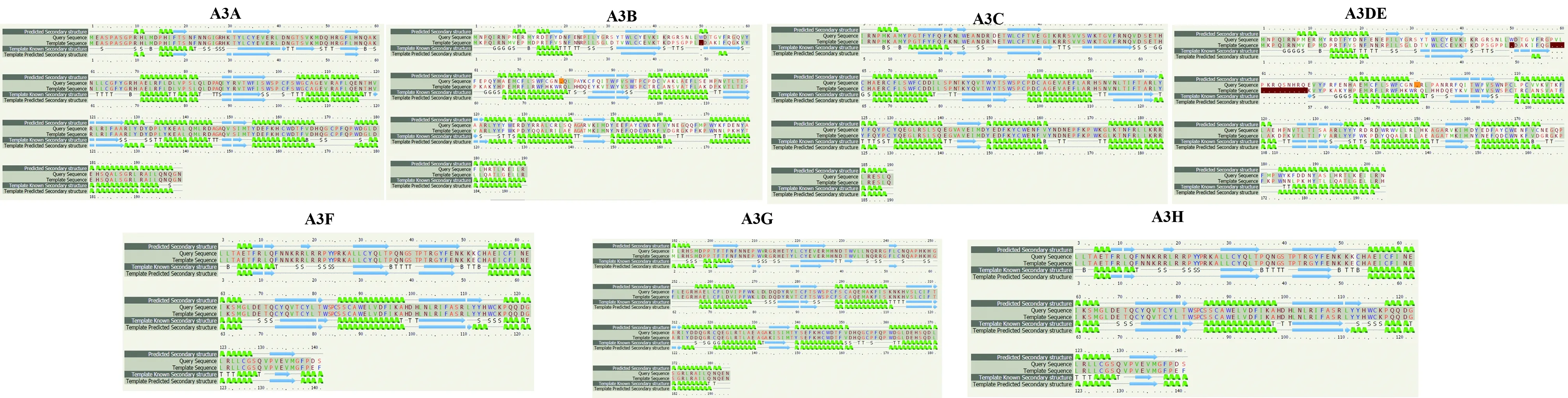
图5 APOBEC3蛋白三维结构下的二级结构Fig.5 Secondary structure of Three-dimensional APOBEC3 protein
2.6APOBEC3蛋白质相互作用分析 利用STRING数据库预测APOBEC3的蛋白相互作用网络,结果如图6。与A3A、A3B和A3C相互作用的蛋白网络是一样的,包括APOBEC1、APOBEC2、APOBEC4等,主要参与胞嘧啶脱氨酶作用(GO:0009972)、DNA去甲基化(GO:0080111)及DNA修饰等过程(GO:0006304),具有有水解酶活性(GO:0016814)、胞嘧啶脱氨酶活性(GO:0004126)及脱氨酶活性(GO:0019239)等。A3A、A3B与A3C都可构成细胞内线粒体呼吸链复合物Ⅳ(GO:0005751),KEGG通路主要是心肌收缩(hsa04260)、亨廷顿氏病(Huntington′s disease,hsa05016)及帕金森病 (Parkinson′s disease,hsa05012)等,反应途径有编辑体的形成(HSA-75094)、TP53调节代谢基因(HSA-5628897)、呼吸链中的电子传输(HSA-611105)。
与A3DE相互作用的蛋白质主要有CYCS(cytochrome c,somatic,简称CYCS)、COX5A(cytochrome c oxidase subunit 5A,简称COX5A)、CUL5(cullin 5,简称CUL5)等,主要参与电子传递链(GO:0022900)、线粒体细胞色素c到氧的电子传递(GO:0006123)以及线粒体ATP合成耦合电子输运(GO:0042775)等,具有电子转移活性(GO:0009055)、水解酶活性(GO:0016814)及细胞色素c氧化酶活性(GO:0004129)等。细胞色素c是一种定位于线粒体内膜的约12 kD血红素蛋白,在多种细胞过程中起着关键作用,例如在线粒体呼吸复合物Ⅲ和Ⅳ之间传递电子,在固有细胞凋亡途径中是必需的[12]。A3DE可能是细胞内呼吸体(GO:0070469)、细胞色素复合体(GO:0070069)及线粒体内膜(GO:0005743)等的组成部分,KEGG通路主要是帕金森病 (Parkinson′s disease,hsa05012)、阿尔茨海默病(Alzheimer′s disease,hsa05010)及非酒精性脂肪性肝病(hsa04932)等,反应途径有TP53调节代谢基因(HSA-5628897)、呼吸链中的电子传输(HSA-611105)及新陈代谢(HSA-1430728)。
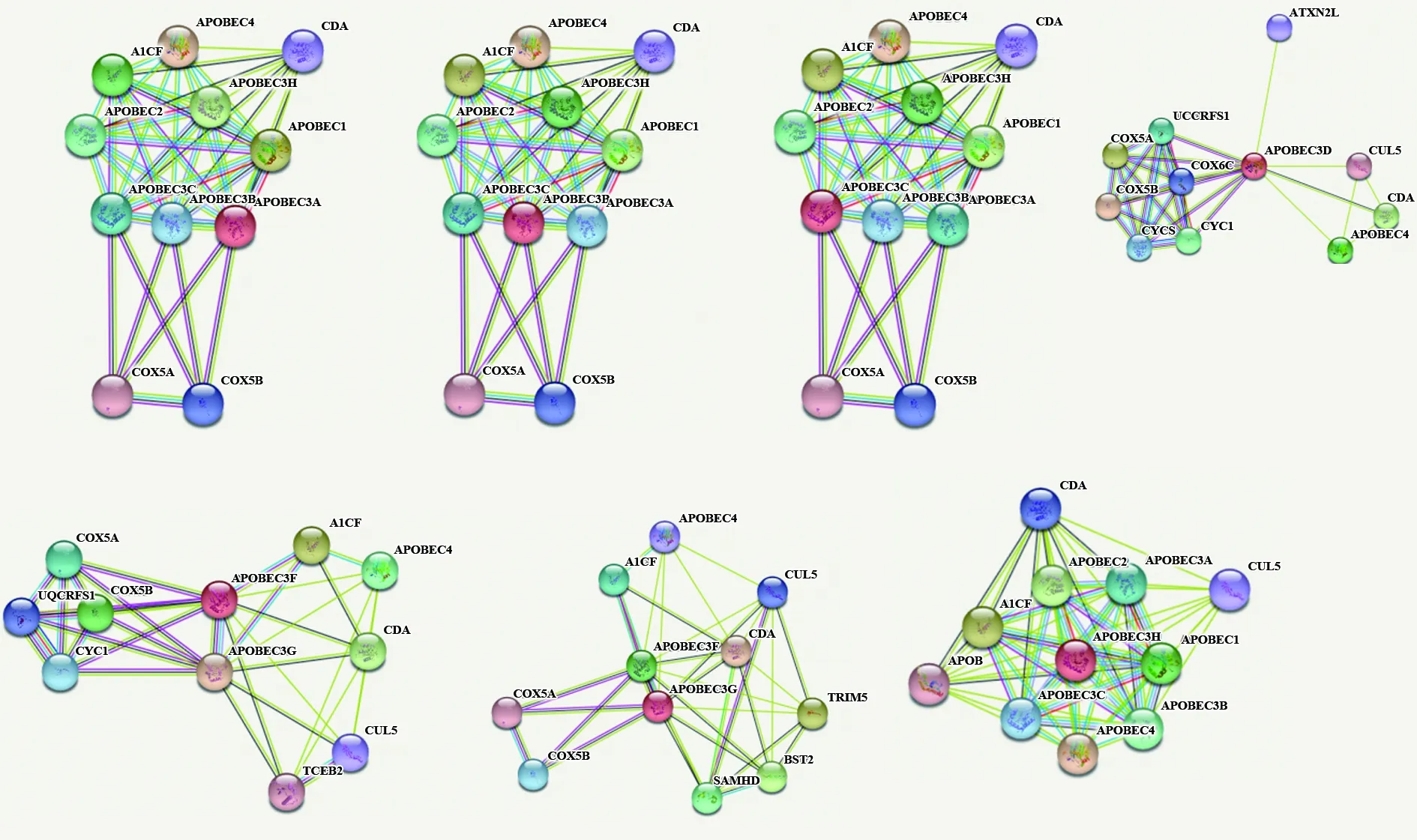
图6 与APOBEC3相互作用的蛋白网络Fig.6 Protein network interacting with APOBEC3
与A3F相互作用的蛋白质有A3G、A1CF(APOBEC1 complementation factor,简称A1CF)、CYC1(cytochrome c1,简称CYC1)等,主要参与碱基转换或替换编辑(GO:0016553)、胞嘧啶脱氨酶作用(GO:0009972)以及电子传递链(GO:0022900)等生物过程,具有水解酶活性(GO:0016814)、胞嘧啶脱氨酶活性(GO:0004126)及电子转移活性(GO:0009055)等。A3F可能是细胞内细胞色素复合体(GO:0070069)、载脂蛋白B mRNA编辑酶复合物(GO:0030895)及呼吸链复合体(GO:0098803)等的组成部分,KEGG通路主要是心肌收缩(hsa04260)、帕金森病 (Parkinson′s disease,hsa05012)及非酒精性脂肪性肝病(hsa04932)等,反应途径有呼吸链中的电子传输(HSA-611105)、编辑体的形成(HSA-75094)及Vif介导的A3G降解(HSA-180585)等。与A3G相互作用的蛋白质有A3F、SAMHD1(Sterile alpha motif and HD domain-containing protein 1,简称SAMHD1)、CDA(cytidine deaminase,简称CDA)等,主要参与病毒生命周期负调节(GO:1903901)、病毒防御应答(GO:0051607)及胞嘧啶脱氨酶作用(GO:0009972)等生物过程,具有水解酶活性(GO:0016814)、胞嘧啶脱氨酶活性(GO:0004126)以及细胞色素c氧化酶活性(GO:0004129)。SAMHD1是一种dNTPs水解酶,通过在非分化细胞降解细胞内dNTPs来限制HIV-1等逆转录病毒及几种DNA病毒的复制[13,14]。人类CDA是一个由完全相同的15 kD亚基组成的四聚体,每个亚基都在活性部位含有必不可少的锌原子,是参与嘧啶修复的酶之一,可利用通过胞嘧啶和脱氧胞嘧啶的水解脱氨基作用分别催化形成尿嘧啶和脱氧尿嘧啶[15]。A3G是细胞内载脂蛋白B mRNA编辑酶复合物(GO:0030895)、细胞质处理小体(P-body,GO:0000932)及线粒体呼吸链复合物Ⅳ(GO:0005751)的组成部分,KEGG通路主要是心肌收缩(hsa04260)、亨廷顿氏病(Hun-tington′s disease,hsa05016)及帕金森病 (Parkinson′s disease,hsa05012)等,反应途径有编辑体的形成(HSA-75094)、干扰素信号(HSA-913531)及TP53调节代谢基因(HSA-5628897)等。
与A3H相互作用的蛋白质有A3A、APOBEC2、A3B等,主要参与胞嘧啶脱氨酶作用(GO:0009972)、DNA去甲基化(GO:0080111)及转座负调控(GO:0010529)等过程,具有脱氧胞苷脱氨酶活性(GO:0047844)、水解酶活性(GO:0016814)及胞嘧啶脱氨酶活性(GO:0004126)等。A3H的反应途径有编辑体的形成(HSA-75094)。
2.7APOBEC3进化分析 利用MEGA 7.0软件分析结果如图7,A3F和A3G的亲缘关系更近一些,A3B和A3C的亲缘关系更近一些,而A3H与A3A亲缘关系要亲近一些,而A3DE与A3B和A3C可能源于亲缘关系更接近的祖先。

图7 人类APOBEC3的进化树Fig.7 Evolution tree of human APOBEC3
3 讨论
本文利用生物信息学方法对人类APOBEC3七个蛋白进行分析和预测,发现A3A、A3C和A3F为弱酸性蛋白质,A3B、A3DE、A3G和A3H为碱性蛋白质,A3A、A3B和A3F为稳定的亲水蛋白,而A3C、A3DE、A3G和A3H为不稳定的亲水性蛋白质;A3H可能主要定位于细胞核外,其余APOBEC3可能主要定位于细胞质,所有APOBEC3蛋白均为不跨膜蛋白。
APOBEC3蛋白的二级结构比例相差不大,结构极为相似,其中α螺旋和无规则卷曲所占比例最高。pfam工具分析发现A3A、A3B、A3C、A3DE、A3F和A3G都属于NAD2家族,这是一个新的AID/APOBEC分支,而A3H属于APOBEC家族;A3A、A3B、A3C、A3H有一个结构域,而A3DE、A3F和A3G有两个结构域,其中A3F和A3G的结构域相似性极高,同时利用Phyre2工具,对APOBEC3蛋白质三维结构进行预测。
利用STRING分析发现APOBEC3除具有已知的胞嘧啶脱氨酶活性外,还具有水解酶活性;A3DE、A3F可能还参与了细胞色素C的电子传递;A3A、A3B、A3C参与DNA修饰过程,A3H参与DNA去甲基化过程,这暗示A3A、A3B、A3C和A3H可能参与基因突变,导致基因组的不稳定与癌症等疾病发生;A3G参与病毒的防御应答,暗示A3G具有广谱抗病毒功能。而最近的研究表明A3G除了有助于抵抗病毒感染外,A3G在癌症中也发挥着重要的非抗病毒功能[16]。利用MEGA 7.0软件分析显示A3F和A3G的亲缘关系更近一些,A3B和A3C的亲缘关系更接近,而A3H与其他A3成员的亲缘关系要远一些。
本文通过对人类APOBEC3七个蛋白理化性质和结构功能的分析,为进一步研究APOBEC3的功能及抗病毒药物的研发提供参考。

