超深渊钩虾Eurythenes gryllus i型溶菌酶的基因克隆与表达
李雪雪,阮灵伟
(1.宁波大学海洋学院,浙江 宁波315000;2.自然资源部第三海洋研究所、自然资源部海洋生物遗传资源重点实验室,福建 厦门 361005)
1922年,英国细菌学家Fleming从人的唾液和眼泪中观察到一种能比较明显溶解细菌细胞壁的成分,因为具有溶菌作用所以将其命名为溶菌酶[1]。1937年,Abraham和Robinson又实现了将溶菌酶晶体从卵蛋白中分离出来,从此开启了对溶菌酶的研究,并不断获得新的发现[2]。
溶菌酶是一种碱性酶,能够水解粘多糖,主要作用机理是通过催化水解细胞壁中N-乙酰氨基葡萄糖和N-乙酰胞壁酸之间形成的β-1,4糖苷键,糖苷键水解后致使细菌细胞壁破裂,破裂的细胞壁会使得内容物逸出从而导致细菌死亡[3]。研究表明溶菌酶发挥作用时有较强的底物特异性,当溶菌酶来源不同时,作用的底物也会不同[4],已知的溶菌酶按来源不同可分为六大类:①鹅型溶菌酶,又称g型溶菌酶(g-type);②鸡型溶菌酶,又称c型溶菌酶(c-type);③T4噬菌体溶菌酶;④无脊椎动物溶菌酶,又称i型溶菌酶(i-type);⑤植物溶菌酶;⑥细菌溶菌酶[5-10]。近年来i型溶菌酶被广泛研究,它作为溶菌酶家族的新成员,在环节动物门、软体动物门、棘皮动物门和节肢动物门中分布比较广泛[11]。有些溶菌酶含有失稳酶结构域。失稳酶是在欧洲医蛭(Haemopissanguisuga)中发现的,最初发现其有异肽酶活性,能特异性水解纤维蛋白ε-(γ-Glu)-Lys 的交联键,进而使纤维蛋白发生水解[12]。后来,人们又在失稳酶中也发现了糖苷酶活力,于是失稳酶被认为是一种新的溶菌酶,并且进一步被认定为 i 型溶菌酶。由于失稳酶既具有异肽酶活性,又具有糖苷酶活性,因此称作DL(Destabilase-lysozyme)[13-14]。溶菌酶在植物、动物和微生物中被普遍的发现,同时它也是一种对机体无毒害、安全性能很高的碱性小分子蛋白,可以选择性地分解植物及微生物细胞壁,并且对没有细胞壁的人体不会产生降解,目前被普遍应用于食品、饲料和医药行业。
超深渊是指在6 000 m以下的深渊带中凹陷深度最大的地带,主要是一系列复杂的海沟组成,目前世界上被发现的独立的超深渊栖息地有46个。超深渊底部的水温在1~4 ℃;底部静水压力在600~1 100 Pa之间;物种的数量和多样性非常低,其中最常见的是端足类、海参类多毛类、腹足类和双壳类。超深渊海沟这种高压、低温、黑暗及寡营养的环境条件使得深海形成了特有的生命现象,由于这种特有环境条件使其成为一个拥有巨大潜力的生物资源宝库[15]。钩虾Eurythenesgryllus又是超深渊最具有代表性物种之一,目前对它的研究还比较少,尤其在分子生物学方面[16]。本研究从E.gryllus克隆表达一个含有失稳酶结构域的i型溶菌酶,成功表达和纯化出较高浓度的表达产物,以期为该蛋白后续生物活性及功能研究奠定基础。
1 材料和方法
1.1 材料与试剂
钩虾E.gryllus采集于雅浦海沟(11°20.0′N, 142°11.5′E, 5 970 m)。采集后的样品放置在RNAlater保护液中,并于-80 ℃保存。
pET-His vector、EscherichiacoliTOP10菌株和E.coliBL21菌株为本实验室保存;DNAase I、RNAase inhibitor、BamH I、EcoR I、PrimerSTAR、Reverse Transcriptase M-MLV、T4DNA连接酶购自TaKaRa公司;DNA胶回收试剂盒、质粒提取试剂盒、His标签蛋白纯化试剂盒购买自GE公司;其余试剂均为国产分析纯。
1.2 实验方法
1.2.1E.gryllus总RNA提取和cDNA合成 按照TRIzol Reagent说明书操作步骤提取组织中的总RNA,经电泳检测无降解后,取RNA 2 μg,加入Oligo(dT)18引物1 mm3,加入DEPC-H20配制到13.5 mm3。70 ℃保温10 min后,迅速在冰上放置2 min。短暂离心数秒后,加入5×M-MLV buffer 4 mm3,10 mmol/dm3dNTP mixture 1 mm3,Reverse Transcriptase M-MLV 1 mm3,RNAase inhibitor 0.5 mm3。混匀后42 ℃保温1 h。70 ℃保温15 min后,冰上冷却,得到cDNA,可用于PCR扩增。
1.2.2 生物信息学分析 根据实验室前期测定的E.gryllus转录组,我们获得了其溶菌酶的全长序列,命名为基因EgLyz。将获得的基因EgLyz用DNAMAN确定正确的开放阅读框,并翻译成氨基酸序列。利用NCBI(美国国家生物技术信息中心)服务器,BLAST比对分析基因EgLyz核酸序列和氨基酸序列;利用SignalP 4.1分析信号肽及切割位点;利用ProtParam预测序列分子式、分子量和等电点;通过Pfam数据库的搜索及SMART软件在线分析蛋白结构域组成;用Predict Protein(https://www.predictprotein.org/)进行空间结构中二硫键及其位置的预测;将获得的E.gryllus溶菌酶氨基酸序列和其他动物不同类型溶菌酶序列进行比较,然后用MEGA程序中邻接法作出溶菌酶的分子系统进化树,节点上的数字表示1 000次复制的自展值,用于检验所计算的进化树分支可信度。
1.2.3EgLyz基因克隆 根据基因EgLyz的开放阅读框除去信号肽序列后设计特异性PCR扩增正反引物,正向引物:5′-CGGGATCCCAAGACTCCACCTGTCTAGGGTG-3′(下划线表示BamH I酶切位点);反身引物:5′-CGGAATTCTCACCCTGTAGCAGCTGTCAACACG-3′(下划线为EcoRI酶切位点)。在50 mm3反应体系中加入cDNA模板量为100 ng, 10 mm35×Primerstar Buffer(含Mg2+),4 mm3dNTP(2.5 mmol/dm3),0.5 mm3正向引物F(20 μmol/dm3),0.5 mm3反向引物R(20 μmol/dm3), 0.5 mm3Primerstar HS DNA Polymerase(2.5 U/dm3),剩余用H2O补齐至总体积50 mm3。PCR反应条件为:98 ℃先预变性1 min;98 ℃再变性10 s;之后58 ℃退火30s;72 ℃延伸1 min,总共30个循环;最后再72 ℃延伸10 min;4 ℃保存。
1.2.4 重组质粒pET-His-EgLyz构建及鉴定 上述PCR产物回收后利用BamH I和EcoR I对产物及pET-His进行双酶切。用琼脂糖凝胶电泳进行酶切产物分离,之后利用DNA胶回收试剂盒分别对EgLyz基因片段和pET-His载体进行回收。将两者16 ℃连接过夜(12~16 h)。构建好的重组表达质粒命名为pET-His-EgLyz。然后将连接产物转化到E.coliTOP10中,在超净工作台中涂布到具有氨苄抗性的固体培养皿上,挑取阳性单菌落进行测序,对测序结果进行分析。
1.2.5 重组质粒pET-His-EgLyz表达 将构建好的重组质粒pET-His-EgLyz转化至E.coliBL21 中,挑取单菌落接种到5 cm3含有氨苄抗性(1∶1 000)的LB液体培养基中(10 g/dm3胰蛋白胨,10 g/dm3NaCl,5 g/dm3酵母提取液),在37 ℃摇床中培养过夜12 h左右,次日取0.5 cm3液体培养基接种到含有氨苄抗性的250 cm3的LB培养基中(1∶1 000),37 ℃震荡培养2 h后(吸光度为0.6~0.8),取出1 cm3作为诱导前对照,然后加入IPTG至终浓度为1.0 mmol/dm3,在摇床中诱导表达5 h。用质量分数为12%的SDS-PAGE电泳鉴定诱导前后样品。
1.2.6 重组质粒pET-His-EgLyz表达纯化 收集上述诱导表达后的菌体使用磷酸盐缓冲液(PBS,0.1 mol/dm3NaH2PO4,0.1 mol/dm3Na2HPO4,pH 7.4)重悬,在冰浴条件下对收集的菌体悬浮液进行超声破碎(超声破碎参数:工作5 s,间隙20 s,全程时间30 min,功率200 W左右)。13 000 r/min离心10 min,将所得上清和包涵体沉淀收集起来,用裂解液(0.5 mol/dm3NaCl,0.02 mol/dm3咪唑,0.02 mol/dm3Na3PO4,8 mol/dm3尿素,pH7.4)重悬包涵体,悬浮液置于室温下孵育过夜。次日于13 000 r/min离心10 min,并收集上清溶液,再与His标签蛋白纯化介质在4 ℃条件下孵育结合3h。去除上清液后,再用含有0.02 mol/dm3咪唑的洗脱液(0.5 mol/dm3NaCl,0.02 mol/dm3咪唑,0.02 mol/dm3Na3PO4,8 mol/dm3尿素,pH 7.4)进行漂洗,最后用含有不同咪唑浓度的洗脱液把目的蛋白从介质上洗脱下来并收集。
将纯化好并且条带单一的目的蛋白转移到透析袋中,在4 ℃条件下依次用含有6.00、4.00、3.00、2.00、1.00、0.50、0.25、0.00 mol/dm3尿素磷酸缓冲液(0.5 mol/dm3NaCl,0.02 mol/dm3咪唑,0.02 mol/dm3Na3PO4,pH 7.4)进行透析复性。复性成功后,将目的蛋白留样进行SDS-PAGE电泳鉴定,剩余保存于-80 ℃备用。
1.2.7 蛋白质印迹法检测重组蛋白 纯化或复性样品电泳结束后,取出凝胶放置于滤纸上,PVDF膜置于凝胶上层,之后放置于预冷电转缓冲液中,于4 ℃冰箱中300 mA下电转1 h;取出PVDF膜,置于含5%脱脂牛奶封闭液中,室温封闭1 h;加入封闭液稀释的一抗,4 ℃孵育过夜;PVDF膜用TBST缓冲液漂洗3次,再用封闭液稀释的二抗室温孵育1 h,再漂洗3次,之后与化学发光底物反应5 min,暗室曝光显影至出现颜色深度适宜的条带。
2 结果与分析
2.1 EgLyz cDNA全长序列的特征分析
EgLyz该基因全长987 bp,5′非编码区为234 bp,3′非编码区为246 bp,其完整的阅读框架为507 bp,编码168个氨基酸。经SignalP 4.1分析,前34个氨基酸为信号肽,从而可以判断其可分泌到胞外。去除信号肽后,成熟肽是由134个氨基酸组成(图1)。ProtParam分析结果表明EgLyz基因成熟肽的相对分子质量为14.5 kDa,理论等电点为5.58。经SMART分析,该基因具有一个失稳酶结构域(36~159 aa)。Predict Proten软件分析表明EgLyz基因含有13个半胱氨酸残基,已检测到1对(Cys56 和 Cys159)二硫键,而二硫键在维持蛋白构象及活性方面起到重要作用[17]。

图1 E.gryllus溶菌酶基因 EgLyz核苷酸序列及其氨基酸序列
通过NCBI对基因EgLyz序列同源性进行比对分析,结果表明基因EgLyz与数据库中已知同源序列相似性都比较低,与果蝇(Drosophilagrimshawi)溶菌酶同源相似性序列最高达到62%,与凡纳滨对虾(Litopenaeusvannamei)溶菌酶一致性达到56%,与克氏原鳌虾(Procambarusclarkii)一致性为50%。进一步,根据基因库中已经报道的凡纳滨对虾、克氏原鳌虾(Procambarusclarkia)、青蟹(Scyllaparamamosain)及其他生物包含的溶菌酶序列,利用MEGA软件建立溶菌酶的进化树。从构建的系统进化树结果可以看出,溶菌酶在不同动物体内可以分为3大组,第一组属于节肢动物体内的溶菌酶;第二组为贻贝、鲍鱼等软体动物的溶菌酶;第三组为老鼠、牛等脊椎动物的溶菌酶。基因EgLyz与斑节对虾(Penaeusmonodon)等节肢动物的溶菌酶聚为一支(图2)。
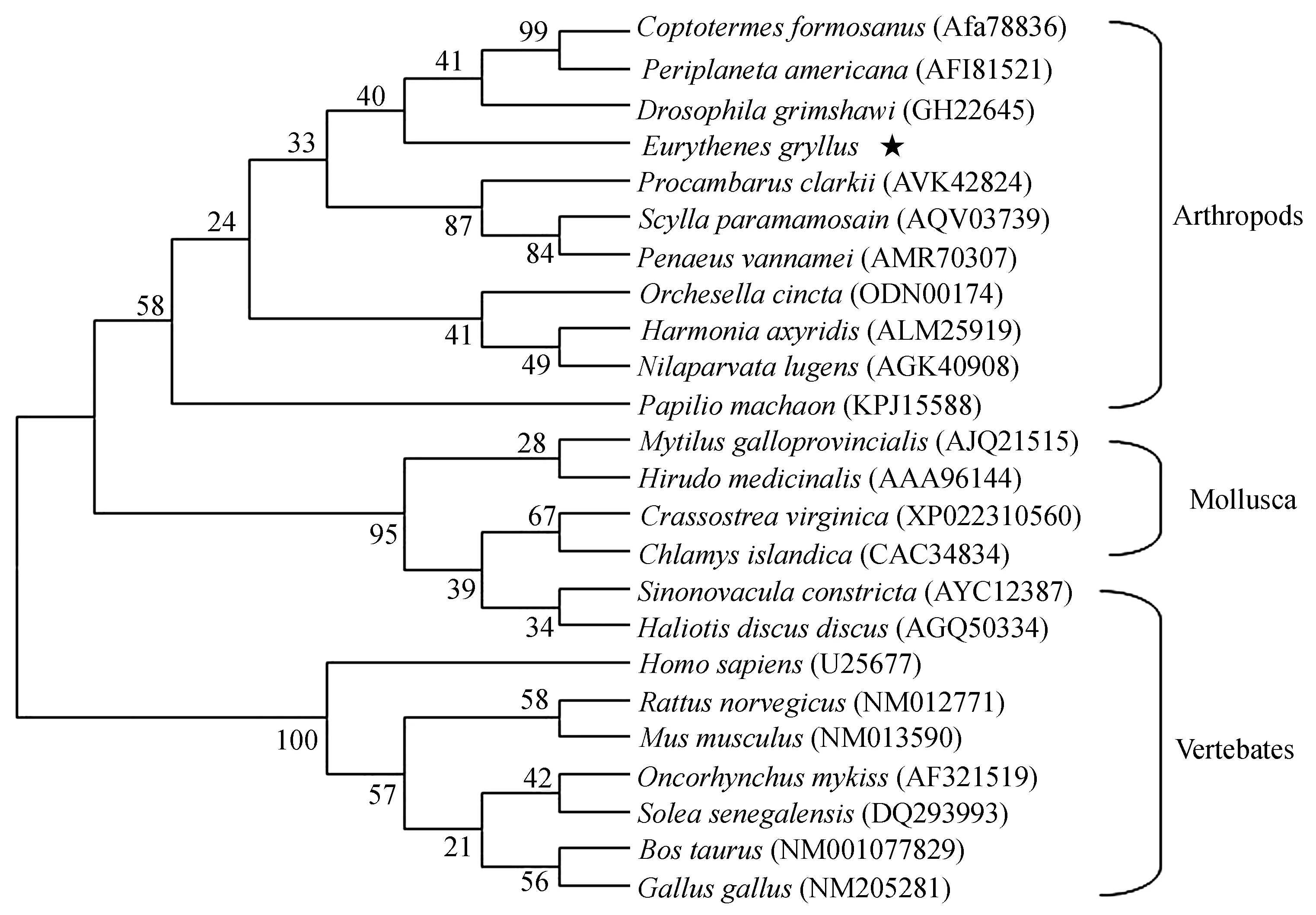
图2 不同生物体内溶菌酶的系统进化树
2.2 pET-His-EgLyz重组质粒的构建
根据基因序列设计引物,对EgLyz基因进行PCR扩增,经1%琼脂糖凝胶电泳检测扩增产物后,能在500 bp左右看到一条非常单一、特异性很高的条带,与实际目的片段大小相符(图3)。

图3 E.gryllus 中溶菌酶基因克隆
将扩增后得到的EgLyz基因与pET-His 连接,构建重组表达质粒pET-His-EgLyz。转化TOP10,挑取单菌落做菌落PCR,挑取目的大小正确的阳性克隆子送测序分析。测序结果表明,我们成功构建了重组表达质粒pET-His-EgLyz。
2.3 EgLyz的重组表达与纯化
将重组表达质粒pET-His-EgLyz转化E.coliBL21中,当OD600在0.6~0.8后,加入IPTG诱导5h,结果显示能够较明显地表达目的蛋白,进一步破碎后分析表明,大量表达的重组蛋白主要以包涵体形式存在(图4)。
在此基础上,利用融合的His 标签对目的蛋白进行纯化,可以看到用分别含有0.2 mol/dm3和0.3 mol/dm3咪唑的两种洗脱液能洗脱出浓度比较高的目的蛋白(图5a)。但是在35 kDa左右的位置也有一条比较弱的单一条带,经Western blot 分析为二聚体条带(图5b)。

图4 pET-His-EgLyz诱导后重组表达产物SDS-PAGE电泳分析结果

图5 SDS-PAGE电泳分析EgLyz的纯化结果
随后在尿素变性的条件下,通过逐渐降低缓冲液中尿素浓度的方法,对纯化的蛋白进行透析,使尿素浓度逐渐减少到0。蛋白在透析袋复性成功后,SDS-PAGE电泳分析检测结果显示目的蛋白能够较好地溶解在缓冲液中,并且在15 kDa左右能够看到一条单一的条带,条带大小与预测EgLyz基因大小相符(图6)。

图6 Western blot 分析EgLyz复性产物
2.4 讨论
溶菌酶是一种天然的蛋白质,作为营养物质可以被人体胃肠消化和吸收,并且不会残留和产生毒害作用,因此被广泛的应用在食品和医学上。在食品方面主要是作用于各种食品如水产品类熟制品、肉类制品的防腐和保鲜;在医学上,溶菌酶作为酶类抗菌药,能参与粘多糖代谢。已有报道,在诊断白血病方面,只要检查病患者的尿或者血清中溶菌酶活性,就可以确诊[18]。在构成机体非特异性免疫功能方面,溶菌酶也是重要的因素之一,因此溶菌酶在体育方面发挥着重要作用,根据俄罗斯巴.戈托夫米夫的报道,他认为运动员在良好状态时,其唾液溶菌酶的滴度值下降,因此溶菌酶可以用来对运动员身体进行评定[19]。而在科研上,溶菌酶作为工具酶在基因工程及细胞工程方面也发挥着重要作用,可以用来生产和提取菌体的活性物质如酶、核酸和活性多肽等[20]。因此,各方面来说溶菌酶都具有广阔的应用前景。由于溶菌酶作用特异性比较强,并且每个类型的溶菌酶最适的反应条件都不同,所以开发新型溶菌酶就显得至关重要。
雅浦海沟存在于地球上的典型的极端环境,其独特的环境条件会使得在这里生存的生物都具有一些特殊的生物活性分子,因此该极端环境中可能蕴含庞大的基因资源库,具有巨大的潜在应用价值。
3 结论
本研究以深海钩虾E.gryllus为材料,首次从E.gryllus中克隆获得i型溶菌酶基因EgLyz。近年由于对超深渊生物研究比较少,深海生物的基因资源库还存在严重缺失,因此基因EgLyz序列分析与已知序列相似性较低,最大相似度与果蝇(Drosophilagrimshawi)也只有62%,因此EgLyz可能是一个新颖的溶菌酶蛋白。EgLyz含有一个失稳酶结构域,13个半胱氨酸残基,已检测到1对二硫键,因此蛋白构象非常稳定。
通过克隆,EgLyz基因成功进行了原核重组表达,并且在复性时对复性条件不断进行优化,最终获取了无其他杂蛋白较高纯度的可溶性表达产物,本研究实验结果为下一步EgLyz基因生物活性及功能分析奠定了坚实的基础,也丰富了i 型溶菌酶基因资源,这将为E.gryllus在深海这种极端环境中的适应机制的研究提供帮助。

