猪热休克蛋白40Hsp40基因的克隆及过表达和RNA干扰载体的构建
卓严玲 颜秋 邓启霞

摘要:热休克蛋白(Hsp)是在各种生物体内广泛分布的一类具有高度保守性、短时性、多样性等特点的热应激蛋白质,参与细胞的多种生理生化活动。已有研究表明,Hsp40在多种病毒感染细胞的过程中发挥重要作用,但其对猪圆环病毒2型(PCV2)复制的影响及机制尚不清楚。为了探究Hsp40在PCV2感染中的生物学功能,在克隆猪Hsp40基因CDS区全基因序列的基础上,采用双酶切位点法分别构建猪Hsp40基因的过表达载体和RNA干扰载体。该研究为猪Hsp40转基因细胞株的构建搭建好平台,同时也为挖掘猪Hsp40对PCV2感染的响应机制提供极大的便利。
关键词:猪热休克蛋白40;过表达载体;RNA干扰载体;猪圆环病毒2型
中图分类号: S852.65+9.2; Q78 文献标志码: A 文章编号:1002-1302(2019)17-0063-04
猪圆环病毒2型是引起猪圆环病毒相关疾病(PCVAD)的主要病原体[1-2],我国于2001年首次报道该病,目前已成为危害我国规模化猪场养猪生产的重要传染病之一[3-4]。然而,直到现在人们对PCV2的复制机理和致病机制仍然不是很清楚,尤其对PCV2感染过程中病毒DNA和蛋白与宿主胞内蛋白之间的相互作用网络及其所蕴含的生物学意义更是知之甚少。PCV2作为能够感染哺乳动物的最小病毒之一,其基因组的编码能力有限,必须依赖于宿主细胞内的酶类才能完成复制增殖,因此PCV2不得不借助其病毒DNA和蛋白与宿主细胞内的各种基因相互作用来调控宿主免疫应答,如导致细胞因子分泌失调、免疫抑制和疾病等,从而实现其对病毒复制和致病机制的调控[5]。其中,有关PCV2衣壳蛋白Cap与猪热休克蛋白40(Hsp40)互作的文献报道最早见于2009年,但至今仍无关于此研究的新进展,特别是它们二者之间是否真的互作以及这种互作所蕴含的生物学意义目前尚不清楚[5-6]。
Hsp40是一类跨物种存在的蛋白家族,存在于各种生物体的线粒体、核糖体、内质网等细胞器中,并对蛋白翻译、折叠、去折叠和转运等起重要作用[7],同时还可以通过与病毒蛋白或病毒复制复合体结合对多数病毒的复制产生影响[8]。其中,不仅有很多动物病毒和植物病毒,而且还涵盖了许多DNA和RNA病毒。因此,既然已有研究表明PCV2 Cap与宿主Hsp40蛋白之间存在相互作用,那么就有必要进一步探究Hsp40对PCV2复制的影响及机制。
目前,研究某一基因的生物学功能往往须要使其正常的表达量上调或者下调,其中最常用的方法就是构建其过表达和RNA干扰载体,其中双酶切位点法又是构建转基因载体的最普遍方法。为此,本研究利用双酶切位点法分别构建猪Hsp40基因的过表达和RNA干扰载体,从而为其进一步的遗传转化和稳定表达细胞系的建立奠定基础,同时为进一步明确猪Hsp40基因在PCV2病毒感染过程中的生物学功能提供了高效便捷的方法。
1 材料与方法
1.1 材料
1.1.1 组织、菌株及载体 脾脏组织采自3日龄美系大白猪,该猪呈CSFV、PRRSV和PCV2阴性;大肠杆菌DH5α,购自天根生化科技(北京)有限公司;真核表达载体pEGFP-C1和RNA干扰载体pCDH-U6-MCS-EF1-GreenPuro(简写为pCDH-U6),均由西北农林科技大学张彦明教授馈赠。本试验于2018年上半年在玉林师范学院生物与制药学院基因工程实验室完成。
1.1.2 主要试剂 限制性内切酶(EcoRⅠ和BamHⅠ)、T4 ligase、PrimeScriptTM RT reagent kit with gDNA Eraser和Trizol,均购自TaKaRa公司;胶回收试剂盒、高纯质粒小量提取试剂盒和UltraPower核酸染料,均购自北京百泰克生公司;DL 2000和Trans 2K plus DNA marker,均购自北京全式金公司;Pfu高保真扩增酶和溴化乙锭(EB),均购自Thermo Fisher Scientific公司;琼脂糖,购自美国BD公司。
1.2 方法
1.2.1 引物的设计与合成 根据GenBank中登录的猪Hsp40基因序列(XM_003131409),利用Primer Premier 6.0软件设计出用于扩增猪Hsp40基因的引物,上游引物Hsp40-F:5′-CGGAATTCTATGGCGGCTGCCGCGGAGTGCGATG-3′(下划线为EcoRⅠ酶切位点),下游引物Hsp40-R:5′-CGGGATCCTTACTTATCGTCGTCATCCTTGTAATCGCCAAACTGGAAAAAGAAATTC-3′(下划线为BamHⅠ酶切位点,斜体为Flag标签序列)。待测出克隆获得的猪Hsp40基因序列后,利用Thermo Fisher Scientific公司的shRNA在线设计软件(https://rnaidesigner.thermofisher.com/rnaiexpress/)设计出猪Hsp40基因的5对干扰序列和1对阴性对照序列[9],引物信息详见表1。为载体构建鉴定需要,本研究参照文献[9]合成了上游引物5′-TTCTTGGGTAGTTTGCAGTT-3′和下游引物5′-CGGAGCCAGTACACGACA-3′,分别命名为U6-F和U6-R。以上引物全由北京华大基因公司合成。
1.2.2 组织总RNA提取与cDNA的制备 取100 mg猪脾脏组织置于研钵中,加入适量液氮并研磨完全后,将样品移入离心管中,利用Trizol法抽提总RNA,具体方法参见文献[10]。待测定出所提取总RNA浓度后,按照PrimeScriptTM RT reagent kit with gDNA Eraser说明书制备cDNA。
1.2.3 猪Hsp40基因的扩增 以上述制备的cDNA为模板,Hsp40-F和Hsp40-R為上、下游引物,利用Pfu高保真酶进行PCR扩增,同时设立阴性对照。反应体系为:Hsp40-F 1.0 μL、Hsp40-R 1.0 μL、2×Pfu高保真酶10.0 μL、cDNA 1.0 μL,ddH2O加至20 μL。反应程序包括:95 ℃ 5 min;94 ℃ 40 s,68 ℃ 40 s,72 ℃ 50 s,35个循环;72 ℃ 10 min;4 ℃ 结束反应。PCR产物用12 g/L琼脂糖凝胶进行电泳检测,然后置于凝胶成像系统中观察并拍照。
1.2.4 猪Hsp40基因真核表达载体和RNA干扰载体的构建与鉴定 将上述含有目的基因片段的凝胶用手术刀快速切下并放置于2.0 mL的离心管中,按照胶回收试剂盒的说明书纯化出PCR产物,置于-20 ℃保存。将纯化后的PCR产物和真核表达载体pEGFP-C1分别用EcoRⅠ和BamHⅠ进行双酶切,反应体系为:10×K buffer 2.0 μL、BamHⅠ 1.0 μL、EcoRⅠ 1.0 μL、PCR产物或pEGFP-C1载体加至20 μL。反应条件为:37 ℃ 2 h,加入2 μL 10×Loading buffer终止酶切反应。酶切产物再次经电泳和胶回收后,利用T4 DNA连接酶于 4 ℃ 过夜条件下将PCR产物连入pEGFP-C1载体,反应体系为:T4 ligase 1.0 μL、T4 ligase buffer 1.0 μL、pEGFP-C1载体1.5 μL、PCR产物加至20 μL,最终得到重组 pEGFP-Hsp40载体。将连接产物转化大肠杆菌DH5α感受态细胞,挑取单克隆菌置于LB液体培养基(Kan+)中扩大培养 12~16 h,按高纯质粒小量提取试剂盒说明书提取细菌质粒,经EcoRⅠ和BamHⅠ双酶切验证后,送由北京华大基因公司测序。应用DNA Star软件(MegAlign)对克隆获得的猪Hsp40基因序列进行比对分析。将测得的猪Hsp40基因序列拷贝进Thermo Fisher Scientific公司的shRNA在线设计软件并按其说明设计出相应的RNA干扰序列,将合成的各shRNA干扰序列按照事先设定好的PCR程序(95 ℃ 30 s;90 ℃ 30 s;85 ℃ 30 s;80 ℃ 30 s;75 ℃ 30 s;70 ℃ 30 s;65 ℃ 30 s;60 ℃ 30 s;55 ℃ 30 s;50 ℃ 30 s)进行退火,然后使用T4 DNA Ligase将其与经Eco RI和Bam HI双酶切过的pCDH-U6干扰载体进行4 ℃过夜连接,分别得到5条干扰载体(pCDH-U6-Hsp40-sh1、pCDH-U6-Hsp40-sh2、pCDH-U6-Hsp40-sh3、pCDH-U6-Hsp40-sh4、pCDH-U6-Hsp40-sh5)和1条阴性对照载体(pCDH-U6-Hsp40-shN),分别简写为Hsp40-sh1、Hsp40-sh2、Hsp40-sh3、Hsp40-sh4、Hsp40-sh5和Hsp40-shN,统称为重组干扰载体。以构建的各重组干扰载体及pCDH-U6空载体为模板,利用引物 U6-F/U6-R 进行PCR扩增,反应条件为:95 ℃ 5 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 10 min;4 ℃ 保存。PCR产物加入12 g/L琼脂糖凝胶中进行核酸电泳检测,检测正确后送由北京华大基因公司测序。
2 结果与分析
2.1 猪脾脏总RNA的提取
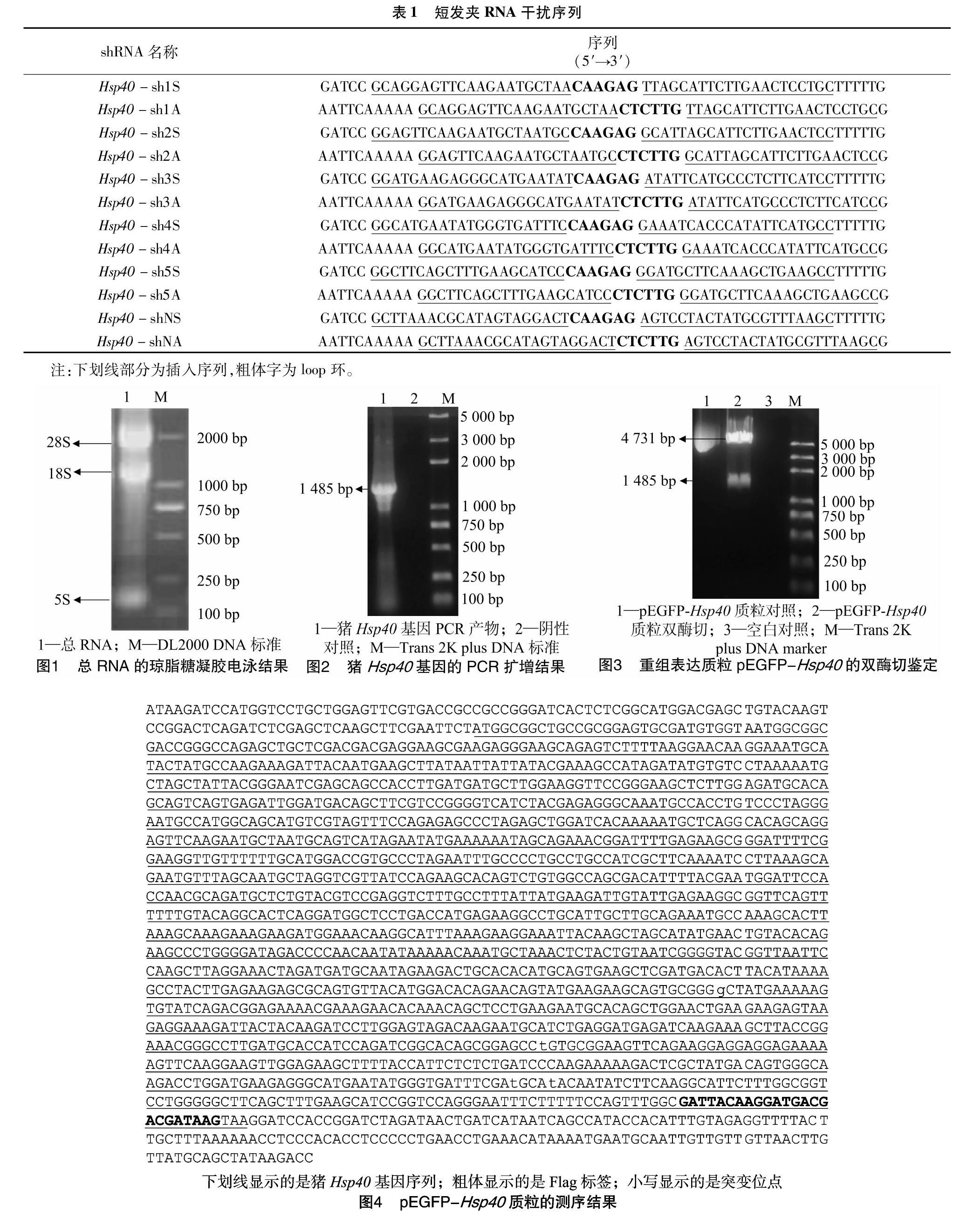
利用Trizol法提取猪脾脏组织总RNA后,取5 μL加入12 g/L琼脂糖凝胶中进行检测分析,结果可见3条清晰的电泳条带,从下往上分别是5S rRNA、18S rRNA和28S rRNA,且条带亮度28S ∶ 18S≥1(图1)。经核酸浓度测定仪检测,其D260 nm/D280 nm=1.96,浓度为780 mg/L,表明所提取的总RNA纯度较高、完整性良好,适用于后续的cDNA制备。
2.2 猪Hsp40基因的克隆
以猪脾脏组织总RNA反转录得到的cDNA为模板,Hsp40-F/Hsp40-R为引物,进行RT-PCR反应。反应结束后,将PCR产物加入12 g/L琼脂糖凝胶中检测分析,结果见图2。可见1条大小约为1 485 bp的DNA条带,与预期结果一致。
2.3 猪Hsp40基因真核表达载体的构建和鉴定
将构建好的重组质粒pEGFP-Hsp40经EcoRⅠ和 BamHⅠ 双酶切,反应体系经12 g/L琼脂糖凝胶电泳检测,见图3。可见1条大小约为4 700 bp载体片段和1条大小约 1 485 bp 的目的基因片段,与预测的片段大小基本一致,说明 pEGFP-Hsp40重组过表达载体构建成功。为了进一步验证其正确性,将酶切鉴定正确的质粒送北京华大基因公司测序并将测序结果与已报道的猪Hsp40基因序列进行比对分析,结果发现二者之间的核苷酸序列同源性为99.8%,氨基酸序列同源性为99.4%,说明猪Hsp40基因克隆成功且构建的pEGFP-Hsp40重組过表达载体成功,测序结果如图4所示。
2.4 猪Hsp40基因RNA干扰载体的构建和鉴定
以构建好的猪Hsp40基因RNA干扰载体和pCDH-U6空载体为模板,U6-F/U6-R为引物,进行PCR扩增,扩增产物置于12 g/L琼脂糖凝胶电泳中检测,结果见图5、图6。可见Hsp40-sh1、Hsp40-sh2、Hsp40-sh3、Hsp40-sh4、Hsp40-sh5、Hsp40-shN和pCDH-U6干扰质粒经引物 U6-F/U6-R PCR扩增后分别得到大小约为359、359、359、359、359、359、300 bp的DNA条带,表明各干扰序列均已被成功插入到pCDH-U6载体,即猪Hsp40基因各重组干扰载体构建成功。
3 讨论与结论
自dsRNA被发现以来,RNA干扰技术逐渐称为研究基因功能的一种有效方法,其形成的发夹RNA通过碱基互补配对识别并降解与之高度保守的同源mRNA,从而使基因沉默[11-14]。近年来,由于RNA干扰技术具有高效、简便和特异性强的特点,其已被广泛应用于各种生物基因表达调控和功能基因的挖掘与鉴定等领域,尤其在动物、植物和微生物等的品质改良中得到成功应用[15-17]。目前,尽管载体构建的方法多种多样,构建策略也逐步完善,但传统的“酶切-连接”方法因其操作简便、成本低廉仍是应用最普遍的方法。本研究利用“酶切-连接”方法成功构建了猪Hsp40基因的真核过表达载体和RNA干扰载体。
由于有关猪Hsp40蛋白生物学功能的研究相对较少,所以目前市场上或各大实验室都还没有该蛋白抗体的文献报道,笔者在构建其真核表达载体时,在猪Hsp40基因的C端添加了1个Flag标签,使将来便于猪Hsp40蛋白的检测。经测序发现,插入的Flag标签序列与当初设计一致,没有突变和移码现象;与此同时,测得的猪Hsp40基因序列与GenBank已发表的猪Hsp40基因的序列同源性达99.8%,氨基酸同源性达99.4%,证明克隆获得的猪Hsp40基因正确,可用于后续RNA干扰序列设计的靶基因。
将克隆获得的猪Hsp40基因序列放入Thermo Fisher Scientific公司的shRNA在线设计软件,笔者设计了其RNA干扰序列,且此干扰序列的设计是一次性合成的,既避免了原始的“酶切-连接”方法无一例外地需要多轮酶切连接反应,又尽可能地降低成本。由于设计出的每一条shRNA干扰序列引物只有59 bp,因此将合成后的各上、下游引物经连续退火后直接与已经双酶切过的pCDH-U6干扰载体相连接。鉴于59 bp这个长度太短,市场上也很少有如此低标准的DNA marker可用,采用PCR方法和基因测序法对插入的各个shRNA干扰序列进行检测验证是可行的。由于笔者是根据pCDH-U6干扰载体的全基因序列在其多克隆位点的上游和下游分别设计引物(U6-F/U6-R)且扩增出来的基因片段大概为300 bp,因此一旦设计出的RNA干扰序列成功插入pCDH-U6干扰载体,那么再用引物(U6-F/U6-R)扩增构建好的各重组干扰载体时就将会得到1条约360 bp DNA电泳条带,由于在DNA凝胶电泳中360 bp显著大于300 bp,因此可以根据PCR扩增条带的高低大致判断出所构建的载体是否正确。图5、图6显示,本研究构建的猪Hsp40基因RAN干扰载体经PCR鉴定是正确的,目前有关其遗传转化和转基因细胞株系建立的研究尚在进行中。本研究的载体构建策略以其步骤简便、效率可观等特点可被广泛运用于各生物表达载体及干扰载体的构建当中。本研究载体构建成功的目的是通过转基因的方式将pEGFP-Hsp40和Hsp40-shRNA转入PCV2宿主细胞中并筛选出其相对应的转基因细胞株系,从而为研究Hsp40对PCV2复制的影响和机制奠定基础。
参考文献:
[1]Dvorak C M,Puvanendiran S,Murtaugh M P. Cellular pathogenesis of porcine circovirus type 2 infection[J]. Virus Research,2013,174(1/2):60-68.
[2]Lv Q Z,Guo K K,Zhang Y M. Current understanding of genomic DNA of porcine circovirus type 2[J]. Virus Genes,2014,49(1):1-10.
[3]郎洪武,王 力,张广川,等. 猪圆环病毒分离鉴定及猪断奶多系统衰弱综合征的诊断[J]. 中国兽医科技,2001,31(3):3-5.
[4]赵明军,闫若潜,盛 敏,等. 猪圆环病毒2型致病机理研究进展[C]//中国畜牧兽医学会家畜传染病分会第八次全国会员代表大会暨第15次学术研讨会论文集. 徐州:中国畜牧兽医学会出版社,2013:81-83.
[5]Meng X J. Porcine circovirus type 2(PCV2):pathogenesis and interaction with the immune system[J]. Animal Biosciences,2013,1(1):43-64.
[6]Finsterbusch T,Steinfeldt T,Doberstein K A,et al. Interaction of the replication proteins and the capsid protein of porcine circovirus type 1 and 2 with host proteins[J]. Virology,2009,386(1):122-131.
[7]李雨轩,包 勇. 热休克蛋白40在肺癌中的研究进展[J]. 生命的化学,2016,36(4):487-490.
[8]Knox C,Luke G A,Blatch G L,et al. Heat shock protein 40(Hsp40)plays a key role in the virus life cycle[J]. Virus Research,2011,160(1/2):15-24.
[9]吕其壮. 猪圆环病毒2型ORF5蛋白功能分析和ORF4蛋白拮抗细胞凋亡机制研究[D]. 杨凌:西北农林科技大学,2016.
[10]刘 伟. 猪Jiv蛋白表达与猪瘟病毒感染相关性研究[D]. 杨凌:西北农林科技大学,2014.
[11]Fire A,Xu S Q,Montgomery M K,et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans[J]. Nature,1998,391(6669):806-811.
[12]Gunsalus K C,Piano F. RNAi as a tool to study cell biology:building genome-phenome bridge[J]. Current Opinion in Cell Biology,2005,17(1):3-8.
[13]Kusaba M. RNA interference in crop plants[J]. Current Opinion in Biotechnology,2004,15(2):139-143.
[14]Perrimon N,Ni J Q,Perkins L. In vivo RNAi:today and tomorrow[J]. Cold Spring Harbor Perspectives in Biology,2010,2(8):36-40.
[15]張瑞杰,苗向阳. 绵羊抑制素shRNA慢病毒干扰载体的构建与鉴定[J]. 华北农学报,2011,26(3):21-27.
[16]郭志鸿,张金文,王 蒂,等. 用RNA干扰技术创造高直链淀粉马铃薯材料[J]. 中国农业科学,2008,41(2):494-501.
[17]刘晓艳. HPV相关miRNA参与HPV免疫逃逸的分子机制及基于miRNA骨架的RNA干扰载体的效应研究[D]. 杭州:浙江大学,2013.

