5种文蛤属贝类线粒体基因密码子偏好性分析
张志东,陈爱华,吴杨平,张 雨陈素华,曹 奕,曹广勇,田 镇
(江苏省海洋水产研究所,江苏省文蛤良种场,江苏省海洋经济贝类研发中心,江苏南通 226007)
文蛤属(Meretrix)贝类隶属于软体动物门(Mollusca),瓣鳃纲(Lamellibrabchia),帘蛤目(Veneroida),帘蛤科(Veneridae),广泛分布于中国、朝鲜半岛、日本、亚丁湾和阿曼湾等海域。据报道,文蛤属贝类在我国主要有文蛤(M.meretrix)、丽文蛤 (M.lusoria)、斧文蛤 (M.lamarckii)、短文蛤(M.petechialis)和皱肋文蛤(M.lyrata)等几种[1],但其外形相似度高,故很多人将文蛤属贝类笼统地称为“文蛤”,忽略了种间进化的差异。长期以来,文蛤分类存在着分歧和混乱现象,已影响和制约了我国文蛤的水产养殖和遗传育种等相关研究[2]。
密码子的偏好性不仅能够反映物种的进化关系,也对研究自然选择、遗传漂变以及基因重组等[3-6]普遍现象具有重要意义。密码子是生命遗传信息传递过程中的基本单位。密码子共有64种,但是实际上编码成的氨基酸只有20种,这就存在某些多个密码子编码同一种氨基酸的现象,被称为密码子的简并性[7]。正是由于该特性,每个氨基酸至少对应1种密码子(如 Met、Trp),最多可以有1种氨基酸对应6种密码子(如Leu、Ser等)。但这些编码相同氨基酸的不同密码子,在不同物种、不同生物体中使用的频率并非完全地平均分布,仅有一部分为常用的密码子,该现象也称为密码子的偏好性。
不同基因密码子使用的偏好性存在一定的差异[2]。线粒体 DNA(mtDNA)基因组是独立于核基因组之外的双链DNA分子,记载着丰富的生物进化过程信息,与核基因组相比,其具有进化速度快、无组内特异性等特点,可作为鉴定物种起源与进化最有利的工具[8]。一般情况下线粒体基因组包含13个蛋白质编码基因,即COX1、COX2、COX3、ND1、ND2、ND3、ND4、ND4L、ND5、ND6、Cytb、ATP6以及 ATP8[9-10]。然而据王健等[11]的研究发现,文蛤线粒体中无ATP8基因,且ND4L与ND3序列长度较短,为保证序列比对的准确性,本文取5种文蛤属贝类与青蛤(Cyclina sinensis)(青蛤作为参照)线粒体基因组COX1、COX2、COX3、ND1、ND2、ND4、ND5、ND6、Cytb、ATP6基因编码蛋白(CDS)序列进行密码子偏好性分析,为探讨种属差异和近缘种之间的亲缘关系提供不同视角,以期为进一步开展文蛤功能基因表达调控研究、蛋白质结构和功能的预测以及分子育种等工作提供理论基础。
1 材料与方法
1.1 实验材料
贝类线粒体基因组CDS序列均来源于NCBI数据库(https://www.ncbi.nlm.nih.gov)。选取各物种mtDNA编码蛋白质的基因序列中长度大于400 bp的 CDS序列,每物种各10个基因(COX1、COX2、COX3、Cytb、ATP6、ND1、ND2、ND4、ND5、ND6)。

表1 6种贝类基因序列号Tab.1 Accession of gene in six species of clams
1.2 分析指标
相对同义密码子使用频率(relative synonymous codon usage,RSCU)是指对某种特定密码子在编码对应氨基酸的同义密码子间的使用概率[12]。当密码子RSCU值大于1时,代表该密码子为使用较多的密码子;当密码子RSCU值小于或等于1时,表示该密码子使用频率偏低。计算公式如下:
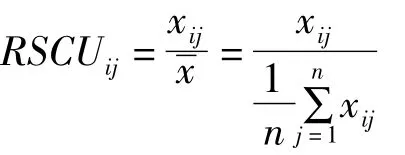
式中,xij是第i个氨基酸的第j个密码子的出现次数;n指编码第i个氨基酸的同义密码子的数量(n为1~6之间的整数);x是指编码第i个氨基酸的同义密码子总出现次数的平均值。
1.3 分析方法
用Codon W软件对5种文蛤线粒体基因组CDS序列分别进行分析,并计算相对同义密码子使用频率(RSCU)。用Excel 2007对数据进行统计,以SPSS 16.0对其密码子的偏好性进行聚类分析,最后用MEGA 5.0分析序列,采用邻接法构建进化树,最后将聚类结果与进化树结果进行对比。
2 结果与分析
2.1 6种贝类线粒体基因组CDS序列的密码子偏好性
A3s、C3s、G3s、U3s分别表示密码子的第 3位碱基中A、C、G、U的含量在第3位碱基总量中所占的比率。5种文蛤属贝类线粒体基因CDS序列中的 U3s分别为文蛤 57.83%、丽文蛤56.87%、皱肋文蛤63.23%、短文蛤58.03%、斧文蛤59.22%,青蛤则为60.34%,均大于 A3s、C3s、G3s(表2),可见5种文蛤属贝类偏爱使用以U结尾的密码子。尤其是UCU、ACU、CCU和GCU,它们的RSCU值均接近3,为5种文蛤属贝类与青蛤最常用的密码子。
用Codon W和Excel计算的结果表明(表3):在文蛤属贝类线粒体基因组中的密码子中UUU、UAU、CAU等24个密码子的RSCU值均大于1,为文蛤属贝类偏好性密码子。但5种文蛤属贝类的密码子偏好性有所不同:丽文蛤与短文蛤编码异亮氨酸(Ile)的偏好密码子只有1个AUU,其余贝类不仅有AUU,还有AUA;皱肋文蛤编码谷氨酸(Glu)的偏好性密码子为GAA,其余贝类为GAG;皱肋文蛤编码甘氨酸(Gly)的偏好密码子中无GGG,与其余贝类存在些差异;斧文蛤编码谷氨酰胺(Gln)的偏好密码子为CAG,其余贝类均为CAA;斧文蛤编码甘氨酸(Gly)的偏好密码子 GAA和 GGG,其余贝类为 GGU与GGG。青蛤编码亮氨酸的偏好密码子无UUG。
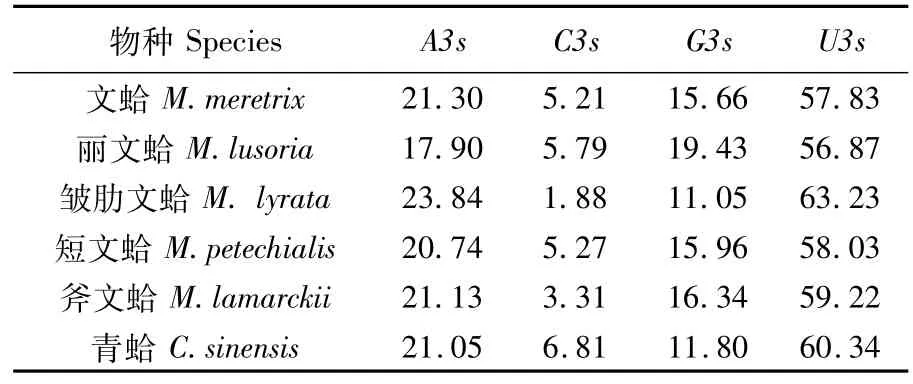
表2 6种贝类 CDS序列的 A3s、C3s、G3s、U3sTab.2 Values of A3s,U3s,G3s,C3s in CDS in six species of clams (%)
2.2 6种贝类RSCU值聚类分析
对5种文蛤属贝类和青蛤的密码子RSCU值进行聚类分析,除去编码色氨酸(Trp)和甲硫氨酸(Met)唯一的密码子的UGG和AUG,其RSCU值始终为1。再除去3个终止密码子UAA、UAG、UGA,取余下的59个密码子的RSCU值对密码子的偏好性分析。聚类结果见图1,文蛤和短文蛤先聚为一类,再与丽文蛤聚为一类;接着和皱肋文蛤及斧文蛤聚为一类,最后与青蛤聚为一类。
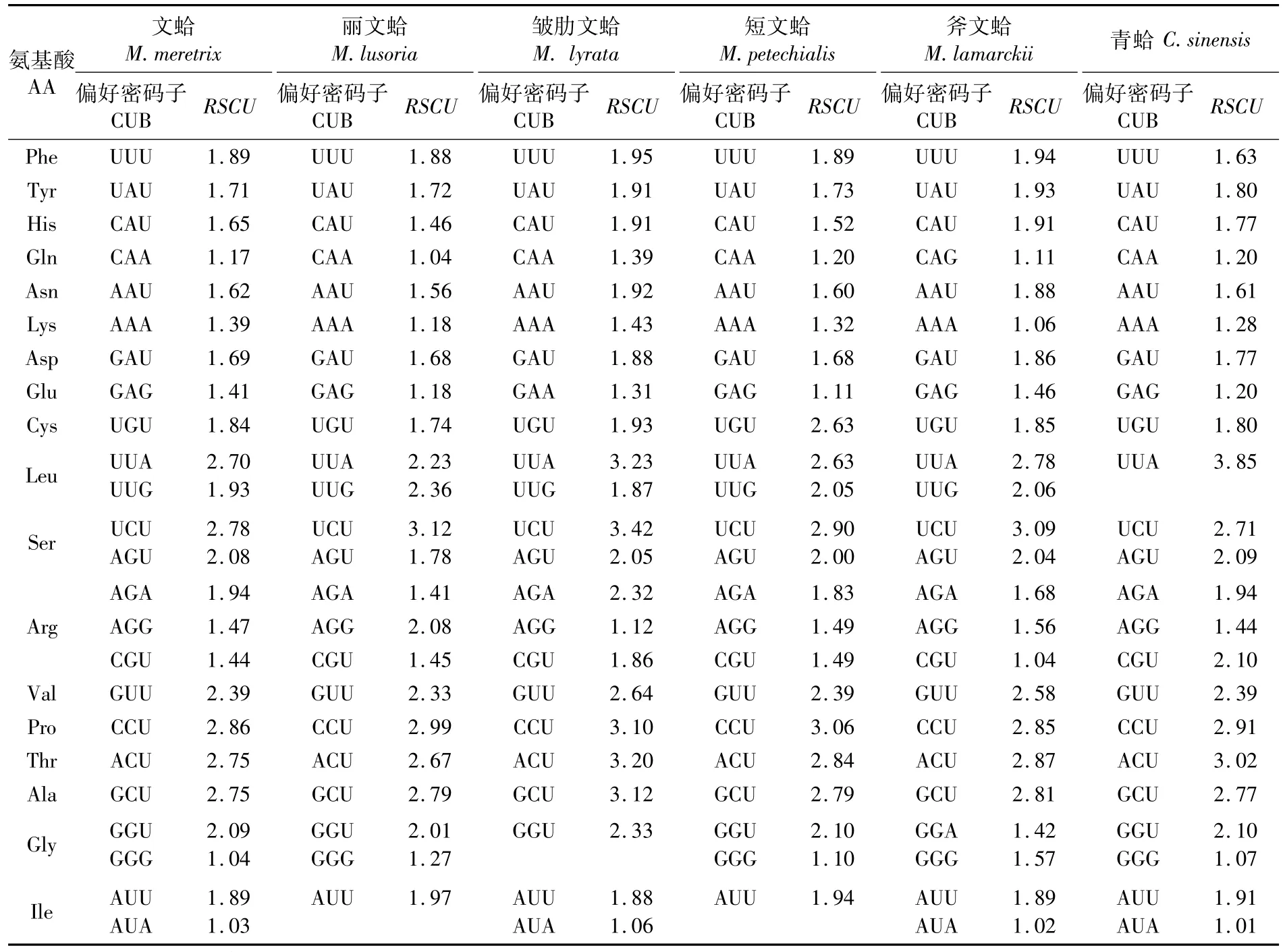
表3 6种贝类线粒体基因CDS序列偏好密码子的RSCUTab.3 RSCU values of mtDNA CDS in six species of clams
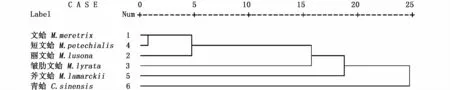
图1 6种贝类线粒体基因CDS序列密码子偏好性聚类图Fig.1 Cluster tree based on RSCU values of mtDNA CDSin six species of clams
2.3 文蛤属贝类线粒体基因序列比对构建进化树
以青蛤为外群,将所研究的5种文蛤属贝类的10个线粒体基因(COX1、COX2、COX3、Cytb、ATP6、ND1、ND2、ND4、ND5和 ND6)CDS序列用邻接法聚类分析,其结果如图2:文蛤与短文蛤在COX2、COX3、Cytb、ATP6、ND1、ND2、ND4、ND5和ND6先聚为一类,再与丽文蛤聚为一类;皱肋文蛤与斧文蛤聚为一类,再与青蛤聚为一类。5种文蛤属贝类线粒体基因系统进化树与密码子偏好聚类分析结果相似。
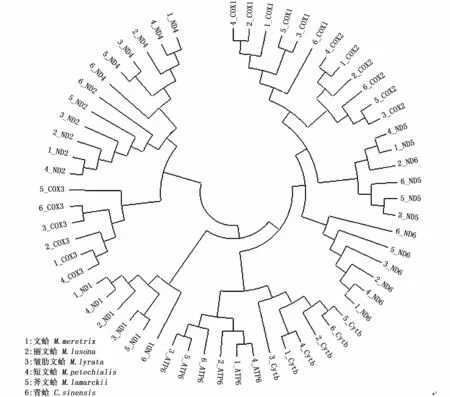
图2 邻接法(NJ)构建的文蛤属贝类与青蛤线粒体基因CDS序列进化树Fig.2 NJ phylogenetic tree based on mtDNA CDS in five Meretrix species and C.sinensis
3 讨论
基因是生物遗传信息的载体,蛋白质是发挥生物功能的主要分子。因此联系两者的密码子使用情况必然具有重要的生物学意义。在长期进化过程中,物种的基因会逐渐形成一套适应基因组的特定密码子用法[13]。文蛤属贝类线粒体基因组CDS序列偏爱使用U结尾的密码子,使用频率最高的是UCU,其次是ACU,而对C结尾的密码子使用频率最低。以丽文蛤为例,27个偏好密码子中以U结尾的有15个,而以C结尾为零,其余4种文蛤属贝类也与之相似。由于线粒体基因组存在着D-loop区,该区域富含腺嘌呤(A)和胸腺嘧啶(T)[14],这可能与文蛤线粒体基因组CDS序列偏爱使用U结尾的密码子有关。文蛤核基因组甚至全基因组是否偏爱使用U结尾的密码子,还需待文蛤全基因组测序完成之后继续研究。
物种的亲缘关系远近与密码子偏好性差异大小密切相关。亲缘关系近,则密码子偏好性差异小;亲缘关系远,则密码子偏好性差异大[15]。宋乔乔等[16]对野牦牛(Bos mutus)、普通牛(Bos taurus)、藏 羚 羊 (Pantholops hodgsonii)、山 羊(Capra hircus)和绵羊(Ovis aries)等哺乳动物线粒体基因密码子偏好性进行聚类分析,发现基于密码子偏好性差异可以揭示物种间的亲缘关系,并用于物种的分类研究。DUAN等[17]比较了14种鲌亚科鱼类密码子偏好性的差异,发现属间物种的密码子偏好性具有物种特异性,主要是受到进化过程中的突变与自然选择的影响。基因突变具有低频性,故近缘物种密码子在使用偏好性上总体相似,又有轻微差别。本研究通过对文蛤属贝类的密码子偏好性分析发现,5种文蛤属贝类密码子偏好性虽有轻微差别,但总体相似,符合上述近缘种密码子使用偏好性特点。而青蛤在密码子使用偏好性方面与文蛤属贝类有所差异,聚类结果和序列比对结果均显示青蛤与文蛤属贝类亲缘关系较远。从密码子使用偏好性来看,文蛤与短文蛤极为相似,与斧文蛤相似度最小,在亲缘关系上很可能亦是文蛤与短文蛤亲缘关系最近,与斧文蛤亲缘关系最远。潘宝平等[18]通过16SrRNA基因及ITS1序列的系统学分析,刘泽浩等[19]通过28SrRNA部分序列研究也得出类似结论。
本研究利用生物信息学的方法,在编码蛋白质水平进行了密码子偏好性分析,明确了文蛤属贝类的密码子偏好性及特征,并比较文蛤与其近缘种的密码子使用偏好性差异。研究结果可为文蛤属贝类在进化中亲缘关系远近的判定提供一定依据,并可为未来文蛤功能基因表达调控研究、蛋白质结构和功能的预测以及分子育种等工作提供理论基础。

