青鱼野生与养殖群体遗传变异的微卫星分析
王 丰 张家华 沈玉帮 , 徐晓雁 , 王荣泉 李家乐 ,
(1. 上海海洋大学农业部淡水水产种质资源重点实验室, 上海 201306; 2. 上海海洋大学上海水产养殖工程技术研究中心, 上海201306; 3. 上海海洋大学水产科学国家级实验教学示范中心, 上海 201306; 4. 苏州市申航生态科技发展股份有限公司农业部大宗淡水鱼类繁育与健康养殖技术重点实验室, 苏州 215221)
青鱼(Mylopharyngodon piceus)是我国重要的淡水养殖鱼类, 也是长江流域重要的渔业资源。随着人类活动对环境的破坏和水利设施的建设, 以及过度捕捞等情况的存在, 长江中的青鱼资源遭到了严重的破坏[1], 青鱼的数量也在急剧下降, 长江水产研究所对四大家鱼早期资源的监测显示, 青鱼的产卵量从1997年的7.68亿粒下降到了2009年的2.90万粒[1]。目前有关青鱼遗传多样性的研究数量甚少,仅有少量基于蛋白质遗传多态[2]、线粒体基因[3,4]、微卫星标记[5]遗传变异的报道。对于青鱼微卫星标记的开发, 除了利用青鱼作为样本[6], 也有使用其他物种的微卫星在青鱼上进行跨物种扩增的报道[7]。
微卫星标记由于其具有高度的多态性, 在整个基因组中随机分布, 数量十分丰富, 且具有较高的保守性, 为共显性遗传, 符合孟德尔遗传定律, 较其他分子标记具有更多的信息量[8], 因此被广泛应用于各种遗传学相关研究中[9]。微卫星标记作为研究工具对水产动物的群体遗传结构、遗传多样性分析、分子育种和种质鉴定等方面[10,11]起到了重要的推动作用。
本研究使用自主开发的12对青鱼微卫星引物对收集到的 4个野生群体和1个养殖群体的遗传多样性和遗传结构进行了分析, 以期为青鱼种质资源的评估和保护提供资料和理论支持。
1 材料与方法
1.1 实验材料
用于遗传分析的青鱼样本来源于国内国家级四大家鱼原良种场, 包括4个野生群体, 1个养殖群体。吴江养殖群体于2016年从江苏吴江四大家鱼良种场收集, 石首、邗江、嘉兴、湘江野生群体于2017年分别从湖北石首老河长江四大家鱼原种场、江苏邗江长江系家鱼原种场、浙江嘉兴长江四大家鱼原种场、湖南省鱼类原种场收集, 具体位置和信息见表 1。采集其鳍条组织, 用无水乙醇固定, 在4 ℃冰箱中保存。

表 1 五个青鱼群体采集信息Tab. 1 Sampling information of five populations of black carp
1.2 基因组DNA提取
本研究使用海洋生物基因组DNA快速提取试剂盒(天根生化科技有限公司, 北京)进行DNA的提取, 用1%的琼脂糖凝胶电泳检测DNA是否降解, 用NanoDrop 2000紫外分光光度计(Thermo, 德国)检测DNA纯度和浓度, 然后将提取的DNA转移至96孔PCR板中, 用硅胶盖封存, 置于-20℃冰箱中保存备用。
1.3 微卫星PCR扩增及分型
本研究所用的微卫星标记来自于自主开发的标记, 引物由上海迈浦生物科技有限公司合成, 上游引物在5′端修饰FAM或HEX荧光基团(表 2)。
PCR扩增体系为25 μL (2×TaqMix 12.5 μL、10 μmol/L primer 1 μL、20 ng/μL DNA 1.5 μL和ddH2O 10 μL), 并调整合适的退火温度以得到清晰和特异性高的条带。PCR反应程序为: 94℃预变性2min, 然后94℃变性30s、退火温度30s、72℃延伸1min, 重复30次循环, 最后72℃延伸10min。扩增反应在Eppendorf梯度PCR仪上进行。
每个位点单独上机, 采用的内标为ROX500, 由上海迈浦生物科技有限公司使用ABI3730XL全自动 DNA 测序仪进行微卫星分型, 获得每个个体在不同位点上的等位基因数据, 用Genemapper Version 4.0[12,13]软件进行扩增产物片段大小的读取。
1.4 数据统计和分析
根据每个位点扩增产物的片段大小确定个体在该位点的基因型, 使用POPGEN32[14]计算得到各位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Shannon多样性指数(I)、近交系数(Fis)、种群间遗传分化系数(FST)、种群间遗传距离和各位点基因频率等信息;用Cervus 3.0[15]计算各位点的多态信息含量(PIC),及Hardy-Weinberg平衡检验。基于群体间的Nei’s遗传距离使用MEGA 5.0构建UPGMA 系统树。
2 结果
2.1 微卫星位点的多态性
本实验共使用了12个位点, 这12对微卫星引物在5个青鱼群体中各个个体进行PCR扩增, 分析扩增结果得到了12个微卫星位点的遗传多样性信息(表 3)。分析结果显示这12个位点在5个青鱼群体中的等位基因数(Na)介于7—29, 有效等位基因数(Ne)介于3.347—13.730, 平均等位基因数和平均有效等位基因数分别为18.429和7.805, 其中MP20和MP65等位基因数最少都为7, MP54位点所具有的等位基因数最多为29。各位点的Shannon多样性指数介于1.442—2.833, 观测杂合度(Ho)介于0.623—0.937, 期望杂合度(He)介于0.703—0.929, 多态信息含量(PIC)介于0.660—0.923, 表明这12个位点均具有高度多态性(PIC>0.5), 种群间遗传分化系数(FST)介于0.034—0.173, Hardy-Weinberg平衡检验显示有3个位点(MP40、MP49和MP62)极显著偏离Hardy-Weinberg平衡(P<0.01)。

表 2 青鱼12个多态性微卫星引物信息Tab. 2 12 pairs of Primers from black carp
2.2 青鱼群体的遗传多样性
5个群体的遗传多样性信息如表 4所示, 结果显示5个群体的平均等位基因数(Na)介于7.917—11.667,其中嘉兴群体平均等位基因最少, 邗江群体最多;平均有效等位基因数(Ne)介于4.837—6.035, 其中石首群体最少, 邗江群体最多; 平均观测杂合度介于0.713—0.861, 其中吴江群体最低, 嘉兴群体最高;平均期望杂合度介于0.749—0.819, 其中湘江群体最低, 邗江群体最高; 平均多态信息含量介于0.711—0.788, 其中湘江群体最低, 邗江群体最高, 且多态信息含量的P值均大于0.5, 表明这5个群体均具有较高的遗传多样性。
5个群体Shannon多样性指数(I)介于1.715—1.949, 其中石首群体最低, 邗江群体最高。除邗江群体相对稍高外, 其他各群体之间相差不大, 且5个群体的shannon多样性指数从高到低排列依次为邗江>吴江>嘉兴>湘江>石首, 与各群体平均有效等位基因数的大小顺序相吻合, 说明在这5个青鱼群体中, 邗江群体具有较高的等位基因丰富度, 而石首群体的等位基因丰富度相对较低。

表 3 青鱼12个微卫星位点的遗传多样性Tab. 3 Genetic diversity information of 12 microsatellites in black carp
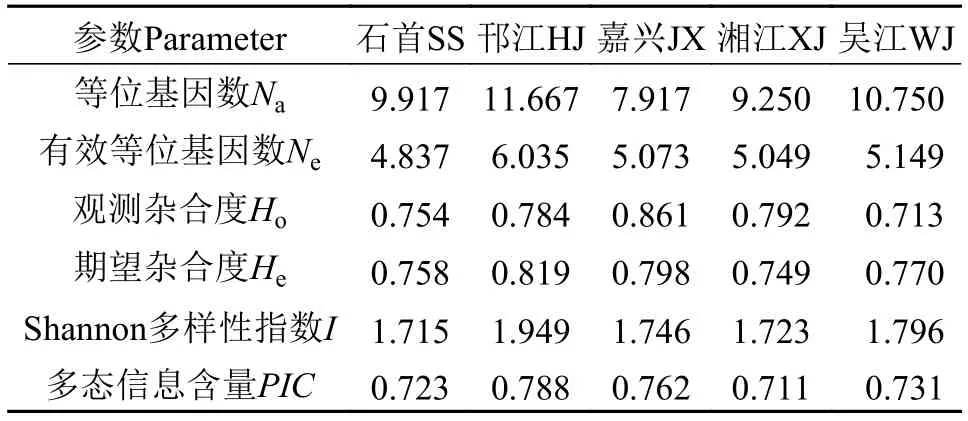
表 4 青鱼5个群体的遗传多样性Tab. 4 Genetic diversity of 5 populations in black carp
2.3 群体间遗传分化及遗传距离分析
根据12对引物的扩增结果对5个青鱼群体间的遗传距离和遗传相似度进行了分析计算(表 5)。5个群体各群体间的遗传距离介于0.207—0.875, 其中邗江群体和嘉兴群体遗传距离最小(0.207), 湘江群体和吴江群体遗传距离最大(0.875)。各群体间的遗传相似度介于0.417—0.813, 其中湘江群体和吴江群体遗传相似度最小(0.417), 邗江群体和嘉兴群体遗传相似度最大(0.813)。
基于群体间Nei’s遗传距离(DA)构建UPGMA聚类树如图 1所示, 结果显示邗江群体和嘉兴群体首先聚为一支, 然后与石首和湘江两个群体聚为一支,最后与吴江群体聚为一支。
3 讨论
3.1 微卫星位点在群体中的遗传多样性水平
多态信息含量(PIC)是评价微卫星位点的重要参考标准, 若PIC>0.5表明该位点具有高度多态性,若0.25<PIC<0.5表明该位点具有中度多态性, 若PIC<0.25表明该位点具有低度多态性。本研究所用的12个微卫星位点等位基因分布相对均匀, 所含等位基因数介于7—29, 平均多态信息含量在5个青鱼群体中均在0.7以上, 观测杂合度及期望杂合度均具有较高水平(Ho>0.7,He>0.7)。本实验中位点MP20在石首群体中(PIC=0.499)以及位点MP36和MP65在湘江群体中(PIC=0.444)表现为中度多态性(0.25<PIC<0.5), 但都接近0.5, 可能与所用群体该位点的多态性相对较低有关, 另外采样的随机性以及群体数量的大小也可能对微卫星位点的多态性分析结果产生一定的影响, 从而导致在单一群体中位点多态性低于应当表现出的高多态性水平。
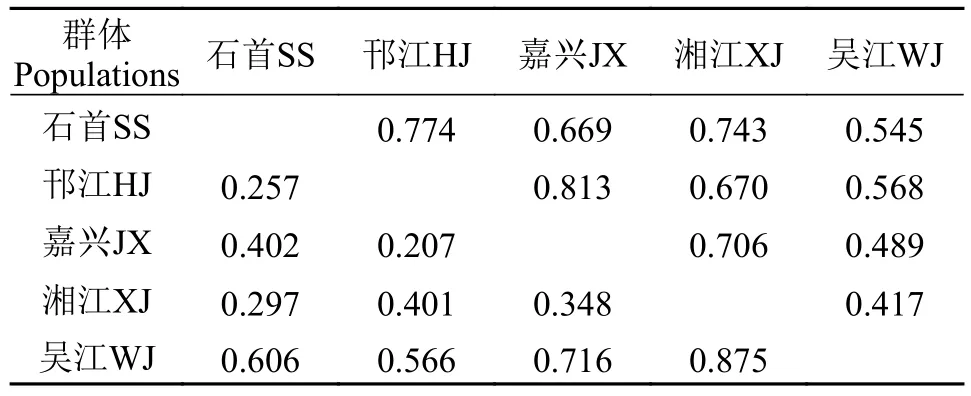
表 5 青鱼5个群体的遗传距离和遗传相似度Tab. 5 Genetic distance and genetic similarity of 5 populations in black carp
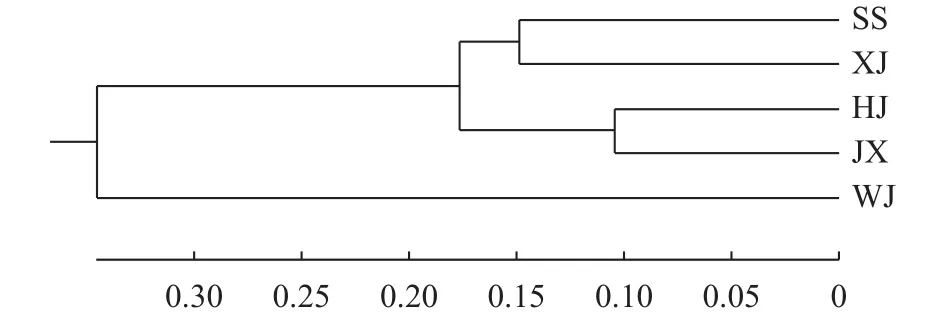
图 1 基于Nei’s 遗传距离构建的5个青鱼群体UPGMA聚类树Fig. 1 UPGMA trees among 5 populations of black carp constructed based on Nei’s genetic distance
Guo等[7]利用草鱼的微卫星标记在青鱼群体中跨物种扩增各位点扩增所得的等位基因数在0—12不等, 分布十分不均匀, 而且许多位点扩增出的等位基因数为0或1, 大多小于5; 本研究中所用微卫星标记各位点在群体中扩增所得的等位基因数明显比之要多, 且均在5以上, 与之相比, 本研究和Zhu等[6]所用的微卫星标记在青鱼群体中扩增结果较为理想。
使用其他物种的微卫星标记跨物种扩增可能出现无扩增结果[16]、所得等位基因数低于预期、扩增非同源产物[17]等问题, 这些问题可能会增加分析过程的复杂程度和降低研究结果的准确性, 在使用中需要进行进一步的筛选和验证, 而使用本物种所属的微卫星标记则可以有效避免类似问题的出现, 因此开发本物种的微卫星标记十分必要。
总体来说, 本研究所用的12个位点均具有较丰富的遗传多样性, 遗传多样性比较丰富, 可用于青鱼的遗传结构、遗传育种、遗传多样性分析等研究。
3.2 群体的遗传多样性水平
生物群体的遗传多样性是评价物种种质资源状况的重要依据之一。维持种内的遗传多样性水平, 是种质保护的最终目的, 也是种质资源能持续利用的基础。群体的有效等位基因数、观测杂合度、期望杂合度以及多态信息含量都是衡量群体遗传多样性的参考标准, 群体的基因丰富度越高,将在这些方面表现出越高的数值[18]。在本研究中,5个青鱼群体的平均观测杂合度、平均期望杂合度以及平均多态信息含量的值均在0.7以上, 说明各群体的遗传多样性水平均处于较高水平。
方耀林等[19]用RAPD法对长江水系青鱼遗传多样性进行分析, 湘江群体的平均观测杂合度为0.832, 其实验所用青鱼湘江群体与本研究样本同样采集于长沙四大家鱼原种场。在本研究中湘江群体的平均观测杂合度为0.792, 与之相比稍低, 表明2004—2017年期间青鱼湘江群体的遗传多样性可能有一定程度的降低; 在本研究中湖北石首群体平均观测杂合度明显低于其研究中湖北金口群体, 可能同样存在遗传多样性降低的情况。但由于实验所用群体数量较少, 采样时间跨度较长, 尚难以形成确定的结论, 还需进行更多相关研究以进行确认。Hunter和Nico[5]报道了引入于美国密西西比河流域青鱼的遗传多样性, 结果显示期望杂合度平均为0.224, 远低于本研究, 可能是当时引入的群体小和分析的样本少的缘故。
等位基因在群体中分布的越均匀,Ne和Na就越接近[20], 在本研究中每个群体的Ne均小于Na, 群体的平均Ne小于Na, 主要是等位基因在群体中分布不均匀, 导致某些等位基因的频率不够平均。这说明各群体均存在等位基因分布不均匀的情况, 且不均匀程度吴江>石首>邗江>湘江>嘉兴。
3.3 群体的遗传结构
基于群体间Nei’s遗传距离构建UPGMA聚类树显示嘉兴群体和邗江群体首先聚为一支, 之后和石首群体和湘江群体聚为一支, 最后与吴江群体聚为一支。从地理分布上来说, 邗江和嘉兴2个群体位于长江下游, 石首和湘江2个群体位于长江中游,这4个群体的遗传距离和地理距离具有较强的相关性, 在聚类树上表现出较为明显的东西分布。但是同处长江下游的吴江群体与邗江和嘉兴两群体的遗传距离反而较远, 并且地理距离最近的吴江群体和嘉兴群体的遗传距离反而较远, 结合采样地点水域环境类型, 推断可能与吴江群体所在水域存在太湖这一大面积湖泊环境, 而其他4个群体所在水域无类似水文环境, 从而导致吴江群体与其他4个群体产生了较大的遗传分化。关于青鱼多个群体的遗传变异的研究目前仅见方耀林等[17]的研究, 但其所做研究仅涉及长江中游3个群体, 因此无法为该情况提供更多参考。在傅建军等[21]的研究中, 在吴江、石首、邗江3个地区的草鱼群体中, 吴江和邗江聚为一支, 石首与之聚为一支, 与本研究中情况并不相同。因此对于该情况, 尚需更多进一步的研究。

