灵芝尿苷二磷酸葡萄糖焦磷酸化酶基因的电子克隆与生物信息学分析
贺望兴,杨普香,李延升,石旭平,黎小萍,张贱根,蔡海兰,蔡 翔
(江西省蚕桑茶叶研究所,江西 南昌 330203)
灵芝属于担子菌纲多孔菌属灵芝科,是极其珍贵的药用真菌。我国古代的名医及其医学巨著记载灵芝能够治疗多种疾病,具有滋补强身、扶正固本等重要功效。研究表明:灵芝主要的一类活性成分是灵芝多糖[1-2],它具有免疫调节抗肿瘤、抗辐射、抗衰老、抗氧化、调节血糖以及保肝等主要药理学作用[3]。
1953年Kalckar等在酵母细胞中首次发现尿苷二磷酸葡萄糖焦磷酸化酶(UGPase)[4],目前在植物的许多组织细胞中都发现了它的存在。UGPase在植物的非光合作用组织,尤其是储藏组织中含量较高,在植物组织细胞中的UGPase绝大多数分布在胞液里[5]。UGPase是调控合成植物多糖过程的关键酶,催化反应Glc-1-P+UTP→UDPG+PPi发生,UDPG即尿苷二磷酸葡萄糖,它是植物活化糖的主要形式,在高等植物中为各类碳水化合物包括蔗糖、纤维素、果胶质以及糖蛋白等的合成提供葡萄糖基[6]。
截至目前登陆GenBank没有发现灵芝UGPase的cDNA序列,本研究基于灵芝EST数据库和生物信息学手段,对灵芝UGPase酶基因进行电子克隆和序列分析,然后利用生物信息学软件对UGPase基因编码蛋白的理化性质、亚细胞定位、亲疏水性、信号肽、蛋白结构域、蛋白高级结构以及系统进化树的构建等方面进行了预测和分析,以期丰富灵芝基因资源,为灵芝多糖生物合成的代谢机理提供理论基础。
1 材料与方法
1.1 灵芝UGPase基因的电子克隆
利用NCBI中的Blast软件,以已知的草茎点霉(Phomaherbarum)UGPase基因(GenBank Accession: ABW96356.1)作为探针与灵芝较完整的表达序列标签(EST)数据库进行序列相似性搜索,选择EST同源性比较高的序列进行聚类、拼接、延伸,然后以新拼接的重叠群(Contig)为新探针,继续搜索数据库,直到没有新的灵芝EST序列可供拼接为止。最后将拼接完成的新基因序列确认为灵芝UGPase基因序列。
1.2 灵芝UGPase基因序列的验证
按照RNA提取试剂盒的步骤提取灵芝细胞的RNA,用cDNA第一链合成试剂盒将mRNA反转录成cDNA。根据电子克隆得到的基因序列,用Primer 5.0程序设计正向引物F:5’-CCGAGAAACAGAGGAGCA-3’,反向引物R:5’-TGAGGACGAGAAGAAGCAC-3’,预计扩增片段长989 bp。以灵芝RNA的反转录成的cDNA作为模板,PCR扩增程序为:94 ℃、5 min,(94℃、30 s, 58 ℃、30 s, 72 ℃、30 s)×30个循环,72 ℃、10 min。PCR产物用1.0%琼脂糖凝胶电泳,并将PCR产物交上海生工测序。
1.3 灵芝UGPase生物信息学分析
利用生物信息学相关软件对UGPase基因编码蛋白从氨基酸组成、理化性质、保守结构域、跨膜结构域、亲水性疏水性、亚细胞定位、信号肽、蛋白结构、同源性分析及系统进化树的构建等方面进行了预测和分析。具体软件信息如表1所示。
2 结果与分析
2.1 灵芝UGPase基因的电子克隆
通过电子克隆方法获得了一条全长为1691 bp的灵芝UGPasecDNA基因。ORF在线软件分析表明,该序列包含一个1515 bp的完整开放阅读框,编码504个氨基酸。图1为电子克隆得到的灵芝UGPase基因序列及其推导的氨基酸序列。
2.2 灵芝UGPase基因的验证
根据电子克隆得到的灵芝UGPase基因序列,用Primer 5.0程序设计引物,经PCR验证,得到一条大小约989 bp的电泳条带。如图2所示,PCR产物电泳分子量大小与基因电子克隆序列理论相吻合,可初步判断该条带是灵芝UGPase基因。该PCR产物经上海生工测序,测序结果与电子克隆的理论序列一致。
2.3 灵芝UGPase生物信息学分析
2.3.1 灵芝UGPase蛋白理化性质分析 利用ExPASy-Protparam在线软件对UGPase蛋白理化性质进行分析,结果如表2所示。灵芝UGPase基因编码504个氨基酸,蛋白质等电点为6.8、分子量大小为56830.1、不稳定系数为30.63、平均疏水性为-0.320、脂肪系数为93.41。
根据Protparam算法,不稳定系数数值小于40时,预测的蛋白是比较稳定,反之则较差[7]。分析结果表明UGPase蛋白较稳定;平均疏水性为负值,表明UGPase蛋白很可能是一个亲水性蛋白;脂肪系数是表征一个蛋白质中脂肪侧链所占的相对值,可作为球蛋白热稳定性增加的阳性因素,脂肪系数越高蛋白质越稳定,预测结果再次证明了UGPase蛋白很可能是一种稳定性蛋白。接着对UGPase基因所编码的氨基酸组成进行分析,结果如表3所示。UGPase蛋白由20种氨基酸组成,其中亮氨酸(Leu)含量最高,占10.7%;其次是赖氨酸(Lys),占7.1%;半胱氨酸(Cys)和色氨酸(Trp)的含量最低,均为0.6%。

图1 电子克隆获得的灵芝UGPase基因的cDNA序列及其推导的氨基酸序列

图2 PCR电泳图
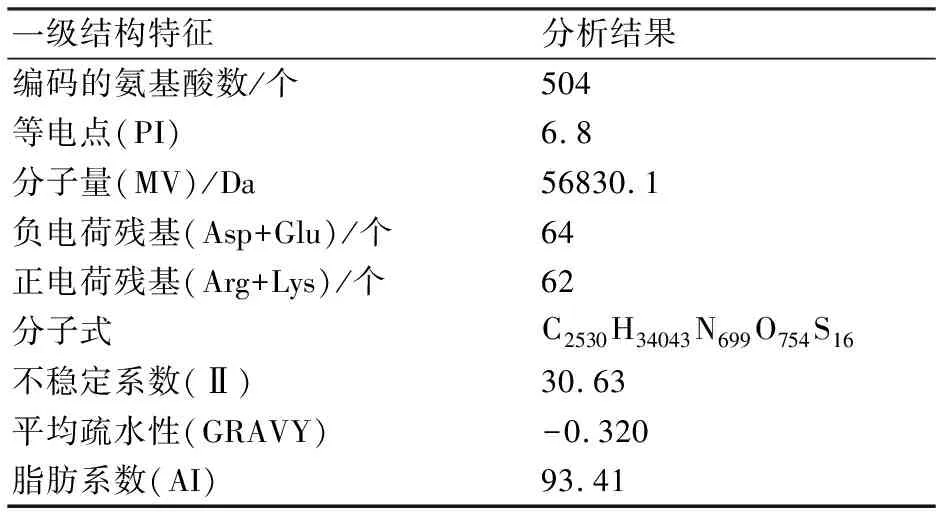
表2 UGPase基因所编码的氨基酸一级结构
2.3.2 亚细胞定位 蛋白质在细胞中的定位与该蛋白质的功能密切有关,利用ExPASy-Psort在线软件对UGPase 蛋白在细胞内的定位进行预测,结果见表4,UGPase 蛋白定位在细胞质内的概率最大为69.6%,这与报道的UGPase 蛋白主要参与植物糖类代谢调控以及纤维素合成等过程相符合[8]。
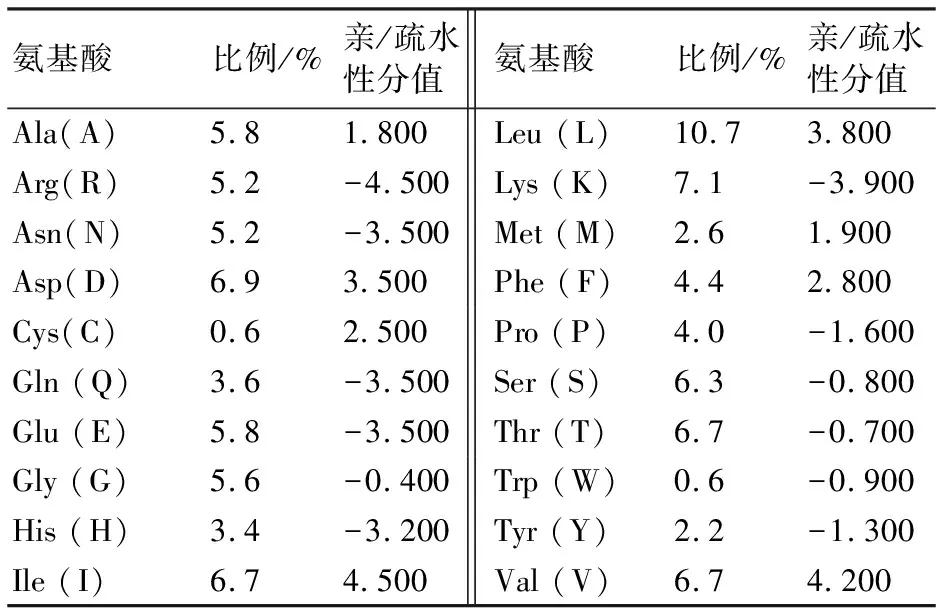
表3 UGPase基因所编码蛋白质的氨基酸组成
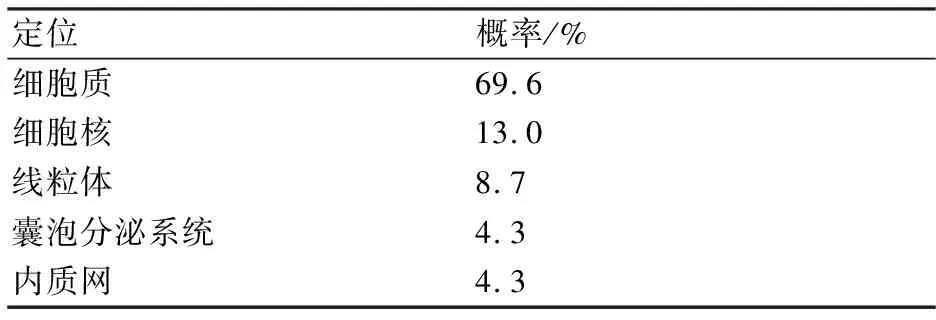
表4 灵芝UGPase蛋白亚细胞定位
2.3.3 灵芝UGPase蛋白跨膜结构域预测分析 跨膜结构域是膜内在蛋白与膜脂相结合的主要部位,一般由20个左右的疏水氨基酸组成,形成α螺旋,它固着于细胞膜上起“锚定”作用[9]。利用TMHMM-2.0在线工具预测UGPase 蛋白的跨膜结构域,结果如图3所示,信号线平直没有变化,可知UGPase 蛋白不是膜蛋白,不存在跨膜结构域,该预测结果与该蛋白定位在细胞质内的预测结果相一致。
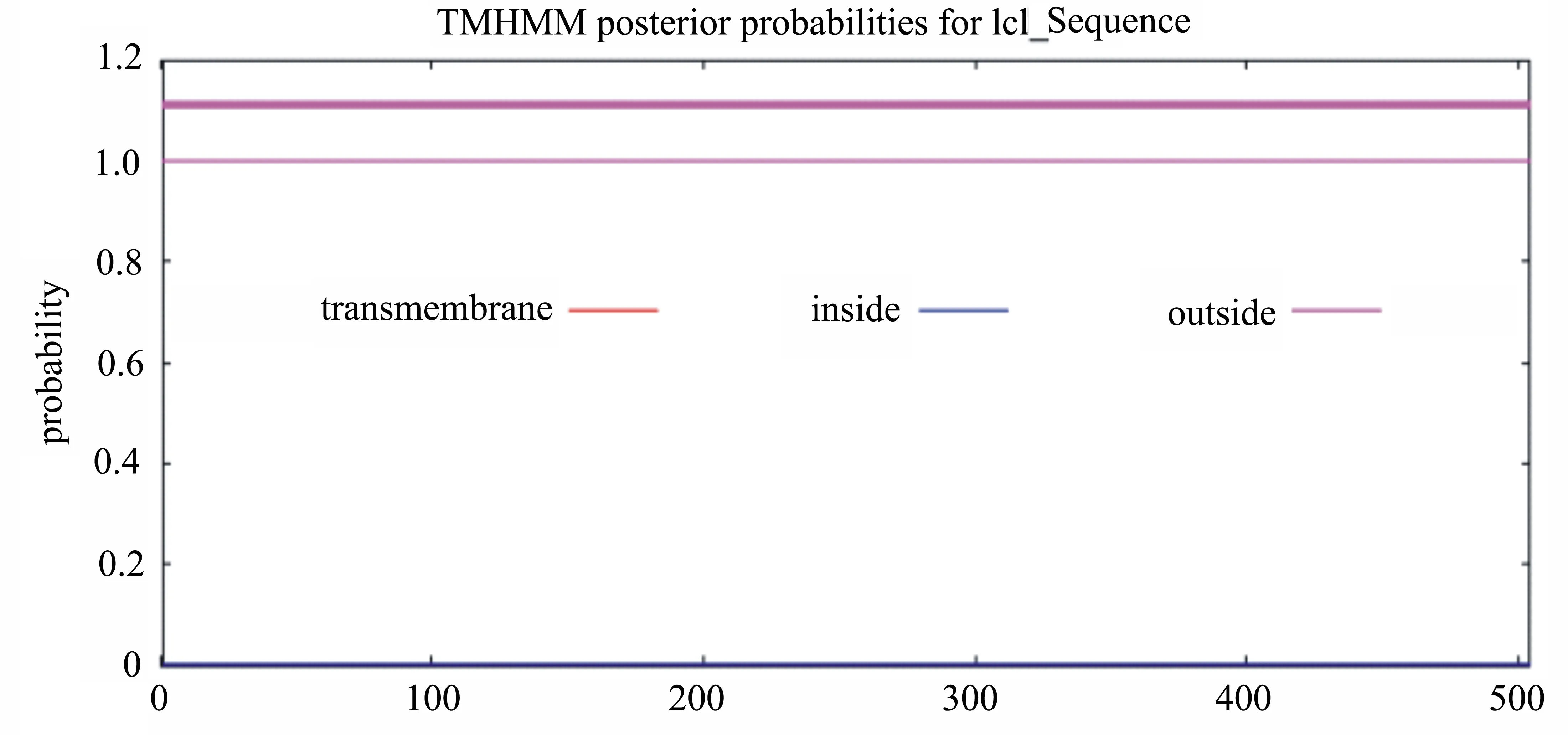
图3 灵芝蛋白跨膜结构域预测
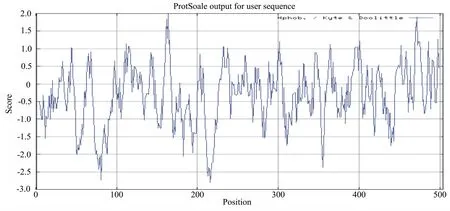
图4 灵芝UGPase蛋白氨基酸疏/亲水性预测
2.3.5 UGPase 蛋白的信号肽结构预测 信号肽是分泌蛋白新生肽链 N端的一段20~30个氨基酸残基组成的肽段,它决定某些氨基酸残基的修饰,常用于指导蛋白质的跨膜转移[11]。利用ExPASy-Signalp 4.1在线软件对灵芝UGPase蛋白信号肽结构进行预测,软件预测结果如图5和表5所示。UGPase蛋白的第23位氨基酸残基具有最高的原始剪切位点分值0.116,第34位氨基酸残基具有最高的信号肽分值为0.119,第1位到第 22位氨基酸残基的信号肽分值为0.101,第23位氨基酸残基具有最高综合剪切位点分值为0.108。由于该氨基酸残基的原始剪切位点和信号肽的分值均较小,所以推断灵芝UGPase基因不存在信号肽,它是一种非分泌蛋白。
图5 灵芝UGPase蛋白信号肽结构预测
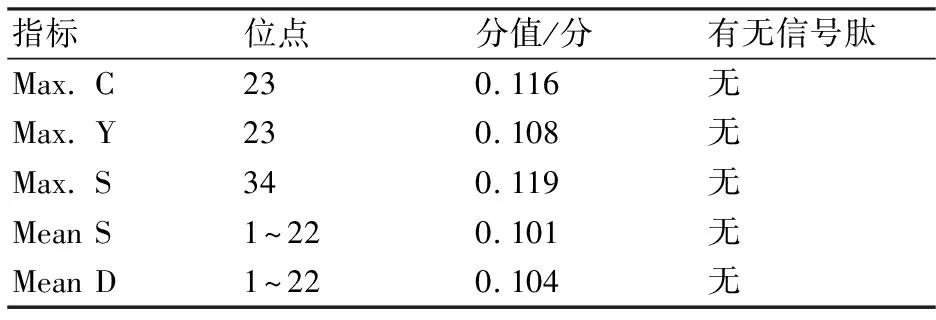
表5 灵芝UGPase蛋白信号肽预测结果
2.3.6 UGPase 蛋白结构域分析 使用 NCBI的CDD (Conserved Domain Database )数据库,对UGPase基因编码蛋白序列进行保守结构域分析,结果如图6所示。UGPase蛋白具有Substrate binding site、UDPGP、UTP-1-磷酸葡萄糖转移酶、UDP-葡萄糖焦磷酸化酶(UDPase)和UDPGlcNAc焦磷酸酶等结合位点和保守域,属于Glyco _tranf _GTA_type超家族蛋白。
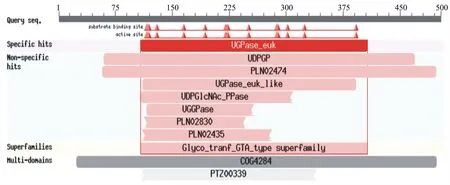
图6 UGPase基因编码蛋白的保守结构域分析
2.3.7 UGPase 蛋白的二级结构预测 利用ExPASy-SOPMA在线软件对UGPase蛋白二级结构进行预测,结果如图 7所示。由表6可知,无规则卷曲的比例最高为37.30%,其次是α螺旋为34.52%,而延伸链和β折叠所占的比例低,分别只有20.04%和8.13%。由此可推测,α螺旋结构和无规则卷曲结构散布于其整个蛋白质中,是构成灵芝UGPase蛋白质二级结构的主要骨架。
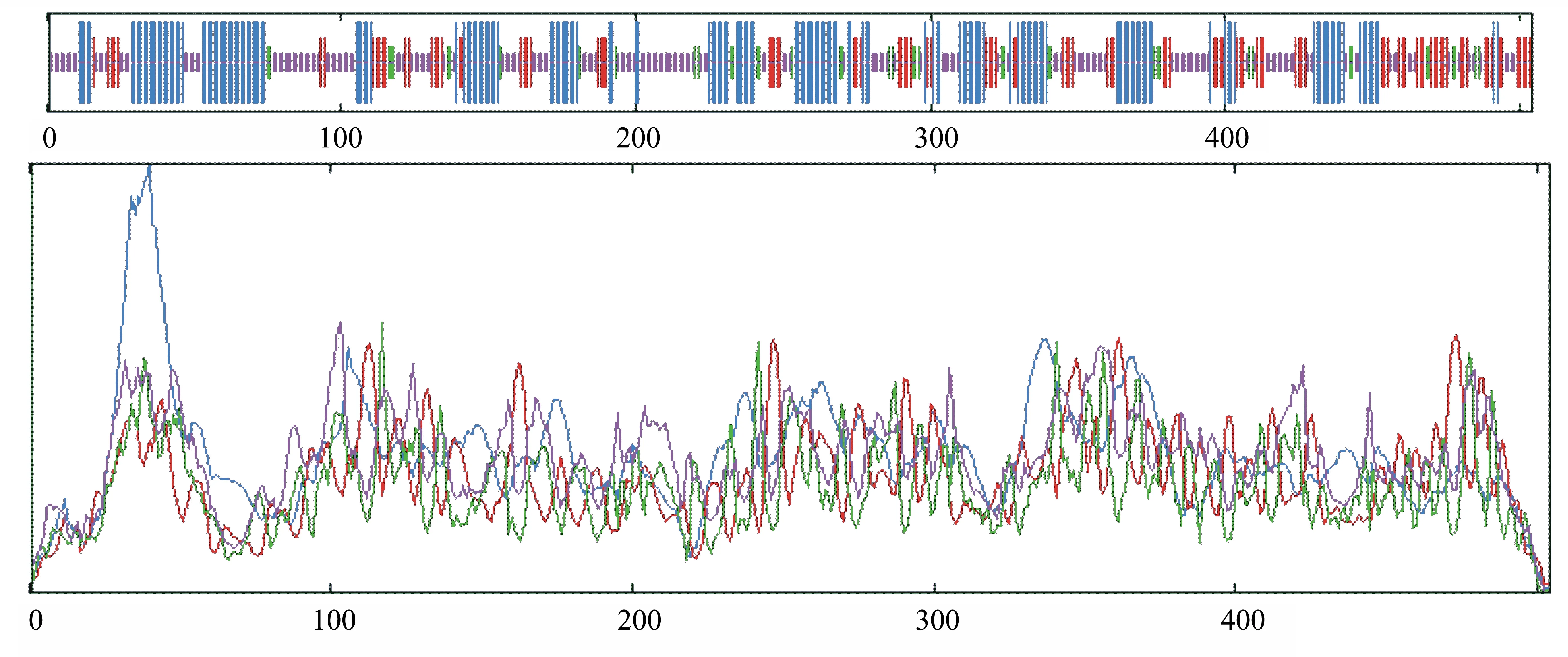
图7 灵芝UGPase蛋白二级结构预测

表6 灵芝UGPase蛋白二级结构
2.3.8 UGPase蛋白的三级结构预测 通过Expasy工具中的在线软件SWISSS-MODEL对UGPase蛋白的三级结构进行预测,从图8可知,UGPase蛋白的空间构象主要是由α螺旋结构和无规则卷曲结构所组成,延伸链数目也很多,而β折叠很少,这与前面得到的二级结构的预测结果相一致。把UGPase蛋白空间构象模拟图与云芝、污叉丝孔菌、灰盖鬼伞菌的空间构象模拟图进行比较,发现其空间结构特点非常相似,表明UGPase蛋白保守性很强。

图8 灵芝、云芝、污叉丝孔菌、灰盖鬼伞菌蛋白空间构象模拟图
2.3.9 UGPase蛋白同源性分析及系统进化树的构建 把灵芝UGPase氨基酸序列与Dichomitussqualens(EJF65102.1)、Trametesversicolor(EIW63033.1)、Coprinopsiscinereaokayama(XP_001830101.1)、Stereumhirsutum(EIM79169.1)、Laccariabicolor(XP_001880115.1)、Coniophoraputeana(EIW80502.1)、Fomitiporiamediterranea(EJD03181.1)、Pucciniagraminis(XP_003336014.1)、Rhodosporidiumtoruloides(EMS18250.1)、Rhodotorulaglutinis(EGU10896.1)、Pseudozymahubeiensis(GAC95340.1)、Dacryopinax(EJU03952.1)、Cryptococcusneoformansvar(AFR93944.1)通过序列处理在线工具包(SMS)进行同源比对,结果如图9显示,灵芝UGPase与Dichomitussqualens同源性最高达到97%,与其他植物的同源性也比较高,表明UGPase蛋白在不同植物中具有比较高的保守性。

图9 灵芝UGPase与其他植物基因编码的氨基酸序列比较
通过MEGA 5.0软件比对分析,构建了灵芝UGPase氨基酸序列与上述物种的氨基酸序列的进化树,如图10所示。灵芝与Dichomitussqualens亲缘关系最近,其次是Trametesversicolor,而与其他物种的亲缘关系相距较远。根据氨基酸序列分析的物种亲缘关系与传统进化亲缘关系相同。

图10 灵芝UGPase与其他植物UGPase酶的氨基酸序列进化树
3 结论
基因的电子克隆是随着基因组计划和EST计划的发展而兴起的,它主要利用不同物种的同类基因之间存在序列保守性这一原理[12-13],相对与传统基因克隆的方法,电子克隆速度快、成本低、针对性强、技术要求低,可以将更多的精力放在克隆基因的功能研究上[14]。
本文利用草茎点霉葡萄糖焦磷酸化酶作为电子探针,通过电子克隆方法得到灵芝UGPase基因,利用生物信息学软件对UGPase基因编码蛋白的理化性质、亚细胞定位、亲疏水性、信号肽、蛋白结构域、蛋白高级结构以及系统进化树的构建等方面进行了预测和分析,结果表明:UGPase基因全长1691 bp,编码504个氨基酸,分子量为56830.1 Da,等电点为6.8。该蛋白不存在信号肽和跨膜结构域,是定位于细胞质内的一种亲水性稳定蛋白酶。该蛋白的高级结构主要是由α螺旋结构和无规则卷曲结构所组成,和其他植物的UGPase酶相比在序列组成、高级结构方面具有一定的相似性,表明该蛋白在进化上较保守。本研究采用电子克隆方法首次得到了灵芝UGPase基因的cDNA序列,为进一步研究UGPase基因在灵芝中的生物学功能奠定了基础。

